CADM1基因甲基化与宫颈鳞癌及癌前病变的相关性研究
2010-01-11谭秋芬胡丹妮梁伟华潘晓琳
谭秋芬,贾 薇,胡丹妮,李 锋,陶 林,梁伟华,潘晓琳
(新疆地方病与民族高发病重点实验室,石河子832002)
本研究采用MassARRY Epi TYPER DNA甲基化分析技术定量分析宫颈鳞癌、CIN2/3、CIN1、正常对照组中CADM1基因启动子区15个CpG位点的甲基化状态,探讨CADM1基因CpG位点甲基化状态与宫颈鳞癌发生机制的关系。
1 材料与方法
1.1 组织样本
石河子大学医学院第一附属医院2007~2009年间石蜡包埋组织,宫颈鳞癌49例,CIN2/347例,CIN128例,对照宫颈组织24例(为子宫肌瘤行全子宫切除术后,经病理诊断证实无宫颈病变)。
1.2 实验方法
1.2.1 提取DNA 将10~15张约5um厚石蜡包埋组织蜡膜置于2.0mL消毒离心管中;经二甲苯脱蜡,无水乙醇脱苯,细胞裂解液及蛋白酶 K消化,苯酚-氯仿-异戊醇抽提,无水乙醇沉淀,适量 TE(pH=8.0)溶解DNA,沉淀12~24h后置于-20℃保存备用。
1.2.2 DNA质量鉴定 所提取DNA均经β-globin PCR扩增检测DNA是否提取成功,使用Nanodrop ND-1000紫外分光光度仪检测DNA浓度和纯度。1.2.3 DNA亚硫酸氢盐处理 使用 EZDNA甲基化处理试剂盒(Zymo Research,Orange County,CA)处理DNA,按照试剂盒提供步骤操作。
1.2.4 CADM1基因甲基化检测 采用MassARRY Epi TYPERDNA甲基化分析技术技术(Sequenom,SanDiego,CA)定量分析CADM1基因的甲基化状态。使用MassCLEAVE试剂盒(Sequenom)应用PCR法在384孔板上扩增目的基因片段,加入虾碱性磷酸酶(shrimp alkaline phosphatase,SAP)混合液去除反应体系中游离的核苷酸,对PCR产物进行体外转录,加入树脂清除离子成分,混匀离心裂解反应液后备用。使用MassARRAY Nanodispenser (Sequenom,SanDiego)从384孔板裂解反应液中取出22nL入基质芯片(Spectro-CHIPs;Sequenom,SanDiego),MassARRAY质谱仪(Bruker-Sequenom)收集质谱图,经 EpiTYPER v1.0软件(Sequenom)进行分析。
1.2.5 HPV16E6DNA检测 采用 PCR法检测HPV16DNA,选用课题组前期经测序确定为HPV16阳性样本为阳性对照,PCR扩增未见特异性目的片段的样本为阴性对照,空白对照以高压消毒双蒸水代替DNA模板,取2μLPCR产物经2%琼脂糖凝胶电泳,BIO-RAD凝胶成像图像分析仪观察结果。引物序列见表1。

表1 引物序列Tab.1Primer sequences
1.3 统计学方法
采用 SPSS16.0软件进行数据处理。运用Kruskal-WallisH检验和相关分析。运用Cluster3.0软件和TreeView软件进行聚类分析。以 P<0.05为差异有统计学意义。
2 结果
2.1 DNA质量鉴定
结果见图1。由图 1可见,β-globinPCR产物经2%琼脂糖凝胶电泳,均在268bp处出现目的片段(图1),DNA纯度 OD260/OD280在1.7~2.0之间,DNA浓度>50ng/μL,样本 DNA均可用于PCR扩增。
图1 β-globin PCR扩增产物电泳图Fig.1 The electrophoretogram ofβ-globin amplified products
2.2 CADM1基因在宫颈鳞癌、CIN2/3、CIN1及正常组中的甲基化检测结果
利用MassARRY Epi TYPER质谱仪分析提供的图谱数据,精确定位到基因片段每个CpG单位的甲基化状态,用甲基化率量化每个CpG单位的甲基化程度。本实验148例样本,每例样本扩增子中包含10个CpG单位(扩增子中包含15个CpG位点,经 T切后分为10个CpG单位,每个CpG单位包含单个或多个CpG位点),CpG单位总数为1480个,其中 1402个 CpG单位可被分析,可分析率达94.7%。1402个可被分析的CpG单位中,平均甲基甲率<30%的CpG单位为1227个,占87.5%,甲基化水平较低,甲基化率>90%的CpG单位为140个,占1.0%。应用双向聚类分析描绘宫颈鳞癌(n=49)、CIN2/3(n=47)、CIN1(n=28)与正常组(n=24)中的CADM1基因10个CpG单位甲基化率的分布趋势,如图2,显示宫颈鳞癌组甲基化率高于CIN2/3、CIN1及正常对照组。
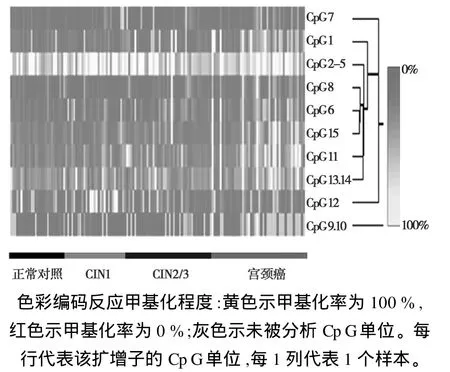
图2 CADM1基因在宫颈148例样本中的甲基化状态聚类分析Fig.2Cluster diagram depicting Cp G Units methylation of CADM1
宫颈不同病变中,CADM1基因各CpG单位呈现不同水平的甲基化,CpG_2·3·4·5位点平均甲基化率在宫颈鳞癌、CIN2/3、CIN1、正常组均较高,平均甲基化率约50%;CpG_7、CpG_9.10位点平均甲基化率在宫颈鳞癌、CIN2/3、CIN1、正常组均较低,平均甲基化率<10%,如图3。
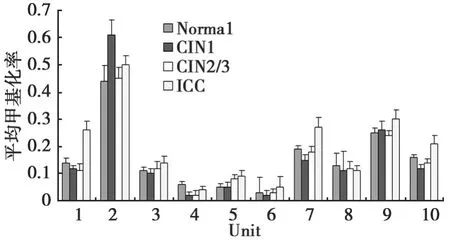
图3 CADM1基因CpG单位在正常、CIN1、CIN2/3、宫颈鳞癌组的平均甲基化率Fig.3 Group mean methylation of the Cp G units of CADM1
应用 Kruskal-WallisH检验,显示宫颈鳞癌组CpG_1、CpG_8、CpG_11、CpG_15位点甲基化率高于CIN2/3、CIN1及正常组,差异有统计学意义(P<0.05),如表2。CIN2/3、CIN1及正常组间甲基化率差异无统计学意义(P>0.05)。
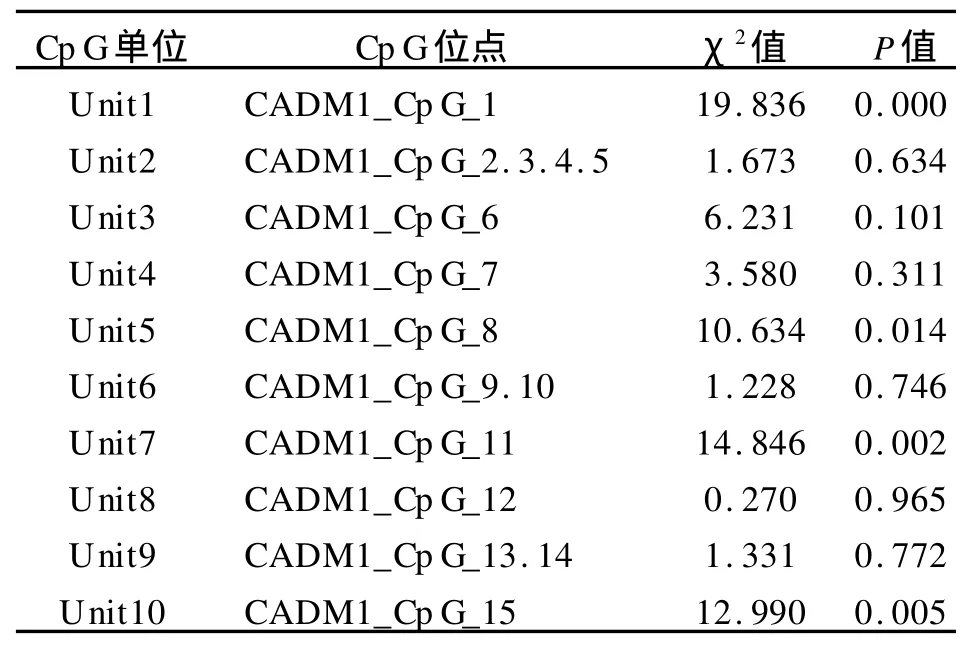
表2 宫颈鳞癌、CIN2/3、CIN1及正常对照组中CADM1基因CpG单位甲基化状态的比较Tab.2 The comparison of individual Gp G units methylation of CADM1 ,bettween different cervical lesions and normal average
2.3 宫颈鳞癌中 CADM1基因甲基化与HPV16感染的相关性
应用相关分析显示,CADM1基因CpG_8、CpG_11、CpG_15甲基化水平与 HPV16感染呈正相关(HPV16阳性样本36例,阴性样本13例),有统计学意义(P<0.05),见表3。
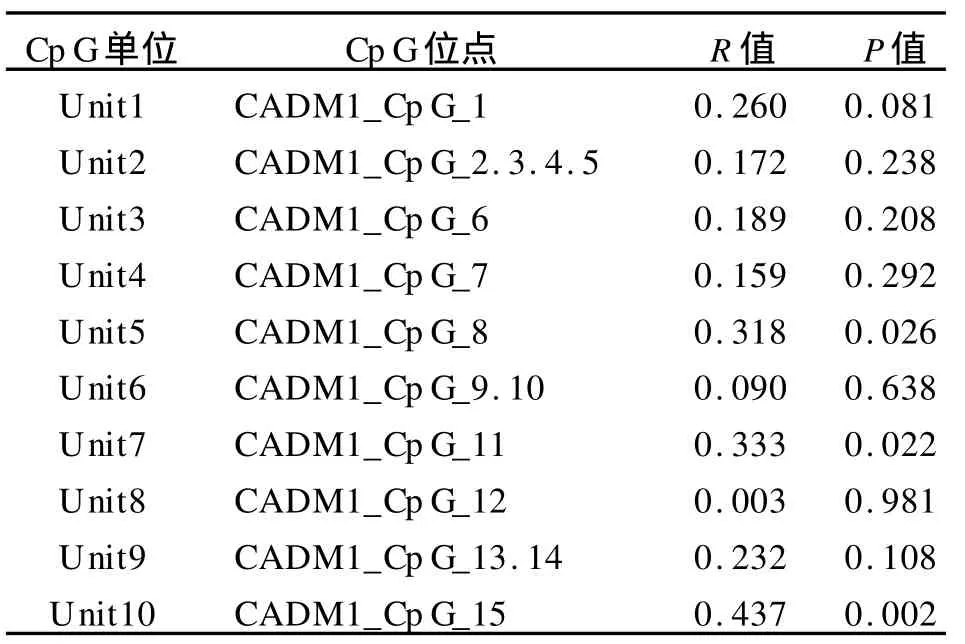
表3 宫颈鳞癌中HPV16检测阳性与阴性样本CADM1基因甲基化状态比较Tab.3 Relationship of individual Cp G units methylation of CADM1 between HPV16 positive samples and negative sample s
3 讨论
本文对CADM1基因启动子区15个CpG位点进行分析发现,在宫颈鳞癌、CIN2/3、CIN1、正常组中,CpG_2·3·4·5位点平均甲基化率均较高,CpG_7、CpG_9.10位点平均甲基化率均较低,不同CpG位点甲基化程度不同。宫颈鳞癌CADM1基因启动子区甲基化水平高于 CIN2/3、CIN1、正常组,其中CpG_1、CpG_8、CpG_11、CpG_15四个位点甲基化率在宫颈鳞癌中明显高于CIN2/3、CIN1及正常组,CIN2/3、CIN1及正常组间CpG位点甲基化水平无显著差异。提示CADM1基因启动子区特异性位点甲基化状态的改变可能在促进CIN向宫颈鳞癌转化的过程中起着一定的作用。CADM1基因编码的跨膜糖蛋白羧基端包括 FERM-和 PDZ-结合模序[10],FERM-和 PDZ-结合模序分别与 MPP3和DAL-1连接形成复合体,并与细胞骨架连接,可能与细胞增殖、细胞粘附、肿瘤抑制和免疫逃逸有关[11~13]。CADM1基因表达沉默在宫颈肿瘤中是频发事件,伴有或不伴有等位基因缺失的启动子区甲基化是CADM1基因失活的主要方式[11]。CpG岛通常位于基因的5′端启动子区,也可延伸至基因的外显子区,启动子区的CpG被甲基化时基因转录受到抑制。有学者提出,基因启动子区CpG甲基化的密度与转录的抑制程度有关[14]。Steenbergen等[11]研究显示,在宫颈鳞癌及 CIN2/3组中CADM1基因启动子区所有CpG岛均发生了甲基化,其检出率分别为58%和35%,认为宫颈鳞癌中CADM1基因甲基化频率高、范围广。Overmeer等[6]以CADM1基因启动子区的三个区域为靶点,结果显示高密度(≥2个区域)甲基化与蛋白表达减低有关,且在宫颈病变≤CIN1组与≥CIN3组之间有显著差异,而单个区域甲基化在正常组和CIN1组中占有较大比例,认为高密度甲基化可以作为诊断宫颈鳞状上皮CIN3及以上病变的有效指标。另有研究认为DNA建立的甲基化模式是有区域特异性的,并非所有的CpG位点甲基化敏感度都一样,少数CpG位点甲基化就可能有效地消弱基因启动子的活性[15,16]。有学者应用MassARRYEpiTYPERDNA甲基化分析技术检测前列腺肿瘤中8个基因的甲基化状态,发现 RARB2基因CpG_10.11位点甲基化评分诊断前列腺癌的灵敏度和特异度分别为90.0%,92.59%,认为基因单个CpG位点甲基化的改变可以用于肿瘤的诊断[17]。因此,本研究结果提示特定CpG位点甲基化状态的改变可能导致了基因表达沉默,造成肿瘤抑制丧失,在宫颈鳞癌的发生发展中起着重要的作用。
本研究中,在宫颈鳞癌中CADM1基因CpG_8、CpG_11、CpG_15三个位点的甲基化水平与HPV16感染呈正相关,提示 CADM1基因特定CpG位点的高甲基化状态可能与 HPV感染协同导致了宫颈鳞癌的发生、发展。近年来研究证实单独HPV感染不足以引起 HPV相关性肿瘤(如宫颈癌)的发生,遗传和其他因素一起导致了侵袭性表型的形成。研究显示 HPVDNA感染细胞必须获得永生化表型和停泊非依赖性生长表型才能具有致瘤性,所有这些表型都为隐性性状,提示这些表型可能与抑癌基因表达缺失有关[18]。另有研究发现在人类纤维母细胞中11p缺失与 HPV16诱导的停泊非依赖性有关,当人类11号染色体被导入到 Hela和SiHa细胞株中,其致瘤性丧失,表明11号染色体上存在抑癌基因[19]。早期发现11q23.2缺失中有一特殊的约700kb的区域能完全抑制肺癌细胞株A549的恶性表型,通过功能互补研究发现其抑制作用主要依赖其中100kb的区域,而CADM1是该区惟一基因[13]。高危型 HPV感染的CIN2/3到宫颈癌的演进过程中经常发现由于启动子甲基化引起的CADM1基因表达沉默,但是CADM1mRNA在非瘤性 HPV永生化细胞株中表达丰富,提示CADM1基因表达沉默可能与 HPV永生化到致瘤性表型的转化有关[11]。
本研究通过对CADM1基因多个CpG位点甲基化状态定量分析,认为CADM1基因特异性位点CpG_1、CpG_8、CpG_11、CpG_15的甲基化可能促进了宫颈鳞癌的发生、发展,其中 CpG_8、CpG_11、CpG_15的甲基化可能与 HPV永生化到致瘤型表型的转化有关。CADM1基因特异性CpG位点甲基化状态的改变可能导致基因转录表达沉默,可能是宫颈鳞癌发生发展的促进因素。
[1]Pisani P ,Bray F , Parkin D M. Estimates of the worldwide prevalence of cancer for 25 sites in the adult population[J] . Int J Cancer ,2002 ,97 :72-81.
[2]Jones P. A ,Baylin S. B. The epigenomics of cancer [J] .Cell 2007 ,128 (4) :683-692.
[3]Ting A H ,McGarvey K M ,Baylin S B ,et al. The cancer epigenome component s and functional correlates [J] .Genes Dev ,2006 ,20 (23):3215-3231.
[4]Brueckner B , Kuck D ,Lyko F ,et al. DNA methyltransferase inhibitors for cancer therapy[J ]. Cancer J ,2007 ,13(1) :17-22.
[5]Egger G,Liang G,Aparicio A ,et al. Epigenetics in human disease and prospects for epigenetic therapy [ J ]. Nature ,2004 ,429 (6990) :457-463.
[6]Overmeer R M ,Henken F E ,Snijders P J ,et al. Association between dense CADM1 promoter methylation and reduced protein expression in high-grade CIN and cervicalSCC[J] . Pathol ,2008 ,215 (4) :388-397.
[7]李 惠,杨永秀,刘 磊.宫颈鳞癌中肺癌肿瘤阻抑基因-1和上皮型钙黏蛋白的定量研究[J].中国现代医学杂志,2008,18(13):1801-1803.
[8]Ehrich M ,Nelson M R ,Stanssens P ,et al. Quantitative high-throughput analysis of DNA methylation patterns by base-specific cleavage and mass spectromet ry [J] .PNAS ,2005 ,102 (44) :15785-15790.
[9]Lukaszuk K,Liss J ,Wozniak I ,et al. Human papillomavirus type 16 status in cervical carcinoma cell DNA assayed by multiplex PCR[J ] . J Clin Microbiol ,2003 ,41(2) :608-612.
[10]Yageta M ,Kuramochi M ,Masuda M ,et al. Direct association of TSLC1 and DAL-1 , two distinct tumor suppressor proteins in 1ung cancer [J ] . Cancer Res ,2002 ,62 :5129-5133.
[11]Steenbergen R D , Kramer D ,Braakhuis B J ,et al. TSLC1 Gene Silencing in Cervical Cancer Cell Lines and Cervical Neoplasia[J ]. Natl Cancer Inst ,2004 ,96 (4) :294-305.
[12]BolesKS,BarchetW,DiacovoT,etal.ThetumorsuppressorTSLC1/NECL-2triggersNK-cellandCD8(+)T-cellresponsesthroughtheceff-surfacereceptor CRTAM[J].Blood,2005,106:779-786.
[13]Boles K S ,Barchet W ,Diacovo T ,et al. The tumor suppressor TSLC1/ NECL-2 t riggers N K-cell and CD8 ( + ) T-cell responses through the ceff-surface receptorCRTAM[J ] .Blood ,2005 ,106 :779-786.
[14]Robertson K D ,Jones P A. DNA methylation :past ,present and future directions [J ]. Carcinogenesis ,2000 ,21 (3) :461-467.
[15]Catteau A ,Harris W H ,Xu C F ,et al. Methylation of the BRCA1 promoter region in sporadic brest and ovarian cancer :correlation with disease characteristics [J ] .Oncogene ,1999 ,18 (11) :1957-1965.
[16]Gonzalgo M L , Hayashida T ,Bender C M , et al. The role of DNA methylation in expression of the p19/ p16 local in human bladder cancer cell lines [ J ]. Cancer ,1998 ,58 (6) :1245-1252.
[17]Vanaja D K,Ehrich M ,Boom D V ,et al. Hypermethylation of Genes for diagnosis and risk stratification of prostatecancer[J ]. Cancer Investigation ,2009 ,32 :1-12.
[18]Chen T M , Pecoraro G,Defendi V. Genetic analysis of in vitro progression of human papillomavirus-transfected humancervical cells[J ]. Cancer Res ,1993 ,53 :1167-1171.
[19]Steenbergen R D ,Kramer D ,Meijer C J ,et al. Telomerase suppression by chromosome 6 in a human papillomavirus type 16-immortalized keratinocyte cell line and in a cervical cancer cell line [J] . J Natl Cancer Inst ,2001 ,93 :865-872.
