苹果自交不亲和基因PCR扩增程序优化v
2009-01-27陈超于葆杰李保国
陈 超 于葆杰 李保国
摘要:通过研究PCR反应体系(DNA、Primer、Mg2+、dNTP、 Taq聚合酶、buffer)及退火温度对苹果自交不亲和基因(S基因)扩增结果影响的分析,构建了适合于苹果自交不亲和基因的PCR扩增程序的优化体系。结果表明:在20 μL的反应体系中,模板DNA的最适含量为40 ng,dNTP最适浓度为0.25 mmol/L,而Primer最适浓度为0.4 μmol/L,Mg2+的最适浓度为2.5 mmol/L,Taq聚合酶最适浓度为1 U,buffer最适浓度为1×buffer,最适退火温度为50 ℃。
关键词:苹果;S基因;PCR体系优化
中图分类号:S661.1; Q503文献标识码:A DOI编码:10.3969/j.issn.1006-6500.2009.04.008
Optimized Study on PCR Amplification Procedure of the Apple Self-incompatibility Gene
CHEN Chao1,YU Bao-jie1,LI Bao-guo2
(1.Forestry Science Institute of Cangzhou,Cangzhou,Hebei 061001,China;2. Forestry College,Hebei Agricultural University,Baoding,Hebei071000,China)
Abstract:Through analyzing the effect of PCR system (DNA, Primer, Mg2+, dNTP, Taq polymerase, and buffer) and annealing temperature on amplification of apple self-incompatibility gene (s-gene), we constructed a optimized system of PCR amplification procedure which was fit for apple self-incompatibility gene. The result showed that the optimized content in 20 μL system contained 40 ng DNA template, 0.25 mmol/L dNTP, 0.4 μmol/L primer, 2.5 mmol/L Mg2+, 1 U Taq polymerase, 1×buffer, and the optimized annealing temperature was 50 ℃.
Key words: apple;s-gene;optimized PCR system
苹果自交不亲和是在长期的生物进化过程中形成的自我保护机制。但是,对苹果栽培生产却有很大影响。随着分子生物学技术的发展,查明苹果的自交不亲和是由S基因控制的[1]。利用PCR技术可以进行特异基因的扩增,但目前对苹果S基因PCR扩增的研究报道极少,采用常规的PCR反应体系难以获得较高浓度的产物,不能进行正常回收鉴定。因此,笔者对苹果S基因PCR扩增程序进行了优化研究。
1材料和方法
1.1材料
2004年5月上旬,于河北农业大学果树标本园采集富士苹果新梢顶端完全展开的第1~2片幼叶,液氮速冻后置于-70 ℃冰箱保存备用。
1.2方法
基因组DNA提取参照CTAB法[2],DNA质量和浓度的检测用萨姆布鲁克[3]的方法。引物按Shogo matsumoto[4]的设计合成,其上游引物为:5'-TTTACGCAGCAATATCAG-3',下游引物为:5'-ACGTTCGGCCAAATA/CATT-3',由宝生物工程(大连)有限公司合成。
PCR反应体系及热循环条件在Ishimizu T[5]的基础上进行优化试验。在20 μL PCR反应体系中,DNA模板含量设5,10,20,40,60,80 ng 6个浓度梯度;Mg2+浓度设1,1.5,2,2.5,3,3.5,4 mmol/L 7个浓度梯度;TaqDNA聚合酶设0.5,1,2.5,5,7,15 U 6个浓度梯度;dNTP浓度设0.1,0.15,0.2,0.25,0.3,0.4 mmol/L 6个浓度梯度;引物浓度分别设0.1,0.2,0.25,0.3,0.4,0.5 μmol/L 6个浓度梯度;buffer浓度设0.5×buffer、1×buffer、2×buffer、3×buffer、4×buffer、5×buffer 6个浓度梯度;退火温度设41,42,43,44,45,46,47,48,49,50,51,52 ℃共12个温度梯度。
2结果与分析
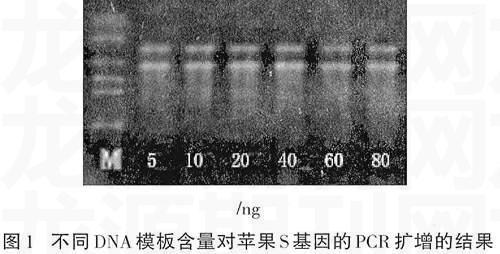
2.1不同DNA模板含量对苹果S基因的PCR扩增的影响
不同DNA模板含量对苹果S基因的PCR扩增结果如图1所示。由此表明,在DNA模板含量5~40 ng都得到了较好的扩增,说明DNA模板含量在此范围内对苹果S基因PCR扩增结果影响较小,5 ng即可满足扩增需要。随着模板含量的增加,扩增条带的亮度有所增强,以40 ng扩增效果最好,当DNA含量达到60~80 ng时扩增条带亮度变弱且宽度变小。因此认为,进行苹果S基因的PCR扩增时DNA模板含量以40 ng为宜。
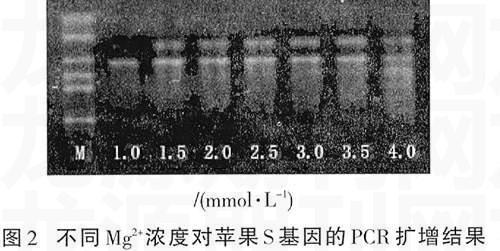
2.2不同Mg2+浓度对苹果S基因的PCR扩增的影响 不同Mg2+浓度条件下苹果S基因的PCR扩增结果如图2所示。由此表明,当Mg2+浓度为1.0 mmol/L时扩增条带少而且弱,随着浓度的增加,特异性结合增强,扩增条带越来越清晰;当浓度达到4 mmol/L时,特异性降低,出现一些非特异性条带,使扩增的条带变宽;Mg2+浓度在1.5~3.0 mmol/L之间均能获得清晰稳定的条带,以浓度在2.5 mmol/L最好。因此认为,进行苹果S基因的PCR扩增时Mg2+浓度以2.5 mmol/L为宜。
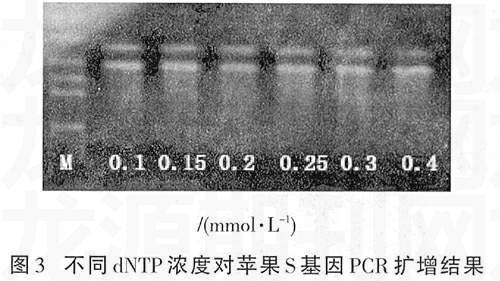
2.3不同dNTP浓度对苹果S基因的PCR扩增的影响
不同dNTP浓度对苹果S基因的PCR扩增结果如图3所示。由此表明,dNTP浓度从0.1~0.3 mmol/L均能扩增清晰的条带,以0.25 mmol/L时扩增条带最清晰,浓度达0.4 mmol/L时扩增条带变弱。因此认为,进行苹果S基因的PCR扩增时dNTP浓度以0.25 mmol/L为宜。
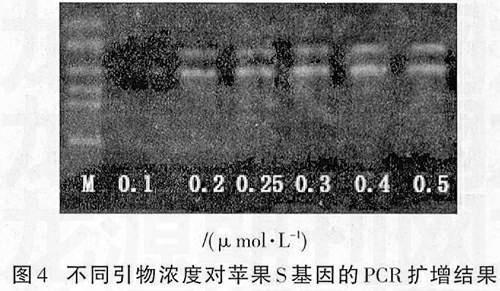
2.4不同引物浓度对苹果S基因的PCR扩增的影响
不同引物浓度对苹果S基因的PCR扩增结果如图4所示。由此表明,引物浓度为0.1 μmol/L时没有扩增产物,浓度为0.2~0.3 μmol/L时扩增条带不清晰,说明在引物浓度较低时,扩增产物效率低;在0.3~0.4 μmol/L时较好,以0.4 μmol/L最佳。因此认为,进行苹果S基因的PCR扩增时引物浓度以0.4 μmol/L为宜。
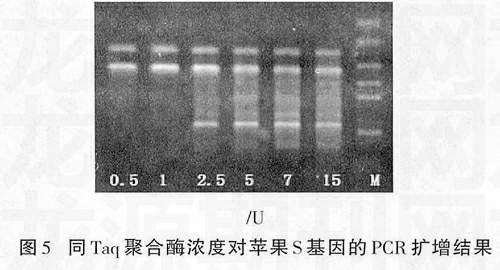
2.5不同Taq聚合酶浓度对苹果S基因的PCR扩增的影响
不同Taq聚合酶浓度对苹果S基因的PCR扩增结果如图5所示。由此表明, Taq聚合酶浓度在0.5~1 U时,能够得到条带清晰、特异性强的扩增产物,以1 U时最佳;在0.25~15 U时,有一些非特异性条带得到了扩增。因此认为,进行苹果S基因的PCR扩增时Taq聚合酶浓度以1 U为宜。
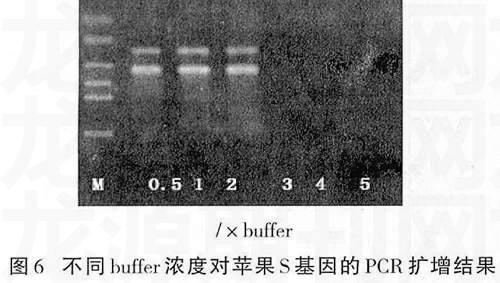
2.6不同buffer浓度对苹果S基因的PCR扩增的影响
不同buffer浓度对苹果S基因的PCR扩增结果如图6所示。由此表明,当buffer浓度为0.5×buffer、1×buffer、2×buffer时,能够得到特异性强而且清晰的条带。以后随着buffer浓度的增加, PCR扩增产率降低。1×buffer时条带清晰,特异性好。因此认为,进行苹果S基因的PCR扩增时buffer浓度以1×buffer为宜。
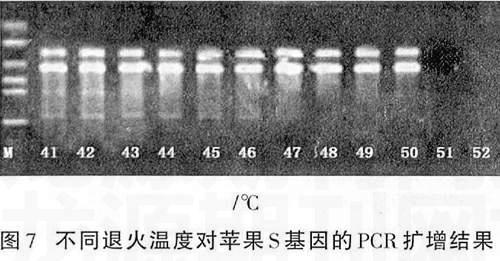
2.7不同退火温度对苹果S基因的PCR扩增的影响
不同退火温度对苹果S基因的PCR扩增结果如图7所示。由此表明,温度在47 ℃以下时扩增条带轮廓不清且出现非特异性条带;在48~50 ℃之间时出现了鲜明的谱带,且特异性很强;在51 ℃以上时,得不到扩增产物。试验结果说明,退火温度太低,有条带模糊不清或扩增效率下降的趋势;而退火温度太高,几乎看不到扩增产物。可见,只有在温度允许的范围内,适当提高退火温度,可以提高PCR反应的特异性,使扩增条带清晰。因此认为,进行苹果S基因的PCR扩增时退火温度以50 ℃为宜。
3结论与讨论
本试验结果表明,苹果S基因PCR最优化反应体系为:总体积20 μL时,DNA模板含量为40 ng,Mg2+浓度为2.5 mmol/L,引物浓度为0.4 μmol/L, dNTP浓度为0.25 mmol/L,Taq聚合酶浓度为1 U,buffer浓度为:1×buffer,退火温度为50 ℃。
PCR对DNA质量要求不高[6],当DNA模板含量在5~40 ng范围内均扩增出清晰条带,说明苹果DNA模板含量在此范围内不会影响苹果S基因的PCR扩增结果。但当DNA含量超出此范围以后,随着DNA含量的提高,扩增条带不清晰。因为,当DNA含量超过60 ng以后,由于引物浓度相对较少,在PCR反应过程中高浓度的DNA对引物有竞争作用,可能对引物有一定的分散效应,使扩增产率降低。
参考文献:
[1] Kobel F,Steinegger P,Anliker J. Wertere untersuchungen uber die befruchung sverhaftnisse der apfel und birnsorien[J].Landw Jbschweiz,1939,53:160-191.
[2] 顾红雅.植物分子生物学实验手册[M].北京:高等教育出版社,1998:3-12.
[3]萨姆布鲁克 J,拉塞尔D W.分子克隆实验指南[M].北京:科学出版社,2002:387-400.
[4] Matsumoto S, Kitahara K. Discovery of a new self-incompatibility allele in apple[J]. HortScience,2000,35(7):1329-1332.
[5] Ishimizu T, Inoue K, Shimonaka M, et al. PCR-based method for identifying the S-genotypes of Japanese pear cultivars[J]. Theor Appl Genet,1999(98):961-967.
[6] Saiki R K, Scharf S, Faloona F, et al. Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia [J].Science,1985, 230(4732):1350-1354.
