CRISPRCas 系统在枯草芽孢杆菌基因组编辑中的研究进展
2025-02-19孙志康李力群郝捷吴晗吴娜郑超季嫱李选陈晨

摘要:枯草芽孢杆菌(Bacillus subtilis)是一种食品安全级微生物,现已广泛应用于工业发酵。CRISPR(clustered regularly interspaced short palindromic repeats)介导的基因组编辑技术在以枯草芽孢杆菌为底盘细胞的微生物代谢工程研究中发挥了重要作用。介绍了CRISPR‐Cas系统的免疫应答机制和分类以及枯草芽孢杆菌中CRISPR-Cas9基因组编辑的3种类型,重点总结了最新的CRISPR开发和设计策略,以期为优化现有的枯草芽孢杆菌基因组编辑系统提供参考,从而提高枯草芽孢杆菌的工业化应用潜能。
关键词:枯草芽孢杆菌;基因编辑;CRISPR-Cas9;CRISPR-Cpf1;PAM无依赖doi:10.13304/j.nykjdb.2023.0260
中图分类号:Q812 文献标志码:A 文章编号:1008‐0864(2025)02‐0024‐09
枯草芽孢杆菌主要分布于土壤及腐败的有机物中[1],具有清晰的遗传背景和成熟的基因工程操作技术,因此一直被用作模式微生物[2‐3]。它具有很强的蛋白分泌能力,并且不会产生内毒素等有害物质,因此被美国食品药品监督管理局(Food and Drug Administration,FDA)认证为GRAS(generally recognized as safe)菌株,是食品安全级微生物[4‐5];除此之外,由于其生长速度快、蛋白分泌能力强、不易被噬菌体感染以及在基因操作过程中没有明显的密码子偏好性,常被用于工业发酵[5-7],特别是在淀粉酶、蛋白酶、果胶酶等酶制剂的生产中有着广泛的应用。
近年来,随着基因工程技术和分子生物学等的发展,启动子工程、辅因子工程、基因回路、途径酶组装和基因编辑等多种研究策略和工具被用以枯草芽孢杆菌底盘细胞的构建,进行透明质酸[8]、七稀甲萘醌[9]、N-乙酰氨基葡萄糖[10]、核黄素[11]和母乳寡糖[12] 等生物制品的高效合成。其中CRISPR (clustered regularly interspaced shortpalindromic repeats)介导的基因编辑系统被广泛用于构建枯草芽孢杆菌底盘细胞以进行酶制剂等生物制品的高效合成。CRISPR是细菌和古菌抵御外来病毒入侵的一种获得性免疫系统,如今已发展成为基因工程中强大的基因编辑工具[13]。CRISPR 基因编辑系统是通过sgRNA 引导Cas蛋白(如Cas9、Cpf1等)定位到靶基因位点,随后Cas蛋白在基因组的特定位点对DNA进行切割,造成DNA双链断裂,在同源性定向修复过程中通过同源臂将特定修饰的片段引入基因组,从而实现基因的敲除、插入或突变[14]。
枯草芽孢杆菌中建立了多种基于CRISPR‐Cas的基因编辑系统(CRISPR‐Cas9/Cpf1 等)。Zhang等[15]通过构建包含靶标特异性的sgRNA、Cas9和同源修复模板的多合一CRISPR‐Cas9编辑系统,敲除了枯草芽孢杆菌中与孢子形成相关的5个基因,获得的突变株在发酵过程中,由于孢子萌发率大幅降低,重组蛋白得以稳定生产,胞外分泌的β-环糊精糖基转移酶活力达到277.8 U·mL-1,是未改造前的2.5倍。张春晓等[16]通过CRISPR/Cpf1编辑系统,对枯草芽孢杆菌中的d-丙氨酸消旋酶基因da1 进行了敲除,实现了β-甘露聚糖酶的食品级表达,酶活力达到17 601.3 U·mL-1。本文介绍了CRISPR-Cas 系统的机制和分类,并总结了3种基于CRISPR-Cas9的枯草芽孢杆菌基因组编辑系统构建方法,最后展望了新兴的CRISPR 系统开发和设计策略,为提高枯草芽孢杆菌的工业化应用提供新的见解。
1 CRISPRCas系统简介
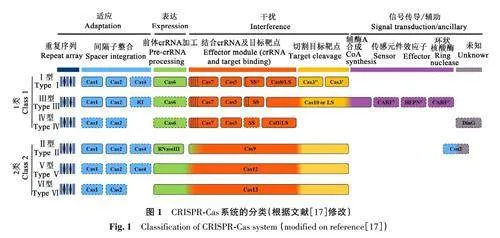
作为新一代基因组编辑工具的关键技术,CRISPR‐Cas系统在细菌和古生菌中发挥着适应性免疫机制作用。CRISPR‐Cas免疫应答包括3个主要阶段:适应、表达和干扰。在适应阶段,外源DNA 片段(protospacer)被识别并整合在CRISPR基因座区域中的2个相邻重复之间,形成CRISPR阵列,protospacer 相邻基序(protospacer adjacentmotif,PAM)是存在于protospacer近端的一小段保守核苷酸,用作获取DNA片段的识别基序。在表达阶段,CRISPR阵列被RNA聚合酶转录成前体CRISPR RNA(pre-crRNA),再被Cas 蛋白进一步切割成小的crRNA。crRNA包含1个部分重复序列和1个来自外源DNA片段并与之互补的单个间隔区。在干扰阶段,Cas蛋白与crRNA‐外源DNA复合物结合并切割DNA片段,干扰病毒复制并为宿主细胞提供免疫力。
如图1所示,根据干扰阶段效应蛋白的组成和具体的响应模式,CRISPR‐Cas系统可分为2类(class),6型(type)[17]。第1类CRISPR‐Cas系统目前多发现于古细菌中,其特点是系统编码的多个Cas蛋白会构成效应复合体与CRISPR 一起发挥作用;与其不同的是,第2类CRISPR‐Cas 系统中的Cas蛋白往往是具有多结构域的单一蛋白,较第1类CRISPR系统的组成更简单。因此,目前基于CRISPR的基因编辑系统绝大部分都是由第2类CRISPR 系统改造而来的,其中应用最为广泛的是第2类的第Ⅱ型——CRISPR‐Cas9系统[18]。
2 基于CRISPRCas9的枯草芽孢杆菌基因组编辑系统构建
枯草芽孢杆菌中建立了多种基于CRISPR‐Cas的基因编辑系统,并且都取得了较好的应用效果。CRISPR‐Cas9系统为许多领域的研究带来了新发展[19],Ⅱ型CRISPR‐Cas9 系统已被证明可以在枯草芽孢杆菌中引入点突变、基因缺失和插入,并被广泛应用于代谢工程和合成生物学领域。根据CRISPR‐Cas9 系统中供体DNA、Cas9 和gRNA 表达的设计策略,可以将枯草芽孢杆菌CRISPR‐Cas9系统分为3种不同类型,分别是单质粒系统、双质粒系统和基因组整合型[20](表1)。
2.1 单质粒CRISPRCas9基因组编辑系统
基于单个质粒编码的基因组编辑系统已经被开发应用于各种原核生物和真核生物。Altenbuchner[21]报道了一种用于单质粒枯草芽孢杆菌基因组的编辑系统,不仅可以高效地进行基因组编辑,还克服了反向筛选方法的局限性。该系统通过将Cas9、gRNA、供体DNA和其他元件共同连接到同一载体骨架,构建了单质粒体系,其中Cas9和gRNA分别由1个诱导型启动子和1个强组成型启动子启动转录。
单质粒基因编辑系统具有较高的基因编辑效率,因此可以应用于工业枯草芽孢杆菌的改造。Zou等[31]构建了一个“多合一”的CRISPR‐Cas9单质粒系统,缩短了枯草芽孢杆菌一轮编辑周期。该系统能够实现100%的单基因缺失和点突变效率以及96%的基因插入效率。由于所有元件均整合在1个质粒载体上,载体太大导致转化效率低,研究人员将质粒消化成片段,使用分子凝结剂PEG4000辅助连接质粒片段后转化入枯草芽孢杆菌,转化效率较直接转化提升了2~3 个数量级。单质粒介导的CRISPR‐Cas9基因组编辑系统在枯草芽孢杆菌中的应用结果表明,该系统在枯草芽孢杆菌的基因组改造中具有巨大的潜力。
单质粒基因编辑系统不仅可以实现枯草芽孢杆菌基因组的高效编辑,更重要的是,可以降低宿主菌株的适应性负担,从而提高工程菌株的存活率。但是使用单质粒系统需要多步、多次构建质粒,大大降低了基因编辑系统的内在效率,而且单质粒系统的同源重组效率在很大程度上取决于同源修复模板的长度。综上所述,单质粒介导的CRISPR‐Cas9基因组编辑系统在枯草芽孢杆菌的基因工程改造中仍存在一定的局限性。
2.2 双质粒CRISPRCas9基因组编辑系统
双质粒基因编辑系统是一种基于CRISPRCas9的、更灵活的基因组编辑系统,已经开始应用于枯草芽孢菌的基因组编辑,在该系统中,Cas9、gRNA 和供体DNA 被分别构建到2 个不同的载体质粒中,最后在宿主菌株体内结合进行基因组编辑,其中第1个质粒用于产生Cas9蛋白,第2 个质粒用于传递gRNA 转录模板和供体DNA模板。
Lim 等[28]开发了一种基于CRISPR‐Cas9的高效双质粒基因编辑系统,并对枯草芽孢杆菌基因组中8 个胞外蛋白酶基因进行了精准的基因缺失。在这个双质粒系统中,Cas9 基因被连接到pHCas9载体中以产生Cas9蛋白,2个sgRNA——靶基因sgRNA (sgRNAct)、自靶向sgRNA (sgRNAst)及用于同源定点修复的供体DNA 被连接到pG2载体中,以对靶位点进行相应操作。将pHCas9载体和pG2载体先后转入枯草芽孢杆菌后发现,在多轮的枯草芽孢基因组编辑中,8个胞外蛋白酶基因的敲除效率都达到了90% 以上,其中bpr、nprB、wprA 的敲除效率达到了100%。由此表明,该系统可以在枯草芽孢杆菌基因组的连续编辑中对靶基因实现高效率删除。
双质粒介导的CRISPR‐Cas9基因组编辑系统极大地促进了枯草芽孢杆菌基因组编辑研究的发展,然而双质粒系统也存在诸多局限性,如在基因工程操作过程中存在不稳定性,可能会增加宿主菌株的适应性负担,并且在迭代基因组编辑中需要耗费大量时间来消除质粒。
2.3 基于CRISPRCas9的基因组整合
为了克服潜在的质粒不稳定、转化效率低和在宿主菌株上引入代谢负担等问题,Westbrook等[30]构建了一个基于CRISPR‐Cas9的基因组整合工具箱来编辑枯草芽孢杆菌基因组,结果表明,该基因组整合工具箱可以对枯草芽孢杆菌实现单基因的插入与敲除以及连续基因组的编辑和靶向转录抑制,是一种连续有效的综合基因组编辑工具箱。在这个基因组整合工具箱中,扩增出了thrC 5’端与3’端间的同源长度,并插入到相应的bpr 同源长度中,构建了gRNA 传递载体PAW002-2[32];为了提高转化效率,并使质粒易于在大肠杆菌中复制繁殖,利用枯草芽孢杆菌的araE/R 启动子系统[33] 构建了载体PAW004-2,以驱动gRNA 的转录;选择lacA 位点进行Cas9 的基因组整合[34]。此外,将每个gRNA分别构建了单gRNA传递载体和多gRNA传递载体,最后,将这些gRNA传递载体线性化后转化枯草芽孢杆菌,结果显示,单基因敲除效率为100%,双基因敲除效率为85%,单基因插入效率为69%(插入片段长度为2.9 kb)[30]。该方法是CRISPR‐Cas9系统在枯草芽孢杆菌中同时敲除2个基因的首次报道。
与其他CRISPR‐Cas9系统相比,该基因组整合工具箱提高了转化效率和编辑效率。然而,尽管基于CRISPR‐Cas9的基因组整合在枯草芽孢杆菌基因组编辑中展现了较高的编辑效率,但该工具箱需要1个酶切连接步骤将新的gRNA序列插入gRNA传递载体,并需要一轮重叠延伸PCR生成编辑模板,其操作过程非常繁琐,因此,基于CRISPR‐Cas9的基因组整合在操作便利性方面仍有改进和优化的余地。
3 枯草芽孢杆菌CRISPR 开发和设计策略
不同遗传操作方法的应用提高了对枯草芽孢杆菌基因组的认识和利用,其中CRISPR‐Cas9系统是最实用、最高效的基因组编辑方法,为点突变、基因敲除、基因插入提供了有效的解决办法[28,30]。然而,利用CRISPR‐Cas9系统对枯草芽孢杆菌进行基因组编辑仍处于初步阶段,由Cas9-gRNA 复合物引起的DNA 双链断裂修复可能很慢,以至于影响宿主细胞在被CRISPR‐Cas9系统转化后的存活。此外,有些系统构建了多个质粒来实现基因组编辑,需要额外的操作来减少这些质粒对宿主菌株的代谢负担,同时确保工程菌的安全性。因此,完善和拓展CRISPR 系统在枯草芽孢杆菌基因组编辑中的应用还有以下几个方面的工作要做。
3.1 开发用于枯草芽孢杆菌基因组编辑的不同CRISPR 核酸酶
Cas9 蛋白与crRNA/tracrRNA 双链结合成复合物,以介导侵入性双链DNA 的序列特异性切割,其中crRNA指导切割特异性,而Cas9蛋白中的2个核酸酶活性位点介导切割以产生DNA双链断裂[18,35]。大量对Cas9蛋白结构特征的研究促进了切割特异性更高的Cas9编辑变体的发展[36-39]。Hirano等[36]对来自新凶手弗朗西斯菌(Francisellanovicida)的FnCas9进行了特征分析,并开发了一个能够识别5’-YG’PAM 而不是原来的5’-NGGPAM的变体,增加了基因组编辑可用目标空间,即任何CG或TG后面的目标序列都容易被gRNA:FnCas9复合物靶向。此外,还可以使用其他具有不同PAM偏好的CRISPR核酸酶来增加基因组编辑的可用目标空间。
与Cas9 蛋白类似,Cpf1 蛋白是2 类CRISPR‐Cas系统中由单链RNA引导的内切酶,它起源于普氏菌属(Prevotella)或弗朗西斯菌属(Francisella)。与Cas9蛋白不同的是,Cpf1蛋白特异性识别前间隔序列5’端的TTTN PAM[40-42],且Cpf1:crRNA 复合物的形成只需要1个crRNA,并在目标DNA上切割产生5个核苷酸突出的黏性末端[42‐43],但Cas9:crRNA/tracrRNA 复合物的形成不仅需要crRNA,还需要1 个tracrRNA,并在目标DNA上切割产生平末端[18]。此外,Ran等[43]发现,CRISPR/Cpf1 的初级转录产物pre-crRNA 由Cpf1蛋白自身加工成熟,不需要RNase Ⅲ的参与,因此使用Cpf1 核酸内切酶进行基因组编辑可以是Cas9核酸内切酶之外的另一种选择。Wu等[27]对CRISPR/Cpf1系统进行工程设计,构建了一种功能强大的枯草芽孢杆菌多基因编辑和调控系统(CAMERS-B),该系统可实现双基因框内敲除、多点突变或单基因一次插入,效率可达100%。同时,Ran 等[43]还发现Cas9 中的1 个核酸酶活性位点(RuvCD10A或HNHH840A)发生突变,会得到不能切割DNA 的dCas9或者只能切割一条DNA 的nCas9,导致不能产生原来的平末端,该特性可以减少脱靶效应,并增强某些生物的同源重组修复。为了完善CRISPR系统在枯草芽孢杆菌基因组编辑中的应用,应针对不同的、可相互替代的CRISPR核酸酶进行全面优化,提高编辑效率并弥补现有基因操作方法的不足。
3.2 开发用于枯草芽孢杆菌基因组编辑的自靶向CRISPRCas系统
尽管CRISPR‐Cas免疫系统能严格区分自身DNA与外源DNA,但仍能够在细菌中发现自靶向间隔子(self-targeting spacers)[44]。一般情况下,宿主细胞的命运是死亡或严重生长迟缓、或去除自身靶点以保证继续存活[44-46],然而,Hale等[47]报道,自靶向CRISPR‐Cas系统及其宿主生物在某些条件下可共同存在。在这种特殊情况下,RNA 切割CRISPR‐Cas系统的crRNA引导其crRNA靶向并切割crRNA转录所在位点的短逆转录本。Zou等[31]构建的枯草芽胞杆菌CRISPR‐Cas9“多合一”单质粒系统,结合自靶向sgRNA 进行迭代编辑,一轮编辑周期仅2.5 d,是单质粒系统中最快的迭代编辑系统,在质粒上设计了Pspo0A严格控制下的自靶向sgRNA 用于清除质粒,效率可达90%,加速了枯草芽胞杆菌的工程细胞构建。Lim等[28]利用双质粒系统构建了一种高效、连续的枯草芽孢杆菌基因组编辑方法,该方法在pG2载体上连接了2个sgRNA—— 靶基因sgRNA (sgRNAct)和自靶向sgRNA (sgRNAst),其中sgRNAct 在生长阶段引导Cas9切割靶基因,sgRNAst在孢子体阶段引导Cas9切割sgRNA以清除质粒,由此产生的无sgRNA细胞进入下一轮编辑。该系统成功地在多轮编辑中删除掉8 个胞外蛋白酶基因,表明自靶向的CRISPR‐Cas系统在枯草芽孢杆菌基因组编辑中有巨大的应用潜力。此外,Zhang等[48]报道了一种基于内源性I-B型CRISPR‐Cas系统的高效酪丁酸梭菌(Clostridium tyrobutyricum)基因组编辑工具,该工具显著降低了CRISPR‐Cas的毒性,并提高了转化效率。虽然这种调控机制和自靶向系统的作用原理尚不清楚,但这些结果揭示了利用内源性CRISPR‐Cas系统调控枯草芽孢杆菌基因表达的可能性。内源性CRISPR‐Cas系统是一种理想的基因组编辑工具,它可以减弱经典CRISPR 系统(如Ⅱ型CRISPR‐Cas9 系统)的潜在脱靶效应和毒性。
3.3 开发不限于PAM 的枯草芽孢杆菌CRISPRCas9系统
CRISPR激活(CRISPR activation, CRISPRa)和CRISPR 抑制(CRISPR interference, CRISPRi)为基因表达调控提供了基础,但主要应用于真核生物,特别是缺乏原核CRISPRa研究[49],这可能是由于细菌激活元件需要精确的空间定位和与目标启动子的距离才能起作用,但基于Cas9的CRISPR工具只与NGG PAM序列相邻的位点结合。这些情况阻碍了Cas9引导的激活元件精确介导细菌中内源基因表达的上调。
Walton等[50]构建了首个几乎可以识别整个基因组序列的spCas9突变体——SpRY,它不再需要特定的PAM 序列。Klanschnig 等[51]用不依赖于PAM 的Cas9 变体SpRY 和噬菌体蛋白MCP 融合转录激活因子SoxS组合成CRISPRa,克服了PAM的限制,该工具被称为SMS;与之前报道的CRISPRa比较,SMS可以不依赖PAM上调大肠杆菌的报告基因质粒库;用scRNA(细胞质小RNA)替换gRNA 破坏MCP 与SoxS 的相互作用,实现SMS 在非NGG PAM 位点上的下调;同时使用scRNA和gRNA实现多基因表达控制;此外,还成功地利用SMS上调了内源基因和整合在大肠杆菌基因组上的gfp 基因。这项研究是CRISPRa在原核生物基因表达调控研究中的重要进展,为枯草芽孢杆菌等原核生物的基因组编辑研究提供了新的方向。
3.4 枯草芽孢杆菌高通量基因组修饰系统的开发
目前,在各种宿主细胞中构建的多种CRISPR-Cas9系统具有高效的基因组编辑能力,是适合开发和优化微生物细胞工厂的高通量技术。最近,Ronda等[52]将CRISPR‐Cas9和MAGE(multiplexautomated genome engineering)技术相结合,创造了一种高效、快速的大肠杆菌基因组工程方法CRMAGE(CRISPR‐Cas9 and λ Red recombineeringbased MAGE technology),使用CRMAGE 对大肠杆菌中3个基因组靶标进行基因重组,重组效率为96.5%~99.7%。Garst等[53]报道了CRISPR‐Cas9结合大量平行寡聚体的合成可以在全基因组范围内实现跟踪编辑,并基于此创造了CREATE(CRISPR enabled trackable genome engineering)基因组编辑方法,这是一种高效的基因组编辑策略,其通过大量平行寡聚体的合成构建CREATE 文库,可以在大肠杆菌的基因组中进行大规模的精准编辑。Liang 等[54]在大肠杆菌中使用CREATE策略生成640个单独的核糖体结合位点变体,然后利用这些单独的变体构建了组合文库用来选择生产异丙醇的最佳变体,与常规方法相比,CREATE策略可在较短时间内完成近1 000株大肠杆菌变体的合理构建和试验,并通过组合修饰提高异丙醇产量。此外,Bao 等[55] 开发了一种CRISPR‐Cas9和同源定向修复辅助的基因组规模工程方法(CHAnGE),该方法可以在酵母中快速输出数万个特定的遗传变异。在CHAnGE系统中,98% 以上的目标序列被有效编辑,平均频率为82%。由此表明,CRISPR‐Cas9 系统在高通量技术的发展中具有广阔的前景,为合理设计的枯草芽孢杆菌菌株的构建和测试以及生产改良提供了技术支持。此外,还应基于高通量技术开发更强大的多基因修饰系统,以增强枯草芽孢杆菌基因组编辑的全域性。
3.5 系统弱化CRISPR-Cas 编辑低重组效率细菌
细菌基因组编辑通常依赖于Cas核酸酶切割未编辑的细胞染色体DNA来反筛出发生编辑的细胞。然而,编辑通常需要高重组和转化效率,这在大多数菌株中是不具备的。即使是在模式菌中,有时也难以实现更大的插入、多重编辑或建库。
近期,Collias等[56]发现CRISPR驱动的同源重组可以通过降低DNA靶向活性提升转化子数量和编辑效率,从而在不同细菌中实现编辑。降低DNA 靶向活性可以通过改变gRNA 的序列结构( 使用CRISPR 阵列加工得到crRNA,再与tracrRNA相互作用,而非直接使用sgRNA)或表达强度;使用切割活性降低的核酸酶或设计减毒gRNAs(attenuated gRNAs,atgRNAs)扰动核酸酶结合支架、非典型PAMs或错配等来实现。这些修饰极大地增加了存活细胞数量,甚至提高了不同类型的Cas9和Cas12a在大肠杆菌和产酸克雷伯氏菌中的编辑效率。其中,最有效的方法是atgRNA,这种方法不需要重组酶,并且在不牺牲编辑效率的情况下增加了存活细胞数量。因此,减弱DNA 靶向提供了一种不同的方法来实现CRISPR驱动的细菌基因编辑,为枯草芽孢杆菌基因组编辑提供了新思路。
3.6 CreLoxP位点重组系统与CRISPRCas9基因编辑系统联用
Cre‐LoxP系统是源于P1噬茵体的DNA重组体系,由Cre重组酶和相应的LoxP位点组成,它能导致重组发生在特定的DNA序列处(LoxP位点),该系统可以将外源基因定点整合到染色体上或将特定DNA片段删除[57],因此在枯草芽孢杆菌基因组的连续编辑中被广泛应用于微生物菌株的代谢改造。
为了提高枯草芽孢杆菌基因组编辑的稳定性,并扩大其应用,Cai等[58]提出了Cre‐Cas系统的新概念,该系统将Cre‐LoxP 和CRISPR‐Cas9结合为一种简便有效的方法。携带表达盒的整合载体通过单交叉同源重组整合到枯草芽孢杆菌染色体上,然后pHT-Cre质粒表达Cre重组酶去除lox66和lox71位点两侧的选择标记,并将lox66和lox71位点之间的复制子重组为单个lox72位点。当不再需要外源基因表达盒时,使用辅助质粒pHPGCas9-gRNA,通过CRISPR‐Cas9 系统将表达盒移除并自我修复至其原始状态。为了验证该系统,使用T7和角蛋白酶表达盒,依据抗性基因的丢失或维持来评估自我修复效率,结果显示自我修复效率都在97%以上[58]。这一系统为枯草芽孢杆菌基因组编辑提供了新思路。
4 结语
枯草芽孢杆菌作为革兰氏阳性菌的重要模式菌种,在医药、食品、农业和生物材料等行业有着广泛的应用,如疫苗、酶制剂、饲料添加剂及其他生物化学品的生产。基于CRISPR 基因编辑系统,构建高效的枯草芽孢杆菌底盘细胞是提高其工业化应用的有效策略之一。然而,尽管枯草芽孢杆菌在工业上有着广泛的应用,但是与大肠杆菌的工业化应用相比,在基因工程改造方法层面还存在不少差距,其中,在对枯草芽孢杆菌基因组编辑的应用中,枯草芽孢杆菌质粒构建效率低,是目前新方法应用需要突破的瓶颈之一。
随着系统生物学、合成生物学及其他相关学科的快速发展,多学科的交叉结合应用将成为枯草芽孢杆菌底盘细胞设计与构建的重要方向。未来的研究应着眼于通过不同辅助技术的创造性和灵活性使用来解决枯草芽孢杆菌基因组编辑目前存在的问题,在保证遗传操作中枯草芽孢杆菌高重组率的同时,提高载体构建效率,并采用创新的方法和策略,使现有的枯草芽孢杆菌基因组编辑系统全面优化,这将大大提升枯草芽孢杆菌的工业应用潜能和范围。
参考文献
[1] EARL A M, LOSICK R, KOLTER R. Ecology and genomics of
Bacillus subtilis [J]. Trends Microbiol., 2008, 16(6):269-275.
[2] DEUTSCHER J. The mechanisms of carbon catabolite repression
in bacteria [J]. Curr. Opin. Microbiol., 2008, 11(2):87-93.
[3] NICOLAS P, MÄDER U, DERVYN E, et al.. Condition-dependent
transcriptome reveals high-level regulatory architecture in Bacillus
subtilis [J]. Science, 2012, 335(6072):1103-1106.
[4] CHEN J Q, ZHAO L Q, FU G, et al .. A novel strategy for
protein production using non-classical secretion pathway in
Bacillus subtilis [J/OL]. Microb. Cell Fact., 2016, 15(1):1-16
[2023-03-03]. https://doi.org/10.1186/s12934-016-0469-8.
[5] LIU Y, LIU L, LI J, et al .. Synthetic biology toolbox and
chassis development in Bacillus subtilis [J]. Trends
Biotechnol., 2019, 37(5):548-562.
[6] GU Y, XU X H, WU Y K, et al .. Advances and prospects of
Bacillus subtilis factories: from rational design to industrial
applications [J]. Metab. Eng., 2018, 50(5):109-121.
[7] XIANG M J, KANG Q, ZHANG D W. Advances on systems
metabolic engineering of Bacillus subtilis as a chassis cell [J].
Syn. Syst. Biotechnol., 2020, 5(4):245-251.
[8] WIDNER B, BEHR R, VON DOLLEN S, et al .. Hyaluronic
acid production in Bacillus subtilis [J]. Appl. Environ.
Microbiol., 2005, 71(7):3747-3752.
[9] CUI S X, LV X Q, WU Y K, et al .. Engineering a bifunctional
Phr60-Rap60-Spo0A quorum-sensing molecular switch for
dynamic fine-tuning of menaquinone-7 synthesis in Bacillus
subtilis [J]. ACS Synth. Biol., 2019, 8(8):1826-1837.
[10] LIU Y F, LIU L, SHIN H D, et al .. Pathway engineering of
Bacillus subtilis for microbial production of N-acetylglucosamine
[J]. Metab. Eng., 2013, 19:107-115.
[11] 张续,班睿,刘露,等.枯草芽孢杆菌基因修饰生产核黄素[J].
微生物学通报,2017,44(1):59-67.
ZHANG X, BAN R, LIU L, et al .. Riboflavin production by a
genetically modified Bacillus subtilis [J]. Microbiol. China,
2017, 44(1):59-67.
[12] DENG J Y, CHEN C M, GU Y, et al .. Creating an in vivo
bifunctional gene expression circuit through an aptamer-based
regulatory mechanism for dynamic metabolic engineering in
Bacillus subtilis [J]. Metab. Eng., 2019, 55:179-190.
[13] DEB S, CHOUDHURY A, KHARBYNGAR B, et al ..
Applications of CRISPR/Cas9 technology for modification of
the plant genome [J]. Genetica, 2022, 150(1):1-12.
[14] 林璐,吕雪琴,林延峰,等.枯草芽孢杆菌底盘细胞的设计、
构建及应用[J].合成生物学,2020,1(2):247-265.
LIN L, LYU X Q, LIN Y F, et al .. Advances in design,
construction and applications of Bacillus subtilis chassis cells
[J]. Synth. Biol. J., 2020, 1(2):247-265.
[15] ZHANG K, DUAN X, WU J. Multigene disruption in
undomesticated Bacillus subtilis ATCC 6051a using the
CRISPR/Cas9 system [J/OL]. Sci. Rep., 2016, 6(1): 27943
[2023-03-03]. https://doi.org/10.1038/srep27943.
[16] 张春晓,王丽丽,陈赟,等.β-甘露聚糖酶突变体食品级枯草
芽孢杆菌表达载体、表达系统、构建方法和应用:
CN114703163A [P].2022-07-05.
[17] MAKAROVA K S, WOLF Y I, IRANZO J, et al .. Evolutionary
classification of CRISPR-Cas systems: a burst of class 2 and
derived variants [J]. Nat. Rev. Microbiol., 2020, 18(2):67-83.
[18] JINEK M, CHYLINSKI K, FONFARA I, et al .. A programmable
dual RNA-guided DNA endonuclease in adaptive bacterial
immunity [J]. Science, 2012, 337(6096):816-821.
[19] LI T X, YANG Y Y, QI H Z, et al .. CRISPR/Cas9 therapeutics:
progress and prospects [J/OL]. Signal. Transduct. Tar., 2023, 8
(1): 36 [2023-04-23]. https://www.nature.com/articles/s41392-
023-01309-7.
[20] 吕秀琴,武耀康,林璐,等.枯草芽孢杆菌代谢工程改造的策
略与工具[J].生物工程学报,2021,37(5):1619-1636.
LYU X Q, WU Y K, LIN L, et al .. Strategies and tools for
metabolic engineering in Bacillus subtilis [J]. Chin. J. Biotechnol.,
2021, 37(5):1619-1636.
[21] ALTENBUCHNER J. Editing of the Bacillus subtilis genome
by the CRISPR-Cas9 system [J]. Appl. Environ. Microbiol.,
2016, 82(17):5421-5427.
[22] TOYMENTSEVA A A, ALTENBUCHNER J. New CRISPRCas9
vectors for genetic modifications of Bacillus species [J/OL].
FEMS Microbiol. Lett., 2019, 366(1):985 [2023-04-23]. https://
doi.org/10.1093/femsle/fny284.
[23] SACHLA A J, ALFONSO A J, HELMANN J D. A simplified
method for CRISPR-Cas9 engineering of Bacillus subtilis [J/OL].
Microbiol. Spectr., 2021, 9(2):e00754-21 [2023-04-23]. https://
doi.org/10.1128/Spectrum.00754-21.
[24] YU S, PRICE M A, WANG Y, et al .. CRISPR-dCas9 mediated
cytosine deaminase base editing in Bacillus subtilis [J]. ACS
Synth. Biol., 2020, 9(7):1781-1789.
[25] GARCÍA-MOYANO A, LARSEN Ø, GAYKAWAD S, et al ..
Fragment exchange plasmid tools for CRISPR/Cas9-mediated
gene integration and protease production in Bacillus subtilis [J/OL].
Appl. Environl. Microb., 2020, 87(1):e02090-20 [2023-04-23].
https://doi.org/10.1128/AEM.02090-20.
[26] HAO W L, SUO F, LIN Q, et al .. Design and construction of
portable CRISPR-Cpf1-mediated genome editing in Bacillus
subtilis 168 oriented toward multiple utilities [J/OL]. Front
Bioeng. and Biotech., 2020, 8: 524676 [2023-04-23]. https://
doi.org/10.3389/fbioe.2020.524676.
[27] WU Y K, LIU Y F, LV X Q, et al .. CAMERS-B: CRISPR/Cpf1
assisted multiple-genes editing and regulation system for
Bacillus subtilis [J]. Biotechnol. Bioeng., 2020, 117(6): 1817-
1825.
[28] LIM H, CHOI S K. Programmed gRNA removal system for
CRISPR-Cas9-mediated multi-round genome editing in
Bacillus subtilis [J/OL]. Front Microbiol., 2019, 10:1140 [2023-
04-23]. https://doi.org/10.3389/fmicb.2019.01140.
[29] JAKUTYTE-GIRAITIENE L, GASIUNAS G. Design of a
CRISPR-Cas system to increase resistance of Bacillus subtilis
to bacteriophage SPP1 [J]. J. Ind. Microbiol. Biot., 2016, 43(8):
1183-1188.
[30] WESTBROOK A W, MOO-YOUNG M, CHOU C P.
Development of a CRISPR-Cas9 tool kit for comprehensive
engineering of Bacillus subtilis [J]. Appl. Environl. Microbiol.,
2016, 82(16):4876-4895.
[31] ZOU Y, QIU L, XIE A W, et al .. Development and application
of a rapid all-in-one plasmid CRISPR-Cas9 system for iterative
genome editing in Bacillus subtilis [J/OL]. Microbiol. Cell
Fact., 2022, 21(1):173 [2023-04-23]. https://doi.org/10.1186/
s12934-022-01896-0.
[32] ZHANG X Z, YAN X, CUI Z L, et al .. mazF, a novel counterselectable
marker for unmarked chromosomal manipulation in
Bacillus subtilis [J/OL]. Nucl. Acids Res., 2016, 34(9): e71
[2023-04-23]. https://doi.org/10.1093/nar/gkl358.
[33] INÁCIO J M, COSTA C, DE SÁ-NOGUEIRA I. Distinct
molecular mechanisms involved in carbon catabolite repression of
the arabinose regulon in Bacillus subtilis [J]. Microbiology, 2003,
149(9):2345-2355.
[34] JIANG W Y, BIKARD D, COX D, et al.. CRISPR assisted editing
of bacterial genomes [J]. Nat. Biotechnol., 2013, 31(3):233-239.
[35] GASIUNAS G, BARRANGOU R, HORVATH P, et al .. Cas9-
crRNA ribonucleoprotein complex mediates specific DNA
cleavage for adaptive immunity in bacteria [J]. Proc. Natl.
Acad. Sci. USA, 2012, 109(39):2579-2586.
[36] HIRANO H, GOOTENBERG J S, HORII T, et al .. Structure
and engineering of Francisella novicida Cas9 [J]. Cell, 2016,
164(5):950-961.
[37] KLEINSTIVER B P, PATTANAYAK V, PREW M S, et al ..
High-fidelity CRISPR-Cas9 nucleases with no detectable
genome-wide off-target effects [J]. Nature, 2016, 529(7587):
490-495.
[38] KLEINSTIVER B P, PREW M S, TSAI S Q, et al .. Engineered
CRISPR-Cas9 nucleases with altered PAM specificities [J].
Nature, 2015, 23(7561):481-485.
[39] SIKSNYS V, GASIUNAS G. Rewiring Cas9 to target new PAM
sequences [J]. Mol. Cell, 2016, 61(6):793-794.
[40] DONG D, REN K, QIU X L, et al .. The crystal structure of
Cpf1 in complex with CRISPR RNA [J]. Nature, 2016, 532
(7600):522-526.
[41] YAMANO T, NISHIMASU H, ZETSCHE B, et al .. Crystal
structure of Cpf1 in complex with guide RNA and target DNA
[J]. Cell, 2016, 165(4):949-962.
[42] ZETSCHE B, GOOTENBERG J S, ABUDAYYEH O O, et al ..
Cpf1 is a single RNA-guided endonuclease of a class 2
CRISPR-Cas system [J]. Cell, 2015, 163(3):759-771.
[43] RAN F A, HSU P D, LIN C Y, et al .. Double nicking by RNAguided
CRISPR Cas9 for enhanced genome editing specificity [J].
Cell, 2013, 154(6):1380-1389.
[44] STERN A, KEREN L, WURTZEL O, et al .. Self-targeting by
CRISPR: gene regulation or autoimmunity? [J]. Trends Genet.,
2010, 26(8):335-340.
[45] GOMAA A A, KLUMPE H E, LUO M L, et al .. Programmable
removal of bacterial strains by use of genome-targeting
CRISPR-Cas systems [J/OL]. MBio, 2014, 5(1): e00928-13
[2023-04-23]. https://doi.org/10.1128/mBio.00928-13.
[46] VERCOE R B, CHANG J T, DY R L, et al .. Cytotoxic
chromosomal targeting by CRISPR/Cas systems can reshape
bacterial genomes and expel or remodel pathogenicity islands
[J/OL]. PLoS Genet., 2013, 9(4):e1003454 [2023-04-23]. https:
//doi.org/10.4161/mge.26831.
[47] HALE C R, MAJUMDAR S, ELMORE J, et al .. Essential
features and rational design of CRISPR RNAs that function
with the Cas RAMP module complex to cleave RNAs [J]. Mol.
Cell, 2012, 45(3):292-302.
[48] ZHANG J, ZONG W M, HONG W, et al .. Exploiting
endogenous CRISPR-Cas system for multiplex genome editing
in Clostridium tyrobutyricum and engineer the strain for
highlevel butanol production [J]. Metab. Eng., 2018, 47:49-59.
[49] CASAS-MOLLANO J A, ZINSELMEIER M H, ERICKSON S E,
et al.. CRISPR-Cas activators for engineering gene expression in
higher eukaryotes [J]. CRISPR J., 2020, 3(5):350-364.
[50] WALTON R T, CHRISTIE K A, WHITTAKER M N, et al ..
Unconstrained genome targeting with near-PAMless engineered
CRISPR-Cas9 variants [J]. Science, 2020, 368(6488):290-296.
[51] KLANSCHNIG M, CSERJAN-PUSCHMANN M, STRIEDNER
G, et al .. CRISPRactivation-SMS, a message for PAM sequence
independent gene up-regulation in Escherichia coli [J]. Nucleic
Acids Res., 2022, 50(18):10772-10784.
[52] RONDA C, PEDERSEN L E, SOMMER M O, et al ..
CRMAGE: CRISPR optimized MAGE recombineering [J/OL].
Sci. Rep., 2016, 6:19452 [2023-04-23]. https://doi.org/10.1038/
srep19452.
[53] GARST A D, BASSALO M C, PINES G, et al .. Genome-wide
mapping of mutations at singlenucleotide resolution for protein,
metabolic and genome engineering [J]. Nat. Biotechnol., 2017,
35(1):48-55.
[54] LIANG L Y, LIU R M, GARST A D, et al .. CRISPR EnAbled
Trackable genome Engineering for isopropanol production in
Escherichia coli [J]. Metab. Eng., 2017, 41(3):1-10.
[55] BAO Z H, HAMEDIRAD M, XUE P, et al .. Genome-scale
engineering of Saccharomyces cerevisiae with single-nucleotide
precision [J]. Nat. Biotechnol., 2018, 36(6):505-508.
[56] CALLIAS D, VIALETTO E, YU J, et al .. Systematically
attenuating DNA targeting enables CRISPR-driven editing in
bacteria [J/OL]. Nat. Commun., 2023, 14(1):680 [2023-04-23].
https://doi.org/10.1038/s41467-023-36283-9.
[57] YAN X, YU H J, HONG Q, et al .. Cre/lox system and PCRbased
genome engineering in Bacillus subtilis [J]. Appl.
Environ. Microbiol., 2008, 74(17):5556-5562.
[58] CAI M Z, CHEN P T. Novel combined Cre-Cas system for
improved chromosome editing in Bacillus subtilis [J]. J. Biosci.
Bioeng., 2021, 132(2):113-119.
