番茄CCCH类锌指蛋白家族的鉴定及其表达分析
2025-01-27李贤国戴麒王泽鹏陈兆龙闫会转李宁





摘要:CCCH(C3H)型锌指蛋白是进化上保守的RNA结合蛋白质,主要与DNA和RNA结合来调控基因表达,不仅参与植物的生长发育和激素调控过程,同时也响应生物和非生物胁迫。运用生物信息学方法,在番茄(Solanum Lycopersicum)中鉴定出47个SlC3H 基因,不均匀地分布在12条染色体上,主要定位于细胞核中。根据系统进化树将其分为3个亚族(Ⅰ~Ⅲ)。共线性分析发现,SlC3H 基因家族存在基因复制现象,表明在进化过程中SlC3H 基因可能通过复制进行家族成员扩增。番茄SlC3H 基因含有1~14个外显子,其启动子区域存在与激素和非生物胁迫响应的元件。Ka/Ks分析表明,SlC3H 基因家族成员和C3H 基因在进化过程中经历了强烈的进化选择。qRT-PCR 分析发现,在盐胁迫下,SlC3H 基因家族有一半以上的基因表达上调,其中SlC3H1 在8 h时表达量最高,是0 h的30倍;用0.1 mol·L−1 2,4-表油菜素内酯(2,4-epibrassinolide,EBR)处理后所有SlC3H 基因在绿熟期都表达上调,表达量是对照组的8~150倍,尤其是SlC3H28 在其他时期表达量几乎为0,但在绿熟期其表达量是对照组的150倍,推测EBR对果实绿熟期的发育有促进作用。上述研究结果为进一步揭示番茄C3H 基因家族在响应非生物胁迫以及果实发育中的功能提供了参考依据。
关键词:CCCH;番茄;生物信息学;表达分析
doi:10.13304/j.nykjdb.2023.0156
中图分类号:S641.2
文献标志码:A
文章编号:1008‑0864(2025)01‑0080‑16
CCCH(C3H)型锌指蛋白是进化上保守的RNA结合蛋白质,含有5个串联的C3H锌指结构域。C3H锌指转录因子(zinc finger,Znf)是一种新型的Znf 基因,其编码的蛋白包含1~6个C3H 类型的锌指基序,基序特征有3个基序半胱氨酸残基和1个组氨酸残基[1-4]。C3H型锌指蛋白主要与DNA和RNA结合,从而调控基因表达,但更多表现出与RNA 结合的特性,在RNA 加工中起作用[5]。C3H锌指基序已在人类和酵母的生物体蛋白中发现[6‑7]。在植物中,C3H型锌指蛋白不仅参与植物的生长发育、激素调控[8‑9],同时也参与生物和非生物胁迫的响应[10]。
拟南芥(Arabidopsis thaliana)中有11 个C3H家族成员,不仅参与植物对干旱、盐、冷和脱落酸(abscisic acid,ABA)等胁迫的响应,而且参与调控花发育[11]。此外,AtC3H14 和AtC3H15 被证实在拟南芥中调控次生细胞壁的生物合成,这也意味着它们的同源物可能也参与了拟南芥次生细胞壁的形成[12]。CCCH-ZFP 基因提高了西兰花对盐和渗透胁迫的耐受性[13]。水稻(Oryza sativa)中C3H 基因OsDOS 主要通过茉莉酸途径的负调控进行过度表达,使叶片延缓衰老[14]。棉花GhZFP1 转基因烟草对盐、干旱、水杨酸(salicylic acid,SA)胁迫和真菌疾病的耐受性显著增强[15]。
番茄(Solanum lycopersicum)是我国重要的经济作物之一,发掘其C3H 基因的潜在功能将为提高番茄产量、品质及抵抗逆境胁迫提供重要的理论支持[16]。目前,已在拟南芥、水稻[17]、玉米(Zeamays)[18]、玫瑰(Rosa rugosa)[19]、烟草(Nicotianatabacum)[20]、茄子(Solanum melongena)[21]、芸苔属(Brassica)[22‑23]、柑橘(Citrus reticulata Blanco)[24]和葡萄(Vitis vinifera)[25]中进行了全基因组C3H 鉴定,但在番茄中鲜有报道。鉴于此,本研究运用生物信息学方法鉴定番茄C3H 基因家族,研究这些基因的结构、保守结构域和组织特异性表达,并从系统进化、染色体分布、蛋白质理化性质等方面分析番茄C3H类锌指蛋白家族成员的特性;同时,分析家族成员基因启动子顺式作用元件,并进行荧光定量分析,为分析C3H参与番茄生长发育的机理提供理论依据,为进一步探究C3H类锌指蛋白在番茄应对非生物胁迫中的功能机理奠定理论基础。
1 材料与方法
1.1 供试材料
以番茄栽培品种‘M82’为研究材料,种子由新疆农业科学院园艺作物研究所提供。将‘M82’种子清洗后放置培养皿中进行萌发,挑选长势一致的番茄幼苗,移入1/2改良型霍兰格营养液中进行培养,待番茄幼苗长至4叶期用200 mol·L−1的NaCl溶液进行胁迫处理,每个处理3次重复,分别在处理0.0、0.5、2.0、4.0、6.0、8.0、12.0 h 时采集叶片。另采集膨大期(expansion period,EP)、绿熟期(green ripe period,GRP)、转色期(veraiso period,VP)、红熟期(red ripe period,RRP)4 个时期的番茄果实作为材料,分别采用0.00(E1)、0.05(E2)、0.10(E3)、0.20 mol·L−1(E4)的外源激素2,4-表油菜素内酯(2,4-epibrassinolide, EBR)处理番茄果实,连续喷施3 d后再自然生长3 d,于第7天进行果实取样。为避免相互干扰,每3株设为1个分区,共有7个区组,各个区组之间距离设置为1.5 m。收集的材料放入液氮中速冻,并贮存在−80 ℃冰箱。
1.2 番茄SlC3H 基因家族鉴定
为鉴定番茄中潜在的C3H 家族基因,从番茄基因组数据库(http://solomics.agis.org.cn/tomato/)下载番茄亨氏1705 基因组数据,并通过EnsemblPlants数据库(http://plants.ensembl.org/)获得拟南芥和烟草的基因组数据;然后从Pfam数据库(http://pfam-legacy.xfam.org/)下载C3H 结构域隐马尔可夫模型(ID:PF00642),并利用Hmmer工具对番茄C3H 全基因组蛋白质进行搜索和结构域比较,E-value≤1×10−5[26]。最后,通过NCBI(https://www.ncbi.nlm.nih.gov/)中Conserved Domain Database程序和Pfam(http://pfam-legacy.xfam.org/)在线软件中Search程序对所有获得的蛋白进行验证。通过Expasy在线软件(https://web.expasy.org/tools/)预测番茄C3H蛋白的理化性质[27]。采用Wolf Psort II(http://www.genscript.com/wolf-psort.html)进行亚细胞定位预测分析[28]。
1.3 番茄SlC3H 家族基因结构分析
通过MEGA 7.0软件中的Muscle对番茄、拟南芥和烟草的C3H 蛋白序列进行比对,并采用邻接法进行分析,采用最大似然法(maximumlikelihood,ML)构建系统进化树,Bootstrap 重复值为1 000,使用在线网站EVOLVIEW(https://www. evolgenius. info/evolview-v2/)美化进化树[29]。采用DNAMAN 进行结构域序列多重比对,利用MEME(https://meme-suite. org/tools/meme/)进行保守基序分析[30],通过TBtools 软件绘制基因结构图[31]。
1.4 番茄SlC3H 基因的染色体定位、共线性和Ka/Ks 分析
根据SlC3H 基因的染色体位置信息,使用TBtools 软件绘制位置分布图。其中采用MCScanX 进行番茄和拟南芥C3H 基因以及番茄和烟草C3H 基因的共线性分析[32];Circos工具用于生成染色体共线性图[33];通过Ka/ks工具获得番茄共线基因对的Ka/Ks比值[34]。
1.5 番茄SlC3H 基因顺式元件分析
利用TBtools软件提取SlC3H 基因上游2 000 bp的序列,通过Plant Care 在线网站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测启动子区域的顺式作用元件。最后用TBtools软件中的HeatMap工具制作表达热图。
1.6 SlC3H 基因的qRT-PCR
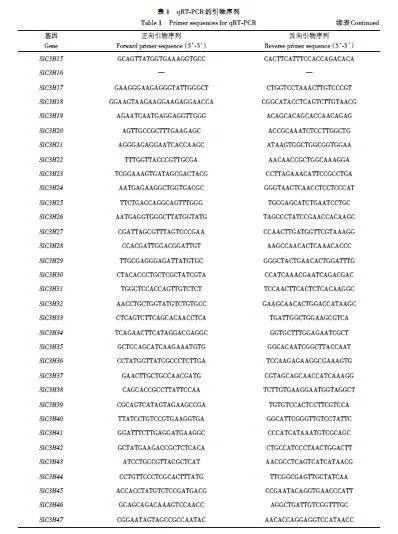
使用多糖多酚植物总RNA 提取试剂盒(DP432,北京天根生化科技有限公司)提取处理后番茄的总RNA, 通过BiomarkerScirpt-1stStrandcDNA-SynthesisKit反转录试剂盒(北京百迈客生物科技有限公司)合成cDNA,利用ChamQSYBR qPCR Master Mix荧光定量试剂盒(南京诺唯赞生物科技股份有限公司)进行qRT-PCR 反应。根据番茄C3H 基因家族成员保守基序设计荧光定量引物( 表1)。qRT-PCR 反应体系20 μL:SuperReal Color PreMix(北京天根生化科技有限公司)10 μL、上下游特异性引物各0.4 μL,cDNA模板1 μL,ddH2O补到20 μL。PCR程序:94 ℃预变性120s;94 ℃变性5 s,(退火)15 s,72 ℃延伸10 s,45个循环。以番茄Actin 为内参基因。采用3个生物重复和2−△△CT法[35]计算,相对表达量为处理组和对照组的相对值。
2 结果与分析
2.1 番茄SlC3H 基因家族成员特征分析
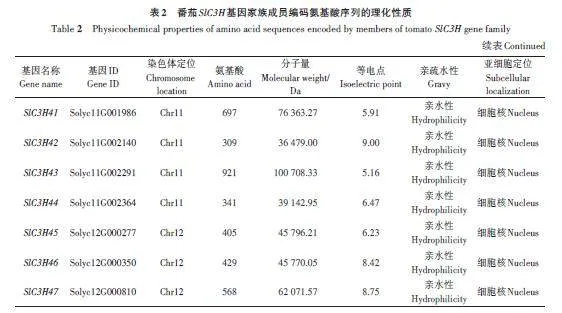
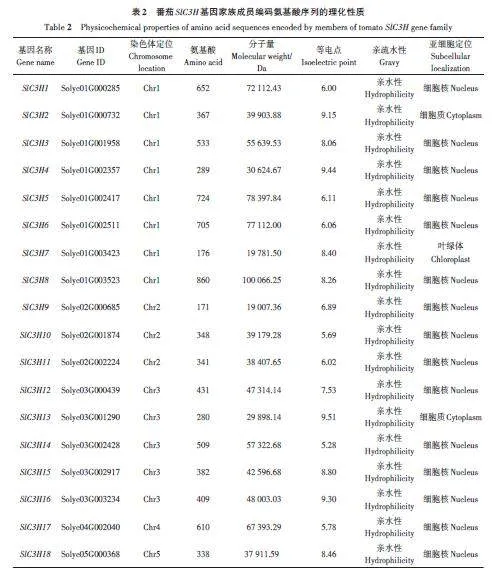
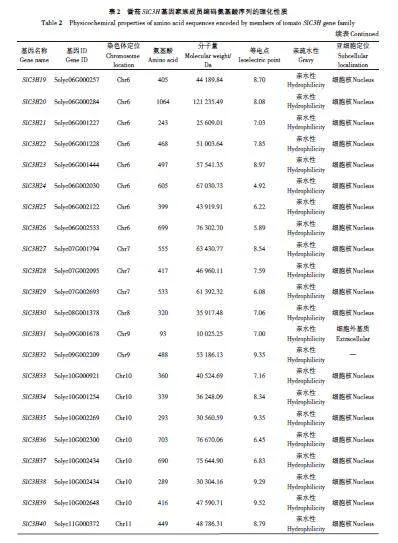
通过全基因组鉴定分析,初步鉴定到47个潜在的SlC3H 基因,依据家族成员在基因组染色体的位置将其命名为SlC3H1~SlC3H47。蛋白质理化性质分析结果(表2)表明,SlC3H 家族成员的氨基酸残基数为93~1 064个,蛋白分子量为10 025.25~121 235.49 Da,其中SlC3H31 最小,SlC3H20 最大;等电点为4.92~9.52,SlC3H24 最低,SlC3H39 最高;SlC3H 基因家族蛋白均表现为亲水性。亚细胞定位预测结果表明,SlC3H2 和SlC3H13 位于细胞质,SlC3H7 位于叶绿体上,SlC3H31 处于细胞外基质,其余均位于细胞核内。
2.2 番茄SlC3H 基因家族染色体定位分析
染色体定位分析结果如图1所示。47个番茄SlC3H 基因家族成员分布在12条染色体上,其中,多数SlC3H 基因主要分布于Chr3(5个)、Chr1(8个)、Chr6(8个)、Chr10(7个)、Chr11(5个),其余7条染色体上C3H基因家族成员数量为1~3个。番茄SlC3H基因家族成员在同一条染色体上的分布也不均匀,大多分布在各染色体的上端和下端,除了Chr6、Chr7、Chr10、Chr11和Chr12染色体上的C3H 基因家族成员分布较为集中外,其余染色体上的C3H基因家族成员分布较为分散。
2.3 番茄SlC3H 基因的系统进化分析
为明确C3H 基因的系统进化关系,对拟南芥(AtC3H,50个)、烟草(NtC3H,49个)和番茄(SlC3H,47个)的146个C3H保守结构域的蛋白序列构建系统进化树,结果如图2所示。将这3个物种的146个C3H基因划分为Ⅰ、Ⅱ、Ⅲ 3个组,同一组内的C3H基因家族成员亲缘关系较近。其中Ⅰ组有62个C3H,包含16个SlC3H、24个AtC3H 和22个NtC3H;Ⅱ组有19个C3H 基因,包含6个SlC3H、9个AtC3H和4个NtC3H;Ⅲ组有65个C3H 基因,包含25个SlC3H、17个AtC3H 和23个NtC3H。同时,Ⅰ组又可划分为Ⅰ-a、Ⅰ-b 2个亚组,分别包含6和10个SlC3H家族成员;Ⅲ组又可划分为Ⅲ-a、Ⅲ-b 2个亚组,分别有11和14个SlC3H。同一组内的基因通常具有相似的功能特征,多数番茄SlC3H 基因处于Ⅰ组与Ⅲ组内。
2.4 番茄SlC3H 基因家族成员基序和基因结构分析
由图3 可知,C3H 蛋白有不同的基序类型。所有SlC3H 蛋白都含有motif 1,SlC3H36、SlC3H45、SlC3H30、SlC3H28、SlC3H25、SlC3H44、SlC3H20、SlC3H16、SlC3H23、SlC3H15、SlC3H9、SlC3H31、SlC3H2、SlC3H40、SlC3H21 只含编码区(coding sequence,CDS),其他基因既有CDS,又有非翻译区(untranslated region,UTR)。基因结构分析发现,番茄SlC3H 基因的内含子数为0~14个,其中有5个基因(SlC3H37、SlC3H26、SlC3H5、SlC3H25、SlC3H44)仅含1 个内含子,有4 个基因(SlC3H36、SlC3H28、SlC3H45、SlC3H30)不含内含子。内含子是阻断基因线性表达的序列,增加了基因的长度,提高了基因间的重组频率,有利于物种进化并具有调控作用。有内含子的5个SlC3H 基因可能具有某项功能,但对番茄进化作用不明显;其余4个不含有内含子的SlC3H 基因推测其可能不具有调控作用,且不利于番茄物种进化。结合进化树分析可知,处于同一组内的基因家族成员亲缘关系较近,例如,SlC3H36、SlC3H37、SlC3H5 和SlC3H26 在同一组内,具有较近的亲缘关系,并且由保守基序可知,这4 个SlC3H 基因具有相似的基因结构,即同组成员的motif 高度相似。由此可知,同一组内成员的关系较近且基因结构相似,可能具有相似的功能。
2.5 番茄SlC3H 基因家族成员共线性和进化压力分析
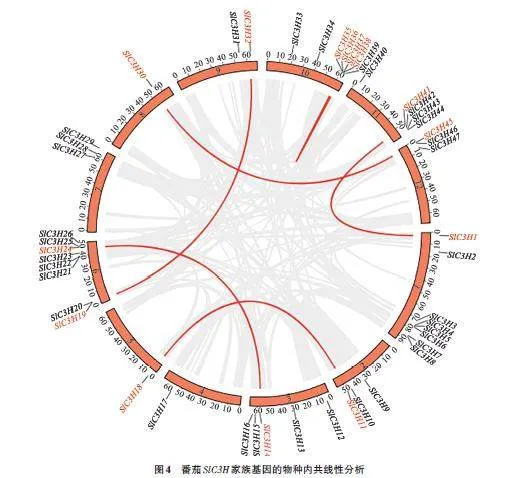
构建番茄SlC3H 基因的共线性图,结果(图4)表明,有7对番茄SlC3H 基因具有同源关系,其成员之间存在共线性关系,分别是SlC3H1 与SlC3H41、SlC3H11与SlC3H18、SlC3H14与SlC3H24、SlC3H19 与SlC3H32、SlC3H30 与SlC3H45、SlC3H35与SlC3H38、SlC3H36 与SlC3H37,其余成员间不存在共线性关系。由上可知,SlC3H 基因家族存在基因复制现象,表明在进化过程中C3H 基因可能通过复制进行家族成员扩增。
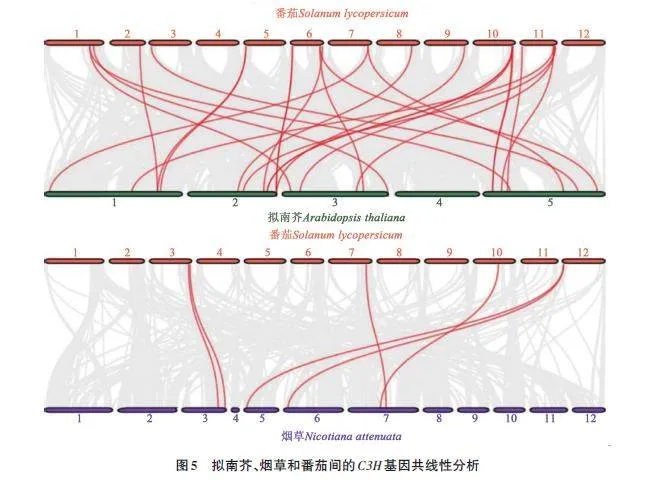
为进一步探索C3H 基因家族成员间的进化关系,构建了番茄SlC3H 与拟南芥AtC3H 家族以及番茄SlC3H 与烟草NtC3H 家族的比较共线性图,结果(图5)表明,番茄、拟南芥和烟草中存在直系同源C3H 基因对,其中番茄与拟南芥之间的同源基因对数量多于番茄与烟草的同源基因对。由此说明,番茄与拟南芥的C3H 基因家族具有更近的同源进化关系。
为探明物种间SlC3H 基因家族和C3H 基因的进化,计算番茄中SlC3H 基因的复制和共线C3H基因之间的Ka/Ks值,结果(表3)显示,6个SlC3H基因对的Ka/Ks值均小于1,表明SlC3H 在进化时受到纯化选择。由此可知,SlC3H 基因家族成员和C3H 基因在进化过程中经历了强烈的选择作用。
2.6 番茄SlC3H 基因家族启动子顺式作用元件分析
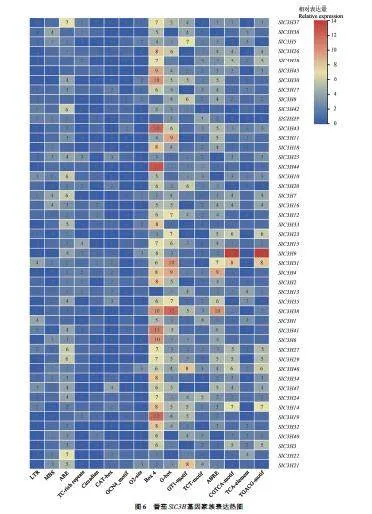
对番茄SlC3H 基因家族成员的顺式作用元件进行分析,按照其功能可分为光响应元件、胁迫响应元件、植物激素响应元件和植物生长发育元件(图6)。光响应元件(Box 4、G-box、GT1-Box 4的数量最多(300个),G-box次之(195个);SlC3H44 基因中的Box 4元件数量最多,SlC3H43与SlC3H19 次之;SlC3H38 基因中的G-box 元件数量最多。Box 4 是参与光反应的一部分DNA模块,G-box 是参与光反应的顺式作用元件,由此推断,这些基因主要调控特征与光有关。在胁迫响应元件中,ARE 元件决定调节蛋白的结合与厌氧诱导的表达,其响应元件数量最多,共142 个,且ARE 元件在所有47 个基因中均被检测到,这说明番茄SlC3H 基因可能与番茄对厌氧响应密切相关。与干旱相关的MBS元件的数量排在第2位,共62个,其中有37个启动子检测到MBS 元件。低温(low temperature response,LTR)响应元件共39 个,防御和应激(TC-richrepeats)反应元件共40 个。除此之外还发现了一些与植物生长发育有关的元件,其中分生组织表达相关的启动子元件最多,说明其可能促使细胞进行分裂分化,进而对番茄生长发育起调控作用;其他还包括玉米醇溶蛋白代谢(O2-site)、胚乳表达(GCN4-motif)和昼夜节律(circadian)相关的顺式作用元件。
与激素响应相关的顺式作用元件有3种,包括脱落酸响应元件(ABRE)、茉莉酸甲酯响应元件(CGTCA-motif、TGACG-motif)以及水杨酸响应元件(TCA-element)。茉莉酸甲酯能够促使番茄植株产生防御反应,从而表现出较强的抗病能力,其响应元件最多,共246个;脱落酸能够有效控制番茄生长、提高抗逆性,其响应元件次之,共154 个;水杨酸既可以诱导番茄细胞分化,又可以参与番茄植株对病原体的抗性,其响应元件最少,共54 个。这说明多种激素可参与调控C3H 基因的表达,共同促进番茄的生长,提高番茄的抗逆性。
2.7 番茄SlC3H 基因响应盐胁迫和激素的表达模式分析
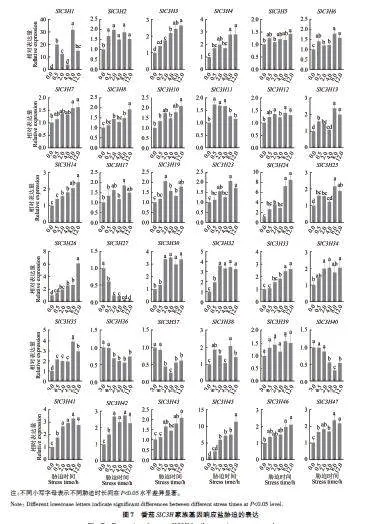
盐胁迫是制约农业生产的重要环境因素,鉴定盐胁迫相关基因以培育耐盐品种是目前亟需解决的问题。为了解番茄在盐胁迫及果实生长发育处理下SlC3H 基因的表达水平,利用qRT-PCR检测番茄各器官中SlC3H 基因及其下游基因的转录水平,挑选了36个SlC3H 基因进行了qRT-PCR分析,结果(图7)显示,在盐胁迫下,SlC3H14、SlC3H10、SlC3H8、SlC3H3、SlC3H46、SlC3H7、SlC3H41、SlC3H42、SlC3H43、SlC3H45、SlC3H23、SlC3H24、SlC3H30、SlC3H32、SlC3H35、SlC3H1、SlC3H33和SlC3H26 表达上调,尤其是SlC3H1 在8h 达到最大值,较0 h 的表达量上升了30 倍;SlC3H27、SlC3H40、SlC3H36 和SlC3H37 表达下调,推测这4个基因在盐胁迫下起负调控作用;剩下的SlC3H2和SlC3H12在盐胁迫下均匀表达。
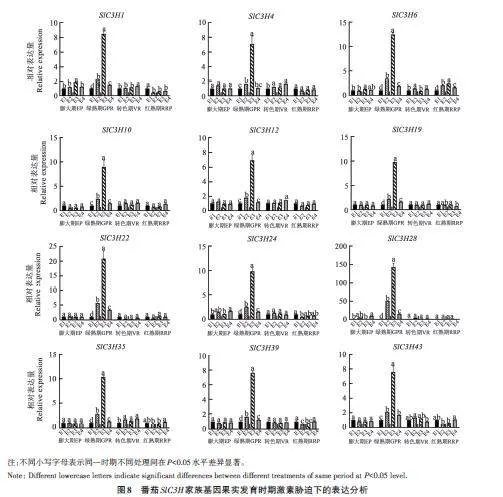
为了进一步研究激素胁迫下番茄SlC3H 基因在果实中表达,挑选12个SlC3H 基因进行了qRTPCR分析,结果(图8)显示,在0.1 mol·L−1 EBR处理下,12个SlC3H 基因在绿熟期均表达上调,表达量是对照组的8~150倍,尤其是SlC3H28,该基因在其他时期的表达量几乎为0,但在绿熟期其表达量是对照组的150倍。因此,推测EBR对果实绿熟期的发育有促进作用,其他时期基因都均匀表达。
3 讨论
C3H蛋白在植物生长发育的许多方面发挥着重要作用。前人对C3H 基因家族成员进行了大量研究,在拟南芥和水稻中分别鉴定出68和67个[17],柑橘中62个[24],杨树中91个[37],香蕉中89个[2],烟草中86个[20],然而在番茄中鲜有报道。对番茄C3H基因进行全基因组分析,有助于更好地了解该基因家族。本研究从番茄基因组中鉴定出47个SlC3H 基因,分析基因结构发现,SlC3H基因的内含子数量为0~14个,其中有5个基因仅含1个内含子,有4个基因不含内含子;序列分析表明,SlC3H 基序高度保守,处于同一亚家族的SlC3H 基因结构域高度相似,表明这些SlC3H 基因可能具有相似的功能;系统发育树分析将47个SlC3H 基因划分为3个亚族,亚族内的C3H基因有较高的同源性,同源基因发生复制时可能来自同一个祖先,推测其具有相似的功能。
基因复制是导致基因家族快速扩张和进化的主要因素[38]。本研究发现7对同源基因,其成员之间存在共线性关系,这与香蕉中的9对相似[2]。这一结果进一步表明,C3H 基因具有高度的同源性,家族成员存在基因复制现象,表明在进化过程中C3H 基因可能通过复制进行家族成员扩增。SlC3H 基因家族成员中有6对同源基因的Ka/Ks值小于1,表明这些基因主要受进化选择的影响。
顺式元件在基因表达的转录调控中发挥着重要作用[39]。本研究分析了SlC3H 基因上游2 000 bp启动子区域的顺式作用元件,检测到多种与植物生长发育相关的元件,包括参与细胞周期调控的顺式作用元件、激素响应元件以及与分生组织表达相关的顺式作用调控元件;同时还检测到与非生物胁迫相关的顺式元件,如参与防御和胁迫响应的顺式作用元件、参与低温响应的顺式作用元件,以及参与干旱诱导的MYB结合位点。因此,顺式作用元件分析为SlC3H 成员的功能研究,特别是相关基因的调控和植物在不同胁迫下的发育提供参考。
本研究运用实时荧光定量PCR 首次对番茄SlC3H 基因家族的表达模式进行了全面分析,结果表明,在NaCl胁迫时,SlC3H基因家族中有一半以上的基因上调表达,这与芝麻中的SiC3H1和香蕉中的CCCH-ZFP 表达相似[2,40],而SlC3H27、SlC3H40、SlC3H36 和SlC3H37 下调表达,这与水稻中的OsC3H30、OsC3H50、OsC3H52 的表达模式一致[41],说明它们可能起负调控作用。拟南芥CCCH类锌指蛋白还参与激素响应[11,42],本研究中SlC3H基因在激素处理后表达量发生变化,说明SlC3H 基因参与番茄果实的生长发育,同时在0.1 mol·L−1的EBR处理后,SlC3H基因在绿熟期表达量显著升高,SlC3H28在激素处理后表达量提升了150倍,这与烟草NtC3H39基因的表达模式相似,进一步说明番茄SlC3H 基因家族的功能与拟南芥CCCH类锌指蛋白相似[43]。
参考文献
[1] GAO G, GUO X, GOFF S P, et al .. Inhibition of retroviral RNAproduction by ZAP, a CCCH-type zinc finger protein [J].Science, 2002, 297(5587):1703-1706.
[2] MAZUMDAR P, LAU S E, WEE W Y, et al .. Genome-wideanalysis of the CCCH zinc-finger gene family in banana (Musaacuminata): an insight in to motif and gene structurearrangement, evolution and salt stress responses [J]. Trop.Plant Biol., 2017, 10(11):177-193.
[3] DRÖGE-LASER W, SNOEK B L, SNEL B, et al .. TheArabidopsis bZIP transcription factor family-an update [J].Curr. Opin. Plant Biol., 2018, 45(PtA):36-49.
[4] SU L Y, XIAO X C, JIANG M Q, et al .. Genome-wide analysisof the CCCH zinc finger family in longan: characteristicidentification and expression profiles in Dimocarpus longanLour [J]. J. Integr. Agric., 2022, 21(1):113-130.
[5] DELANEY K J, XU R, ZHANG J, et al .. Calmodulin interactswith and regulates the RNA-binding activity of an Arabidopsispolyadenylation factor subunit [J]. Plant Physiol., 2006, 140 (4):1507-1521.
[6] CARRICK D M, LAI W S, BLACKSHEAR P J. The tandemCCCH zinc finger protein tristetraprolin and its relevance tocytokine mRNA turnover and arthritis [J]. Arthritis Res. Ther.,2004, 6 (6):248-264.
[7] DE J, LAI W S, THORN J M, et al .. Identification of four CCCHzinc finger proteins in Xenopus, including a novel vertebrateprotein with four zinc fingers and severely restricted expression[J]. Gene, 1999, 228(1-2):133-145.
[8] BOGAMUWA S P, JANG J C. Tandem CCCH zinc fingerproteins in plant growth, development and stress response [J].Plant Cell Physiol., 2014, 55(8):1367‑1375.
[9] ZHANG L Y, BAI M Y, WU J, et al .. Antagonistic HLH/bHLHtranscription factors mediate brassinosteroid regulation of cellelongation and plant development in rice and Arabidopsis [J].Plant Cell, 2009, 21(12):3767-3780.
[10] ZHANG H, GAO X, ZHI Y, et al .. A non‑tandem CCCH‑typezinc-finger protein, IbC3H18, functions as a nuclear transcriptionalactivator and enhances abiotic stress tolerance in sweet potato [J].New Phytol., 2019, 223(4):1918‑1936.
[11] KIM D H, YAMAGUCHI S, LIM S, et al .. SOMNUS, a CCCHtypezinc finger protein in Arabidopsis, negatively regulateslight-dependent seed germination downstream of PIL5 [J].Plant Cell, 2008, 20(5):1260-1277.
[12] LI J, JIA D, CHEN X. HUA1, a regulator of stamen and carpelidentities in Arabidopsis, codes for a nuclear RNA bindingprotein [J]. Plant Cell, 2001, 13(10):2269-2281.
[13] JIANG M, JIANG J J, MIAO L X, et al.. Over-expression of a C3Htypezinc finger gene contributes to salt stress tolerance intransgenic broccoli plants [J]. Plant Cell, 2017, 130(2):239-254.
[14] PRADHAN S, KANT C, VERMA S, et al .. Genome-wideanalysis of the CCCH zinc finger family identifies tissuespecific and stress responsive candidates in chickpea (Cicerarietinum L.) [J/OL]. PloS One, 2017, 12(7):e0180469 [2023-02-05]. https://doi.org/10.1371/journal.pone.0180469.
[15] GUO Y H, YU YP, WANG D, et al .. GhZFP1, a novel CCCHtypezinc fifinger protein from cotton, enhances salt stresstolerance and fungal disease resistance in transgenic tobaccoby interacting with GZIRD21A and GZIPR5 [J]. New Phytol.,2009,183(1):62-75.
[16] 刘雨轩.番茄WRKY 基因家族成员鉴定及表达分析[D].沈阳:沈阳农业大学,2020.
LIU Y X. Identification and expression analysis of tomatoWRKY gene family members [D]. Shenyang: ShenyangAgricultural University, 2020.
[17] WANG D, GUO Y, WU C, et al .. Genome-wide analysis ofCCCH zinc finger family in Arabidopsis and rice [J]. BMCGenomics, 2008, 9(1):44-64.
[18] PENG X, ZHAO Y, CAO J, et al .. CCCH-type zinc finger family in maize: genome-wide identification, classification andexpression profiling under abscisic acid and droughttreatments [J/OL]. PloS One, 2012, 7(7): e40120 [2023-02-05].https://doi.org/10.1371/journal.pone.0040120.
[19] LI C, FANG Q, ZHANG W, et al.. Genome-wide identification ofthe CCCH gene family in rose (Rosa chinensis Jacq.) reveals itspotential functions [J]. Biotechnol. Equip., 2021, 35(1):517-526.
[20] 唐春闺,邓兆龙,刘琼,等.普通烟草CCCH类锌指蛋白家族的全基因组鉴定和表达分析[J]. 河南农业科学, 2022, 51(4): 48-58.
TANG C G, DENG Z L, LIU Q, et al .. Genome wide identificationand expression analysis of CCCH zinc finger like protein family intobacco [J]. J. Henan Agric. Sci., 2022, 51(4):48-58.
[21] XU R. Genome-wide analysis and identification of stressresponsivegenes of the CCCH zinc finger family in Solanumlycopersicum [J]. Mol. Genet. Genomics, 2014, 289(5):965-979.
[22] PI B, HE X, RUAN Y, et al .. Genome-wide analysis and stressresponsiveexpression of CCCH zinc finger family genes inBrassica rapa [J/OL]. BMC Plant Biol., 2018, 18(1):7 [2023-02-05]. https://doi.org/10.1186/s12870-018-1608-7.
[23] PI B, PAN J, XIAO M, et al .. Systematic analysis of CCCH zincfinger family in Brassica napus showed that BnRR-TZFs areinvolved in stress resistance [J/OL]. BMC Plant Biol., 2021, 21(1):8 [2023-02-05]. https://doi.org/10.1186/s12870-021-03340-8.
[24] LIU S, KHAN M R G, LI Y, et al .. Comprehensive analysis ofCCCH-type zinc finger gene family in citrus (Clementinemandarin) by genome-wide characterization [J]. Mol. Genet.Genomics, 2014, 289(5): 855-872.
[25] WANG X L, ZHONG Y, CHENG Z M. Evolution andexpression analysis of the CCCH zinc finger gene family inVitis vinifera [J]. Plant Genome, 2014, 7(3):16-25.
[26] PRAKASH A, JEFFRYES M, BATEMAN A, et al .. TheHMMER web server for protein sequence similarity search [J].Curr. Prot. Bioinf., 2017, 60:15-23.
[27] GASTEIGER E, GATTIKER A, HOOGLAND C, et al ..ExPASy: the proteomics server for in-depth protein knowledgeand analysis [J]. Nucl. Acids Res., 2003, 31(13):3784-3788.
[28] HORTON P, PARK K J, OBAYASHI T, et al .. WoLF PSORT:protein localization predictor [J]. Nucl. Acids Res., 2007, 35:585-587.
[29] KUMAR S, STECHER G, LI M, et al .. MEGA X: molecularevolutionary genetics analysis across computing platforms [J].Mol. Biol. Evol., 2018, 35(6):1547-1579.
[30] BAILEY T L, BODEN M, BUSKE F A, et al .. MEME SUITE:tools for motif discovery and searching [J]. Nucl. Acids Res.,2009, 37:202-208.
[31] CHEN C, CHEN H, ZHANG Y, et al .. TBtools: an integrativetoolkit developed for interactive analyses of big biological data [J].Mol. Plant, 2020, 13(8):1191-1202.
[32] WANG Y P, TANG H B, DEBARRY J D, et al .. MCScanX: atoolkit for detection and evolutionary analysis of gene syntenyand collinearity [J/OL]. Nucl. Acids Res., 2012, 40(7): e49[2023-02-05]. https://doi.org/10.1093/nar/gkr1293.
[33] KRZYWINSKI M, SCHEIN J, BIROL I, et al .. Circos: aninformation aesthetic for comparative genomics [J]. GenomeRes., 2009, 19(9):1639-1645.
[34] WANG D, ZHANG Y, ZHANG Z, et al.. KaKs_Calculator 2.0: atoolkit incorporating gamma-series methods and sliding windowstrategies [J]. Genom Proteom. Bioinf., 2010, 8(1):77-80.
[35] LIVAK K J, SCHMITTGEN T D. Analysis of relative geneexpression data using real-time quantitative PCR and the2-ΔΔCT method [J]. Methods, 2001, 25(4):402-408.
[36] YAO Q, BAI Y, KUMAR S, et al.. Minimal residual diseasedetection by next-generation sequencing in multiple myeloma: Aomparison with real-time quantitative PCR [J/OL]. Front. Oncol.,2020, 10: 611021 [2023-02-05]. https://doi.org/10.1111/bjh.15075.
[37] CHAI G, HU R, ZHANG D, et al .. Comprehensive analysis ofCCCH zinc finger family in poplar (Populus trichocarpa ) [J/OL].BMC Genomics, 2012, 13(1):253 [2023-02-05]. https://doi.org/10.1186/1471-2164-13-253.
[38] JOUFFREY V, LEONARD A S, AHNERT S E. Geneduplication and subsequent diversification strongly affectphenotypic evolvability and robustness [J]. Roy Soc. Open Sci.,2021, 8(6):1636-1642.
[39] WANG W, JIANG W, LIU J G, et al .. Genome-widecharacterization of the aldehyde dehydrogenase gene superfamilyin soybean and its potential role in drought stress response [J/OL].BMC Genomics, 2017, 18(1): 518 [2023-02-05]. https://doi. org/10.1186/s12864-017-3908-y.
[40] 魏梦园,刘爱丽,黎冬华,等. 芝麻CCCH 锌指蛋白基因SiC3H1 的克隆及表达分析[J].分子植物育种,2020,18(24):7982-7988.
WEI M Y, LIU A L, LI D H, et al .. Cloning and expressionanalysis of CCCH zinc finger protein gene SiC3H1 fromsesame [J]. Mol. Plant Breed., 2020, 18(24):7982-7988.
[41] 刘小艳,孙艳侠,王亚男,等.水稻CCCH型锌指蛋白亚家族Ⅰ基因的表达分析[J]. 山东农业科学,2015,47(2):7-11.
LIU X Y, SUN Y X, WANG Y N, et al .. Expression analysis ofCCCH zinc finger protein subfamily Ⅰ genes in rice [J].Shandong Agric. Sci., 2015, 47(2):7-11.
[42] VERMA V, RAVINDRAN P, KUMAR P P. Plant hormonemediatedregulation of stress responses [J]. BMC Plant Biol.,2016, 16:1-10.
[43] LIN P C, POMERANZ M C, JIKUMARU Y, et al .. TheArabidopsis tandem zinc finger protein AtTZF1 affects ABA‑andGA‑mediated growth, stress and gene expression responses [J].Plant J., 2011, 65(2):253-268.
基金项目:国家自然科学基金项目(32260763);新疆农业科学院科技创新专项孵化项目(xjkcpy-2021001)。
