嗜热毁丝霉木糖代谢调控机制研究
2024-12-31陈美欣顾淑莹刘佳刘浩李金根田朝光






摘要:为提高发酵菌株的木糖利用速率,以嗜热毁丝霉为体系,利用遗传学手段及转录组学技术,探究丝状真菌嗜热毁丝霉木糖代谢的调控机制。结果表明,在嗜热毁丝霉中,转录因子XlnR能够正向调控木糖代谢和转运以及半纤维酶的表达,是木糖条件下孢子萌发的必需因子。在木糖条件下,突变体ΔxlnR孢子丧失了萌发的能力,并且菌丝木糖利用速率显著降低,而过表达xlnR 使得木糖利用速率提升14.2%。转录组数据表明,敲除xlnR导致木聚糖水解酶基因、木糖转运蛋白及代谢相关基因的转录水平显著降低。全局调控因子Cre-1是木糖及葡萄糖代谢的抑制因子,敲除cre-1 后显著提升嗜热毁丝霉木糖和葡萄糖利用速率;同时,Cre-1可直接抑制xlnR 及自身的表达。通过过表达木糖代谢激活因子XlnR编码基因xlnR,同时敲除抑制因子Cre-1编码基因cre-1,嗜热毁丝霉木糖利用速率提高100%。以上研究结果为生物质高效利用底盘细胞的创建提供了良好的出发菌株。
关键词:嗜热毁丝霉;木糖代谢;转录调控;Cre-1;XlnRdoi:10.13304/j.nykjdb.2023.0151
中图分类号:S182;Q933 文献标志码:A 文章编号:10080864(2024)12007711
生物质资源是地球上最丰富的可再生有机原料,实现生物质转化生产生物燃料以及生物化学品,是解决目前发酵原料短缺、实现双碳目标的关键。木质纤维素由纤维素、半纤维素以及木质素构成,三者之间通过共价键和氢键紧密缠绕连接[1],形成坚硬的、难降解的刚性结构。在生物质中纤维素含量为38%~50%,半纤维素含量为23%~32%[2],二者通过多种碳水化合物活性酶协同作用进行降解[3],降解后产生的纤维寡糖由β-葡萄糖苷酶 (β-glucosidase, BGLs)降解成葡萄糖,木寡糖由β-木糖苷酶 (β-xylosidase, BXLs) 降解成木糖。传统生物质利用技术主要包括预处理、纤维素酶生产和木质纤维素酶解糖化以及产品发酵等工艺[4]。目前,国内外纤维素酶工业生产主要体系均为丝状真菌,包括里氏木霉(Trichoderma reesei)[5]、嗜热毁丝霉(Myceliopthorathermophila ) 以及青霉菌 (Penicillium)[67]。经过遗传改造以及人工诱变后,工业菌株嗜热毁丝霉和里氏木霉的纤维素酶产量均达到100 g·L-1[8-10],显著降低了生物质发酵成本。生物质水解液的主要成分为葡萄糖和木糖,实现生物质全糖的高效率利用是生物质高效转化的关键。尽管研究者已经鉴定了木糖主要代谢途径[11],但对于其调控网络尚未明晰。
丝状真菌是纤维素酶的主要生产菌,同时也被开发为重要的有机酸等工业发酵菌种[7, 1213]。丝状真菌在利用生物质水解糖的过程中,由于碳分解代谢物阻遏效应(carbon cataboliterepression,CCR)会优先利用葡萄糖,而木糖利用则严重滞后[14]。研究发现,碳阻遏调控因子Cre-1(CreA)是Cys2His2型转录因子[1516],已被证实存在于多种丝状真菌中,如里氏木霉(T. reesei)[17]。Cre-1通过调控糖转运蛋白基因、关键转录因子、糖代谢途径基因和纤维素酶基因的表达来实现对生物质中纤维素和半纤维素降解的调控[13,17],同时,Cre-1可直接与木聚糖酶、半乳糖苷酶和阿拉伯聚糖酶基因及其代谢途径关键酶基因的启动子结合[15,17]。但是,Cre-1对丝状真菌木糖代谢的调控机制尚未明晰,有待进一步研究。在丝状真菌中,XlnR (Xyr1/Xlr1)是木聚糖酶和纤维素酶的主要调控因子[1819]。在粗糙脉孢菌和嗜热毁丝霉中,XlnR 主要参与木聚糖降解和木糖代谢[2021]。在黑曲霉中,XlnR 的同源蛋白Xyr1[22]参与调控20多个编码半纤维素酶基因和纤维素酶基因的表达[23],被认为是纤维素酶和半纤维素酶基因表达的主要激活因子[15],该基因缺失后,导致所有诱导物丧失对纤维素酶的诱导作用,同时削弱诱导物对木聚糖水解酶基因和阿拉伯聚糖水解酶基因的诱导作用[2425]。
嗜热毁丝霉为天然高效降解纤维素菌种,具有发酵温度高、纤维素降解能力强、蛋白分泌水平高、能够天然利用五碳糖和六碳糖等优势,是生产工业酶和生物基化学品的优秀底盘微生物[26]。目前,研究者已开发了基于CRISPR/Cas9 和CRISPR/Cpf1的基因组编辑技术[27-29],为嗜热毁丝霉的遗传改造奠定了技术基础。嗜热毁丝霉已被开发为有机酸及有机醇细胞工厂,实现了生物质直接发酵合成目标产品[30],即真菌整合生物炼制(consolidated bioprocessing,CBP)技术[31-33]。在真菌CBP 技术中,产酶、生物质酶解和目标产品合成均由单一菌种来完成,极大降低了生物质炼制成本。因此,本研究以嗜热毁丝霉为体系,研究其木糖代谢调控机制,通过人工干预使嗜热毁丝霉的木糖利用效率显著提升,为实现生物质全糖高效率利用奠定了理论及技术基础。
1 材料与方法
1.1 材料
1.1.1 菌种和试剂 嗜热毁丝霉ATCC 42464购于美国模式培养物集存库(American Type CultureCollection,ATCC);质粒ppK2-Bar-GFP、pAN52-PtrpC-TN、pGEX-4T-1和AT-U6-gRNA均保藏于本实验室;大肠杆菌感受态Mach1-T1和BL21 (DE3)购于北京全式金生物技术股份有限公司;PCR试剂盒、PCR 产物回收试剂盒、质粒小提试剂盒和GST磁珠纯化试剂盒均购于康为世纪生物科技股份有限公司;限制性内切酶和T4 DNA 连接酶均购于南京诺唯赞生物科技股份有限公司;GibsonAssembly®克隆试剂盒和RNA纯化回收试剂盒购于纽英伦生物技术(北京)有限公司;非变性聚丙烯酰胺凝胶购于赛默飞世尔科技公司;新霉素(neomycin)和草铵膦 (glufosinate ammonium) 购于北京吉普赛生物技术有限公司,氨苄青霉素购于Amresco;异丙基-β-D-硫代半乳糖苷 (isopropylthio-β-galactoside, IPTG) 购于上海阿拉丁生化科技股份有限公司;葡萄糖、木糖和阿拉伯糖购于Sigma公司;木二糖和木三糖购于Megazyme公司。
1.1.2 培养基 LB 培养基:酵母提取物5 g,胰蛋白胨10 g,氯化钠10 g;定容至1 L,121 ℃灭菌20 min。
50×vogel’salt:柠檬酸钠130 g,硝酸钾126 g,磷酸二氢铵144 g,磷酸二氢钾80 g,七水硫酸镁10 g,二水氯化钙5 g,微量元素5 mL。100 mL微量元素溶液含5 g C6H8O·7H2O,5 g ZnSO4·7H2O,1 g Fe(NH4)2(SO4)·6H2O,0.25 g CuSO4·5H2O,0.05 gMnSO4·H2O,0.05 g H3BO3,0.05 g NaMnO4·2H2O,0.1 mg·mL-1的生物素溶液2.5 mL;定容至1 L,加入1 mL氯仿作为保护剂,室温保存。
2%葡萄糖/木糖/阿拉伯糖培养基:1 × vogel’salt 1 L, 碳源(葡萄糖、木糖或阿拉伯糖)20 g,115 ℃灭菌25 min。
1.2 试验方法
1.2.1 质粒构建 靶基因敲除质粒构建:在联合基因组研究所(Joint Genome Institute,JGI)网站查找Mycth_2310145 (xlnR) 和Mycth_2310085 (cre-1)序列信息,按照文献[31]方法确定PAM序列,以嗜热毁丝霉基因组DNA为模板,利用表1中引物扩增xlnR 和cre-1 的5’和3’片段。以p0380-bar 质粒为模板,设计引物(bar-F/R)并扩增PtrpC-bar 片段;以p0380-neo 质粒为模板,设计引物(neo-F/R)并扩增PtrpC-neo 片段。使用Gibson Assembly®克隆试剂盒分别将xlnR 的5’片段、3’片段和PtrpC-neo片段连接到用 BamHⅠ和EcoRⅠ 切开的ppK2-Bar-GFP 载体中,获得质粒 donor-xlnR-neo。类似地,利用cre-1 基因的5’和3’片段以及PtrpC-bar 片段,构建质粒donor-cre-1-bar。靶向xlnR 和cre-1的sgRNA由嗜热毁丝霉的U6启动子控制,以ATU6-gRNA 质粒为模板,以引物 (U6p-F/U6p-cre-1-R、cre1-gRNA-F/gRNA-R)扩增U6p-cre-1 和cre-1-gRNA 片段。二者融合获得sgRNA 片段后,将其克隆到pJET1.2/blunt 载体上,得到U6p-cre-1-sgRNA重组质粒。类似的,设计引物 U6p-F/U6pxlnR-R 和xlnR-gRNA-F/gRNA-R 分别获得U6pxlnR和 xlnR-gRNA片段,二者融合获得sgRNA片段后,将其插入pJET1.2/blunt载体,得到重组质粒U6p-xlnR-sgRNA。以Cas9 为模板,引物为cas-F/R,PCR扩增出Cas9表达盒。
靶基因过表达质粒构建:设计引物OE xlnRF/R,以嗜热毁丝霉cDNA为模板,扩增xlnR 片段;将Ptef-1 启动子、翻译延长因子Tef-1(translationelongation factor Tef-1)启动子和xlnR 片段插入pAN52-PtrpC-TN 载体(Spe Ⅰ 和 BamH Ⅰ 双酶切),获得过表达质粒pAN52-ptef-1-xlnR-PtrpCneo-TN。
1.2.2 嗜热毁丝霉原生质体转化及验证 嗜热毁丝霉原生质体转化参照文献[3132]。利用CRISPR/Cas9系统进行基因敲除,将sgRNA片段、donor片段和Cas9片段以1∶1∶1混合,浓缩后转化到野生型或突变体原生质体中。在靶基因过表达过程中,将10 μg线性化质粒转化到嗜热毁丝霉原生质体中,并用相应的抗生素进行筛选,在35 ℃培养5 d 后,挑取少量菌丝于裂解缓冲液中,沸水浴20 min,高速离心(10 000 r·min-1,10 min)后加入150 μL灭菌水,作为模板使用。后续利用PCR验证获得阳性转化子,验证引物详见表2。
1.2.3 突变体的形态学表征 将3 μL孢子悬液(1×104 cell·μL-1)接种到含有2%碳源(葡萄糖、木糖或阿拉伯糖)的固体培养基中,置于45 ℃条件下培养,观察菌丝长势,实时拍照记录。
1.2.4 嗜热毁丝霉糖利用分析 将成熟孢子接种到2% 葡萄糖或木糖液体培养基,接种量为106cell·mL-1,45 ℃下培养18 h,而后收集菌丝并用灭菌水洗涤3次。将菌丝接种于24孔板中,每孔接种3 mL 菌丝,分别加入木糖 (xylose)、木二糖(xylobiose) 和木三糖 (xylotriose),终含量为5 g·L-1,45 ℃、 700 r·min-1培养24 h。
1.2.5 凝胶迁移试验 (electrophoretic mobilityshift assay, EMSA)
①获取融合蛋白:以嗜热毁丝霉野生型DNA为模板,用表1中的引物( cre-1-emsa-F/R 和xlnR-emsa-F/R) 扩增xlnR 和cre-1 片段,而后插入经BamHⅠ/XhoⅠ双酶切后的载体pGEX-4T-1 中,分别获得重组质粒pGEX-4T-1-xlnR 和pGEX-4T-1-cre-1。将重组质粒及对照质粒pGEX-4T-1 转入到大肠杆菌BL21(DE3) 中,37 ℃ 200 r·min-1 培养至OD 值为0.38~0.42;而后加入20 μL 1 mmol·L-1 IPTG,15 ℃ 200 r·min-1 低温诱导20 h;收集菌体后低温破碎,并使用GST磁珠纯化试剂盒纯化出融合GST标签的目的蛋白。
②获取探针:以嗜热毁丝霉野生型基因组作为模板,利用PCR 扩增相应基因的启动子区片段,引物详见表3。
③ 反应体系:50 mmol·L-1 MgCl2 2 μL、10 mg·mL-1 BSA 1 μL、50%甘油2 μL、Protein (0~200 nmol·L-1) 1 μL、探针(10 ng·μL-1) 1 μL,最后用ddH2O补到20 μL。将反应体系在25 ℃条件下孵育30 min。
④反应程序:预电泳150 V 40 min,冲洗胶孔;正式电泳150 V 50~60 min;电泳结束后,小心取出变性胶,使用SYBR Green 染色10 min,结束后,清水冲洗1次,而后在凝胶成像仪下成像。
1.2.6 糖耗分析 按时间点取发酵液1 mL,经高速离心(10 000 r·min-1,10 min)后,取出上清液过0.22 μm 滤膜,利用高效液相色谱(highperformance liquid chromatography,HPLC)测定发酵液中糖含量。检测器为 Waters 2414示差检测器;分离柱为Aminex HPX-87H 柱 (Bio-Rad,Hercules,CA,USA);检测温度45 ℃ ;流速0.5 mL·min-1;流动相为5 mmol·L-1 H2SO4。
1.2.7 RNA提取及转录组数据分析
①RNA提取:将嗜热毁丝霉成熟孢子接种于2%葡萄糖培养基中,孢子终水平为106个·mL-1,于45 ℃ 150 r·min-1培养18 h,而后收集菌丝并用灭菌水洗涤3 次(40 mL·次-1);将菌丝转移至2%木糖培养基中诱导 4 h,而后利用布氏漏斗收集菌丝,置于-80 ℃冰箱保存。利用液氮预冷的研钵,将样品磨至粉末,而后按照RNA纯化回收试剂盒和文献[27]方法提取纯化RNA。
②转录组数据分析:将检测合格的RNA[OD260/OD280 gt; 1.8,RIN (RNA integrity number)gt;7.0]送至由诺禾致源公司(天津),利用IlluminaHiSeqTM 2000平台进行测序,每个样本2个平行,根据文献[34]方法处理和分析转录组数据。
2 结果与分析
2.1 转录因子XlnR 在嗜热毁丝霉木糖利用中的功能分析
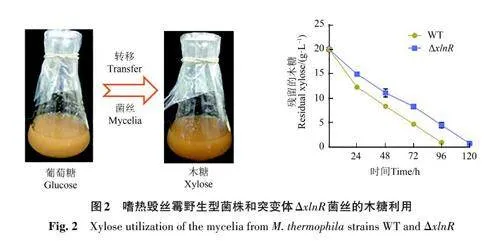
转录激活因子XlnR及同源蛋白具有调控半纤维素酶基因表达及木糖利用的功能[20]。为了研究XlnR (Mycth_2310145) 在嗜热毁丝霉木糖利用中的调控作用,利用CRISPR/Cas9 基因组编辑技术[31]构建了嗜热毁丝霉突变体ΔxlnR。对突变体ΔxlnR 在木糖、阿拉伯糖及葡萄糖液体培养基中的糖利用速率进行检测,结果(图1)显示,敲除xlnR 后,嗜热毁丝霉丧失了木糖利用能力(图1A)。阿拉伯糖与木糖同为五碳糖,二者均是半纤维素的主要构成单元,且具有相似的代谢途径。糖消耗试验显示,突变体ΔxlnR 的阿拉伯糖利用能力与野生型(WT)菌种相比显著下降(图1B);而葡萄糖的消耗速率上升(图1C)。突变体ΔxlnR 对木二糖和木三糖的利用能力与木糖消耗相似,xlnR 失活后,嗜热毁丝霉丧失了在木二糖和木三糖上生长的能力(图1D);同时,突变体ΔxlnR 在木糖固体平板上生长状况显著弱于野生型(图1E)。以上结果表明,转录因子XlnR在嗜热毁丝霉木糖代谢中起激活作用。
将丝状真菌孢子接种至液体培养基中后,首先是孢子的萌发,而后菌丝体生长。xlnR 的敲除可能影响了嗜热毁丝霉孢子在木糖条件下的萌发,进而导致突变体丧失木糖利用能力。为了验证这一推断,将野生型嗜热毁丝霉和突变体ΔxlnR 在葡萄糖液体培养基中培养18 h 后转接到木糖液体培养基中,测定木糖利用速率。结果(图2)显示,突变体ΔxlnR 菌丝仍然具备木糖利用的能力,但其糖耗速率显著低于野生型菌株,说明XlnR不仅参与调控木糖代谢,而且对嗜热毁丝霉孢子在木糖条件下萌发具有重要作用。
2.2 转录因子Cre-1 在嗜热毁丝霉木糖代谢中的功能分析
在丝状真菌中,Cre-1是介导碳源降解物代谢阻遏的主要调控因子,参与调控转运蛋白、转录因子、糖分解代谢相关因子、纤维素酶以及半纤维素酶基因的表达[17]。在嗜热毁丝霉中,利用RNAi技术抑制cre-1 的表达后,纤维素酶及半纤维素酶基因的表达水平显著提高[31]。为了研究Cre-1在嗜热毁丝霉木糖代谢中的调控功能,利用基因组编辑技术构建了突变体Δcre-1,并检测突变体木糖利用速率。结果(图3)显示,敲除cre-1 后,嗜热毁丝霉的木糖利用速率显著提升,与野生型菌株相比,突变体Δcre-1 的木糖利用速率提升25%。相似的是,在葡萄糖培养基中,突变体Δcre-1 的糖耗速率较野生型提升33%。由此表明,转录因子Cre-1参与调控嗜热毁丝霉中葡萄糖和木糖的利用,是葡萄糖和木糖代谢途径的抑制因子。
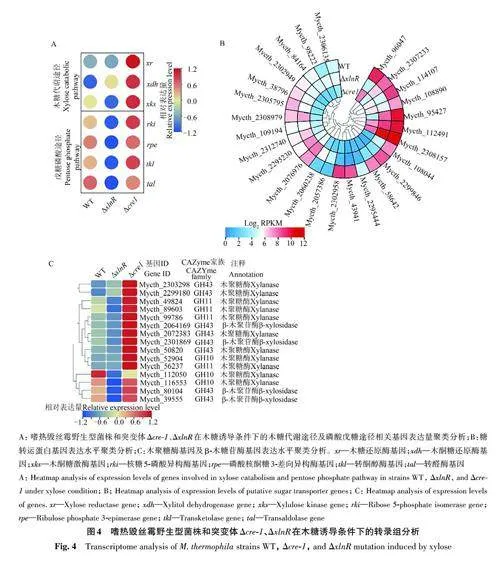
2.3 在木糖条件下突变体ΔxlnR 和Δcre-1 的转录组分析
在丝状真菌中,木糖在木糖还原酶 (xylosereductase,XR) 和木糖醇脱氢酶(xylitoldehydrogenase,XDH) 催化作用下形成木酮糖;在木酮糖激酶 (xylulokinase,XKS) 的作用下,形成5-磷酸木酮糖,然后进入戊糖磷酸途径 (pentosephosphate pathway,PPP) 非氧化阶段。为探究转录因子XlnR 和Cre-1 调控木糖代谢的分子机制,系统分析了嗜热毁丝霉在木糖条件下转录组数据,结果(图4A)表明,突变体ΔxlnR 中xr 和xdh的表达水平与野生型菌株相似,但木酮糖激酶基因xks 和磷酸戊糖途径非氧化阶段相关基因(rki、rpe、tkl 和tal)在突变体中的表达水平显著低于野生型菌株,暗示XlnR可能通过调控木酮糖激酶基因表达以及PPP途径来调节木糖代谢。在突变体Δcre-1 中,木糖代谢途径及PPP途径相关基因的表达水平显著高于野生型,这与突变体Δcre-1 显著提升的木糖利用速率一致。
糖转运是胞内糖代谢的前提,在木糖诱导条件下,嗜热毁丝霉中有3 个木糖转运蛋白基因(Mycth_2308157、Mycth_96047 和Mycth_112491)被高效诱导表达,其中,Mycth_96047在粗糙脉孢菌中的同源蛋白NCU06138 为高效木糖转运蛋白[35]。敲除xlnR 后,Mycth_96047在嗜热毁丝霉中的表达水平下降约10倍,与突变体ΔxlnR 变弱的木糖代谢一致。另外,转录组数据(图4B)表明,在突变体Δcre-1 中,11个糖转运蛋白基因的表达水平相比野生型菌株WT显著提高,其中11个基因的表达水平上调超过10倍。前期研究表明,粗糙脉孢菌中的Cre-1可以通过抑制糖转运蛋白的表达,进而调控胞内糖代谢[36]。
木糖作为半纤维素构成单元,具有诱导木聚糖水解酶基因表达的作用。在木糖条件下,有5个木聚糖水解酶基因(Mycth_112050、Mycth_89603、Mycth_116553、Mycth_80104 和Mycth_39555)被高效诱导表达 (RPKMgt;100)。分析比较不同突变体木聚糖水解酶基因表达,结果(图4C)表明,在木糖条件下,突变体ΔxlnR 中木聚糖水解酶基因的表达水平较野生型菌株显著下调,说明XlnR在嗜热毁丝霉中具有调控半纤维素酶基因表达的功能,这与其同源蛋白在粗糙脉孢菌[21]及里氏木霉[37]中调控作用相似;与之相反的是,敲除cre-1 后,13个木聚糖水解酶基因的表达水平显著上调,表明Cre-1具有抑制嗜热毁丝霉半纤维素酶基因表达的功能。
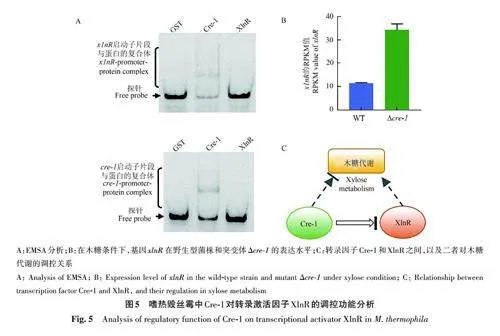
2.4 木糖利用阻遏因子Cre-1 直接抑制转录激活因子XlnR 的表达
为了研究木糖利用调控因子Cre-1 与XlnR的之间的调控关系,将融合纯化标签GST 的蛋白Cre-1-GST 和XlnR-GST 在E. coli BL21 (DE3) 中表达,结果(图5A)显示,Cre-1可直接与xlnR 启动子区域结合,而XlnR则不能结合于cre-1 启动子区域。另外,比较木糖条件下嗜热毁丝霉的转录组数据(图5B)发现,与野生型菌株相比,突变体Δcre-1 中xlnR 基因 的转录水平上调3倍。在嗜热毁丝霉中Cre-1能够直接抑制调控激活因子基因xlnR 的表达(图5C)。
2.5 嗜热毁丝霉木糖代谢调控网络的重构
为了提升嗜热毁丝霉木糖代谢速率,将转录激活因子基因xlnR 在野生型嗜热毁丝霉中过表达,过表达突变体菌株OE_xlnR 的木糖利用速率较野生型菌株提高14.2%,完全消耗20 g·L-1木糖所需时间由96 h 降至 84 h(图6),进一步说明转录因子XlnR对木糖代谢具有激活作用。
为了进一步强化木糖代谢速率,敲除突变体OE_xlnR 中基因cre-1,以实现木糖代谢的解阻遏,获得突变体OE_xlnRΔcre-1。在木糖液体培养基中,突变体OE_xlnRΔcre-1 木糖消耗速率较对照菌株OE_xlnR 提升75%,较野生型菌种提升100%,消耗20 g·L-1 木糖所需时间缩短至48 h(图6)。
3 讨论
丝状真菌是目前国内外纤维素酶工业生产的主要体系,同时也是众多关键工业化学品的重要生产者[7]。实现丝状真菌高效利用生物质糖是降低生物质炼制经济成本的关键之一。木糖是生物质中半纤维素的重要组成部分,但由于碳代谢物阻遏效应的存在,木糖在丝状真菌中的代谢往往受到严重的抑制。嗜热毁丝霉为高效的纤维素降解菌种,同时能够高效利用六碳糖和五碳糖,目前已被开发为工业纤维素酶和化学品细胞工厂[38]。本研究以嗜热毁丝霉为体系,利用遗传学手段及转录组学技术研究了其木糖代谢调控机制,并结合基因组编辑技术对其进行了改造,显著提升了嗜热毁丝霉的木糖代谢速率。
在木霉和曲霉中,转录因子XlnR及其同源蛋白是纤维素酶和半纤维素酶基因的主要激活因子, XlnR的失活将导致纤维素不能诱导纤维素酶和半纤维素酶基因的表达[18- 19,25]。而在粗糙脉孢菌中,XlnR 仅调控半纤维素酶基因的表达,是半纤维素利用相关基因表达所必需的[35]。此外,在曲霉和木霉中,XlnR具有调控木糖代谢的功能,参与调控木糖还原酶基因的表达,敲除xlnR 后将大幅降低突变体利用木糖和木聚糖的能力[15]。本研究利用基因编辑技术构建丝状真菌嗜热毁丝霉突变体ΔxlnR,表型分析发现,突变体ΔxlnR 的阿拉伯糖利用能力显著下降,但葡萄糖利用能力显著提升;而在木糖条件下,突变体ΔxlnR 的孢子丧失了萌发能力,且菌丝木糖的利用速率显著降低,过表达xlnR 使得木糖利用速率提升14.2%;分析突变体ΔxlnR 在木糖条件下的转录组发现,敲除xlnR 导致木聚糖水解酶基因、木糖转运蛋白基因以及磷酸戊糖途径相关基因的转录水平显著降低,说明在嗜热毁丝霉中XlnR参与调控菌丝木糖代谢和半纤维素酶诱导,同时,对木糖条件下孢子萌发具有重要作用。
在丝状真菌中,全局调控因子Cre-1 属于Cys2His2 型锌指家族,是酿酒酵母中Mig1的同源蛋白[14,39]。Cre-1 作为重要碳源代谢阻遏调控因子,在丝状真菌生长、碳代谢及纤维素水解酶的表达中起重要调控作用[17]。在粗糙脉孢菌及里氏木霉中,敲除cre-1 能显著提升纤维素酶和半纤维素酶的表达[16]。本研究的转录组数据分析结果表明,Cre-1的失活导致突变体Δcre-1 中木聚糖水解酶基因的表达水平显著上调,暗示Cre-1对嗜热毁丝霉半纤维素酶表达有抑制作用;同时,突变体Δcre-1 木糖消耗速率显著提高以及木糖代谢和转运蛋白基因高效表达,暗示Cre-1抑制嗜热毁丝霉木糖代谢。EMSA分析表明,Cre-1能够直接结合xlnR 启动子区域,结合xlnR 在突变体Δcre-1 中表达水平下调,证明Cre-1能够直接抑制xlnR 的表达;另外Cre-1能够结合至其自身编码基因启动子区域,暗示Cre-1具有自我调控的功能。研究表明,在葡萄糖条件下,里氏木霉Cre-1的表达能被自身负调控[16]。这也解释了嗜热毁丝霉突变体Δcre-1 具有较高葡萄糖利用能力的原因。
通过代谢网络重构可以有效地改善发酵菌株的碳源利用速率。本研究利用基因组编辑技术过表达木糖代谢激活因子XlnR编码基因,同时敲除抑制因子Cre-1编码基因,显著提升了嗜热毁丝霉木糖代谢速率,为改善发酵菌株木糖利用速率、提升生物质全糖利用提供了新思路。
参考文献
[1] TAKKELLAPATI S, LI T, GONZALEZ M A. An overview of biorefinery derived platform chemicals from a cellulose and hemicellulose biorefinery [J]. Clean Technol. Environ. Policy,2018, 20(7):1615-1630.
[2] ALI S S, NUGENT B, MULLINS E, et al .. Fungal-mediated consolidated bioprocessing: the potential of Fusarium oxysporum for the lignocellulosic ethanol industry [J/OL]. AMB Express, 2016, 6(1):13 [2023-02-10]. https://doi.org/10.1186/s13568-016-0185-0.
[3] ZNAMEROSKI E A, CORADETTI S T, ROCHE C M, et al .. Induction of lignocellulose-degrading enzymes in Neurospora crassa by cellodextrins [J]. Proc. Natl. Acad. Sci. USA, 2012,109(16):6012-6017.
[4] BALAT M. Production of bioethanol from lignocellulosic materials via the biochemical pathway: a review [J]. Energy Convers. Manage., 2011, 52(2):858-875.
[5] VITIKAINEN M, ARVAS M, PAKULA T, et al .. Array comparative genomic hybridization analysis of Trichoderma reesei strains with enhanced cellulase production properties [J/OL].BMC Genomics, 2010, 11(1):441 [2023-02-10]. https://doi.org/10.1186/1471-2164-11-441.
[6] ALI S S, NUGENT B, MULLINS E, et al .. Insights from the fungus Fusarium oxysporum point to high affinity glucose transporters as targets for enhancing ethanol production from lignocellulose [J/OL]. PLoS One, 2013, 8(1):e54701 [2023-02-10]. https://doi.org/ 10.1371/journal.pone.0054701.
[7] LIU G, QU Y. Engineering of filamentous fungi for efficient conversion of lignocellulose: tools, recent advances and prospects [J]. Biotechnol. Adv., 2019, 37(4):519-529.
[8] VISSER H, JOOSTEN V, PUNTPETER J, et al .. Development of a mature fungal technology and production platform for industrial enzymes based on a Myceliophthora thermophila isolate, previously known as Chrysosporium lucknowense C1 [J].Ind. Biotechnol., 2011, 7:214-223.
[9] DRUZHININA I S, KUBICEK C P. Genetic engineering of Trichoderma reesei cellulases and their production [J]. Microb.Biotechnol., 2017, 10(6):1485-1499.
[10] LI N, LIU Y, LIU D, et al .. MtTRC-1, a novel transcription factor, regulates cellulase production via directly modulating the genes expression of the Mthac-1 and Mtcbh-1 in Myceliophthora thermophila [J/OL]. Appl. Environ. Microbiol.,2022, 88(19):e0126322 [2023-02-10]. https://doi.org/10.1128/aem.01263-22.
[11] HECTOR R E, QURESHI N, RHUGHES S, et al .. Expression of a heterologous xylose transporter in a Saccharomyces cerevisiae strain engineered to utilize xylose improves aerobic xylose consumption [J]. Appl. Microbiol. Biotechnol., 2008, 80(4):675-684.
[12] WU V W, THIEME N, HUBERMAN L B, et al .. The regulatory and transcriptional landscape associated with carbon utilization in a filamentous fungus [J]. Proc. Natl. Acad. Sci. USA, 2020, 117(11):6003-6013.
[13] DENG H, BAI Y, FAN T P, et al .. Advanced strategy for metabolite exploration in filamentous fungi [J]. Crit. Rev.Biotechnol., 2020, 40(2):180-198.
[14] ADNAN M, ZHENG W, ISLAM W, et al .. Carbon catabolite repression in filamentous fungi [J/OL]. Int. J. Mol. Sci., 2017,19(1):48 [2023-02-10]. https://doi.org/ 10.3390/ijms19010048.
[15] TAMAYO E N, VILLANUEVA A, HASPER A A, et al .. CreA mediates repression of the regulatory gene xlnR which controls the production of xylanolytic enzymes in Aspergillus nidulans[J]. Fungal Genet. Biol., 2008, 45(6):984-993.
[16] ANTONIETO A C, DOS SANTOS CASTRO L, SILVAROCHAR, et al .. Defining the genome-wide role of CRE1 during carbon catabolite repression in Trichoderma reesei using RNA-Seq analysis [J]. Fungal Genet. Biol., 2014, 73:93-103.
[17] PORTNOY T, MARGEOT A, LINKE R, et al .. The CRE1 carbon catabolite repressor of the fungus Trichoderma reesei: a master regulator of carbon assimilation [J/OL]. BMC Genomics,2011, 12:269 [2023-02-10]. https://doi.org/10.1186/1471-2164-12-269.
[18] FURUKAWA T, SHIDA Y, KITAGAMI N, et al .. Identification of specific binding sites for XYR1, a transcriptional activator of cellulolytic and xylanolytic genes in Trichoderma reesei [J].Fungal Genet. Biol., 2009, 46(8):564-574.
[19] PORTNOY T, MARGEOT A, SEIDL-SEIBOTH V, et al .. Differential regulation of the cellulase transcription factors XYR1, ACE2, and ACE1 in Trichoderma reesei strains producing high and low levels of cellulase [J]. Eukaryot. Cell,2011, 10(2):262-271.
[20] DOS SANTOS GOMES A C, FALKOSKI D, BATTAGLIA E, et al .. Myceliophthora thermophila Xyr1 is predominantly involved in xylan degradation and xylose catabolism [J/OL].Biotechnol. Biofuels, 2019, 12: 220 [2023-02-10]. https://doi.org/10.1186/s13068-019-1556-y.
[21] CRAIG J P, CORADETTI S T, STARR T L, et al .. Direct target network of the Neurospora crassa plant cell wall deconstruction regulators CLR-1, CLR-2, and XLR-1 [J/OL]. Mbio, 2015, 6(5):e01452-15 [2023-02-10]. https://doi.org/10.1128/mBio.01452-15.
[22] HASPER A A, TRINDADE L M, VAN DER VEEN D, et al .. Functional analysis of the transcriptional activator XlnR from Aspergillus niger [J]. Microbiology, 2004, 150(Pt5):1367-1375.
[23] HASPER A A, VISSER J, DE GRAAFF L H. The Aspergillus niger transcriptional activator XlnR, which is involved in the degradation of the polysaccharides xylan and cellulose, also regulates d-xylose reductase gene expression [J]. Mol. Microbiol.,2000, 36(1):193-200.
[24] MACH-AIGNER R, PUCHER E, STEIGER G, et al.. Transcriptional regulation of xyr1, encoding the main regulator of the xylanolytic and cellulolytic enzyme system in Hypocrea jecorina [J]. Appl. Environ. Microbiol., 2008,74(21):6554-6562.
[25] MACH-AIGNER A R, OMONY J, JOVANOVIC B, et al ..D-Xylose concentration-dependent hydrolase expression profiles and the function of CreA and XlnR in Aspergillus niger [J]. Appl. Environ. Microbiol., 2012, 78(9):3145-3155.
[26] BERKA R M, GRIGORIEV I V, OTILLAR R, et al ..Comparative genomic analysis of the thermophilic biomassdegrading fungi Myceliophthora thermophila and Thielaviaterrestris [J]. Nat. Biotechnol., 2011, 29(10):922-927.
[27] XU J, LI J, LIN L, et al .. Development of genetic tools for Myceliophthora thermophila [J/OL]. BMC Biotechnol., 2015, 15:35 [2023-02-10]. https://doi.org/10.1186/s12896-015-0165-5.
[28] SONG R, ZHAI Q, SUN L, et al .. CRISPR/Cas9 genome editing technology in filamentous fungi: progress and perspective [J].Appl. Microbiol. Biotechnol., 2019, 103(17):6919-6932.
[29] SWIAT M A, DASHKO S, DEN RIDDER M, et al .. FnCpf1:a novel and efficient genome editing tool for Saccharomyces cerevisiae [J]. Nucleic Acids Res., 2017, 45(21):12585-12598.
[30] PATEL H, RAWAT S. Thermophilic fungi: diversity, physiology, genetics, and applications [J]. New Future Dev.Microb. Biotechnol. Bioeng., 2021, 13(6):69-93.
[31] LIU Q, GAO R, LI J, et al .. Development of a genome-editing CRISPR/Cas9 system in thermophilic fungal Myceliophthora species and its application to hyper-cellulase production strain engineering [J/OL]. Biotechnol. Biofuels, 2017, 10:1 [2023-02-10]. https://doi.org/ 10.1186/s13068-016-0693-9.
[32] LI J, GU S, ZHAO Z, et al .. Dissecting cellobiose metabolic pathway and its application in biorefinery through consolidated bioprocessing in Myceliophthora thermophila [J/OL]. Fungal Biol. Biotechnol., 2019, 6: 21 [2023-02-10]. https://doi.org/10.1186/s40694-019-0083-8.
[33] XU Q, SINGH A, HIMMEL M E. Perspectives and new directions for the production of bioethanol using consolidated bioprocessing of lignocellulose [J]. Curr. Opin. Biotechnol.,2009, 20(3):364-371.
[34] WANG B, LI J, GAO J, et al .. Identification and characterization of the glucose dual-affinity transport system in Neurospora crassa: pleiotropic roles in nutrient transport, signaling, and carbon catabolite repression [J/OL]. Biotechnol.Biofuels, 2017, 10: 17 [2023-02-10]. https://doi. org/10.1186/s13068-017-0705-4.
[35] SUN J, TIAN C, DIAMOND S, et al .. Deciphering transcriptional regulatory mechanisms associated with hemicellulose degradation in Neurospora crassa [J]. Eukaryot.Cell, 2012, 11(4):482-493.
[36] SUN J, GLASS N L. Identification of the CRE-1 cellulolytic regulon in Neurospora crassa [J/OL]. PLoS One, 2011, 6(9):e25654 [2023-02-10]. https://doi.org/10.1371/journal.pone.0025654.
[37] FURUKAWA T, SHIDA Y, KITAGAMI N, et al .. Identification of specific binding sites for XYR1, a transcriptional activator of cellulolytic and xylanolytic genes in Trichoderma reesei [J].Fungal Genet. Biol., 2009, 46(8):564-574.
[38] LI J, LIN L, SUN T, et al .. Direct production of commodity chemicals from lignocellulose using Myceliophthora thermophila [J].Metab. Eng., 2020, 61:416-426.
[39] WEINHANDL K, WINKLER M, GLIEDER A, et al .. Carbon source dependent promoters in yeasts [J/OL]. Microb. Cell Fact., 2014, 13: 5[2023-02-10]. https://doi.org/10.1186/1475-2859-13-5.
