3种滨海碱蓬属植物串联重复序列与转座元件比较分析
2024-11-21陈静黄永吉


摘要:为了解析3种常见的滨海碱蓬属物种的重复序列类型、丰度、染色体分布模式及种间差异,采用基于序列相似性聚类分析和荧光原位杂交技术,对它们的重复序列进行比较分析。结果表明:3个物种中均存在串联重复序列和转座元件。其中,端粒序列、5S rDNA、35S rDNA为3个物种保守的串联重复序列,另外有2个串联重复序列是碱蓬中特异的,这些串联重复序列倾向于在染色体端粒、亚端粒、染色体臂富集。此外,3个碱蓬属物种的转座元件主要由反转录转座子和DNA转座子组成,其中Ty3/gypsy反转录转座子的基因组占比较高,而Ty1/copia反转录转座子和DNA转座子的基因组含量则相对较低。除了CRM谱系主要在着丝粒区域聚集,大多数转座元件在染色体上呈现散布分布,而一些基因组丰度较低的转座元件则未检测到信号。根据重复序列和染色体分布分析结果表明,与碱蓬相比,南方碱蓬和盐地碱蓬的基因组关系更近。这些发现将为理解3种滨海碱蓬属物种重复序列的特征和种间进化关系提供了依据。
关键词:碱蓬属;串联重复序列;端粒;着丝粒;rDNA;荧光原位杂交
中图分类号:Q943文献标志码:A文章编号:0253−2301(2024)08−0016−07
DOI:10.13651/j.cnki.fjnykj.2024.08.002
陈静,黄永吉.3种滨海碱蓬属植物串联重复序列与转座元件比较分析[J].福建农业科技,2024,55(8):16−22.
Comparative Analysis of Tandem Repeats and Transposable Elements in Three Coastal Suaeda Species
CHEN Jing,HUANG Yong-ji*
(College of Geography and Oceanography,Minjiang University,Fuzhou,Fujian 350108,China)
Abstract:In order to analyze the types,abundance,chromosomal distribution patterns,and interspecific differences of repetitive sequences in three common species of coastal Suaeda,the sequence similarity-based clustering analysis and fluorescence in situ hybridization techniques were used for the comparative analysis of their repetitive sequences.The results revealed that there were tandem repeats and transposable elements in all the three species.Among them,the telomeric sequences,5S rDNA,and 35S rDNA were identified as the conserved tandem repeats across the three species,while the other two tandem repeats were identified as being specific to Suaeda glauca.These tandem repeats tended to be enriched in the chromosome telomeres,subtelomeres,and chromosomal arms.Furthermore,the transposable elements in the three Suaeda species were mainly composed of retrotransposons and DNA transposons.Notably,the genome of Ty3/gypsy retrotransposons were found to have a higher genomic proportion,while the Ty1/copia retrotransposons and DNA transposons were relatively less abundant.Except for the CRM lineage,which was mainly clustered in the centromeric region,most of transposable elements exhibited a dispersed distribution across the chromosomes,while some transposable elements with lower genomic abundance were not detected.According to the analysis results of repetitive sequence and chromosomal distribution,the genomic relationship between Suaedaaustralis and Suaeda salsa was closer than that of Suaeda glauca.These findings would provide a basis for understanding the characteristics of repetitive sequences and the evolutionary relationships among the three species of coastal Suaeda.
Key words:Suaeda;Tandem repeat;Telomere;Centromere;rDNA;Fluorescence in situ hybridization
福建省的海岸线蜿蜒曲折,其滨海盐碱地面积广阔,达到5 240 hm2[1]。这些盐碱地土壤中盐分含量过高,限制了大多数植物的正常生长和发育[2]。因此,开发适应福建省盐碱地环境的植物种质资源,对于促进福建滨海盐碱地的农业产业发展和生态修复具有重要的战略意义。碱蓬属植物是一种耐盐性强、生物产量高、经济价值高的盐生植物[3]。其中,南方碱蓬Suaeda australis、盐地碱蓬Suaeda salsa和碱蓬Suaedaglauca是福建滨海盐碱地3种常见的碱蓬属物种[4−5]。随着盐生植物作物化和盐碱农业研究的不断深入,碱蓬属植物在经济价值和盐碱环境生态修复方面的潜力越来越受到重视[3]。
在真核生物的基因组中,重复序列占据了相当大的比例,它们曾被错误地看作“垃圾DNA”[6]。然而,越来越多的研究表明,这些重复序列在基因调控、染色体完整性维持以及基因组稳定性方面发挥着至关重要的作用[6−8]。与编码序列相比,重复序列展现出快速进化的特性,并在不同物种间表现出明显的物种特异性[9−10]。重复序列主要分为2大类:串联重复序列和转座元件[11]。串联重复序列倾向于在染色体的特定区域如着丝粒、端粒和染色体臂聚集[12−17]。有些转座元件广泛分布于所有染色体上,而另外一些转座元件则在某些特定的染色体位置如着丝粒处聚集,它们通过剪切粘贴或复制粘贴机制在基因组内移动[6]。对碱蓬属植物重复序列进行解析,有助于加深人们对碱蓬属植物的重复序列类型、丰度、染色体分布特征及其它们的种间进化关系的理解。
由于重复序列在基因组中的含量较高,通过低深度测序即可对特定物种的基因组DNA中的重复序列进行研究[18−19]。Novak等[20]开发的Repeat-Explorer2软件提供了一种简单、易操作且开源的全面分析重复序列的生物信息学工具。利用低深度测序结合生物信息学分析方法,已经揭示了包括甘蔗、棉花、小麦、马铃薯等复杂基因组植物的重复序列类型、丰度和结构[17,21−25]。因此,基于低深度测序的生物信息学分析方法是一种研究碱蓬属重复序列的高效且经济的策略。因此,本研究采用低深度测序技术和荧光原位杂交技术对3种滨海碱蓬的重复序列进行鉴定和染色体定位,比较分析它们的重复序列多样性、拷贝数差异和染色体分布特征,这些研究结果将为理解3种滨海碱蓬属物种的重复序列特征及其种间进化关系提供重要的理论依据。
1材料与方法
1.1植物材料
本研究中使用的植物材料包括3种滨海碱蓬属植物:南方碱蓬S.australis、盐地碱蓬S.salsa和碱蓬S.glauca,均在自然条件下生长。
1.2全基因组重复序列鉴定
首先,使用Illumina HiSeq平台对3种碱蓬属植物基因组DNA进行低深度双端测序。随后,使用FastQC对测序结果进行质量过滤。接下来,随机选取200万对测序数据,并将序列文件上传至Galaxy平台上的RepeatExplorer2软件服务器,并使用默认参数进行基于序列相似性的聚类分析[20]。为了评估重复序列在这3个物种之间丰度的变化,随机选取这3个物种的各自200万对双端测序reads,并将其合并为一个数据集,并使用默认参数使用RepeatExplorer2进行基于序列相似性的比较聚类分析[20]。
1.3 PCR扩增和探针制备
通过PCR扩增目标重复序列,PCR反应体系为20μL,包括200 ng基因组DNA、2.5 mmol·L−1 dNTPs、1×ExTaq缓冲液、10μmmol·L−1引物和5 U ExTaq DNA聚合酶。PCR条件为98℃预变性3 min,随后95℃变性30 s,58℃退火30 s,72℃延伸30 s,共35个循环,最后72℃终延伸10 min。最后,通过凝胶电泳检测扩增产物,并用Digo-xigenin-11-dUTP通过缺口平移法进行探针标记。
1.4染色体制备
染色体制备参照Huang等[14]的方法。首先,将幼叶放入2 mmmol·L−1 8-羟基喹啉中孵育3 h,然后在卡诺氏固定液中固定24 h。使用纤维素酶和果胶酶混合酶液在37℃下消化叶片2 h。将得到的细胞悬浮液滴到载玻片上展片,直到载玻片干燥。
1.5荧光原位杂交
荧光原位杂交参照Huang等[14]的方法。首先,将含有探针的杂交液在96℃下变性7 min,然后滴到70℃下变性的载玻片上。将载玻片置于37℃的杂交盒中过夜。用2×SSC洗脱3次,每次5 min,再用1×PBS洗脱5 min。将抗地高辛荧光素抗体添加到载玻片上,并在37℃下孵育1 h。随后,用1×PBS洗涤载玻片3次,每次5 min。干燥后,用4',6-二脒基-2-苯基吲哚(DAPI)对载玻片进行复染。最后,使用显微镜和相机捕获染色体图像,并使用Adobe Photoshop CS软件进行图像处理。
2结果与分析
2.1 3种滨海碱蓬属植物基因组中重复序列鉴定
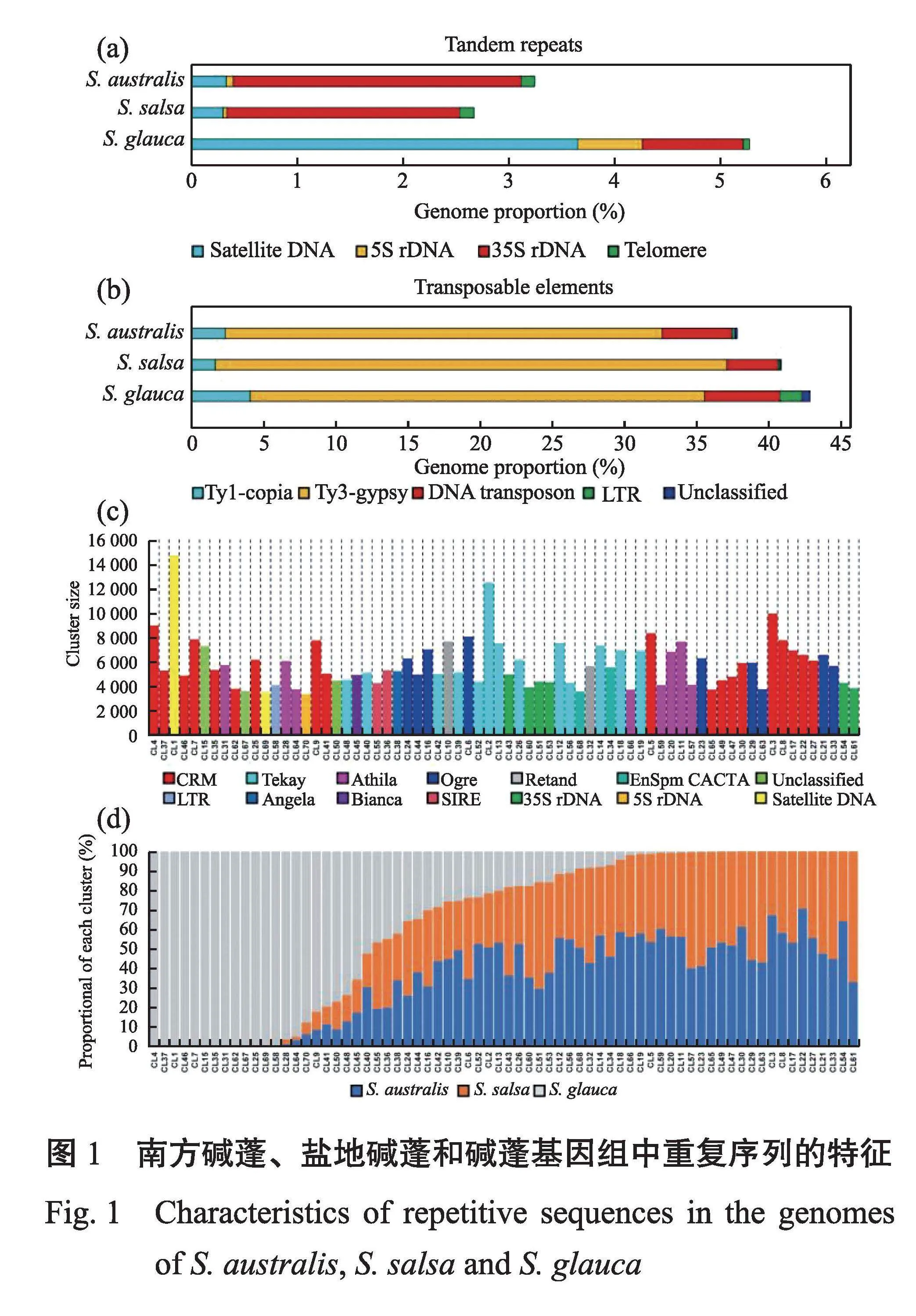
由图1可知,碱蓬、盐地碱蓬和南方碱蓬基因组中,串联重复序列(包括卫星序列、端粒序列、5S rDNA和35S rDNA)分别比的5.27%、2.84%和3.25%;转座元件分别占比41.64%、40.21%、38.22%,其中Ty3/gypsy家族反转录转座子的含量最高,Ty1/copia反转录转座子和DNA转座子含量则相对较低,同时还鉴定到一些未分类的重复序列。利用基于序列相似性的比较聚类分析,结果表明碱蓬具有大量物种特异的重复序列,而盐地碱蓬和南方碱蓬则共享大多数重复序列,暗示着与碱蓬相比,南方碱蓬和盐地碱蓬的基因组关系更近。其中,2个卫星序列为碱蓬物种特异串联重复序列,而端粒序列、5S rDNA、35S rDNA为3个碱蓬属物种保守的串联重复序列。此外,在转座元件中,Ty3/gypsy的CRM谱系和Athila谱系在3个碱蓬属物种中发生显著分化,Ty3/gypsy的Tekay谱系则在3个碱蓬属物种中更加保守。
2.2 3种滨海碱蓬属植物串联重复序列比较分析
由图2可知,在3个碱蓬属物种的基因组中,2个卫星序列和端粒序列显示出典型的星状图形,5S rDNA呈双环状形状,35S rDNA则呈线性排列。FISH试验结果表明,卫星序列Satellite 1定位于大多数碱蓬染色体的末端,而卫星序列Satellite 2则分布在碱蓬的4条染色体的末端。然而,在盐地碱蓬和南方碱蓬中并未检测到信号。端粒序列定位于这3个物种的所有染色体末端。在这3个物种中,5S rDNA均定位于2条染色体臂上。35SrDNA在南方碱蓬和盐地碱蓬中存在多个信号,包括染色体臂、染色体末端以及几个游离的脆弱位点信号。但是在碱蓬中,35S rDNA则在2条染色体的臂上产生明亮的信号。
2.3 3种滨海碱蓬属植物反转录转座子比较分析
由图3可知,在3个碱蓬属物种的基因组中,Ty3/gypsy转座元件可分为Tekay、CRM、Retand、Athila和Ogre谱系。FISH试验结果表明,CRM谱系主要集中在着丝粒区域,其他4个谱系的信号则散布分布在所有染色体上。此外,本试验发现来自CRM谱系的CL4为碱蓬物种特异的反转录转座子。
由图4可知,Ty1/copiay转座元件包括5个不同的谱系:Ale、Angela、Bianca、SIRE和TAR。FISH试验结果表明,来自Angela、Bianca和TAR谱系的反转录转座子探针在3个碱蓬属物种的整个染色体组中显示出相似分布的杂交信号。相比之下,SIRE和Ale谱系的信号通过FISH无法检测到或者信号极其微弱,这与其在基因组中较低的含量一致。
2.4 3种滨海碱蓬属植物DNA转座子比较分析
由图5可知,在3种碱蓬属物种中,DNA转座子主要由CACTA-TPase、Harbinger-TPase、hAT-TPase、MuDR-TPase谱系组成。FISH试验结果表明,来自CACTA-TPase谱系等转座元件分散在所有染色体上,其他谱系未检测到FISH信号。
3讨论与结论
重复序列在绝大多数真核生物基因组中构成了基因组的大部分[26]。例如,在玉米和小麦的基因组中,至少80%的序列由重复序列构成[27−28]。在本研究中,在3种碱蓬属物种中鉴定出5个串联重复序列。其中3个串联重复序列在3种滨海碱蓬中是保守的,包括5S rDNA、35S rDNA和端粒序列。值得注意的是,虽然它们在3个物种间是保守的,但它们在种间的拷贝数却存在相当大的差异。众所周知,端粒作为位于真核染色体末端的保护结构发挥作用,在保持DNA复制的保真度和保证基因组稳定性方面发挥着关键作用[29]。端粒长度异质性是包括植物、酵母、小鼠和人类在内的各种生物的共同特征[29]。同样,本研究发现3种滨海碱蓬物种不同染色体的端粒信号不同,表明了它们的染色体间端粒长度存在差异。此外,还观察到南方碱蓬和盐地碱蓬端粒的基因组比例大于碱蓬,其丰度的变化似乎与进化适应生态环境有关。此前的一些研究表明环境压力和地理分布可能会影响拟南芥、水稻和玉米等植物的端粒长度[30−32]。例如,在拟南芥等植物中端粒长度与纬度之间存在负相关[32],同时具有长端粒的拟南芥ku70突变体在高温条件下表现出增强的生殖适应性,但这种优势在较冷的环境中会减弱[30]。
反转录转座子作为植物基因组中普遍存在的一类主要重复序列元件,在功能变异的塑造、基因组可塑性以及进化过程中发挥重要作用[6]。反转录转座子在不同物种间的序列特征、结构组成以及染色体分布方面展现出显著的异质性。例如,在拟南芥基因组中,反转录转座子约占9.9%,而在玉米中,约85%的基因组序列由反转录转座子构成[9,27]。同样,本研究表明反转录转座子是3种碱蓬属物种基因组中的主要成分,在碱蓬、盐地碱蓬基因组中的占比分别为41.64%、40.21%、38.22%。基因组中反转录转座子的含量变化,部分可归因于这些元件扩增与删除过程之间的动态平衡[6]。反转录转座子可以是自主型的,即能够编码自身扩增所需的蛋白质,也可以是非自主型的,依赖于其他自主元件编码的蛋白质进行扩增[6]。本试验观察到Athila谱系在南方碱蓬和盐地碱蓬中丰度远高于碱蓬,表明该谱系在南方碱蓬和盐地碱蓬基因组发生扩增。此外,大多数反转录转座子随机分散在植物的基因组中,这一模式与本研究的结果一致。另外有些反转录转座子亚家族则聚集在特定的染色体区域,例如已知Ty3/gypsy反转录转座子的CRM谱系主要在大多数植物基因组的着丝粒区域聚集[14,33−35]。在几种植物物种中已经鉴定出许多着丝粒反转录转座子,如水稻中的RIRE7和CRR、玉米中的CRM、野生小麦中的CRW、甘蔗中的CRS、黑麦中的Bilby和大麦中的Cereba[34]。在本研究中,在3种碱蓬属物种中也检测到了CRM定位于着丝粒区域。但是,在莲花和拟南芥等少数物种的着丝粒区域则富集的是Ty1/copia反转录转座子[9,36]。植物着丝粒区域富含Ty3/gypsy反转录转座子的现象被广泛报道,这暗示在着丝粒的进化过程中,Ty3/gypsy可能较Ty1/copia具有更显著的进化优势[36]。
综上,本研究通过基于序列相似性聚类分析与荧光原位杂交技术对3种滨海碱蓬重复序列进行鉴定和染色体定位,揭示了这些物种的重复序列类型、丰度、染色体分布特征,根据重复序列和染色体分布分析结果表明,与碱蓬相比,南方碱蓬和盐地碱蓬的基因组关系更近。这些发现将为理解3种滨海碱蓬属物种重复序列的特征和种间进化关系提供了理论依据。
参考文献:
[1]高志强.福建滨海盐土客土改良效果研究[J].土壤学报,1995,32(1):101−107.
[2]YU Y,HUANG W,CHEN H,et al.Identification of differentially expressed genes in flax(Linum usitatissimum L.)under saline-alkaline stress by digital gene expression[J].Gene,2014,549(1):113−122.
[3]CHENG Y,YANG P,ZHAO L,et al.Studies on genome size estimation,chromosome number,gametophyte development and plant morphology of salt-tolerant halophyte Suaeda salsa[J].BMC Plant Biol,2019,19(1):473.
[4]LI J,HUSSAIN T,FENG X,GUO K,et al.Comparative study on the resistance of Suaeda glauca and Suaeda salsa to drought,salt,and alkali stresses[J].Ecol Eng,2019,140:105593.
[5]PARK J-S,JIN D-P,CHOI B-H.Insights into genomic structure and evolutionary processes of coastal Suaeda species in East Asia using cpDNA,nDNA,and genome-wide SNPs[J].Sci Rep,2020,10(1):20950.
[6]WICKER T,SABOT F,HUA-VAN A,et al.A unifiedclassification system for eukaryotic transposable elements[J].Nat Rev Genet,2007,8(12):973−982.
[7]WELLS J N,FESCHOTTE C.A Field Guide to Eukaryotic Transposable Elements[J].Annu Rev Genet,2020,54:539−561.
[8]CORDAUX R,BATZER M A.The impact of retrotransposons on human genome evolution[J].Nat Rev Genet,2009,10(10):691−703.
[9]NAISH M,ALONGE M,WLODZIMIERZ P,et al.The genetic and epigenetic landscape of the Arabidopsis centromeres[J].Science,2021,374(6569):eabi7489.
[10]ANAMTHAWAT-JóNSSON K,WENKE T,THóRSSON AT,SVEINSSON S,ZAKRZEWSKI F,SCHMIDT T.Evolutionary diversification of satellite DNA sequences from Leymus(Poaceae:Triticeae)[J].Genome,2009,52(4):381−390.
[11]BISCOTTI M A,OLMO E,HESLOP-HARRISON J S.Repetitive DNA in eukaryotic genomes[J].Chromosome Res,2015,23(3):415−420.
[12]ALTEMOSE N,LOGSDON G A,BZIKADZE A V,et al.Complete genomic and epigenetic maps of human centromeres[J].Science,2022,376(6588):eabl4178.
[13]CHENG Z,DONG F,LANGDON T,et al.Functional rice centromeres are marked by a satellite repeat and a centromere-specific retrotransposon[J].Plant Cell,2002,14(8):1691−1704.
[14]HUANG Y,DING W,ZHANG M,et al.The formation and evolution of centromeric satellite repeats in Saccharum species[J].Plant J,2021,106(3):616−629.
[15]MAHESHWARI S,ISHII T,BROWN C T,et al.Centromere location in Arabidopsis is unaltered by extreme divergence in CENH3 protein sequence[J].Genome Res,2017,27(3):471−478.
[16]WLODZIMIERZ P,RABANAL F A,BURNS R,et al.Cycles of satellite and transposon evolution in Arabidopsis centromeres[J].Nature,2023,618(7965):557−565.
[17]YANG X,ZHAO H,ZHANG T,et al.Amplification and adaptation of centromeric repeats in polyploid switchgrass species[J].New Phytol,2018,218(4):1645−1657.
[18]NOVáK P,ÁVILA R L,KOBLíŽKOVáA,et al.TAREAN:a computational tool for identification and characterization of satellite DNA from unassembled short reads[J].Nucleic Acids Res,2017,45(12):e111−e111.
[19]NOVáK P,NEUMANN P,MACAS J.Graph-based clustering and characterization of repetitive sequences in next-generation sequencing data[J].BMC Bioinformatics,2010,11(1):378.
[20]NOVáK P,NEUMANN P,MACAS J.Global analysis of repetitive DNA from unassembled sequence reads using RepeatExplorer2[J].Nat Protoc,2020,15(11):3745−3776.
[21]ZHANG H,KOBLIZKOVA A,WANG K,et al.Boom-bust turnovers of megabase-sized centromeric DNA in Solanum species:Rapid evolution of DNA sequences associated with centromeres[J].Plant Cell,2014,26(4):1436−1447.
[22]HUANG Y,CHEN H,HAN J,et al.Species-specific abundantretrotransposons elucidate the genomic composition of modern sugarcane cultivars[J].Chromosoma,2020,129(1):45−55.
[23]GONG Z,WU Y,KOBLIZKOVA A,et al.Repeatless and repeat-based centromeres in potato:implications for centromere evolution[J].Plant Cell,2012,24(9):3559−3574.
[24]SU H,LIU Y,LIU C.Centromere satellite repeats have undergone rapid changes in polyploid wheat subgenomes[J].Plant Cell,2019,31(9):2035−2051.
[25]HAN J,MASONBRINK R E,SHAN W,et al.Rapid proliferation and nucleolar organizer targeting centromeric retrotransposons in cotton[J].Plant J,2016,88(6):992−1005.
[26]GEMMELL N J.Repetitive DNA:genomic dark matter matters[J].Nat Rev Genet,2021,22(6):342−342.
[27]CHEN J,WANG Z,TAN K,et al.A complete telomere-to-telomere assembly of the maize genome[J].Nat Genet,2023,55(7):1221−1231.
[28]MIDDLETON C P,STEIN N,KELLER B,et al.Comparative analysis of genome composition in Triticeae reveals strong variation in transposable element dynamics and nucleotide diversity[J].Plant J,2013,73(2):347−356.
[29]SRINIVAS N,RACHAKONDA S,KUMAR R.Telomeres and Telomere Length:A General Overview[J].Cancers(Basel),2020,12(3):558.
[30]CAMPITELLI B E,RAZZAQUE S,BARBERO B,et al.Plasticity,pleiotropy and fitness trade-offs in Arabidopsis genotypes with different telomere lengths[J].New Phytol,2022,233(4):1939−1952.
[31]ABDULKINA L R,KOBAYASHI C,LOVELL J T,et al.Components of the ribosome biogenesis pathway underlie establishment of telomere length set point in Arabidopsis[J].Nat Commun,2019,10(1):5479.
[32]CHOI J Y,ABDULKINA L R,YIN J,et al.Natural variation in plant telomere length is associated with flowering time[J].Plant Cell,2021,33(4):1118−1134.
[33]NEUMANN P,NOVáK P,HOŠTáKOVáN,et al.Systematic survey of plant LTR-retrotransposons elucidates phylogenetic relationships of their polyprotein domains and provides a reference for element classification[J].Mob DNA,2019,10:1.
[34]NEUMANN P,NAVRáTILOVáA,KOBLíŽKOVáA,et al.Plant centromeric retrotransposons:a structural and cytogenetic perspective[J].Mob DNA,2011,2(1):4.
[35]GAO D,GILL N,KIM H R,et al.A lineage-specific centromere retrotransposon in Oryza brachyantha[J].PlantJ,2009,60(5):820−831.
[36]ZHU Z,GUI S,JIN J,et al.The NnCenH3 protein and centromeric DNA sequence profiles of Nelumbo nucifera Gaertn.(sacred lotus)reveal the DNA structures and dynamics of centromeres in basal eudicots[J].Plant J,2016,87(6):568−582.
(责任编辑:柯文辉)
