中国南瓜CmoAux/IAA基因的克隆和表达特征分析
2024-10-25任广乾刘正响韩圆圆陈碧华李新峥李庆飞






摘 要:在前期对中国南瓜败育雌蕊和正常雌蕊进行转录组分析发现,CmoAux/IAA基因可能参与了雌花发育,为进一步探究其生物学功能,利用同源序列克隆法从中国南瓜中克隆得到全长为600 bp的CmoAux/IAA基因,该基因编码199个氨基酸;保守结构域分析结果表明,其属于Aux/IAA家族,包含结构域Ⅰ、Ⅲ、Ⅳ;该蛋白无信号肽和跨膜结构。多序列比对和系统进化树分析结果显示,CmoAux/IAA氨基酸序列与印度南瓜XP_022986212.1的氨基酸序列、美洲南瓜XP_023513171.1的氨基酸序列亲缘关系较近,相似度高达98.01%、96.50%。拟南芥原生质体亚细胞定位结果显示,CmoAux/IAA蛋白定位于细胞核和细胞质。实时荧光定量结果显示,该基因具有组织表达特异性,主要在南瓜植株生长点及雌花柱头中表达,柱头中的相对表达量是雄蕊的137倍,在叶和根中不表达,其可能在植株生长点幼嫩组织分化或雌花发育过程中发挥着重要作用。
关键词:中国南瓜;Aux/IAA;基因克隆;生物信息学分析;亚细胞定位
中图分类号:S642.1 文献标志码:A 文章编号:1673-2871(2024)10-030-08
收稿日期:2024-04-29;修回日期:2024-07-07
基金项目:国家自然科学基金(32302559);河南省科技攻关项目(242102110312);河南省高等学校青年骨干教师培养计划(2024GGJS102);河南省重大科技专项(241100110200)
作者简介:任广乾,男,助理研究员,主要从事瓜类品种选育研究工作。E-mail:1491748031@qq.com
共同第一作者:刘正响,男,在读硕士研究生,主要从事南瓜重要农艺性状基因挖掘研究工作。E-mail:liuzhengxiang33@163.com
通信作者:李庆飞,副教授,主要从事蔬菜种质资源创新与利用研究工作。E-mail:lqf_20131025@126.com
Cloning and expression analysis of CmoAux/IAA gene in Cucurbita moschata
REN Guangqian1, LIU Zhengxiang2, HAN Yuanyuan2, CHEN Bihua2, LI Xinzheng2, LI Qingfei2
(1. Xinxiang City Academy of Agricultural Sciences, Xinxiang 453003, Henan, China; 2. College of Horticulture and Landscape, Henan Institute of Science and Technology, Xinxiang 453003, Henan, China)
Abstract: In the previous study, transcriptomic analysis of abortive pistil and normal pistil of Cucurbita moschata found that CmoAux/IAA gene might be involved in female flower development. In order to further explore its biological function, a 600 bp gene(CmoAux/IAA) was cloned from Cucurbita moschata by homologous sequence cloning method and it encodes 199 amino acids. The results of conserved domain analysis showed that it belongs to the Aux/IAA family and includes domains Ⅰ, Ⅲ, and Ⅳ. The protein has no signal peptide and transmembrane structure. Multiple sequence alignment and phylogenetic tree results showed that the amino acid sequence of CmoAux/IAA were closely related to XP_022986212.1 of Cucurbita maxima and XP_023513171.1 of Cucurbita pepo, and the similarity was 98.01% and 96.50%, respectively. The results of subcellular localization of Arabidopsis protoplasts showed that the protein was located in nucleus and cytoplasm. Real-time fluorescence quantitative results showed that CmoAux/IAA has tissue expression specificity, mainly expressed in the growing points and stigma of female flowers in pumpkin plants, and the expression level of CmoAux/IAA in stigma was 137 fold that of in stamen, but not expressed in leaves and roots and it may play an important role in the differentiation of young tissue or the development of female flowers.
Key words:Cucurbita moschata; Aux/IAA; Gene cloning; Bioinformatics analysis; Subcellular localization
南瓜为葫芦科南瓜属一年生蔓生草本植物,是一种集食用、药用、观赏兼加工的蔬菜作物[1],在世界各地广泛栽培,主要栽培种有中国南瓜(Cucurbita moschata)、印度南瓜(Cucurbita maxima)、美洲南瓜(Cucurbita pepo)和黑籽南瓜(Cucurbita ficifolia),其中,中国南瓜栽培范围最广泛。我国是南瓜生产大国,据2022年FAO(https://www.fao.org/faostat/zh/#data/QCL)统计,我国南瓜年栽培面积约39.8万hm2,年总产量737.8万t,均居世界第一位。提高南瓜产量是增加经济效益的有效途径,而雌花与产量息息相关,因此,研究南瓜雌花发育相关基因具有重要意义。
生长素是植物最重要的激素之一,在生长发育过程中具有重要作用,包括细胞分裂、叶片延伸、开花模式、顶端优势、向性生长和抗逆性等[2]。植物生长素信号转导途径包括信号识别、下游生长素相关基因的表达以及植物的生理反应等,下游生长素相关基因又称为早期/原初响应基因,主要包括生长素/吲哚-3-乙酸(auxin/indole-3-acetic acid,Aux/IAA)、生长素响应因子(ARF)、GH3(gretchen hagen 3)和SAUR(small auxin-up RNA)等基因家族[3-4]。Aux/IAA蛋白通常包含4个保守结构域[3,5]。结构域Ⅰ含有“LxLxLx”(亮氨酸重复单元)基序,该基序与乙烯响应因子相关,是Aux/IAA蛋白转录抑制功能不可缺少的一部分[6]。结构域II具有保守的“GWPP”基序,主要负责Aux/IAAs的生长素依赖性降解[7-8]。结构域III和结构域IV包含一个羧基端PB1(Phox和Bem1)结构域,该结构域也位于ARFs的羧基端,负责Aux/IAA和ARF蛋白之间的同源和异源二聚化[9-10];结构域Ⅲ由三部分组成(β、α1和α2)[11]。结构域IV包含1个保守基序(GDVPW)和1个保守的SV40型核定位信号(NLS:KRxRxxK),有助于二聚化[12-13]。在结构域I和II之间发现的双向保守NLS将Aux/IAA蛋白引导到细胞核中,比SV40型NLS更有效[11,14]。同时具有4个结构域的蛋白称为典型的Aux/IAA蛋白,而至少缺1个结构域的Aux/IAA蛋白被称为是非典型的Aux/IAA蛋白,这种非典型的Aux/IAA蛋白在植物中普遍存在,且在植物生长发育过程中发挥着重要作用[15-17]。Aux/IAA是一种转录抑制因子,通过与参与生长素基因表达响应的ARFs结合抑制下游基因的表达[18]。生长素调控着Aux/IAA蛋白的降解,低浓度生长素抑制ARF转录,调节生长素诱导的基因表达;高浓度生长素使Aux/IAA发生降解,减轻了Aux/IAA对ARF的抑制作用,使其参与到下游靶基因的调控[19-20]。在拟南芥中,Aux/IAA家族基因IAA8与ARF6和ARF8互作,影响下游生长素诱导基因的表达[21];而ARF6和ARF8在柱头、萼片、花丝及花瓣等花器官中均有表达[22],通过影响茉莉酸含量可调控花药开裂影响授粉[23];因此,Aux/IAA间接参与调节拟南芥花器官的发育。在番茄中,SIIAA27表达下调会降低胚珠和花粉育性,而敲除SIIAA27基因则在影响根发育的同时也会降低叶片中的叶绿素含量[24]。与野生型相比,稳定的IAA16突变体使得花丝伸长率下降[25]。
南瓜是雌雄异花同株植物,是研究雌雄花发育的有利材料,但相比黄瓜、甜瓜等同科作物,南瓜雌花发育研究相对落后。河南科技学院南瓜种质资源研究课题组前期研究发现,1个CmoAux/IAA基因在中国南瓜败育雌蕊中下调表达,推测其可能与雌花发育有关,且南瓜中尚未见有关CmoAux/IAA基因的报道。笔者对该基因进行克隆、亚细胞定位及生物信息学分析,并研究了该基因在不同组织和不同花结构中的表达模式,研究结果将有助于深入探究CmoAux/IAA基因的生物学功能。
1 材料与方法
1.1 植物材料
试验于2021年5月至2022年5月在河南科技学院园艺园林学院实验室及基地进行。试验所用植物材料为河南科技学院南瓜课题组提供的中国南瓜自交系2018-3-1。
1.2 方法
1.2.1 中国南瓜CmoAux/IAA基因的克隆 采用试剂盒进行南瓜材料总RNA提取以及cDNA第一链合成,依据中国南瓜基因组数据库(http://cucurbitgenomics.org/organism/9)中同源基因的CDS序列设计引物,引物序列为:CmoAux/IAA-F(CATCTAGAATGGAGCCTAATCAGTTCT)、CmoAux/IAA-R(ACCTCGAGATTTTGATGAATGGCA)。以cDNA为模板进行PCR扩增,用1%的琼脂糖凝胶电泳检测,回收CmoAux/IAA基因片段连接至pMD19-T载体,转入大肠杆菌DH5α,将阳性菌液进行测序,选择测序无误的菌液进行后续试验。
1.2.2 CmoAux/IAA基因的生物信息学分析 利用Deep TMHMM预测跨膜结构;利用ExPASy ProParam tool预测CmoAux/IAA蛋白的分子质量、理论等电点pI、平均亲水性和不稳定系数等理化性质;Net Phos3.1 Serve预测CmoAux/IAA蛋白的磷酸化位点;NPS@:SOPMA预测CmoAux/IAA蛋白的二级结构;SignalP 6.0 Server预测CmoAux/IAA基因编码蛋白的信号肽;NCBI-CDS预测保守结构域;NCBI BLAST进行同源性比对分析;DNAMAN 6.0软件进行多序列比对分析,MEGA 7.0构建系统进化树。所用工具网站见表1。
1.2.3 CmoAux/IAA蛋白的亚细胞定位 利用WoLF PSORT和Plant-mPLoc server预测CmoAux/IAA亚细胞位置,并构建GFP融合表达载体转化拟南芥原生质体,确定CmoAux/IAA蛋白表达的亚细胞位置。原生质体的制备及重组质粒转化原生质体参照Yoo等[26]、陈培雯等[27]的方法。
1.2.4 CmoAux/IAA在不同组织和花不同结构中的表达分析 为了研究CmoAux/IAA基因在中国南瓜中的表达模式,利用基因特异性荧光定量引物(qCmoAUX-IAA-F:ATTCCAATCTCGCACTT;qCmoAUX-IAA-R:CTCAGCCTCTTTACATCC),采用实时荧光定量qRT-PCR对基因在同一植株根、茎、叶、生长点以及相邻节位的雌花(柱头、雌花萼片、子房)、雄花(雄蕊、雄花萼片)中的表达水平进行分析。当植株处于开花结果期时,采取完全随机设计,随机选取长势一致、生长健壮无病害的植株。具体取样部位:根为主根不含侧根,茎为由下至上第5到第6片真叶之间,叶为由下至上第5片真叶,生长点为主蔓生长点;花不同结构取自2018-3-1同一植株上当天开放的雌花和雄花,用解剖刀将雌花分为雌花萼片、柱头和子房三部分,雄花分为雄花萼片和雄蕊两部分。取样后立即投入液氮中冷冻,存于-80 ℃超低温冰箱待用。样本的RNA提取、cDNA反转录参考李庆飞等[28]的方法。每个样品设3个生物学重复,相对表达量计算采用2−ΔΔCt法[29],利用DPS 7.0.5.8软件进行差异显著性分析。
2 结果与分析
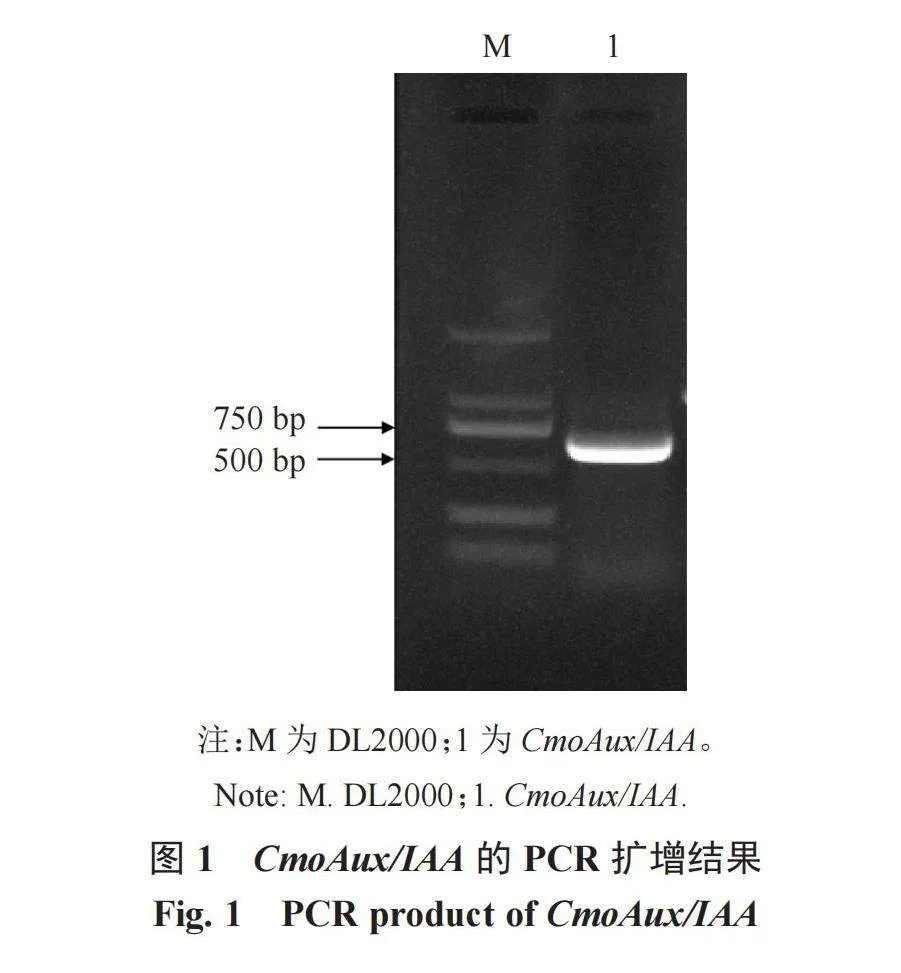
2.1 中国南瓜CmoAux/IAA基因的获得
以南瓜自交系2018-3-1雌花的cDNA为模板,用CmoAux/IAA-F和CmoAux/IAA-R引物进行PCR扩增,电泳结果如图1。CmoAux/IAA基因的CDS全长为600 bp。
2.2 CmoAux/IAA蛋白的生物信息学分析
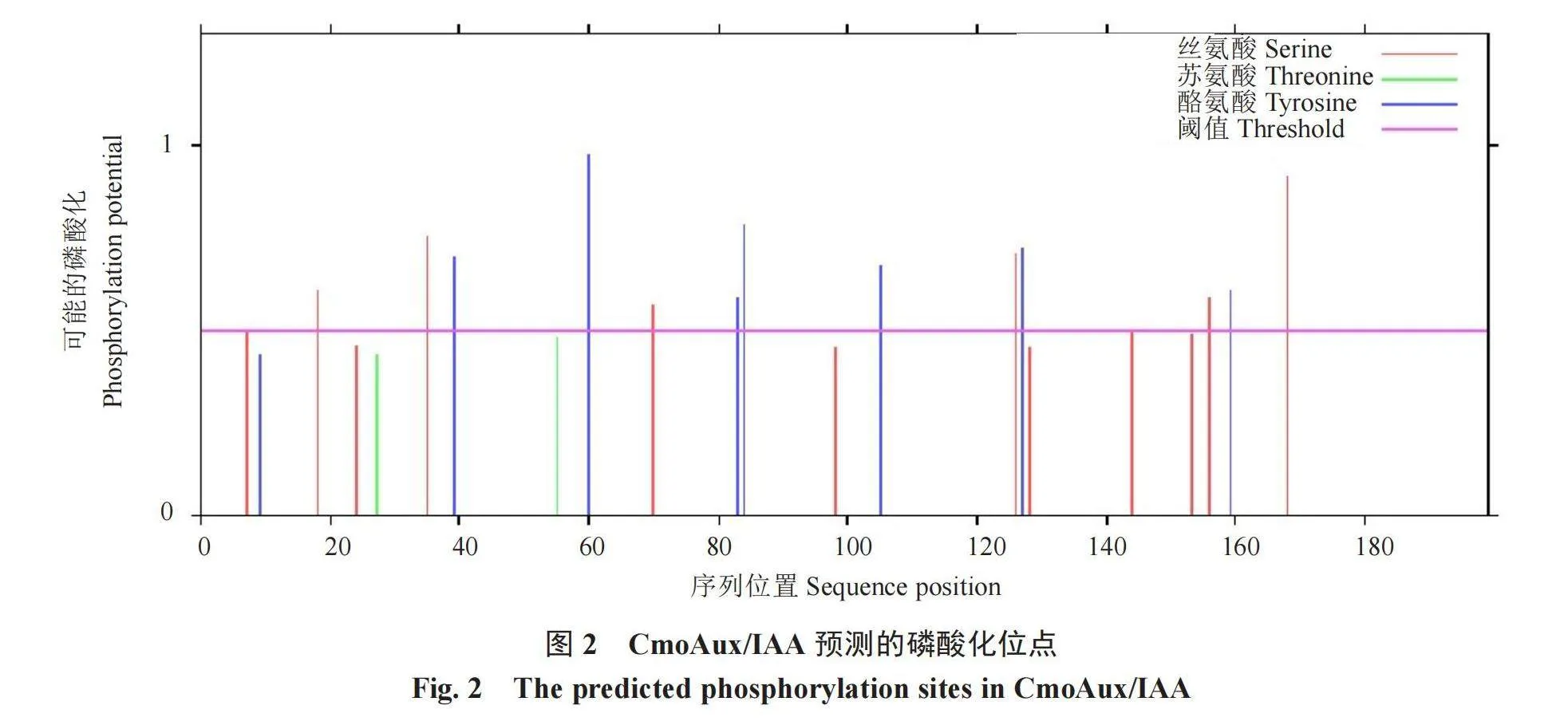
2.2.1 CmoAux/IAA蛋白的理化性质 ProParam对CmoAux/IAA蛋白的理化性质预测结果表明,CmoAux/IAA蛋白分子质量为22.70 kDa,分子式为C992H1509N273O320S10,由199个氨基酸组成,理论等电点pI为4.57,消光系数34 170,正电荷残基(精氨酸+赖氨酸)18个,负电荷残基(天冬氨酸+谷氨酸)36个,脂肪氨基酸系数73.37,不稳定系数为45.93,平均亲水性为-0.595。NetPhos 3.1 server预测CmoAux/IAA蛋白包含2个苏氨酸位点、8个酪氨酸位点、12个丝氨酸位点共22个磷酸化位点,详见图2。
2.2.2 CmoAux/IAA的蛋白结构分析 利用NPS@:SOPMA对CmoAux/IAA蛋白预测,发现CmoAux/IAA蛋白199个氨基酸中,无规则卷曲占比最高,为37.19%,其次是α-螺旋占比27.64%,延伸链占比26.13%,β-转角占比最低,为9.05%(图3)。
SignalP 6.0 server预测结果显示CmoAux/IAA蛋白无信号肽(图4-a);Deep TMHMM预测结果显示CmoAux/IAA蛋白无跨膜结构(图4-b)。
2.2.3 CmoAux/IAA蛋白保守结构域分析 利用NCBI在线预测CmoAux/IAA的保守结构域,结果显示,CmoAux/IAA基因所编码的氨基酸序列含有1个Aux/IAA(104~188位氨基酸)结构域,如图5所示。
2.3 CmoAux/IAA蛋白的同源序列比对及进化关系分析
同源序列比对发现,CmoAux/IAA氨基酸序列与中国南瓜数据库中基因(XP_022957191.1)的氨基酸序列完全相同,与印度南瓜(Cucurbita maxima,XP_022986212.1)相似度最高,为98.01%,与美洲南瓜(Cucurbita pepo,XP_023513171.1)相似度为96.50%,与冬瓜(Benincasa hispida,XP_038893192.1)相似度为62.25%,与黄瓜(Cucumis sativus,XP_004135388)相似度为52.22%,与甜瓜(Cucumis melo,XP_008446639.1)的相似度最低,为50.96%。同时发现氨基酸序列中包含结构域Ⅰ的“LxLxL”基序、结构域Ⅲ的β、α1、α2及结构域Ⅳ的保守基序“GDVPW”和核定位信号“KRLR”基序(图6)。
由图7系统进化树可知,中国南瓜CmoAux/IAA与印度南瓜亲缘关系最近,其次为美洲南瓜,与甜瓜、黄瓜和冬瓜等的亲缘关系较远。
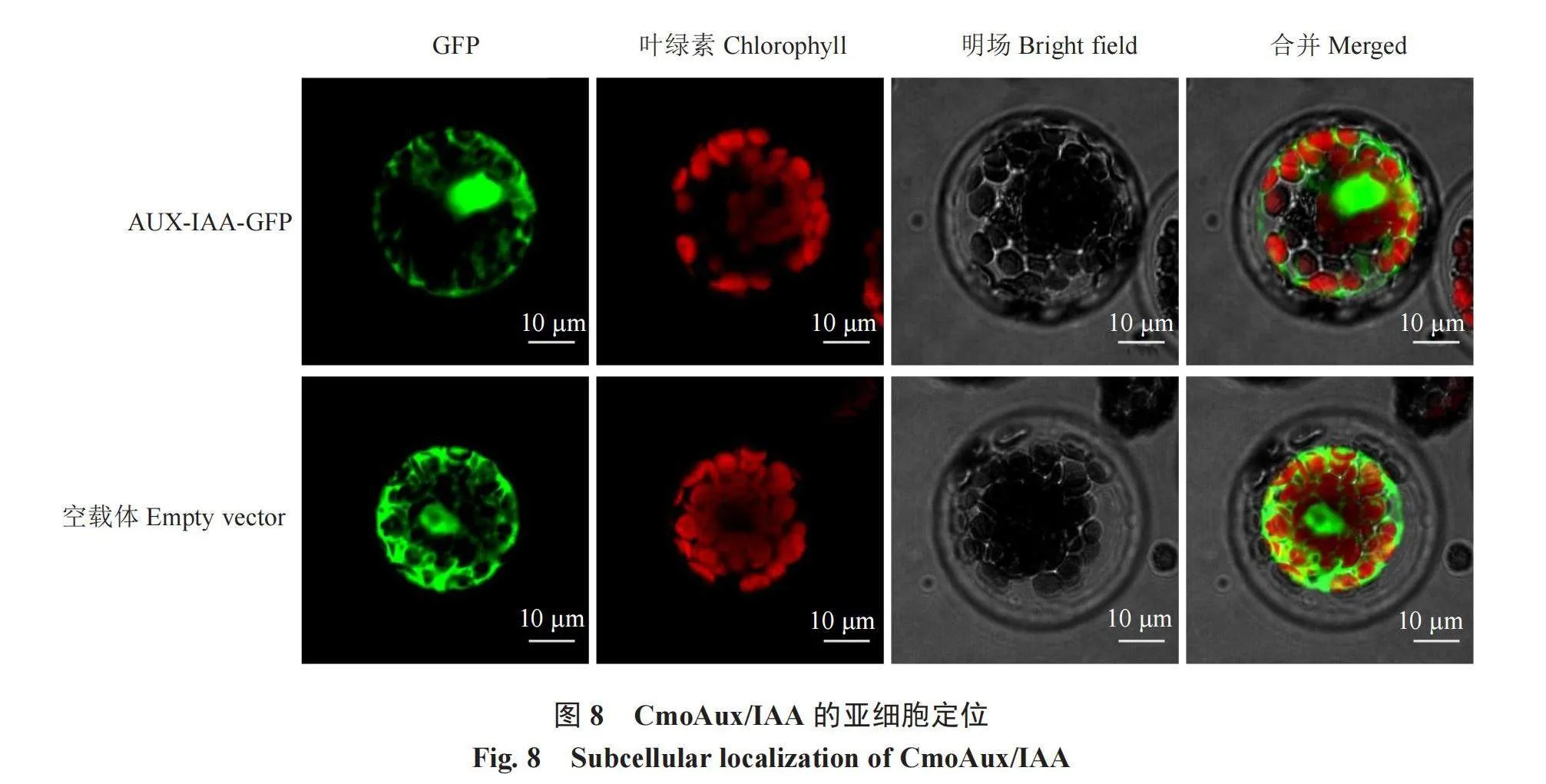
2.4 CmoAux/IAA蛋白的亚细胞定位
WoLF PSORT Prediction和Plant-mPLoc server预测结果显示,CmoAux/IAA蛋白可能位于细胞核和细胞质。如图8所示,CmoAux/IAA-GFP融合蛋白在细胞核和细胞质中有明显的绿色荧光,说明CmoAux/IAA蛋白位于细胞核和细胞质中。
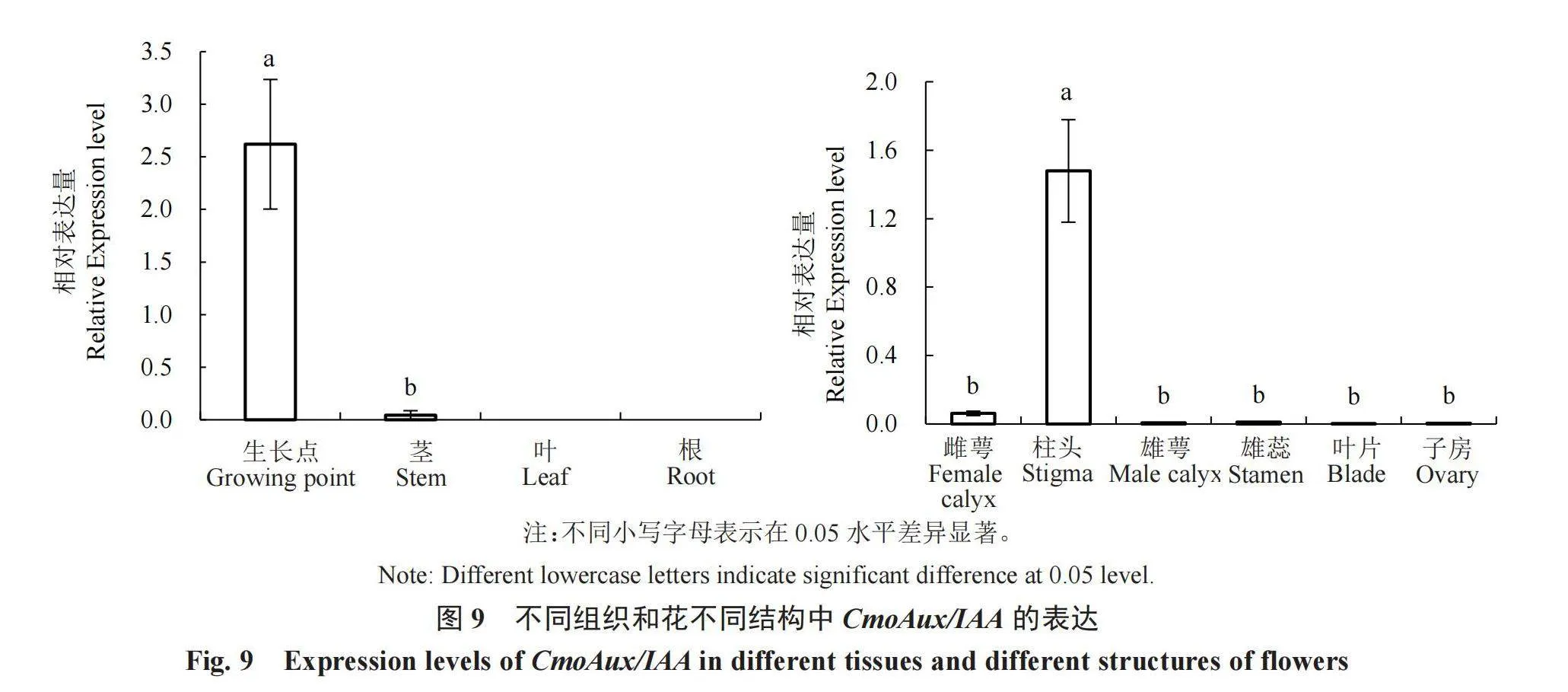
2.5 CmoAux/IAA在植株不同组织和花不同结构中的表达
对该基因的表达模式进行分析,结果显示CmoAux/IAA在生长点中表达量最高,是茎中的58倍,在叶和根中不表达。进一步分析其在花不同结构中的表达模式,发现该基因在雌花柱头及雌花萼片结构中的表达水平较高,在柱头中的相对表达量显著高于雄花各个结构。其中,在雌花柱头中的表达量最高,其次为雌花萼片,柱头中的相对表达量是雄蕊的137倍(图9),表明该基因具有明显的组织表达特异性。
3 讨论与结论
Aux/IAA是生长素信号转导通路的转录抑制因子,抑制生长素响应因子的表达,在植物根、芽、花和果实的生长发育中发挥多效性作用[2,6,30-32]。南瓜雌花发育是否正常直接关系到能否正常授粉,子房是否膨大结实,因此,研究南瓜雌花发育的关键基因具有重要意义。笔者所在课题组前期研究发现了1个CmoAux/IAA基因可能与中国南瓜雌花发育有关。
笔者通过克隆CmoAux/IAA基因进行生物信息学分析发现,CmoAux/IAA蛋白等电点值为4.54,与大多数Aux/IAA蛋白一致,表现为酸性[33-34]。CmoAux/IAA基因编码的蛋白包含1个Aux/IAA超家族结构域,与三月李中PsIAA1、PsIAA2、PsIAA4等9个蛋白质一致[35]。研究发现,多数Aux/IAA家族基因预测在细胞核中[3,33-36]。三月李中PsIAA8和PsIAA10预测在细胞核和细胞质中[35],与本试验基因定位结果一致。同源多序列比对和进化树分析发现,美洲南瓜和印度南瓜的氨基酸序列与CmoAux/IAA氨基酸序列高度相似,说明该蛋白在进化过程中比较保守。多序列比对分析发现,CmoAux/IAA蛋白不含结构域Ⅱ的“GWPP”基序且只含有1个NLS;在番茄中发现Sl-IAA4、Sl-IAA22、Sl-IAA29基因严格靶向细胞核,表明退化的NLS足以将蛋白质特异性驱动到细胞核,而Sl-IAA32定位延伸到核外区室可能是由于缺乏部分NLS或缺乏负责蛋白质降解的结构域Ⅱ,从而提出了Aux/IAA可能参与独立于传统生长素信号通路机制的假设[36],这与本试验结果相一致。测序结果显示第348位碱基T突变为C,但是由于密码子具有简并性,并未引起氨基酸序列的变化。许多研究表明,同源基因在不同物种间可能具有功能上的相似性[3]。笔者通过实时荧光定量发现CmoAux/IAA基因在雌花柱头中的表达量显著高于其他花结构,表明CmoAux/IAA基因可能在雌花生长发育过程中扮演重要角色,推测可能与雌花发育相关。在玉米中,ZmIAA29的过表达显著提前了玉米的雄穗授粉和吐丝时间,这意味着ZmIAA29在生殖转化和花形态建成中起着关键作用[37]。在拟南芥中,IAA8可能与ARF6/8蛋白存在相互作用,影响花器官的发育[38]。在铁皮石斛中,DoIAA4、DoIAA5、DoIAA6、DoIAA8分别在花瓣、花柱、萼片和花蕾中高表达,表明它们在花发育中的作用,与本试验结果相呼应[39]。在典型的被子植物中,花发育过程有许多快速、有序和短暂的事件,这些事件包括萼片打开露出内部器官,花瓣、雄蕊和花柱伸长,花药开裂释放花粉,柱头成熟使花粉萌发[38]。Aux/IAA是一种短寿命的核蛋白[40],生长素响应基因Aux/IAA1和Aux/IAA2在草莓果实发育早期阶段强烈上调,但在整个成熟过程中显著降低[6]。不同组织中表达模式分析发现,CmoAux/IAA基因在生长较为旺盛的生长点中高表达,在茎中的表达显著低于生长点,而在叶中未检测到CmoAux/IAA基因表达,说明CmoAux/IAA是一种短寿命蛋白,与前人研究结果相符。本研究结果为后续深入探索CmoAux/IAA的生物学功能提供了参考。
本研究中的CmoAux/IAA基因CDS全长600 bp,编码蛋白理论等电点pI为4.57,是不稳性定亲水蛋白,位于细胞核和细胞质中,共22个磷酸化位点,无信号肽和跨膜结构,缺少结构域Ⅱ且只有1个核定位信号,是非典型的Aux/IAA蛋白。该蛋白与印度南瓜(XP_022986212.1)亲缘关系最近。CmoAux/IAA基因在南瓜植株生长点及雌花柱头中的表达量较高,具有明显的组织表达特异性。
参考文献
[1] 赵一鹏,李新峥,周俊国.世界南瓜生产现状及其种群多样性特征[J].内蒙古农业大学学报(自然科学版),2004,25(3):112-115.
[2] SU Y L,HE H H,WANG P,et al.Genome-wide characterization and expression analyses of the auxin/indole-3-acetic acid (Aux/IAA) gene family in apple (Malus domestica)[J].Gene,2021,768:145302.
[3] XU H W,LIU Y,ZHANG S M,et al.Genome-wide identification and expression analysis of the Aux/IAA gene family in turnip (Brassica rapa ssp.rapa)[J].BMC Plant Biology,2023,23(1),342.
[4] ZHU H Y,LI H J,YU J Y,et al.Regulatory mechanisms of ArAux/IAA13 and ArAux/IAA16 in the rooting process of Acer rubrum[J].Genes,2023,14(6):1206.
[5] HU Q,YANG J J,MENG L H,et al.Genome-wide identification of Aux/IAA genes in watermelon reveals a crucial role for ClIAA16 during fruit ripening[J].Horticulturae,2023,9(11):1167.
[6] IQBAL S,HAYAT F,MUSHTAQ N,et al.Bioinformatics study of Aux/IAA family genes and their expression in response to different hormones treatments during Japanese apricot fruit development and ripening[J].Plants-Basel,2022,11(15):1898.
[7] LUO J,ZHOU J J,ZHANG J Z.Aux/IAA gene family in plants:Molecular structure,regulation,and function[J].International Journal of Molecular Sciences,2018,19(1):259.
[8] WANG J,XUE L,ZHANG X,et al.A new function of MbIAA19 identified to modulate Malus plants dwarfing growth[J].Plants-Basel,2023,12(17):3097.
[9] REDDY V A,SAJU J M,NADIMUTHU K,et al.A non-canonical Aux/IAA gene MsIAA32 regulates peltate glandular trichome development in spearmint[J].Frontiers in Plant Science,2024,15:1284125.
[10] FAN J M,DENG M J,LI B B,et al.Genome-wide identification of the Paulownia fortunei Aux/IAA gene family and its response to witches' broom caused by phytoplasma[J].International Journal of Molecular Sciences,2024,25(4):2260.
[11] CHENG W H,ZHANG M,CHENG T R,et al.Genome-wide identification of Aux/IAA gene family and their expression analysis in Prunus mume[J].Frontiers in Genetics,2023,14:1208488.
[12] OUELLET F,OVERVOORDE P J,THEOLOGIS A.IAA17/AXR3:Biochemical insight into an auxin mutant phenotype[J].Plant Cell,2001,13(4):829-841.
[13] GUIFOYLE T J,HAGEN G.Getting a grasp on domain III/IV responsible for auxin response factor-IAA protein interactions[J].Plant Science,2012,190:82-88.
[14] WU J,PENG Z,LIU S Y,et al.Genome-wide analysis of Aux/IAA gene family in solanaceae species using tomato as a model[J].Molecular Genetics and Genomics,2012,287(4):295-311.
[15] KORASICK D A,WESTFALL C S,LEE S G,et al.Molecular basis for AUXIN RESPONSE FACTOR protein interaction and the control of auxin response repression[J].Proceedings of the National Academy of Sciences of the United States of America,2014,111(14):5427-5432.
[16] LV B S,YU Q Q,LIU J J,et al.Non-canonical Aux/IAA protein IAA33 competes with canonical Aux/IAA repressor IAA5 to negatively regulate auxin signaling[J].Embo Journal,2020,39(1):e101515.
[17] GUILFOYLE T J.The PB1 domain in auxin response factor and Aux/IAA proteins:A versatile protein interaction module in the auxin response[J].Plant Cell,2015,27(1):33-43.
[18] WANG S C.Light promotes protein stability of Auxin Response Factor 7[J].Phyton-International Journal of Experimental Botany,2023,92(4):1153-1160.
[19] GALLI M,KHAKHAR A,LU Z F,et al.The DNA binding landscape of the maize auxin response factor family[J].Nature Communications,2018,9(1):4526.
[20] WEI S Y,CHEN Y N,HOU J,et al.Aux/IAA and ARF gene families in Salix suchowensis:Identification,evolution,and dynamic transcriptome profiling during the plant growth process[J].Frontiers in Plant Science,2021,12:666310.
[21] 王婧.拟南芥Aux/IAA家族基因IAA8参与花器官发育的研究[D].武汉:武汉大学,2018.
[22] ISHIGURO S,KAWAI-ODA A,UEDA J,et al.The DEFECTIVE IN ANTHER DEHISCIENCE1 gene encodes a novel phospholipase A1 catalyzing the initial step of jasmonic acid biosynthesis,which synchronizes pollen maturation,anther dehiscence,and flower opening in Arabidopsis[J].Plant Cell,2001,13(10):2191-2209.
[23] NAGPAL P,ELLIS C M,WEBER H,et al.Auxin response factors ARF6 and ARF8 promote jasmonic acid production and flower maturation[J].Development,2005,132(18):4107-4118.
[24] 李艳艳,齐艳华.植物Aux/IAA基因家族生物学功能研究进展[J].植物学报,2022,57(1):30-41.
[25] RINALDI M A,LIU J,ENDERS T A,et al.A gain-of-function mutation in IAA16 confers reduced responses to auxin and abscisic acid and impedes plant growth and fertility[J].Plant Molecular Biology,2012,79(4/5):359-373.
[26] YOO S D,CHO Y H,SHEEN J.Arabidopsis mesophyll protoplasts:A versatile cell system for transient gene expression analysis[J].Nature Protocols,2007,2(7):1565-1572.
[27] 陈培雯,张立,陈学进,等.中国南瓜CmNPR1基因的克隆和表达分析[J].西北植物学报,2021,41(12):2038-2045.
[28] 李庆飞,王梦梦,郭卫丽,等.南瓜转录因子基因CmRAV的克隆与表达分析[J].植物生理学报,2021,57(12):2337-2345.
[29] LIVAK K J,SCHMITTGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△C T Method[J].Methods,2001,25(4):402-408.
[30] JIANG L,LI Z N,YU X M,et al.Bioinformatics analysis of Aux/IAA gene family in maize[J].Agronomy Journal,2021,113(2):932-942.
[31] BREITEL D A,CHAPPELL-MAOR L,MEIR S,et al.Auxin response factor 2 intersects hormonal signals in the regulation of tomato fruit ripening[J].PLoS Genetics,2016,12(3):e1005903.
[32] JIA Y C,CHEN C X,GONG F F,et al.An Aux/IAA family member,RhIAA14,involved in ethylene-inhibited petal expansion in Rose (Rosa hybrida)[J].Genes,2022,13(6):1041.
[33] 刘湘楠,胡祥祥,乔梦燚,等.红花Aux/IAA基因家族鉴定及盐胁迫响应分析[J].江苏农业科学,2024,52(6):68-76.
[34] 冯曦,豆浩,孙佳佳,等.桑树Aux/IAA基因家族鉴定与IBA处理下表达模式分析[J].河南农业大学学报,2023,57(6):936-948.
[35] 方智振,姜翠翠,周丹蓉等.三月李及其红肉突变体Aux/IAA家族基因鉴定及分析[J].核农学报,2020,34(2):247-255.
[36] AUDRAN-DELALANDE C,BASSA C,MILA I,et al.Genome-wide identification,functional analysis and expression profiling of the Aux/IAA gene family in tomato[J].Plant and Cell Physiology,2012,53(4):659-672.
[37] MA C H,DANG K T,XIE Q K,et al.Over-expression of ZmIAA29,an Aux/IAA transcription factor,improved maize flowering time[J].Agronomy-Basel,2023,13(8):2028.
[38] WANG J,YAN D W,YUAN T T,et al.A gain-of-function mutation in IAA8 alters Arabidopsis floral organ development by change of jasmonic acid level[J].Plant Molecular Biology,2013,82(1/2):71-83.
[39] SI C,ZENG D Q,TEIXEIRA DA SILVA J A,et al.Genome-wide identification of Aux/IAA and ARF gene families reveal their potential roles in flower opening of Dendrobium officinale[J].BMC Genomics,2023,24(1):199.
[40] ABEL S,OELLER P W,THEOLOGIS A.Early auxin-induced genes encode short-lived nuclear proteins[J].Proceedings of the National Academy of Sciences of the United States of America,1994,91(1):326-330.
