西瓜TALE基因家族鉴定及表达分析
2024-10-25陈筱瑶冯梦娇黄欣张瑞青代雪王家发张显李军华霍治邦魏春华


摘 要:TALE基因家族在调控花器官发育、花分生组织形成、器官形态发生、果实发育、植物非生物胁迫响应及激素响应中发挥重要作用。利用生物信息学的方法鉴定了西瓜 TALE基因家族成员,并分析了其基因结构、系统发育树、保守基序、顺式作用元件、组织特异性表达,以及低温胁迫和干旱胁迫后基因的表达情况,为西瓜TALE基因家族的功能研究和利用提供了理论依据。结果表明,西瓜TALE基因家族共包含19个成员。系统发育分析将ClTALE家族分为BEL1-like和KNOX两个亚家族,KNOX亚家族又可进一步划分为两个分支Class I和Class II。顺式作用元件分析表明,ClTALE基因的启动子中富含光、植物激素和非生物胁迫响应的顺式元件,其中光响应元件最多。西瓜组织特异性表达分析表明,ClBLH09在根中高表达,ClBLH01在卷须、雌花和雄花中高表达,因此西瓜TALE基因家族可能参与了西瓜的生长发育过程。低温胁迫(0、3、6、12、24、36和48 h)和干旱胁迫(0、1、2、3、4、5、6、7和8 d)不同时间后基因的表达分析表明,ClBLH08和ClKN02在低温胁迫下呈现上调趋势,ClBLH04和ClBLH05在干旱胁迫下呈上调的趋势,因此西瓜TALE基因家族可能在低温胁迫和干旱胁迫中发挥作用。
关键词: 西瓜;TALE基因家族;生物信息学;表达分析
中图分类号:S651 文献标志码:A 文章编号:1673-2871(2024)10-018-12
收稿日期:2024-02-21;修回日期:2024-08-13
基金项目:西北农林科技大学种业创新专项(2452022116);现代农业产业技术体系建设专项(CARS-25);杨凌种业创新中心重点研发项目(Ylzy-sc-01)
作者简介:陈筱瑶,女,在读硕士研究生,研究方向为蔬菜育种与生物技术。E-mail:1173538169@qq.com
通信作者:霍治邦,男,研究员,研究方向为西瓜育种。E-mail:huozhibang@163.com
魏春华,男,副教授,研究方向为蔬菜育种与生物技术。E-mail:xjwend020405@nwafu.edu.cn
Identification and expression analysis of TALE gene family in watermelon
CHEN Xiaoyao1, FENG Mengjiao1, HUANG Xin1, ZHANG Ruiqing1, DAI Xue1, WANG Jiafa1, ZHANG Xian1, LI Junhua2, HUO Zhibang2, WEI Chunhua1
(1. College of Horticulture, Northwest A&F University, Yangling 712100, Shaanxi, China; 2. Kaifeng Academy of Agriculture and Forestry, Kaifeng 475004, Henan, China)
Abstract: The TALE gene family plays a pivotal role in regulating various aspects of plant growth and development, including floral organ development, floral meristem formation, organ morphogenesis, and fruit development, as well as mediating plant responses to abiotic stress and hormone signals. This study utilized bioinformatics methods to identify members of the TALE gene family in watermelon and conducted a comprehensive analysis of their gene structure, phylogenetic relationships, conserved motifs, cis-acting elements, tissue-specific expression patterns, and gene expression profiles under low temperature and drought stress conditions. This analysis provides a theoretical foundation for further functional studies and utilization of the TALE gene family in watermelon. The results revealed that the watermelon TALE gene family consists of a total of 19 members. Phylogenetic analysis classified the ClTALE family into two distinct subfamilies: BEL1-like and KNOX, with the KNOX subfamily further subdivided into two classes, Class I and Class II. Cis-acting element analysis demonstrated that the promoters of ClTALE genes are enriched in cis-elements that respond to light, plant hormones, and abiotic stresses, with light-responsive elements being the most prevalent. Tissue-specific expression analysis in watermelon indicated that ClBLH09 is highly expressed in roots, while ClBLH01 exhibits high expression levels in tendrils, female flowers, and male flowers, implying that the watermelon TALE gene family may be involved in the growth and development of watermelon. Gene expression analysis at various time points under low temperature stress(0, 3, 6, 12, 24, 36, and 48 h) and drought stress(0, 1, 2, 3, 4, 5, 6, 7, and 8 d) revealed that ClBLH08 and ClKN02 exhibited an upward trend in expression under low temperature stress, while ClBLH04 and ClBLH05 showed an upward trend under drought stress. These findings suggested that the watermelon TALE gene family may play a significant role in mediating responses to low temperature and drought stress.
Key words: Watermelon; TALE gene family; Bioinformatics; Expression analysis
同源异型盒基因是一类在植物生长发育过程中扮演重要角色的转录调控因子。早期研究将植物的同源异型盒基因分为11个类别,其中包括HD-ZIP、WOX、NDX、PHD、PLINC、LD、DDT、SAWADEE、PINTOX、KNOX和BEL1-like[1]。根据蛋白序列和进化关系的分析结果,BELL和KNOX被归类为TALE基因家族[2-3]。重要的是,KNOX和BEL1-like同源盒结构可以通过3个额外的氨基酸残基进行环状连接,形成了三氨基酸环延伸(TALE)超级家族同源盒结构。这种结构对调控植物多样化生物过程至关重要[4-6]。TALE基因家族在调节植物生长发育[7-10]、调控孢子体程序[11]、植物分生组织的形成[2]以及器官形态[12]、器官位置[13]、激素调节[14]、信号转导[15]和块茎形成的维持[16]等方面发挥着至关重要的作用。研究表明,BEL1-like蛋白和KNOX蛋白是两类参与植物发育的关键蛋白,BELL和KNOX蛋白质可以特异性地相互识别并结合形成BELL-KNOX异源二聚体[17],这对两种转录因子蛋白的核定位和结合靶基因的活性至关重要[18-19]。BEL1-like蛋白质含有1个称为Homeodomain(也称为Homeobox)的结构域,该结构域与Homeobox_KN结构域共享序列。此外,它还包含1个由SKY和BEL1区域组成的POX结构域(也称为MID)。BELL在胚珠发育、叶片发育和果实发育中起重要作用[20-21]。研究发现,BELL蛋白ATH1会对开花位点C(FLC)的表达产生影响,导致花朵生成受阻[22]。相反地,BLH3和BLH6则被识别为花朵发育的促进因子。当BLH6的表达水平升高时,花朵的开放时间会推迟,而当BLH3的表达水平升高时,花朵的开放时间则会提前[23]。此外,现有研究结果表明,PNY和ATH1之间的相互影响对开花产生一定的抑制作用,而PNF和ATH1之间的相互影响则呈现相反的效果[24]。KNOX基因家族包括KNOX1、KNOX2、ELK和同源域,除了1个不含同源域的基因KNATM[25-26]。其中,KNOX1和KNOX2是MEINOX结构域的2个子域,而MEINOX结构域则是介导KNOX或BEL1-like蛋白与其他蛋白相互作用的关键。研究表明,KNOX1在分生组织的发育和维持中具有重要作用;而KNOX2则参与调节植物细胞壁的次生生长,对根、茎、种皮和心材的发育具有至关重要的调节作用[27-30]。
TALE基因家族不仅在调节植物生长发育中起重要作用,而且在植物胁迫中也起作用。例如,GmBHL4蛋白与gmshbh1蛋白异源二聚体,调控大豆对干旱、高温和湿度胁迫的响应[31]。后来对大豆TALE基因家族的研究表明,TALE基因可能在应对非生物胁迫中发挥作用,并有助于大豆抗盐和脱水胁迫的遗传改良[32]。杨树35个TALE基因中,约有1/3的基因响应盐胁迫[33]。小麦TALE基因家族中的 TaKNOX11-A 增强了拟南芥的耐旱性和耐盐性。TaKNOX11-A 过表达的植物丙二醛含量降低,脯氨酸含量增加,使植物更好地适应不利环境[34]。
笔者利用西瓜种质M08(果皮有清晰条纹的品种)研究TALE基因在西瓜中的功能和调控机制,有助于调控西瓜的生长模式和花果发育。西瓜基因组数据的完成为西瓜基因功能的研究提供了重要的数据支持。笔者对西瓜TALE基因家族进行了全面鉴定,并对其结构、保守结构域、理化性质、系统发育关系、染色体定位、顺式作用元件和基因组织表达进行了详细分析。通过对ClTALE的系统鉴定,为未来基因功能研究奠定基础,并促进其在干旱和低温胁迫下西瓜遗传改良中的应用。
1 材料与方法
1.1 材料
试验材料西瓜种质M08由西北农林科技大学园艺学院瓜类种质创新与遗传改良实验室收集保存,性状稳定遗传。试验于2022年11月至2023年12月在西北农林科技大学园艺学院瓜类种质创新与遗传改良实验室进行。
取籽粒饱满的西瓜种质M08种子,55~60 ℃温汤浸种催芽后播种于50孔穴盘中,放置在人工气候箱(28 ℃,16 h光照/8 h黑暗,相对湿度70%)内。于3叶1心时定植在温室大棚中,并合理实施浇水及病虫害防治等栽培管理措施,开花期严格自交授粉。授粉后26 d分别取根、茎、叶、卷须、雌花和雄花这6个组织部位,采用随机区组法取样,每个样本设置3次重复。所有样品用锡箔纸包裹后放于液氮中速冻,随后存放于-80 ℃超低温冰箱备用。
西瓜种质M08植株3叶1心时在人工气候箱(4 ℃,16 h光照/8 h黑暗,相对湿度70%)进行低温胁迫(0、3、6、12、24、36和48 h),取胁迫处理后的真叶叶片组织,采用随机区组法取样;所有样品用锡箔纸包裹后放于液氮中速冻,随后存放于-80 ℃超低温冰箱备用。西瓜种质M08植株3叶1心时在培养架上(28 ℃,16 h光照/8 h黑暗)进行干旱胁迫处理(0、1、2、3、4、5、6、7和8 d),取胁迫处理后的真叶叶片组织,采用随机区组法取样。
1.2 西瓜TALE基因家族鉴定
根据拟南芥[26]TALE基因家族研究结果,从拟南芥数据库(https://www.arabidopsis.org/)中下载拟南芥的TALE蛋白序列;通过TBtools软件提取最具代表性的ClTALE蛋白序列,获得的西瓜TALE蛋白序列通过NCBI网站上的在线工具BLASTp(https://www.ncbi.nlm.nih.gov/)进一步验证。使用NCBI网站上的在线工具Conserved Domain(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)探索西瓜TALE蛋白的保守结构域。通过Pfam在线网站 (http://pfam.xfam.org/)和SMART在线网站(http://smart.embl-heidelberg.de/)确定候选西瓜TALE蛋白序列是否拥有植物TALE基因家族保守结构域,通过筛选得到西瓜中TALE基因家族的全部蛋白序列。通过利用Expasy网站上提供的在线工具ProtParam(http://web.expasy.org/protparam/),对西瓜TALE基因家族的相对分子质量、等电点和蛋白质长度等信息进行分析。
1.3 西瓜TALE基因在染色体上的分布
从葫芦科数据库(http://cucurbitgenomics.org/v2/)中获取ClTALEs在染色体上的位置信息,根据染色体位置进行重命名,使用TBtools软件进行数据可视化,得到了西瓜TALE基因家族在染色体上的分布情况。
1.4 西瓜TALE基因家族进化关系分析
使用MEGA 7软件中的Clustal W序列比对程序,对西瓜和拟南芥的TALE蛋白序列进行了多序列比对,并利用邻接法(neighbour-joining, NJ)构建了进化树(设置Bootstrap值为1000,model为P-distance,partial deletion值为50%,其余参数按照默认设置)。将得到的进化树文件通过Evolview在线网站(https://evolgenius.info//evolview-v2/)进行美化处理。
1.5 西瓜TALE基因家族基因结构与保守基序分析
利用MEME在线网站(https://meme-suite.org/meme/doc/meme.html)对西瓜TALE蛋白序列进行保守基序分析,将最大基序检索值设置为12,其他参数使用默认值,下载得到了结果文件meme.xml。利用ClTALEs的gff文件、系统进化树与MEME在线预测文件 meme.xml,在 TBtools 软件上进行可视化。利用葫芦科基因组数据库网站Batch query工具下载西瓜 TALE蛋白对应的基因序列和cDNA序列,利用Gene Structure Display Server 2.0 (http://gsds.gao-lab.org)分析其内含子及外显子并进行可视化。
1.6 西瓜TALE基因启动子区域的顺式元件分析
利用TBtools提取西瓜TALE基因上游2000 bp序列。然后将提取的序列提交给PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/),分析基因启动子区域的顺式元件,选取与胁迫及激素响应有关的顺式作用元件,由TBtools绘制西瓜TALE基因启动子区域的顺式元件图。
1.7 西瓜TALE基因表达分析
在本研究中,西瓜种质M08的根、茎、叶、卷须、雌花和雄花这6个组织分别于授粉后26 d取样,以这6个组织提取总RNA,随后使用FastKing一步法基因组 cDNA 第一链合成预混试剂盒进行反转录获得cDNA模板,以管家基因ClACT(cl007792)为内参。使用NCBI在线网站设计定量引物(表1),通过qRT-PCR试验获取西瓜TALE基因在6个组织中的表达量数据,利用Excel标准化处理以上数据,基因相对表达量的计算采用2-∆∆CT法,每个样品3次重复,对基因相对表达量数据进行Log2处理,通过TBtools软件绘制基因表达热图进行聚类分析。在西瓜种质M08植株3叶1心时期进行低温胁迫(0、3、6、12、24、36和48 h)和干旱胁迫(0、1、2、3、4、5、6、7和8 d处理),取胁迫处理后的叶片组织提取 RNA 进行反转录获得 cDNA 模板,通过qRT-PCR试验获取西瓜TALE基因在低温胁迫和干旱胁迫中的表达量数据,利用Excel标准化处理以上数据,基因相对表达量的计算采用2-∆∆CT法,每个样品3次重复,对基因相对表达量数据进行Log2处理,使用TBtools软件的Heatmap功能绘制基因表达的热图,并使用TBtools软件的Show Control Dialog进行图片美化。
2 结果与分析
2.1 西瓜TALE基因家族成员的鉴定
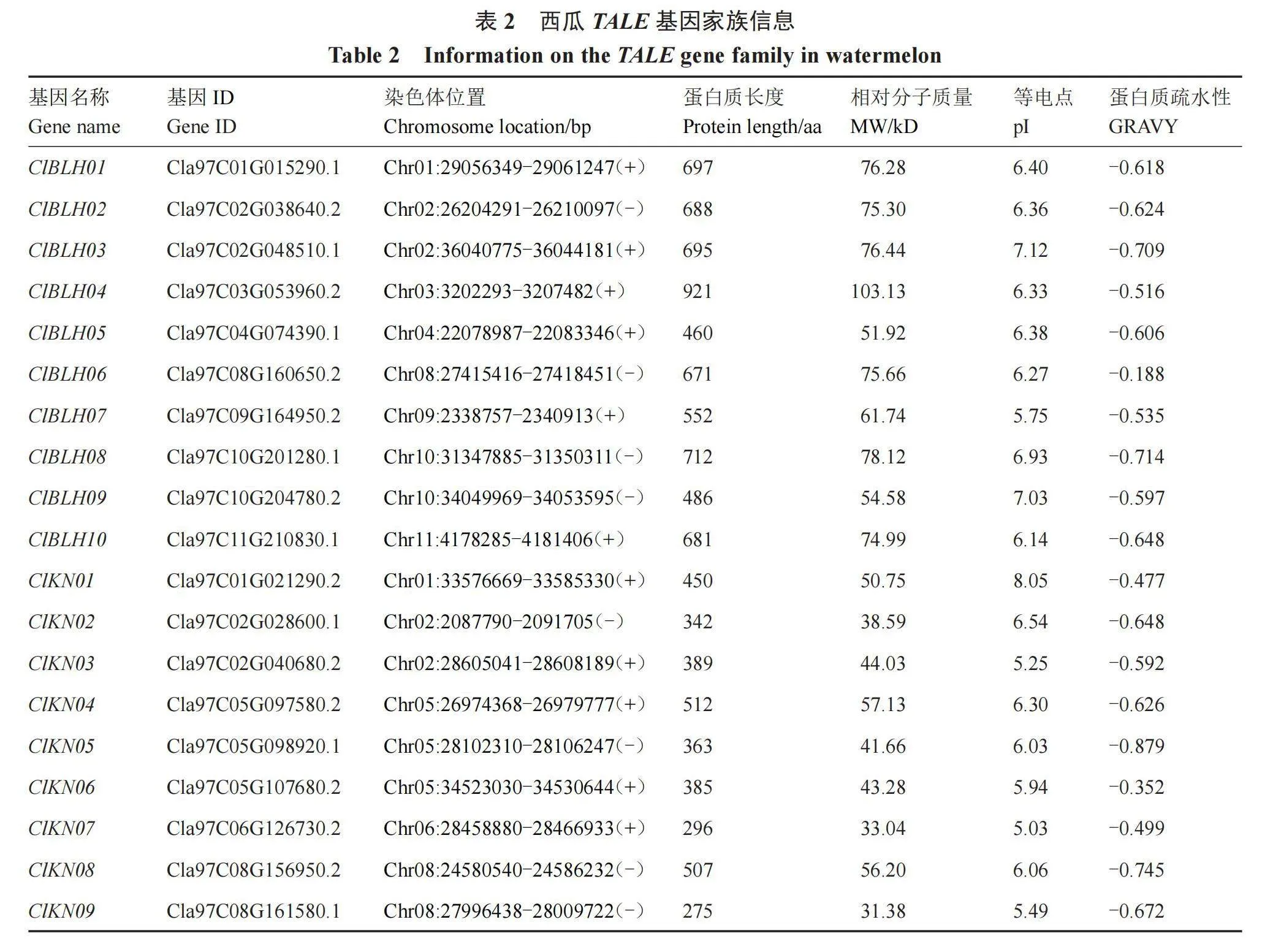
参照拟南芥TALE基因家族的分类,西瓜TALE基因家族分为BEL1-like和KNOX两个亚家族。采用生物信息学方法对西瓜TALE基因家族的成员进行了鉴定,共鉴定出19个成员,根据其在染色体上的排列位置系统命名ClBLH01~ClBLH10、ClKN01~ClKN09(表2)。结果显示,ClTALE蛋白的氨基酸数目在275~921 aa (ClBLH01~ClBLH10、ClKN01~ClKN09) 之间,相对分子质量31.38~103.13 kD(ClBLH01~ClBLH10、ClKN01~ClKN09)之间,除ClBLH03、ClBLH09和ClKN01的蛋白等电点大于7,其余ClTALE 蛋白等电点均在5.03~6.93之间,所有ClTALE蛋白疏水性都小于0。研究发现,ClTALE蛋白具有较大的氨基酸数目和相对分子质量差异。此外,这些蛋白主要是酸性蛋白,并且具有亲水性特点。
2.2 西瓜TALE基因的染色体定位
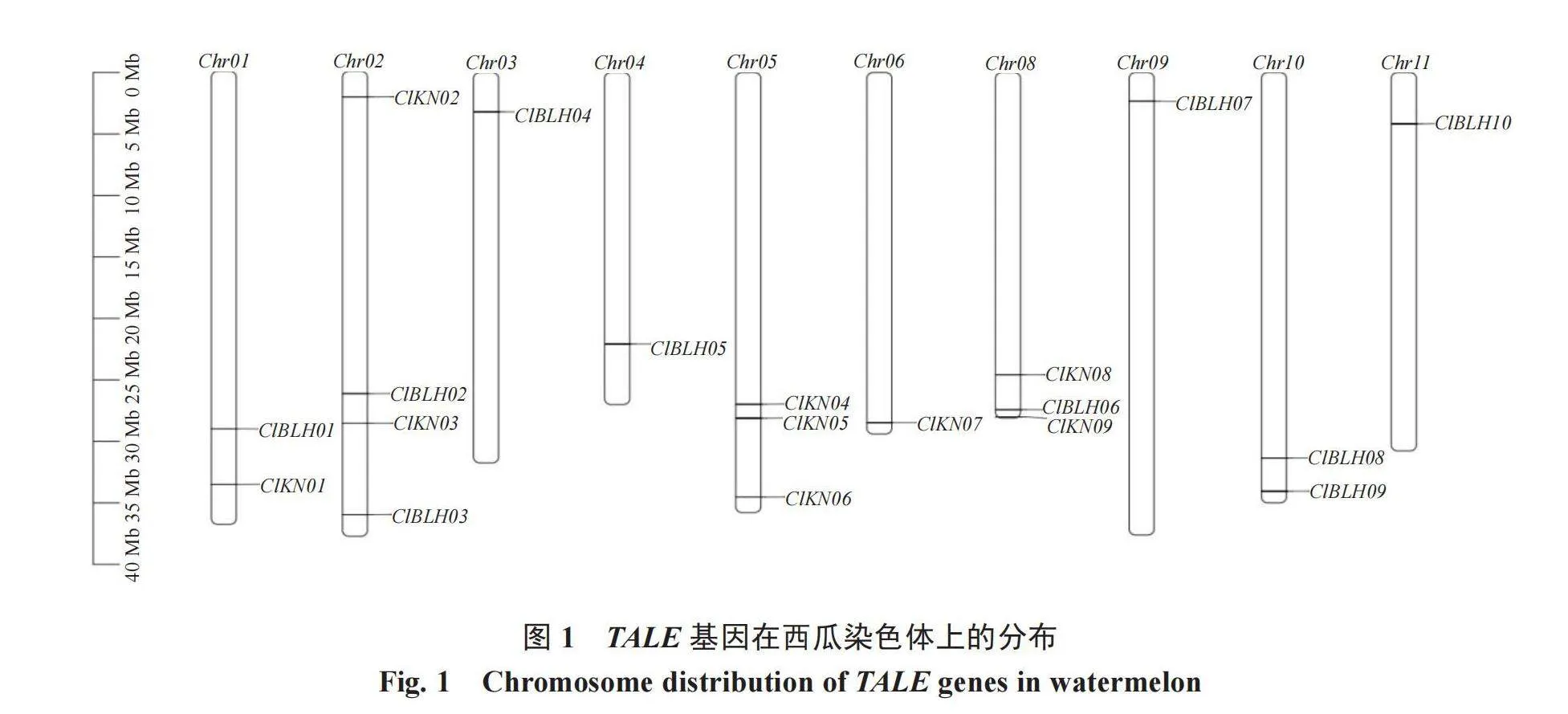
根据在葫芦科数据库中得到的ClTALEs基因组注释文件,用TBtools软件分析了19个西瓜TALE基因在染色体上的具体位置(图1)。结果显示,西瓜ClTALEs不均匀地分布在10条染色体上,其中第03、第04、第06、第09和第11号染色体上仅存在1个TALE基因,第01和第10号染色体上各有2个基因,第05和第08号染色体上各有3个基因,第02号染色体上分布了最多的TALE基因,共计4个(表2,图1)。
2.3 西瓜和拟南芥TALE基因进化关系分析
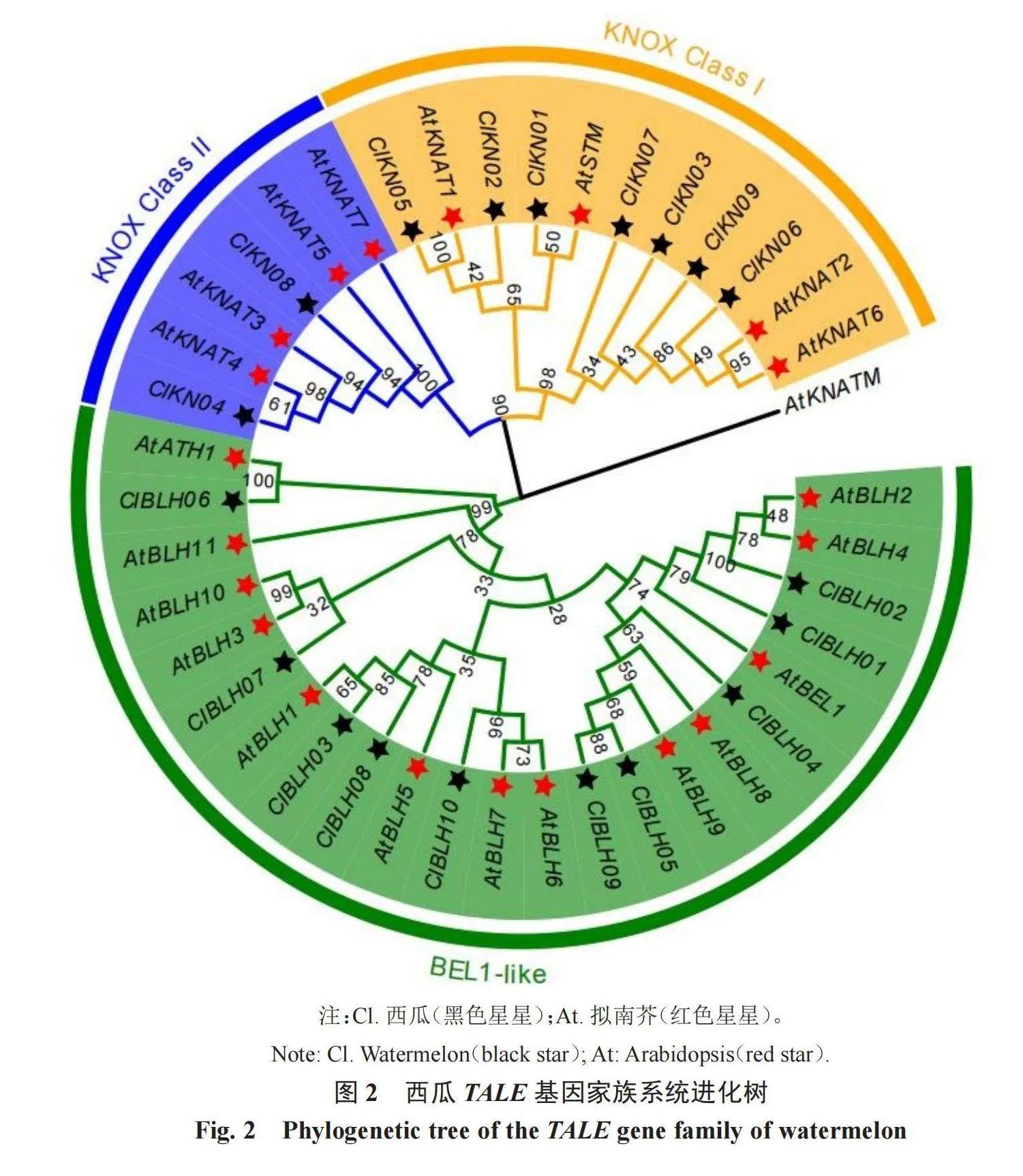
利用MEDGA7软件分析了22个拟南芥TALE基因和19个西瓜TALE基因的进化关系,对19个西瓜TALE基因蛋白序列和22个拟南芥TALE基因蛋白序列构建系统进化树(图2)。参考拟南芥TALE基因家族的研究结果,将西瓜TALE基因家族分为BEL1-like和KNOX 2个亚家族。根据亲缘关系的远近,将41个TALE基因分为3个组:第1组为BEL1-like亚家族,共包括23个基因,其中13个TALE基因来自拟南芥,而另外10个为西瓜TALE基因;第2、第3组为KNOX转录因子,又可进一步划分为2个分支Class I和Class II。第1个分支Class I共包括11个基因,其中4个TALE基因来自拟南芥,而另外7个为西瓜TALE基因;第2个分支Class II共包括6个基因,其中4个TALE基因来自拟南芥,而另外2个为西瓜TALE基因。
2.4 西瓜TALE基因家族的结构特征分析
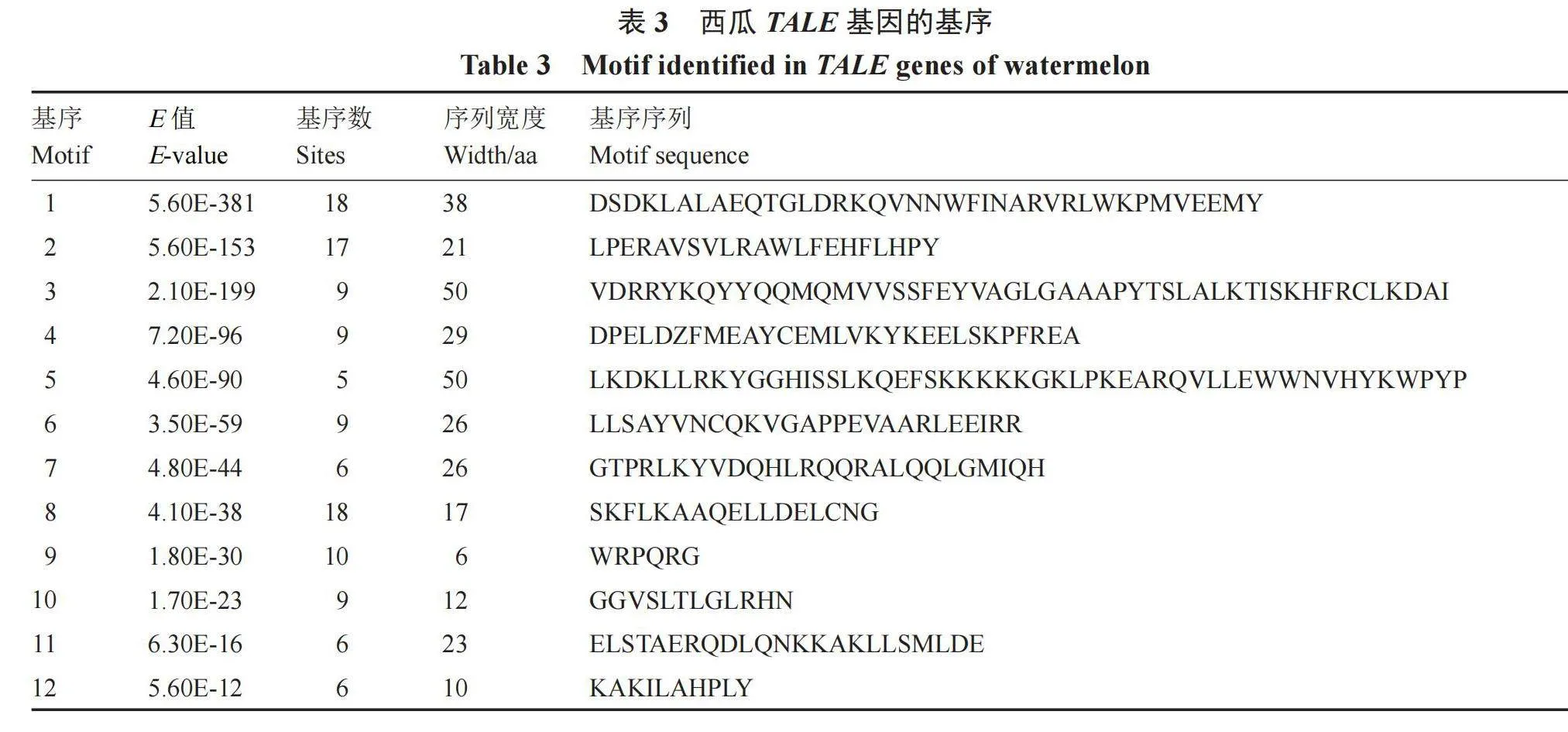
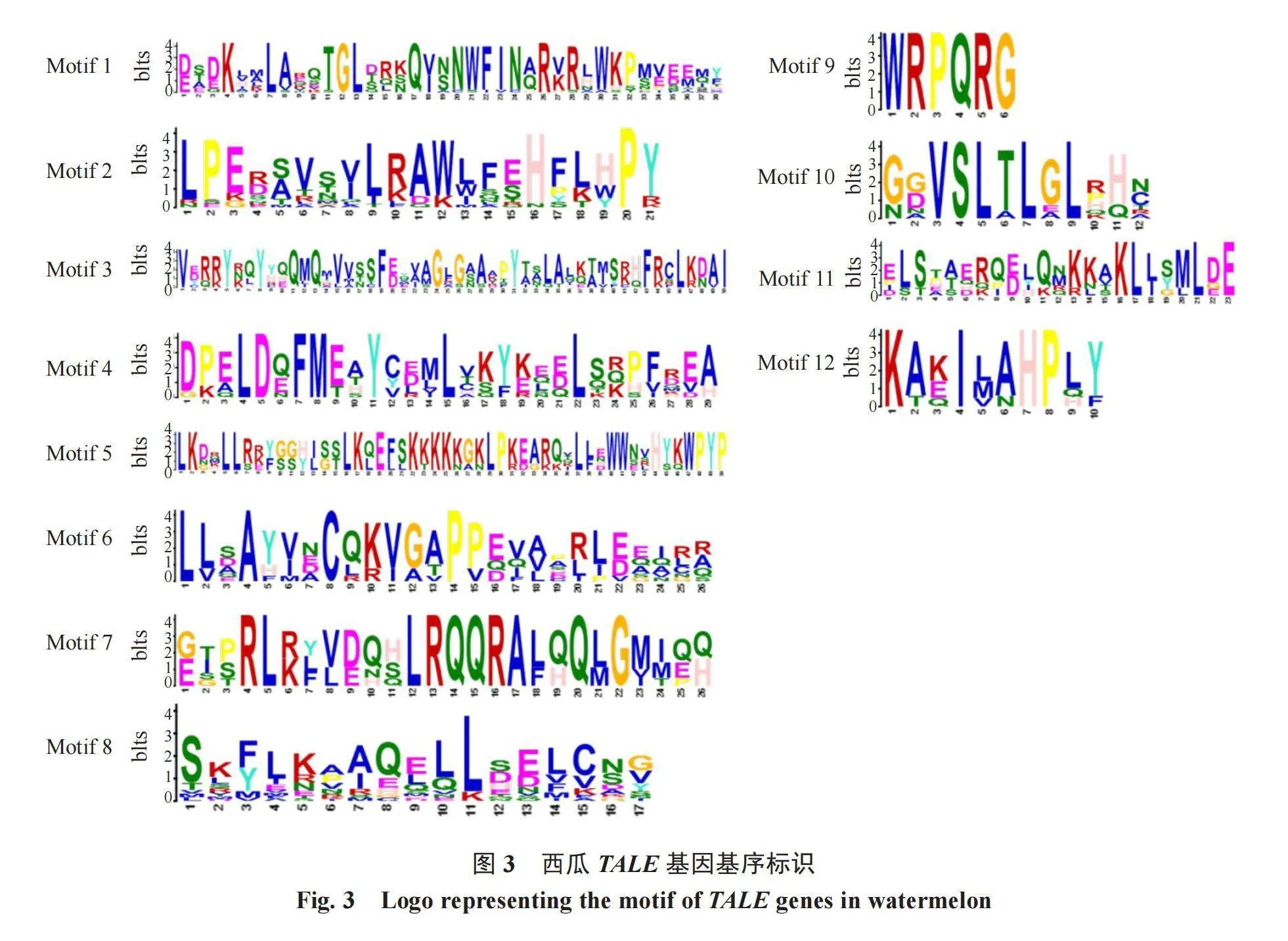
为了更好地理解西瓜TALE基因家族成员之间的进化关系,首先通过构建带有ClBEL1-like和ClKNOX家族基因DNA序列的系统发育树(图2)来进行分析。利用MEME在线网站获得西瓜TALE基因的12个保守基序(motif 1~motif 12)类型(表3)及具体基序标识(图3)。同时,为了鉴定西瓜中TALE基因家族的结构特征,将cDNA序列与其相应的基因组DNA序列进行比较,并利用基因结构显示服务器(GSDS)对ClBEL1-like亚家族基因和ClKNOX亚家族基因的外显子/内含子结构进行了分析(图4-a、图4-c)。还利用Multiple Em for Motif Elicitation(MEME)程序,分别分析了ClBEL1-Like和ClKNOX成员的蛋白质序列(图4-b)。
从基因结构分析结果(图4-c)发现,所有的ClTALE成员的外显子数量在4~10,平均数为5.37。其中,ClBEL1-like亚家族基因的外显子数量主要集中在4~6,只有ClBLH06含有7个外显子。ClKNOX亚家族基因的外显子数量大多在4~6,但ClKN06含有8个外显子,而ClKN01则含有10个外显子。这些结果表明,在西瓜TALE基因家族中,基因结构普遍展现出外显子和内含子的高度保守分布。
BEL1-Like蛋白和KNOX蛋白是两种关键的植物发育相关蛋白。BEL1-like蛋白包含1个Homeodomain结构域(也称为Homeobox),这个结构域与Homeobox_KN结构域共享相似序列。此外,BEL1-like蛋白还包括1个POX结构域(也称为MID),其中包含SKY和BEL1区域。KNOX蛋白含有1个MEINOX结构域,由KNOX1和KNOX2子域组成,它们之间被柔性连接子、ELK结构域和TALE同源结构域分隔。MEINOX结构域能够促进KNOX和BELL蛋白之间的相互作用。基因结构分析证明,所有的ClBEL1-like成员都保守地包含TALE同源结构域和POX结构域(图4-d),而所有的ClKNOX成员都保守地包含KNOX1和KNOX2(MEINOX)结构域,但是ClKN03缺失了ELK结构域(图4-e)。
2.5 西瓜TALE基因启动子的顺式作用元件分析
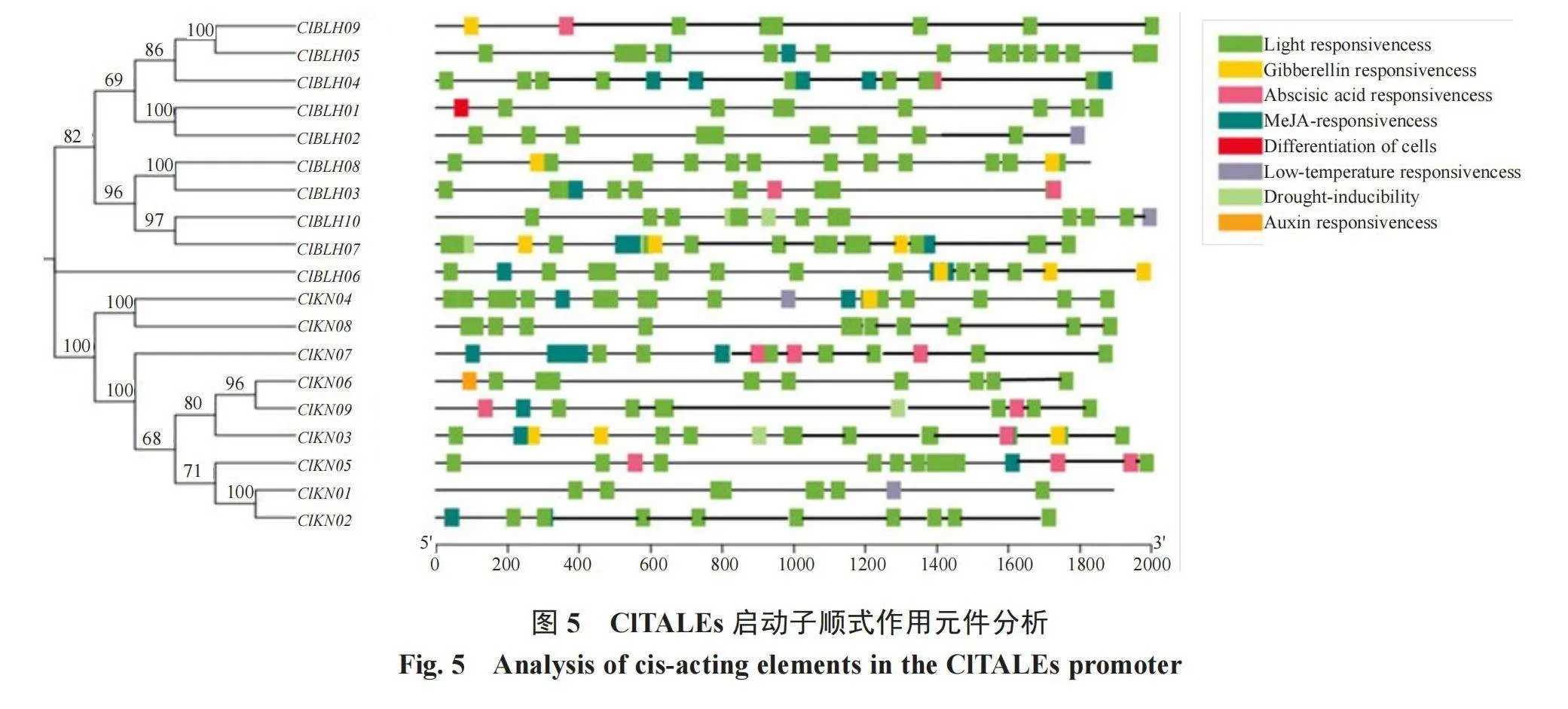
前人的研究表明,TALE基因家族参与调控花器官发育、花分生组织形成、器官形态发生、果实发育、植物非生物胁迫响应及激素响应。使用在线软件PlantCARE对西瓜TALE基因家族上游2000 bp启动子区的顺式作用元件进行预测,并选择与植物生长发育、胁迫响应以及激素响应相关的顺式作用元件进行分析。这些元件主要包括光响应、低温响应、干旱响应、生长素响应、脱落酸响应、茉莉酸甲酯响应和赤霉素响应等(图5)。笔者旨在探究西瓜TALE基因家族在生物学过程中以及对植物激素和非生物胁迫反应中的重要作用。分析表明,分布最广的是光响应相关元件(Light responsiveness)(含元件基因数/总基因数为 19/19),其次是茉莉酸甲酯响应元件(MeJA-responsiveness)(11/19)、赤霉素响应元件(gibberellin responsiveness)(7/19)、脱落酸响应元件(abscisic acid responsiveness)(6/19)、低温响应元件(low-temperature responsiveness))(4/19)、干旱响应元件(drought-inducibility)(4/19);分布最少的为生长素响应元件(1/19)和调控植物细胞分化元件(1/19),前者仅在ClKN06中分布,后者在ClBLH01中分布。由此可以推断,ClTALEs在应对光照、激素信号以及逆境胁迫等方面可能扮演着重要的角色,并且对西瓜的生长发育进行调控。
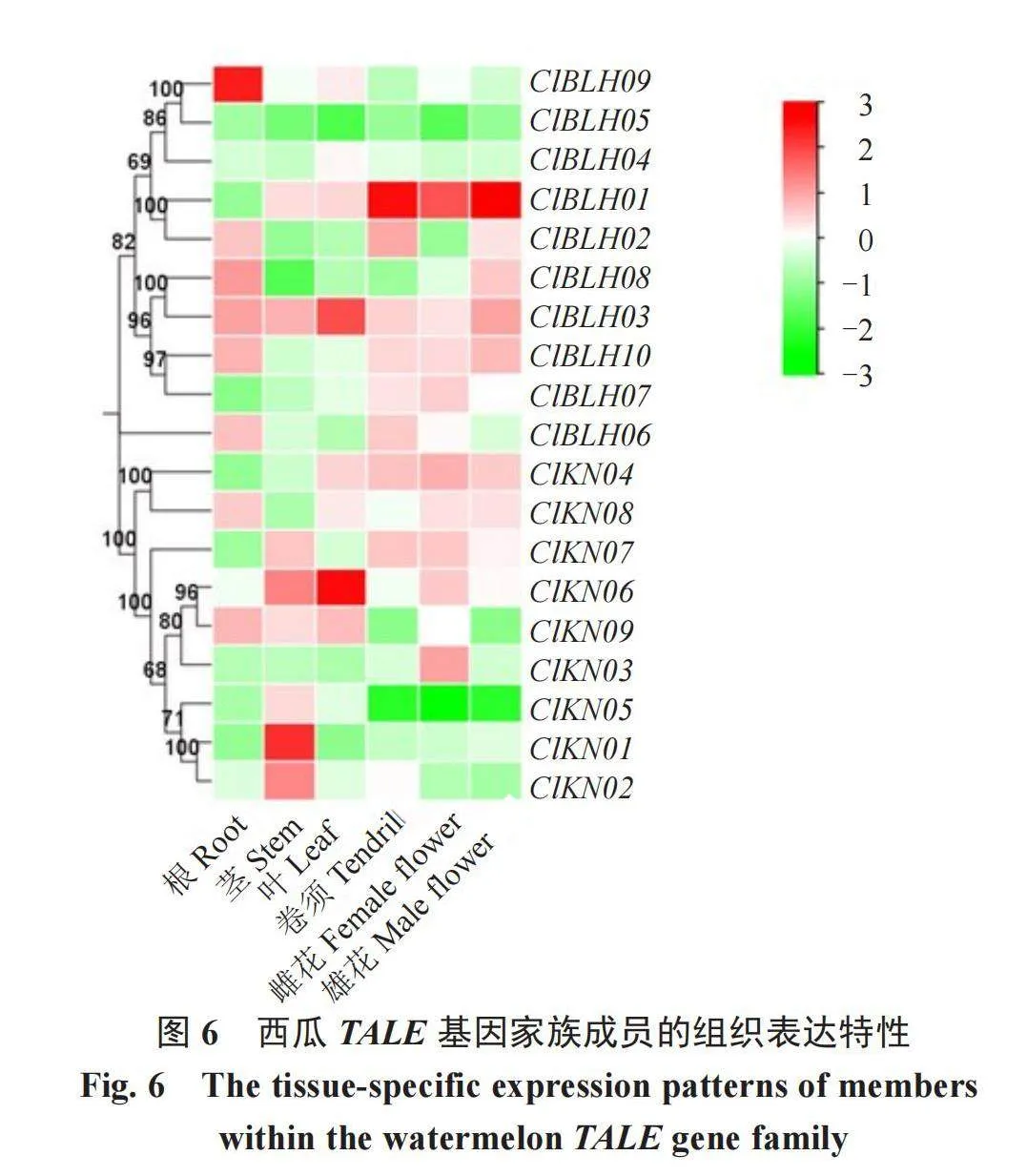
2.6 西瓜TALE基因的组织表达特性
在对19个西瓜TALE基因进行的表达特点分析中(图6)发现,它们在西瓜的不同器官中表达存在差异。具体结果显示,大部分BEL1-like亚家族基因在根、卷须、雌花和雄花中表达,而大部分KNOX亚家族基因则在茎、叶、雌花和雄花中表达。其中,ClBLH09在根中高表达,ClBLH01在卷须、雌花和雄花中高表达,ClBLH03在叶中高表达,ClKN06在茎和叶中高表达,ClKN01和ClKN02在茎中高表达,由此推测西瓜TALE基因家族可能参与了西瓜的生长发育过程。
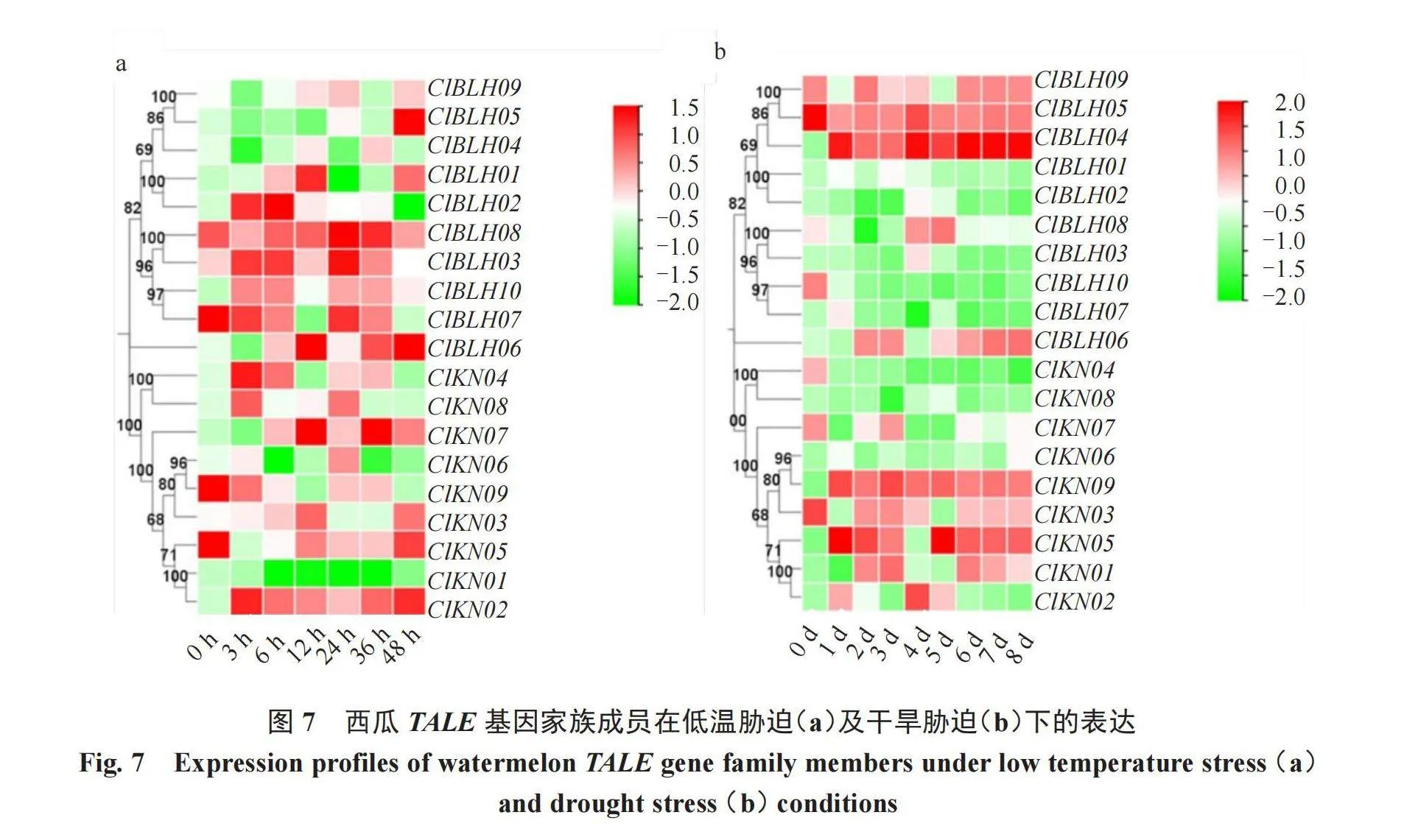
2.7 西瓜TALE基因对低温胁迫、干旱胁迫的响应表达
利用实时荧光定量试验分析低温胁迫和干旱胁迫下西瓜TALE家族基因的表达特性,结果(图7-a)显示,在低温0 h时,除了ClBLH03、ClBLH07、ClBLH08、ClKN05和ClKN09这5个基因呈现上调趋势外,其余基因呈现下调趋势;在低温3 h后,除了ClBLH02、ClBLH03、ClBLH07、ClBLH08、ClBLH10、ClKN02、ClKN04、ClKN08和ClKN09这9个基因呈现上调趋势外,其余基因呈现下调趋势;在低温6 h后,除了ClBLH01、ClBLH02、ClBLH03、ClBLH06、ClBLH07、ClBLH08、ClBLH10、ClKN02、ClKN03、ClKN04、ClKN07和ClKN09这12个基因呈现上调趋势外,其余基因呈现下调趋势;在低温12 h后,除了ClBLH05、ClBLH07、ClKN01、ClKN04、ClKN06和ClKN09这6个基因呈现下调趋势外,其余基因均呈现上调趋势;在低温24 h后,除了ClBLH01、ClBLH04、ClKN01和ClKN03这4个基因呈现下调趋势外,其余基因均呈现上调趋势;在低温36 h后,除了ClBLH01、ClBLH05、ClBLH09、ClKN01、ClKN03、ClKN06和ClKN08这7个基因呈现下调趋势外,其余基因均呈现上调趋势;在低温48 h后,除了ClBLH02、ClBLH03、ClBLH04、ClBLH07、ClKN01、ClKN04、ClKN06、ClKN08和ClKN09这9个基因呈现下调趋势外,其余基因呈现上调趋势。ClBLH08和ClKN02这2个基因在低温胁迫下基本呈现上调趋势,ClKN01这个基因在低温胁迫下基本呈现下调趋势,因此西瓜TALE基因家族可能在低温胁迫中发挥作用。
结果(图7-b)显示,在干旱0 d时,除了ClBLH05、ClBLH08、ClBLH09、ClBLH10、ClKN03、ClKN04和 ClKN07这7个基因呈现上调趋势外,其余基因呈现下调趋势;在干旱1 d后,除了ClBLH04、ClBLH05、ClBLH07、ClKN02、ClKN05和ClKN09这6个基因呈现上调趋势外,其余基因均呈现下调趋势;在干旱2 d后,除了ClBLH04、ClBLH05、ClBLH06、ClBLH09、ClKN01、ClKN03、ClKN05和ClKN09这8个基因呈现上调趋势外,其余基因呈现下调趋势;在干旱3 d后,除了ClBLH04、ClBLH05、ClBLH06、ClBLH09、ClKN01、ClKN03、ClKN05、ClKN07和ClKN09这9个基因呈现上调趋势外,其余基因呈现下调趋势;在干旱4 d后,除了ClBLH03、ClBLH04、ClBLH05、ClBLH08、ClBLH09、ClKN02、ClKN03和ClKN09这8个基因呈现上调趋势外,其余基因呈现下调趋势;在干旱5 d后,除了ClBLH04、ClBLH05、ClBLH06、ClBLH08、ClKN02、ClKN05和ClKN09这7个基因呈现上调趋势外,其余基因呈现下调趋势;在干旱6 d、7 d和8 d后,除了ClBLH04、ClBLH05、ClBLH06、ClBLH09、ClKN01、ClKN03、ClKN05和ClKN09这8个基因呈现上调趋势外,其余基因呈现下调趋势。因此,西瓜TALE基因家族可能在干旱胁迫中发挥作用。
3 讨论与结论
TALE基因家族在植物发育中起着至关重要的作用,在植物界分布广泛,但在不同的植物物种中,这些基因的数量差异很大。TALE基因家族已经在越来越多的植物中被详细鉴定,其中包括拟南芥[26]、石榴[35]、番茄[36]、大豆[37]、萝卜[38]、玉米[39]等,但在葫芦科作物中尚未见到相关的研究报道。通过生物信息学方法,笔者对西瓜基因组中的TALE基因家族进行了鉴定,共发现了19个西瓜TALE基因家族成员。这些基因在10条染色体上呈不均匀分布,与拟南芥中鉴定到的22个基因数量大致相符[23]。根据西瓜TALE基因家族结构、保守基序分布及与拟南芥进化关系分析,可将其分为BEL1-like和KNOX 两个亚家族。其中BEL1-like蛋白包含1个Homeodomain结构域(也称为Homeobox),这个结构域与Homeobox_KN结构域共享相似序列。此外,BEL1-Like蛋白还包括1个POX结构域(也称为MID),其中包含SKY和BEL1区域。KNOX蛋白含有一个MEINOX结构域,由KNOX1和KNOX2子域组成,它们之间被柔性连接子、ELK结构域和TALE同源+REZAMndXzUdeA+IHEyZ80XiItVVXWQAer+IrNBQONw=结构域分隔开来。MEINOX结构域能够促进KNOX和BELL蛋白之间的相互作用。基因结构分析证明,所有的ClBEL1-like成员都保守地包含TALE同源结构域和POX结构域,而所有的ClKNOX成员都保守地包含KNOX1和KNOX2(MEINOX)结构域,但是ClKN03缺失了ELK结构域。西瓜TALE基因家族中同一亚家族基因结构相似,保守基序和内含子外显子的分布相似,这一结果与拟南芥TALE家族特征相似[23],这一结果表明,同一亚家族的基因可能具有相似的功能。
顺式作用元件通过参与基因表达的调控,对植物的生长发育以及对生物和非生物胁迫的响应产生影响。笔者使用在线软件PlantCARE,针对西瓜TALE基因家族上游2000 bp启动子区域,进行了顺式作用元件的预测。选择了与植物生长发育、胁迫响应以及激素响应相关的顺式作用元件进行了分析。这些元件包括光响应、低温响应、干旱响应、生长素响应、脱落酸响应、茉莉酸甲酯响应和赤霉素响应等。分析结果显示,光响应相关元件的分布最为广泛,其次为茉莉酸甲酯响应元件、赤霉素响应元件、脱落酸响应元件、低温响应元件和干旱响应元件;而生长素响应元件和调控植物细胞分化元件的分布则较为稀少。
通过综合分析顺式作用元件、组织表达和非生物胁迫表达等结果,推测西瓜TALE基因家族在植物的生长发育、花器官发育以及抵抗非生物胁迫方面扮演着重要角色。在本研究中发现,西瓜TALE基因家族在光反应、激素反应和非生物胁迫中扮演着至关重要的角色,这与在番茄[36]和石榴[37]中的研究结论一致。笔者利用西瓜种质M08的不同组织提取RNA进行反转录获得cDNA模板,通过qRT-PCR试验获取西瓜TALE基因的表达量数据,结果显示,大部分BEL1-like亚家族基因在根、卷须、雌花和雄花中表达,而大部分KNOX亚家族基因则在茎、叶、雌花和雄花中表达。其中,ClBLH09在根中高表达,ClBLH01在卷须、雌花和雄花中高表达,ClBLH03在叶中高表达,ClKN06在茎和叶中高表达,ClKN01在茎中高表达,由此推测西瓜TALE基因家族可能参与了西瓜的生长发育过程。
TALE基因家族广泛存在于植物中,在植物生长发育和非生物胁迫响应中发挥重要作用。番茄SlBELL的启动子区域包含许多与植物生长发育和应激反应相关的顺式元件。SlBELL基因在组织中具有不同的特异性表达模式,并且对冷、热和干旱胁迫有响应,暗示它们在调控植物生长的多个方面以及响应非生物胁迫方面具有潜在的功能[40]。研究表明CsKNOX在茶叶中的转录水平与黄烷-3-醇和原花青素的积累密切相关。通过转录组学数据和启动子元件分析发现,大多数CsKNOX基因对干旱、盐、低温胁迫以及外源MeJA和GA3均有响应[41]。在棉花TALE基因家族中GhKNOX4-A和GhKNOX22-D都调节植物耐受性;沉默这2个基因会诱导氧化应激,表现为SOD活性降低和叶片细胞死亡,并增强气孔开放和水分流失。因此,GhKNOX4-A和GhKNOX22-D可能通过调节气孔开放和氧化应激来参与干旱响应[42]。在苜蓿TALE基因家族中,KNAT3/4/5-like转录因子可能通过抑制脯氨酸脱氢酶基因(MtPDH)表达来加速脯氨酸积累以应对胁迫和保护细胞膜在免受干旱诱导的渗透胁迫中发挥作用[41]。在拟南芥中TaKNOX11-A增强了拟南芥的耐旱性和耐盐性。TaKNOX11-A过表达的植物丙二醛含量降低,脯氨酸含量增加,使植物更有效地适应不利环境[43]。通过对西瓜TALE基因家族在低温胁迫和干旱胁迫下的表达量进行分析,发现这些基因可能在这两种胁迫情况下发挥作用。这一结论与之前的相关研究所得到的结果相一致[31-33]。
在本试验中共鉴定得到了19个西瓜TALE基因,可分为2个亚家族,根据各基因在染色体上的位置依次命名为ClBLH01~ClBLH10、ClKN01~ClKN09。通过综合分析顺式作用元件、组织表达和非生物胁迫表达等结果,推测西瓜TALE基因家族在植物的生长发育、花器官发育以及抵抗非生物胁迫方面扮演着重要的角色。本研究结果可以为西瓜TALE基因家族的功能研究打下基础,并促进其在低温和干旱胁迫下西瓜遗传改良中的应用。然而,对各个基因的具体功能仍需要进一步研究。笔者对西瓜TALE基因家族成员进行了全基因组的鉴定和分析,为将来对该基因家族功能的验证提供了理论基础。
参考文献
[1] BÜRGLIN T R,AFFOLTER M.Homeodomain proteins:An update[J].Chromosoma,2016,125(3):497-521.
[2] ARNAUD N,PAUTOT V.Ring the BELL and tie the KNOX:Roles for TALEs in gynoecium development[J].Frontiers in Plant Science,2014,5:93.
[3] MA Q,WANG N H,HAO P B,et al.Genome-wide identification and characterization of TALE superfamily genes in cotton reveals their functions in regulating secondary cell wall biosynthesis[J].BMC Plant Biology,2019,19(1):432.
[4] CHEN H,ROSIN F M,PRAT S,et al.Interacting transcription factors from the three-amino acid loop extension superclass regulate tuber formation[J].Plant Physiology,2003,132(3):1391-1404.
[5] HAY A,TSIANTIS M.KNOX genes:Versatile regulators of plant development and diversity[J].Development,2010,137(19):3153-3165.
[6] BERTOLINO E,REIMUND B,WILDTPERINIC D,et al.A novel homeobox protein which recognizes a TGT core and functionally interferes with a retinoid-responsive motif[J].Journal of Biological Chemistry,1995,270(52):31178-31188.
[7] MAHAJAN A,BHOGALE S,KANG I H,et al.The mRNA of a Knotted1-like transcription factor of potato is phloem mobile[J].Plant Molecular Biology,2012,79(6):595-608.
[8] SAKAKIBARA K,ANDO S,YIP H K,et al.KNOX2 genes regulate the haploid-to-diploid morphological transition in land plants[J].Science,2013,339(6123):1067-1070.
[9] LIN T,SHARMA P,GONZALEZ D H,et al.The impact of the long-distance transport of a BEL1-Like Messenger RNA on development[J].Plant Physiology,2013,161(2):760-772.
[10] FURUMIZU C,ALVAREZ J P,SAKAKIBARA K,et al.Antagonistic roles for KNOX1 and KNOX2 genes in patterning the land plant body plan following an ancient gene duplication[J].PLoS Genetics,2015,11(2):e1004980.
[11] RUIZ-ESTÉVEZ M,BAKKALI M,MARTIN-BLAZQUEZ R,et al.Identification and characterization of TALE homeobox genes in the endangered fern Vandenboschia speciosa[J].Genes,2017,8(10):275.
[12] BELLES-BOIX E,HAMANT O,WITIAK S M,et al.KNAT6:An Arabidopsis homeobox gene involved in meristem activity and organ separation[J].Plant Cell,2006,18(8):1900-1907.
[13] AIDA M,ISHIDA T,TASAKA M.Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis:Interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes[J].Development,1999,126(8):1563-1570.
[14] SHANI E,YANAI O,ORI N.The role of hormones in shoot apical meristem function[J].Current Opinion in Plant Biology,2006,9(5):484-489.
[15] CNOPS G,NEYT P,RAES J,et al.The TORNADO1 and TORNADO2 genes function in several patterning processes during early leaf development in Arabidopsis thaliana[J].Plant Cell,2006,18(4):852-866.
[16] KONDHARE K R,VETAL P V,KALSI H S,et al.BEL1-like protein (StBEL5) regulates CYCLING DOF FACTOR1 (StCDF1) through tandem TGAC core motifs in potato[J].Journal of Plant Physiology,2019,241:153014.
[17] BHATT A M,ETCHELLS J P,CANALES C,et al.Vaamana-a BEL1-like homeodomain protein,interacts with KNOX proteins BP and STM and regulates inflorescence stem growth in Arabidopsis[J].Gene,2004,328:103-111.
[18] KIM D,CHO Y H,RYU H,et al.BLH1 and KNAT3 modulate ABA responses during germination and early seedling development in Arabidopsis[J].Plant Journal,2013,75(5):755-766.
[19] SMITH H M S,BOSCHKE I,HAKE S.Selective interaction of plant homeodomain proteins mediates high DNA-binding affinity[J].Proceedings of the National Academy of Sciences of the United States of America,2002,99(14):9579-9584.
[20] BYRNE M E,GROOVER A T,FONTANA J R,et al.Phyllotactic pattern and stem cell fate are determined by the Arabidopsis homeobox gene BELLRINGER[J].Development,2003,130(17):3941-3950.
[21] MENG L H,FAN Z Q,ZHANG Q,et al.BEL1-LIKE HOMEODOMAIN 11 regulates chloroplast development and chlorophyll synthesis in tomato fruit[J].Plant Journal,2018,94(6):1126-1140.
[22] PROVENIERS M,RUTJENS B,BRAND M,et al.The Arabidopsis TALE homeobox gene ATH1 controls floral competency through positive regulation of FLC[J].Plant Journal,2007,52(5):899-913.
[23] COLE M,NOLTE C,WERR W.2006.Nuclear import of the transcription factor SHOOT MERISTEMLESS depends on heterodimerization with BLH proteins expressed in discrete sub-domains of the shoot apical meristem of Arabidopsis thaliana[J].Nucleic Acids Research,2006,34(4):1281-1292.
[24] RUTJENS B,BAO D P,VAN ECK-STOUTEN E,et al.Shoot apical meristem function in Arabidopsis requires the combined activities of three BEL1-like homeodomain proteins[J].Plant Journal,2009,58(4):641-654.
[25] MAGNANI E,HAKE S.KNOX lost the OX:: The Arabidopsis KNATM gene defines a novel class of KNOX transcriptional regulators missing the homeodomain[J].Plant Cell,2008,20(4):875-887.
[26] HAMANT O,PAUTOT V.Plant development: A TALE story[J].Comptes Rendus Biologies,2010,333(4):371-381.
[27] LI E Y,BHARGAVA A,QIANG W Y,et al.The Class II KNOX gene KNAT7 negatively regulates secondary wall formation in Arabidopsis and is functionally conserved in Populus[J].New Phytologist,2012,194(1):102-115.
[28] ZHONG R Q,LEE C H,ZHOU J L,et al.A battery of transcription factors involved in the regulation of secondary cell wall biosynthesis in Arabidopsis[J].Plant Cell,2008,20(10):2763-2782.
[29] BHARGAVA A,MANSFIELD S D,HALL H C,et al.MYB75 functions in regulation of secondary cell wall formation in the Arabidopsis inflorescence stem[J].Plant Physiology,2010,154(3):1428-1438.
[30] LI E Y,WANG S C,LIU Y Y,et al.Ovate family protein4 (OFP4) interaction with KNAT7 regulates secondary cell wall formation in Arabidopsis thaliana[J].Plant Journal,2011,67(2):328-341.
[31] TAO Y,CHEN M,SHU Y J,et al.Identification and functional characterization of a novel BEL1-LIKE homeobox transcription factor GmBLH4 in soybean[J].Plant Cell Tissue and Organ Culture,2018,134(2):331-344.
[32] WANG L,YANG X Y,GAO Y Q,et al.Genome‑wide identification and characterization of TALE superfamily genes in soybean (Glycine max L.)[J].International Journal of Molecular Science,2021,22(8):4117.
[33] ZHAO K,ZHANG X M,CHENG Z H,et al.Comprehensive analysis of the three‑amino‑acid‑loop‑extension gene family and its tissue‑differential expression in response to salt stress in poplar[J].Plant Physiology and Biochemiatry,2019,136:1-12.
[34] HAN Y X,ZHANG L L,YAN L Y,et al.Genome-wide analysis of TALE superfamily in Triticum aestivum reveals TaKNOX11-A is involved in abiotic stress response[J].BMC Genomics,2022,23(1):89.
[35] WANG Y Y,ZHAO Y J,YAN M,et al.Genome-wide identification and expression analysis of TALE gene family in pomegranate (Punica granatum L.)[J].Agronomy-Basel,2020,10(6):829.
[36] WANG J,ZHAO P,CHENG B H,et al.Identification of TALE transcription factor family and expression patterns related to fruit chloroplast development in tomato (Solanum lycopersicum L.)[J].International Journal of Molecular Sciences,2022,23(9):4507.
[37] WANG L,YANG X Y,GAO Y Q,et al.Genome-wide identification and characterization of TALE superfamily genes in soybean (Glycine max L.)[J].International Journal of Molecular Sciences,2021,22(8):4117.
[38] 赵伟,李锡香,王海平,等.萝卜TALE转录因子家族的鉴定与分析[J].生物工程学报,2022,38(1):343-358.
[39] 赵长江,宋巨奇,都梦翔,等.玉米KNOX基因家族鉴定及组织和逆境表达分析[J].西北植物学报,2021,41(7):1109-1119.
[40] HE Y,YANG T W,YAN S W,et al.Identification and characterization of the BEL1-like genes reveal their potential roles in plant growth and abiotic stress response in tomato[J].International Journal of Molecular Sciences,2022,200:193-205.
[41] DAI H W,ZHENG S T,ZHANG C,et al.Identification and expression analysis of the KNOX genes during organogenesis and stress responseness in Camellia sinensis (L.) O.Kuntze[J].Molecular Genetics and Genomics,2023,298(6):1559-1578.
[42] SUN R R,QIN T F,WALL S B,et al.Genome-wide identification of KNOX transcription factors in cotton and the role of GhKNOX4-A and GhKNOX22-D in response to salt and drought stress[J].International Journal of Biological Macromolecules,2023,226:1248-1260.
[43] IANNELLI M A,NICOLODI C,CORAGGIO I,et al.A novel role of Medicago truncatula KNAT3/4/5-like class 2 KNOX transcription factors in drought stress tolerance[J].International Journal of Molecular Sciences,2023,24(16):12668.
