花椒BAHD酰基转移酶基因家族鉴定及表达差异分析
2024-10-18熊文玮蔡炼苏靖杨银迢冯世静



摘 要:
麻味物质是评价花椒(Zanthoxylum bungeanum Maxim.)品质的重要特征性成分之一,为了挖掘花椒麻味物质合成途径酰化反应的候选基因,本研究基于花椒全基因组数据,通过生物信息学方法,对花椒BAHD酰基转移酶基因家族进行了研究。研究结果共鉴定出50个花椒BAHD酰基转移酶基因家族成员,这些基因可分为两大亚家族,均编码不稳定亲水性蛋白;亚细胞定位分析表明大部分花椒BAHD成员主要定位于细胞质,其它成员定位于细胞核、叶绿体等细胞器中。基因表达分析显示,BAHD酰基转移酶基因在花椒果皮发育各阶段均有特异性的表达,且在果梗、刺、茎和花中表达高于种子和叶。不同花椒品种间,即使相同基因也表现出表达差异,甚至在某些品种中未检测到表达。此研究为花椒麻味物质合成途径提供了新的基因资源,并将为其它高等植物BAHD酰基转移酶基因家族的研究提供参考。
关键词:
花椒;麻味物质;BAHD酰基转移酶基因家族;基因表达
中图分类号:S573.9
文献标识码:A
文章编号:1008-0457(2024)05-0073-14
国际DOI编码:10.15958/j.cnki.sdnyswxb.2024.05.012
花椒(Zanthoxylum bungeanum Maxim.)为芸香科花椒属落叶小乔木多年生植物,是著名的“八大调味品”之一,也位于“十三香”之首,具有重要的经济、药用及生态价值。中国是花椒的原产国,其产量和种植面积均居世界首位。以山椒素为代表的一系列链状不饱和脂肪酸酰胺类化合物,是花椒中呈现麻味的最基本的物质基础[1-2],主要分布在花椒果皮表面的凸起上,而在花椒中产生最具代表性的辛麻感的化合物是羟基-α-山椒素,花椒主要通过酰胺基团使人感受到麻辣的感觉,麻味的原理是花椒麻素对触觉神经和口腔黏膜造成的一种轻微的电击效应,这种效应可以产生50 Hz的振动频率。辛麻味的物质是花椒树植物的特征成分,是其重要的品质评价指标[3-4]。辛麻味物质是花椒属植物的特征成分,是其重要的品质评价指标,也是吸引消费者和提高市场竞争力的重要因子。目前对于花椒麻味物质的研究则主要侧重于对麻味物质的提取分离、生物活性和呈麻机理的研究,而对于花椒麻味物质生物合成途径的研究较少,通过对花椒麻味物质的合成途径的研究则将有助于进一步筛选出对花椒麻味物质有影响的基因,为之后通过现代生物技术手段改良花椒品种,从而提高其麻味物质的含量及品种提供一定依据与借鉴。
花椒中的麻味物质——不饱和脂肪酸酰胺类化合物则与植物中存在的酰基化结构修饰过程有关。酰化是生物体内的重要反应,涉及催化含氧、含氮及其含硫化合物的合成,以产生相应的酯和酰胺产物[5]。植物中催化酰基化反应的酶即为酰基转移酶,这一类能够催化活性酰基供体转移至特定受体的酶家族就是BAHD酰基转移酶家族。BAHD酰基转移酶家族是植物中修饰初生和次生代谢物的关键蛋白质。它们以酰基辅酶A为供体,生成多种挥发性脂质、修饰花青素等,与植物对病原微生物的抗性紧密相关,进而在信号转导、胁迫防御和代谢稳态中扮演重要角色[6]。BAHD酰基转移酶家族是以其前4种生化表征酶的首字母来命名的,包括苄醇O-乙酰转移酶(BEAT)、花青素O-羟基肉桂酰转移酶(AHCT)、邻氨基苯甲酸N-羟基肉桂酰/苯甲酰转移酶(HCBT)和脱乙酰乙酸麒多啉4-O-乙酰转移酶(DAT)[6]。
据报道,BAHD酰基转移酶家族成员广泛存在于多种植物中,如模式植物拟南芥、大麦、水稻和杨树,以及具有药用和经济价值的植物,如梨(Pyrus spp.)、苦皮藤(Celastrus angulatus)、茉莉花(Jasminum sambac)、茶(Camellia sinensis)和红豆杉(Taxus wallichiana var. chinensis)[7-16]。这些BAHD蛋白在植物体内扮演着重要角色,它们参与了多种活性酰化天然产物及其前体的形成过程,其产物包括生物碱、芳香醇/胺、脂肪醇/胺、萜类化合物以及糖分解剂等[17]。
本研究将利用生物信息学技术,依托全基因组数据资源,对花椒中的BAHD酰基转移酶家族的成员进行筛选鉴定,通过对花椒中BAHD酰基转移酶基因家族的系统发育、进化及理化性质进行分析,同时利用花椒转录组数据来对花椒不同品种、不同部位、不同时期果皮中花椒的BAHD酰基转移酶基因表达情况进行分析,对鉴定出的BAHD基因进行进一步分析与筛选,以期找出与花椒麻味物质合成相关的候选基因。本研究为花椒BAHD酰基转移酶基因家族的研究提供了理论依据,并且将为其他植物BAHD酰基转移酶基因家族的研究提供一定依据和借鉴。
1 材料与方法
1.1 花椒BAHD基因家族成员的鉴定
为了鉴定花椒基因组中的BAHD酰基转移酶基因家族成员,首先从Pfam数据库中获取了BAHD酰基转移酶家族的HMM模型(PF02458),并用此模型通过隐马尔科夫模型软件(HMMER)在花椒基因组数据库[18]中筛选出潜在的BAHD酰基转移酶基因家族成员。此外,采用拟南芥BAHD蛋白序列[19]作为查询序列,通过BLAST软件对花椒蛋白序列进行比对,获取所有潜在的BAHD酰基转移酶基因家族成员的蛋白序列。最后,使用SMART软件[20]和INTERPRO[21]在线工具栏中pfam软件来对这些候选基因的保守结构域进行分析,并验证这些候选基因的保守结构域的完整性,剔除掉其中不含BAHD家族特征结构域的候选序列,最终得到拥有BAHD酰基转移酶家族特征结构域的花椒BAHD家族成员的序列。
1.2 花椒BAHD酰基转移酶基因结构及系统发育分析
从花椒基因组中共鉴定出50个BAHD酰基转移酶蛋白序列。采用ClustalX软件[19]对大麦、拟南芥、东北红豆杉、苜蓿、大豆、瓜叶菊、甜瓜等(附表1)34个物种[20]的BAHD酰基转移酶蛋白序列和花椒鉴定出的蛋白序列(共168个)进行多序列比对,生成ALN格式的文件(附表1)。该文件被导入MEGA v.11软件[21]中,采用邻接法(Neighbor-Joining)进行系统发育树的构建,自展值设置重复次数2000次。随后,采用ITOL[21]美化构建好的进化树。
1.3 花椒BAHD基因的理化性质与蛋白3D结构分析
利用EXPASY[21]分析花椒BAHD基因的氨基酸数量(aa)、分子量(MW)、等电点(pI)和不稳定系数(II)。同时使用WOLF[21] 预测花椒BAHD基因的亚细胞定位。通过SWISS-MODEL[21]构建花椒BAHD蛋白的三维结构模型。
1.4 花椒BAHD基因的保守基序及基因结构分析
为阐明花椒BAHD蛋白motif序列的进化,采用MEME[21]进行蛋白保守基序分析,最大值设置为10,基序长度范围为6~50之间。利用GSDS网站[21]绘制基因结构图。
1.5 花椒BAHD基因的表达量
利用TBtools软件[22]中的Gene Location Visualize from GTF/GFF工具绘制花椒BAHD基因在染色体上的定位信息。使用本实验室获得的花椒转录组数据(附表2),分析花椒的不同发育时期、不同部位和不同品种中的表达模式。使用TBtools软件中的HEATMAP工具可视化花椒 BAHD酰基转移酶基因的表达水平。
2 结果与分析
2.1 花椒基因家族成员序列属性分析
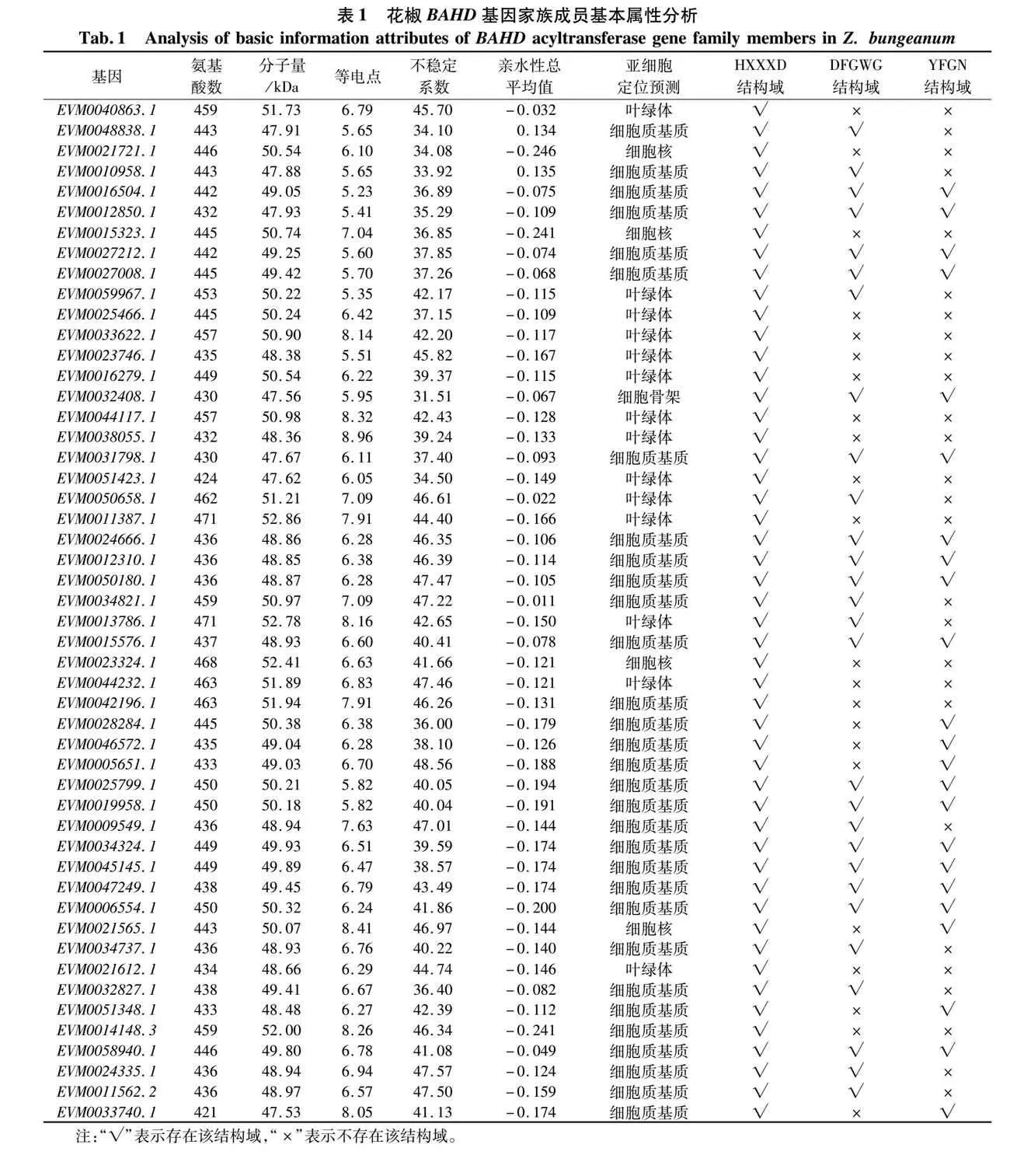
根据基因酰基转移酶基因家族的保守结构域和隐马尔可夫模型文件(HMM),使用(PFAM:PF02458)对已有全基因组测序结果进行搜索识别,最终得到BAHD酰基转移酶基因共计50个(表1)。花椒BAHD酰基转移酶基因家族成员蛋白分子量在47~53 kDa之间,其编码的氨基酸序列长度在420~470 aa之间,等电点在5.23~8.96之间,对其蛋白质亲水性预测结果表明,只有其中两个蛋白质具有疏水性,其余蛋白均为亲水蛋白,不稳定系数在31.51~48.56之间,因此花椒中的BAHD酰基转移酶家族编码蛋白为不稳定亲水蛋白。亚细胞定位预测表明,花椒BAHD酰基转移酶家族成员中约有62%的蛋白定位于细胞质基质中,28%和8%的蛋白分别定位于叶绿体和细胞核中,仅有1个蛋白定位于细胞骨架中。
HXXXD和DFGWG为BAHD酰基转移酶家族的特征保守结构域[23]。花椒BAHD基因家族成员保留了HXXXD结构域,而DFGWG保守结构域具有变异性,只在28个成员中检出,而其余22个成员中的DFGWG保守结构域分化为NFGWG、DLGWG、DFGEG和DFGSG。对于花椒中这22个存在相同DXGXG分化的成员,可能与水稻酚胺合成途径中关于脂肪族苯酚酰胺的生物合成的BAHD酰基转移酶有一定联系[24]。
在BAHD酰基转移酶基因家族中与花青素合成有关的植物中鉴定出了YFGNC结构域[25],植物花青素合成途径属于类黄酮途径的一条重要分支[26]。而花椒的BAHD基因家族部分成员中鉴定出了23个成员拥有YFGN结构域,与程俊等[27]鉴定出胡椒中的YFGN结构域相同,并且花椒中还存在YYGN、FYGN、FFGN变种。
2.2 花椒BAHD酰基转移酶基因家族进化分析
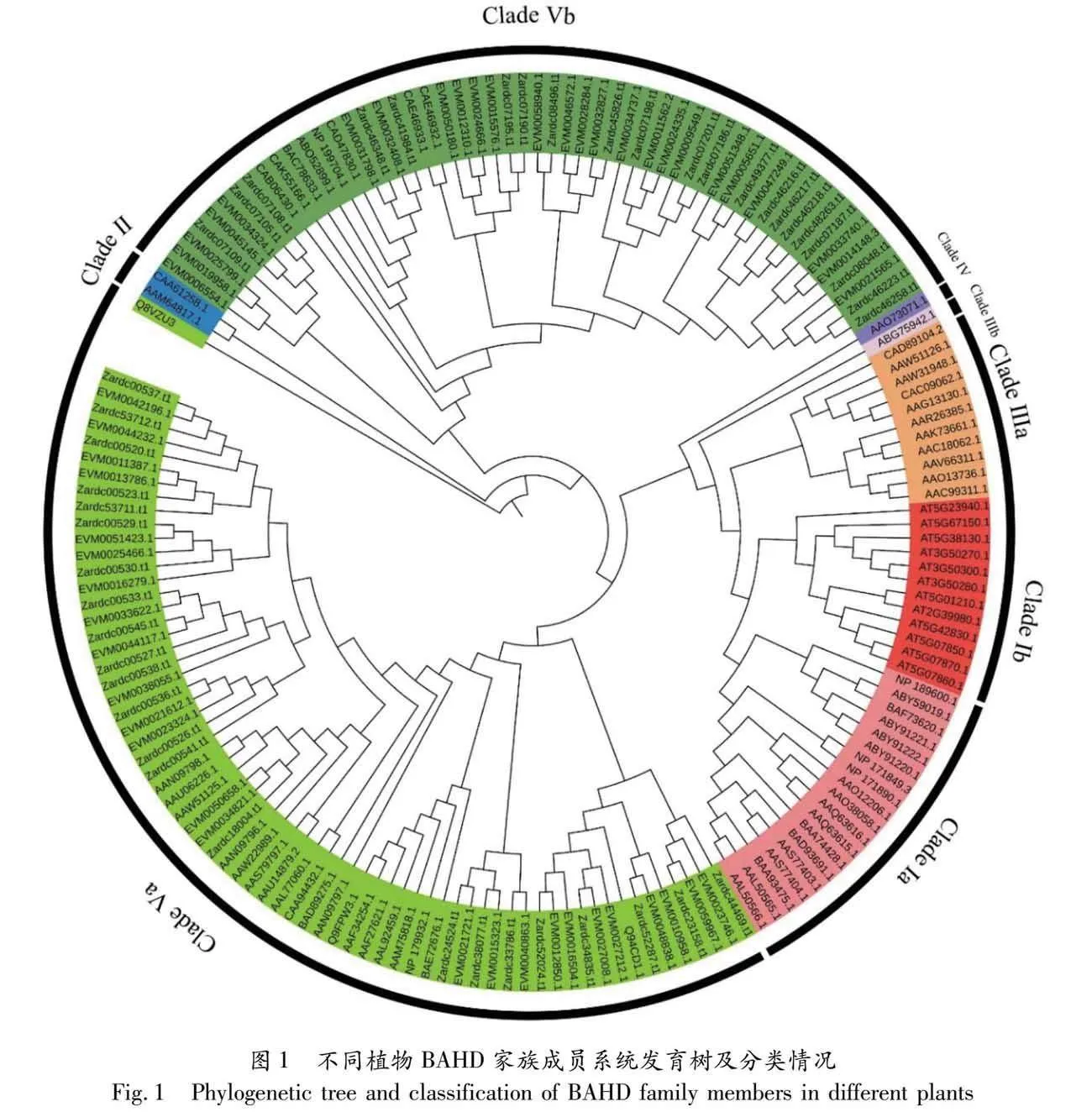
为分析花椒BAHD酰基转移酶基因家族各成员间的进化关系,本研究分别下载了大麦、拟南芥、红豆杉、蒺藜苜蓿、大豆、瓜叶菊等34个物种的BAHD酰基转移酶基因家族蛋白序列,同本研究鉴定的50个花椒BAHD酰基转移酶基因家族蛋白进行序列比对,并构建系统发育树。结果显示,这些序列可以划分为5大分支(图1)。
Clade Ⅰ分支中的序列聚类为2个亚支,在Clade Ⅰa亚支中,所包含的序列大部分都与青花素代谢过程有着紧密的关联,这些序列已经得到了广泛的研究和验证,如:NP_171849,BAA74428,ABY59019等[6, 28-29],该分支中的拟南芥BAHD酰基转移酶基因家族成员也均是属于花色苷分支成员且拥有YFGN蛋白质结构域,说明该分支主要与植物花瓣中存在的花青素合成途径有一定联系。Clade Ⅰb主要由拟南芥BAHD家族基因组成,均为转移蛋白酶家族成员,但该类中无功能已知成员,尽管在分支命名上存在差异,但本研究中该分支的聚合类型及其与家族成员种类的关系与Luo等[30]的研究结果相吻合。在很大程度上支持了本研究的分类方法是可靠和准确的。
Clade Ⅱ主要由AAM64817与CAA61258组成,二者虽然根据同源性被归类为BAHD转移酶,但其似乎并不具有BAHD家族成员的催化机制,反而参与植物蜡质的合成,如AAM64817在超长链脂肪酸(VLCFA)的合成中发挥作用,是C28脂肪酸延长的必要材料[28, 31-32]。但由于该分支成员与花椒BAHD家族基因距离较远,因此推测在花椒的BAHD酰基转移酶一般不参与植物蜡物质的合成。
Clade Ⅲa分支主要由乙酰转移酶组成,功能各异,如AAN09796主要参与乙酸苄酯的生物合成,是植物花香的重要来源[33]。但由于花椒BAHD家族基因成员与其关系较远,说明可能花椒并不具备类似功能,且该分支BAHD家族成员底物种类多样化,而对于花椒BAHD家族成员的底物,则需要通过进一步试验来确定。Clade Ⅲb是由来自茄科的碧冬茄的ABG75942蛋白单独组成,其参与花中挥发性异丁香酚的生物合成,并促进苯乙醛、苯乙醇、乙酸苯乙酯、苯乙基苯甲酸酯和乙酸苄酯的形成,催化针叶醇的乙酰化以产生针叶树乙酸酯[34],但由于其亲缘关系与花椒BAHD家族基因较远,故推断花椒的BAHD家族基因并不具备与该功能相似的功能。
Clade Ⅴ所含成员最多,且与花椒的BAHD家族成员关系最近,功能最为相似,故根据功能性的不同,将Clade Ⅴ分为两个亚分支。在Clade Ⅴa种,因其具有许多亚分支,且每个分支功能各异,如根据拟南芥的NP_179932基因为亚精胺二芥子酰基转移酶,介导种子中二萘酰亚精胺偶联物的积累,并催化N1,N8-二戊酰基亚精胺生物合成所需的2个偶联步骤[35-36],得出该分支可能与亚精胺的生物合成有关;亦通过分析拟南芥的Q94CD1基因,我们了解到其结构域参与了木栓蛋白聚合物中芳烃的合成。基于这一发现,我们推测花椒中的BAHD家族成员可能也参与木栓蛋白的合成过程[29, 37]。同时,碧冬茄的AAU06226基因的功能验证表明,该基因编码的酶主要催化苯甲酰基和乙酰基转移到多种潜在的底物醇上,参与形成挥发性酯,如苯甲酸苄酯和苯乙基苯甲酸酯[38-40]。因此,我们推断花椒中的这一分支的BAHD家族成员可能主要负责催化这些基团的转移。这些反应的主要产物包括羟基肉桂酰基酯、苯甲酸苄酯以及其他相关的苯甲酸类物质的衍生物[41]。Clade Ⅴb中的花椒BAHD酰基转移酶以羟基肉桂酰辅酶A奎宁酸转移酶和羟基肉桂酰转移酶有关联,具有催化以奎宁酸为底物的酰化反应的功能。产物多为羟基肉桂酰基酯、奎宁酸底物合成出的相关产物,故推测此分支中的花椒BAHD家族成员可能也具有相似功能。
2.3 花椒BAHD酰基转移酶基因结构与保守基序分析
花椒BAHD酰基转移酶基因家族分析结果如图2所示,在Motif1中可见BAHD酰基转移酶家族完全保守结构域HXXXD,这是BAHD酰基转移酶家族基因重要鉴定条件之一,并且在蛋白序列中间的位置负责催化活性[42],在图2中可见DFGWG保守结构域,该基序是催化所必须的,也具有结构功能[42]。BAHD酰基转移酶基因家族的DFGWG特征结构域不完全保守,花椒中存在的部分DFGWG变种与NFGWG、DLGWG、DFGEG和DFGSG变种,部分变种与杨树中存在的变种一致[43],因此在植物BAHD酰基转移酶基因家族中存在一定相似进化方向的趋势。
基于MEME在线工具鉴定出花椒BAHD酰基转移酶基因家族有10个motif(图2-b)。所有分支都包含motif 1、motif 6和motif 8;CladeⅤa、CladeⅤb的成员都包含motif 3、motif 5、motif 9;Clade Ⅴa分支存在缺少miotif 2、motif 7、motif 10的情况,缺少的小亚支中成员均出现该情况;而Ⅴb分支中10个motif均有分布,不存在缺少某一个motif的情况。
在真核生物的进化过程中,内含子的丢失和获得扮演着至关重要的角色。对花椒BAHD基因的外显子和内含子组成(图2-c)进行分析,结果表明:不同分支间的内含子和外显子构成存在一定差异性,其中大部分部分基因含有2个外显子,有一个分支只含有1个外显子,也有其他亚支拥有3个外显子的现象,但都不具有明显规律;在花椒BAHD酰基转移酶基因家族成员中,大部分成员都拥有1个内含子,但亚分支上的成员有些出现了无内含子,也有出现拥有2个内含子的现象,规律性不强但与亚支的不同存在一定联系;花椒BAHD基因家族成员中有成员出现不含有UTR区域的现象,但该现象具有分支上的规律性。
2.4 花椒BAHD酰基转移酶基因的表达量分析
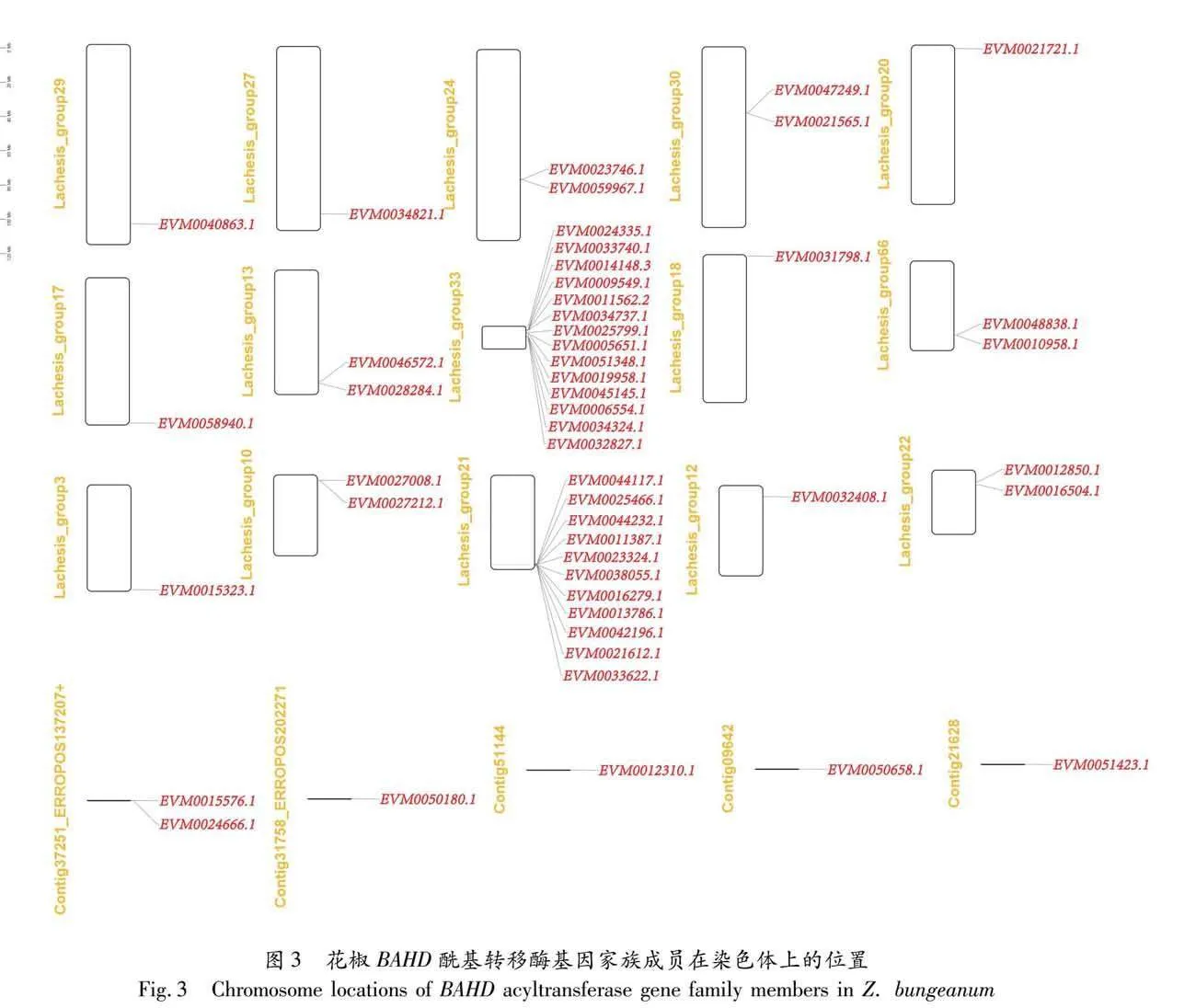
花椒BAHD酰基转移酶基因家族成员在染色体上的位置信息如图3所示,50个BAHD酰基转移酶基因序列分布在20条染色体上,其中第33条染色体上分布了14个BAHD基因,第21条染色体上分布了11条BAHD基因。其余染色体上的BAHD基因的分布比较稀疏,每个染色体上分布1~3个基因。
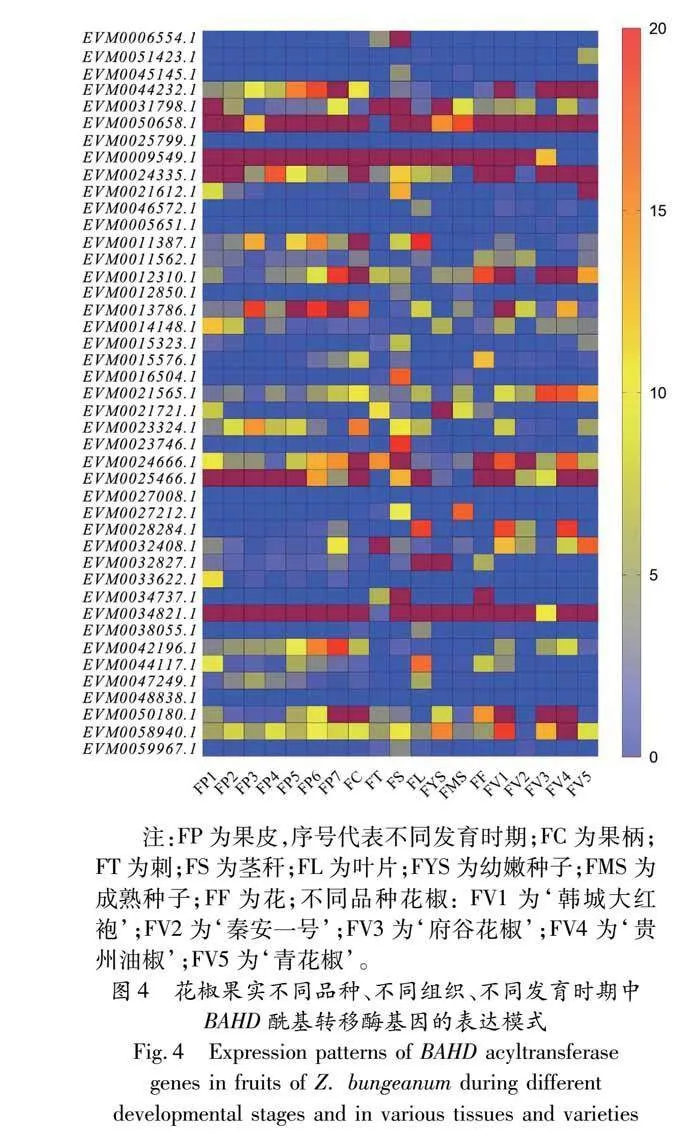
基于花椒果实不同发育时期、不同组织和不同品种的转录组数据分析表明(图4),在花椒果实不同发育过程中,不同BAHD酰基转移酶基因在其不同发育阶段中的表达存在几种变化趋势:
(1)在果实不同发育时期内表达量均高于其他基因的EVM0009549、EVM0050658、EVM0034821和EVM0025466 4个基因都在某一时期达到表达量最高的峰值后出现下降;也有EVM0024335基因在FP1时期就达到了表达量高峰,后面几个时期表达量出现下降趋势的;而EVM0044232基因在前几个时期表达较少,最后FP7时期出现表达量高峰。总体上来说BAHD基因在花椒不同时期的果皮中表达存在一定规律。根据基因的不同,其表达量高峰分布于不同阶段中,不同基因在同一时期的表达量也存在一定差异。
(2)花椒BAHD酰基转移酶基因在果柄、刺、茎秆、叶片、花、幼嫩种子和成熟种子中均有表达,并且在茎秆、刺、果柄和花中表达的基因的数量要比叶片和种子中表达的基因的数量要多,在不同部位中表达量高的基因的数量最多的部位是茎秆,其次是叶片和果柄,花与种子表达量高的基因数量少于前三者。
(3)BAHD酰基转移酶基因在种子中表达的基因的数量要少于其余部位,并且表达量高的基因的数量少于其他部位,但接近刺中的基因表达量高的基因数目。幼嫩种子中部分基因的表达量要略大于成熟种子,但也存在部分基因在成熟种子中表达量要大于幼嫩种子中的表达量的现象;幼嫩种子中表达的基因数量要少于成熟种子中表达的基因的数量,但是幼嫩种子中表达量高的基因的数量要大于成熟种子中的数量;存在两个时期的种子中相同BAHD酰基转移酶基因的表达量发生变化的现象。
(4)不同品种的花椒对花椒中不同的BAHD酰基转移酶基因的表达存在影响,同种基因在不同种花椒中的表达量存在较为明显的差异。如在不同部位、不同时期花椒中均拥有高表达量的EVM0009549基因,在贵州油椒‘FV4’和青花椒‘FV5’中出现了表达量不明显的情况;在5种花椒中‘FV1’表达的基因的数量最多,表达量高的基因数量也最多;而‘FV5’表达的基因的数量最少,但表达量高的基因与其他种数量差距小;‘FV3’中表达量不明显的基因数量最多。相同基因在不同种花椒中的表达存在差异,在表达量上也存在差异。
2.5 花椒BAHD酰基转移酶3D结构预测
BAHD家族的蛋白是一类具有共同特征的球状蛋白,它们大多数位于细胞质中,少数则定位于细胞核[20]。尽管这些蛋白质的一级结构各不相同,但它们普遍含有HXXXD和DFGWG这两个关键的保守序列。HXXXD序列位于活性中心,而DFGWG序列则位于活性位点之外,这两个序列对于蛋白质的催化功能和与供体及底物的结合至关重要。
通过对花椒中来自两个Clade Ⅴ亚支的4个BAHD基因的氨基酸序列进行三级结构预测分析,我们发现了以下模式:EVM0025466的预测蛋白为羟基肉桂酰转移酶,具有17个α螺旋和18个β链(图5-a);EVM0059967的预测蛋白为ω-羟基棕榈酸酯和O-阿魏酰转移酶,含有15个α螺旋和19个β链(图5-b);EVM0034324的预测蛋白为亚精胺羟基肉桂酰转移酶,同样具有15个α螺旋和19个β链(图5-c);而EVM0050180的预测蛋白为羟基肉桂酰辅酶A莽草酸酯/奎宁酸酯羟基肉桂酰转移酶,具有17个α螺旋和18个β链(图5-d)。这些结构特征为理解这些BAHD家族成员的功能提供了重要线索。
综上所述,Clade Ⅴa的两个蛋白(EVM0025466、EVM0059967)的区别主要是α和β螺旋数目不同,在整体结构上不存在太大的差异;Clade Ⅴb的2个蛋白(EVM0034324、EVM0050180)的主要差异也是α和β螺旋数目不同。这4个蛋白的3D结构预测结果在整体上是相似的,可能是由于这四个蛋白同处于一个Clade Ⅴ分支。
3 讨论与结论
本研究采用多种植物的BAHD酰基转移酶基因为参考,通过生物信息学分析了花椒基因组中的BAHD酰基转移酶基因的结构特征,有助于对植物BAHD基因家族的注释,并在一定程度上揭示了花椒BAHD基因家族的成员以及基因结构特征,也作为进一步研究花椒BAHD家族提供一定依据和借鉴。
BAHD酰基转移酶在植物体内扮演着多重角色,不仅参与植物生长发育的调控,还在胁迫反应以及次生代谢产物的合成和修饰过程中发挥着至关重要的作用[17]。本研究在花椒基因组中共鉴定了50个BAHD酰基转移酶基因家族成员。
3.1 花椒BAHD酰基转移酶基因家族成员的系统发育关系
对花椒与其他物种的BAHD酰基转移酶基因家族成员进行系统发育树构建,得到花椒的BAHD成员主要分步于1个大分支Clade Ⅴ,这一分支又可分为两个亚支Clade Ⅴa与Clade Ⅴb。Clade Ⅴa分支中的花椒蛋白可能参与以笨甲酰辅酶和乙酰辅酶A作为酰基供体参与亚精胺或木栓蛋白聚合物的芳烃的合成[29, 37],其产物多为羟基肉桂酰基酯、苯甲酸苄酯以及其他苯甲酸类物质的衍生物[41]。Clade Ⅴb中的花椒分支与羟基肉桂酰辅酶A奎宁酸转移酶和羟基肉桂酰转移酶有关联,具有催化以奎宁酸为底物的酰化反应的功能[17]。Clade Ⅴa分支中存在缺少motif 2、motif 7、motif 10的现象出现,并且缺少motif的情况的基因所处小分支中均出现该情况,因此Ⅴa中的小亚支可能存在由于植物学上的功能的差异从而导致的motif丢失现象,具体情况还需进一步探索。在花椒BAHD基因家族成员的外显子和内含子组成中,Clade Ⅴa、Clade Ⅴb两个分支中都出现了在组成上存在差异的现象。不同亚分支间的内含子和外显子的组成上存在差异,其中大部分基因含有2个外显子,而有一个分支,其组成上只有1个外显子;其余成员大部分都拥有1个内含子,但在CladeⅤa、CladeⅤb中也都存在部分亚分支无内含子、拥有2个内含子的现象;并且存在亚分支基因结构中不含有UTR区域的现象,而根据花椒BAHD家族成员的基因结构差异存7BrYARMunGUBUB3YXaf5lA==在分支规律性,可以根据此来为一步探索不同小分支之间的功能性差异提供一种思路。
花椒的BAHD酰基转移酶基因家族中的HXXXD结构域高度保守,而DFGWG结构域在花椒中并不完全保守,22个成员存在DXGXG的分化与其他物种的植物拥有相同变异方向[43]可能还会拥有相似的进化趋向。在花椒的BAHD基因家族部分成员中鉴定出了YFGN结构域,还存在YYGN、FYGN、FFGN变种,这些拥有YFGN结构域的花椒BAHD家族成员可能与花青素合成途径存在一定联系,且鉴定出YFGN结构域的花椒基因可为鉴定花椒BAHD家族基因提供一种思路。
3.2 花椒BAHD酰基转移酶基因家族成员的理化性质与蛋白结构
Suzuki等[44]的研究表明BAHD家族的分子量在48~55 kDa之间,氨基酸平均数为445个,且主要定位于细胞质中,少数定位于细胞核中。本研究的花椒的BAHD基因家族成员蛋白分子量在47~53 kDa之间,氨基酸长度在420~470 aa之间,理论等电点在5.23~8.96之间,与其研究结果基本一致。对其蛋白质亲水性预测结果表明,只有两个蛋白具有疏水性,其余蛋白均为亲水蛋白,并且蛋白的不稳定系数在31.51~48.56间,所以花椒的BAHD家族成员的蛋白为不稳定亲水蛋白。
BAHD家族成员的第一个表征晶体结构是在植物蛇根木(Rauvolfia serpentina)中发现的酶,属于BAHD(苄醇乙酰基转移酶、花青素-O-羟基肉桂酰基转移酶、邻氨基苯甲酸-N-羟基肉桂酰基/苯甲酰基转移酶、去乙酰基长春新碱乙酰基转移酶)酶超家族的一员(Rauvolfia serpentina vinorine,RsVS)(PDB ID:2BGH),由14个β链和13个α螺旋组成[46],BAHD家族中N-酰基转移酶的第一个表征晶体结构是大麦芽氨酸香豆酰转移酶(Hordeum vulgare agmatine coumaroyltransferase,HvACT)(PDB ID:7CYS)该结构包含18个β链和13个α螺旋[45]。目前发表的BAHD成员的晶体结构为26种。花椒中的BAHD成员的蛋白3D结构便是使用这些已发表晶体结构进行预测构建。选取4个花椒的BAHD酰基转移酶基因的氨基酸进行三级结构预测,Clade Ⅴa的2个蛋白主要是α和β螺旋数目不同,在整体形状上不存在特别大的差异,Clade Ⅴb的2个蛋白之间的差异也主要是α和β螺旋数目不同,这4个蛋白在3D结构上没有明显的差异可能是由于同处于一个大亚族Clade Ⅴ。
3.3 花椒BAHD酰基转移酶基因的表达特征
花椒BAHD家族基因在花椒果皮中展现出表达差异,在表达达到高峰后,其表达水平通常呈现下降趋势。此外,这些成员在不同植物部位的表达情况亦有所区别,具体表现为茎秆、刺、果柄和花的表达量普遍高于叶片和种子。值得注意的是,如果一个基因在某一特定部位的表达量较高,那么在其他部位的表达量则相对较低。这些现象共同揭示了花椒BAHD家族基因的表达情况受到花椒不同部位的显著影响。
花椒BAHD基因种子发育的不同阶段呈现出差异化的表达模式。相较于成熟种子,幼嫩种子中BAHD家族成员的基因表达量较低,这揭示了花椒BAHD基因的表达在种子发育过程中存在动态变化。并且部分基因在幼嫩种子中处于未表达状态,可能与种子发育过程中的不同生物学功能及代谢需求相关。
不同品种的花椒对BAHD酰基转移酶基因的表达具有显著影响,导致同种基因在不同品种的花椒中表现出明显的表达量差异。在研究的5个品种中,‘FV1’品种的花椒显示出了BAHD酰基转移酶基因表达数量的优势,其表达量相对较高。相反,‘FV5’品种的花椒则表现出较低的基因表达数量。值得注意的是,‘FV3’品种虽然表达基因数量正常,但由于其表达量不高,因此基因表达情况并不明显。这些结果表明,花椒的品种差异是导致BAHD酰基转移酶基因表达差异的重要因素之一,对于理解和利用花椒的遗传特性具有重要意义。
花椒BAHD基因的表达受到多种因素的影响,包括组织部位、种子发育阶段以及花椒品种等。这些差异可能与花椒的生长发育、次生代谢产物的合成以及环境适应性等生物学过程密切相关。并且我们发现不同品种的花椒对BAHD酰基转移酶基因的表达有显著影响,花椒的遗传资源可能在很大程度上决定了其生理特性和代谢产物的积累,从而导致不同BAHD基因的表达情况存在明显差异。
3.4 结论
本研究从花椒基因组中筛选鉴定了50个BAHD家族成员,分析发现所有成员均具有HXXXD结构;并且拥有DFGWG结构的成员中有些出现变种;部分成员还具有YFGN结构。这50个成员在发育进化树中根据已鉴定功能蛋白的聚类结果分为两个分支,每个分支下的小分支间在基因结构上存在一定差异。花椒BAHD家族基因在表达情况上存在差异:(1)BAHD基因在果皮不同时期存在特异高表达;(2)BAHD基因在不同部位表达存在差异,表达量最高的部位是茎秆、刺和花;(3)成熟种子的表达量大于幼嫩种子;(4)花椒BAHD基因在不同品种花椒中表达情况也有明显差异。
(责任编辑:严秀芳)
参 考 文 献:
[1]
Zhang L L,Zhao L,Wang H Y,et al.The relationship between alkylamide compound content and pungency intensity of Zanthoxylum bungeanum based on sensory evaluation and ultra-performance liquid chromatography-mass spectrometry/ mass spectrometry (UPLC-MS/MS) analysis[J].Journal of the Science of Food and Agriculture,2019,99(4):1475-1483.
[2]高夏洁,钟葵,赵镭,等.不同产区花椒油的椒麻感官特性及物质组成[J].食品科学,2022,43(8):281-287.
[3]Sugai E,Morimitsu Y,Kubota K.Quantitative analysis of sanshool compounds in Japanese pepper (Xanthoxylum piperitum DC.) and their pungent characteristics[J].Bioscience,Biotechnology,and Biochemistry,2014,69(10):1958-1962.
[4]Menozzi C,Riera C E,Munari C,et al.Synthesis and evaluation of new alkylamides derived from α-hydroxysanshool,the pungent molecule in Szechuan pepper[J].Journal of Agricultural and Food Chemistry,2009,57(5):1982-1989.
[5]Wang L L,Chen K,Zhang M,et al.Catalytic function,mechanism,and application of plant acyltransferases[J].Critical Reviews in Biotechnology,2022,42(1):125-144.
[6]Auria J C.Acyltransferases in plants:a good time to be BAHD[J].Current Opinion in Plant Biology,2006,9(3):331-340.
[7]Grienenberger E,Besseau S,Geoffroy P,et al.A BAHD acyltransferase is expressed in the tapetum of Arabidopsis anthers and is involved in the synthesis of hydroxycinnamoyl spermidines[J].The Plant Journal,2009,58(2):246-259.
[8]Zhang Z,Xu L.Arabidopsis BRASSINOSTEROID INACTIVATOR2 is a typical BAHD acyltransferase involved in brassinosteroid homeostasis[J].Journal of Experiment Botany,2018,69(8):1925-1941.
[9]Liu C X,Qiao X,Li Q H,et al.Genome-wide comparative analysis of the BAHD superfamily in seven rosaceae species and expression analysis in pear (Pyrus bretschneideri)[J].BMC Plant Biology,2020,20(1):14.
[10]Yamane M,Takenoya M,Yajima S,et al.Crystal structure of barley agmatine coumaroyltransferase,an N-acyltransferase from the BAHD superfamily[J].Acta Crystallographica Section F Structural Biology Communications,2020,76(12):590-596.
[11]Yan X G,Qin X Y,Li W G,et al.Functional characterization and catalytic activity improvement of BAHD acyltransferase from Celastrus angulatus Maxim[J].Planta,2020,252(1):6.
[12]Wang T,Li L Y,Zhuang W B,et al.Recent research progress in taxol biosynthetic pathway and acylation reactions mediated by Taxus acyltransferases[J].Molecules,2021,26(10):2855.
[13]Zhao Y J,Yu X H,Lam P Y,et al.Monolignol acyltransferase for lignin p-hydroxybenzoylation in Populus[J].Nature Plants,2021,7(9):1288-1300.
[14]Aktar S,Bai P,Wang L,et al.Identification of a BAHD acyltransferase gene involved in plant growth and secondary metabolism in tea plants[J].Plants Basel,2022,11(19):2483.
[15]Wang Y,Zhang H,Wan C,et al.Characterization of two BAHD acetyltransferases highly expressed in the flowers of Jasminum sambac (L.).Aiton[J].Plants (Basel),2021,11(1):13.
[16]Yuan Z,Yang H,Pan L,et al.Systematic identification and expression profiles of the BAHD superfamily acyltransferases in barley (Hordeum vulgare)[J].Scientific Reports,2022,12(1):5063.
[17]Xu D H,Wang Z,Zhuang W B,et al.Family characteristics,phylogenetic reconstruction,and potential applications of the plant BAHD acyltransferase family[J].Frontiers in Plant Science,2023,14:1218914.
EyaF+MqKFL9xRmGWMV8f4w==[18]Feng S J,Liu Z S,Cheng J,et al.Zanthoxylum-specific whole genome duplication and recent activity of transposable elements in the highly repetitive paleotetraploid Z.bungeanum genome[J].Horticulture Research,2021,8(1):205.
[19]Larkin M A,Blackshields G,Brown N P,et al.clustal W and clustal X version 2.0[J].Bioinformatics,2007,23(21):2947-2948.
[20]Tuominen L K,Johnson V E,Tsai C J.Differential phylogenetic expansions in BAHD acyltransferases across five angiosperm taxa and evidence of divergent expression among Populus paralogues[J].BMC Genomics,2011,12(1):236.
[21]Tamura K,Stecher G,Kumar S.MEGA11:molecular evolutionary genetics analysis version 11[J].Molecular Biology and Evolution,2021,38(7):3022-3027.
[22]Chen C,Chen H,Zhang Y,et al.TBtools:an integrative Toolkit developed for interactive analyses of big biological data[J].Molecular Plant,2020,13(8):1194-1202.
[23]Unno H,Ichimaida F,Suzuki H,et al.Structural and mutational studies of anthocyanin malonyltransferases establish the features of BAHD enzyme catalysis[J].Journal of Biological Chemistry,2007,282(21):15812-15822.
[24]Peng M,Gao Y Q,Chen W,et al.Evolutionarily distinct BAHD N-acyltransferases are responsible for natural variation of aromatic amine conjugates in rice[J].Plant Cell,2016,28(7):1533-1550.
[25]Nakayama T,Suzuki H,Nishino T.Anthocyanin acyltransferases:specificities,mechanism,phylogenetics,and applications[J].Journal of Molecular Catalysis B:Enzymatic,2003,23(2):117-132.
[26]赵海菲,王天娅,余坤江,等.MYB转录因子调控植物花青素生物合成的研究进展[J].山地农业生物学报,2022,41(3):49-56.
[27]程俊,后思宇,李靖,等.胡椒碱合成相关BAHD酰基转移酶基因的克隆与分析[J].分子植物育种,2023,21(2):437-453.
[28]Lai C,Kunst L,Jetter R.Composition of alkyl esters in the cuticular wax on inflorescence stems of Arabidopsis thaliana cer mutants[J].The Plant Journal,2007,50(2):189-196.
[29]Waki T,Yoo D,Fujino N,et al.Identification of protein-protein interactions of isoflavonoid biosynthetic enzymes with 2-hydroxyisoflavanone synthase in soybean(Glycine max(L.)Merr.)[J].Biochemical and Biophysical Research Communications,2016,469(3):546-551.
[30]Luo J,Nishiyama Y,Fuell C,et al.Convergent evolution in the BAHD family of acyl transferases:identification and characterization of anthocyanin acyl transferases from Arabidopsis thaliana[J].The Plant Journal,2007,50(4):678-695.
[31]Haslam T M,Maas-Fernández A,Zhao L,et al.Arabidopsis ECERIFERUM2 is a component of the fatty acid elongation machinery required for fatty acid extension to exceptional lengths[J].Plant Physiology,2012,160(3):1164-1174.
[32]Pascal S,Bernard A,Sorel M,et al.The Arabidopsis cer26 mutant,like the cer2 mutant,is specifically affected in the very long chain fatty acid elongation process[J].The Plant Journal,2013,73(5):733-746.
[33]D'Auria J C,Chen F,Pichersky E.Characterization of an acyltransferase capable of synthesizing benzylbenzoate and other volatile esters in flowers and damaged leaves of Clarkia breweri[J].Plant Physiology,2002,130(1):466-476.
[34]Dexter R,Qualley A,Kish C M,et al.Characterization of a petunia acetyltransferase involved in the biosynthesis of the floral volatile isoeugenol[J].The Plant Journal,2007,49(2):265-275.
[35]Luo J,Fuell C,Parr A,et al.A novel polyamine acyltransferase responsible for the accumulation of spermidine conjugates in Arabidopsis seed[J].Plant Cell,2009,21(1):318-333.
[36]Wang C Y,Li J X,Ma M L,et al.Structural and biochemical insights into two BAHD acyltransferases (AtSHT and AtSDT) involved in phenolamide biosynthesis[J].Frontiers in Plant Science,2021,11:610118.
[37]Gou J Y,Yu X H,Liu C J.A hydroxycinnamoyl transferase responsible for synthesizing suberin aromatics in Arabidopsis[J].Proceedings of the National Academy of Sciences,2009,106(44):18855-18860.
[38]Boatright J,Negre F,Chen X,et al.Understanding in vivo benzenoid metabolism in petunia petal tissue[J].Plant Physiology,2004,135(4):1993-2011.
[39]Orlova I,Marshall-Coloón A,Schnepp J,et al.Reduction of benzenoid synthesis in petunia flowers reveals multiple pathways to benzoic acid and enhancement in auxin transport[J].The Plant Cell,2006,18(12):3458-3475.
[40]Colón A M,Sengupta N,Rhodes D,et al.A kinetic model describes metabolic response to perturbations and distribution of flux control in the benzenoid network of Petunia hybrida[J].The Plant Journal,2010,62(1):64-76.
[41]孙燕铭,李亿红,程曦,等.梨BAHD家族成员的鉴定、序列特征及表达分析[J].核农学报,2018,32(10):1917-1930.
[42]Molina I,Kosma D.Role of HXXXD-motif/BAHD acyltransferases in the biosynthesis of extracellular lipids[J].Plant Cell Reports,2015,34(4):587-601.
[43]Yu X H,Gou J Y,Liu C J.BAHD superfamily of acyl-CoA dependent acyltransferases in Populus and Arabidopsis:bioinformatics and gene expression[J].Plant Molecular Biology,2009,70(4):421-442.
[44]Suzuki H,Sawada S,Watanabe K,et al.Identification and characterization of a novel anthocyanin malonyltransferase from scarlet sage (Salvia splendens) flowers:an enzyme that is phylogenetically separated from other anthocyanin acyltransferases[J].The Plant Journal,2004,38(6):994-1003.
[45]Yamane M,Takenoya M,Yajima S,et al.Crystal structure of barley agmatine coumaroyltransferase,an N-acyltransferase from the BAHD superfamily[J].Acta Crystallographica.Section F,Structural Biology Communications,2020,76(12):590-596.
Identification and Differential Expression Analysis of BAHD Acyltransferase Gene Family in Zanthoxylum bungeanum Maxim.
Xiong Wenwei, Cai lian, Su Jing, Yang Yintiao, Feng Shijing*
(College of Forestry, Guizhou University, Guiyang 550025, Guizhou, China)
Abstract:
Alkyamides are one of the important characteristic components for evaluating the quality of Zanthoxylum bungeanum Maxim. In order to explore candidate genes for the acylation reaction related to the synthesis of alkyamides in Z. bungeanum, the BAHD acyltransferase gene family of Z. bungeanum was studied by bioinformatics method based on the whole genome data of Z. bungeanum. The results showed that a total of 50 members of the Z. bungeanum BAHD acyltransferase gene family were identified. These genes can be divided into two subfamilies, all of which encode unstable hydrophilic proteins. Subcellular localization analysis showed that most of the Z.bungeanum BAHD members were mainly located in the cytoplasm, and other members were located in the nucleus, chloroplasts and other organelles. Gene expression analysis showed that the genes of BAHD family members were highly expressed in all stages of pericarp development of Z. bungeanum, and the expression in pedicels, spines, stems and flowers was higher than that in seeds and leaves. Among different pepper varieties, even the same gene also showed expression differences, and even no expression was detected in some varieties. This paper provides a new gene resource for the synthesis pathway of numb-taste compounds in Z. bungeanum, and will provide a reference for the study of BAHD acyltransferase gene family in other higher plants.
Keywords:
Zanthoxylum bungeanum Maxim.; alkylamides; BAHD acyltransferase gene family; gene expression
