基于线粒体 12S基因分析福建省棘胸蛙不同地理种群遗传多样性
2024-10-18吴锐琼张雪萍杨理程陈友铃黄镇





摘 要:棘胸蛙Quasipaa spinosa为福建省特色分布的大型蛙类,被列入三有保护动物之列,开发新的辨别福建省内棘胸蛙不同地理种群的快速方法对于该物种的保护极为重要。通过选取福建省内14个地区的野生棘胸蛙样本,提取其DNA并通过PCR扩增出线粒体12SrRNA基因,然后进行一代测序,基于获得的12SrRNA基因序列构建了进化树。结果表明:福建省内的棘胸蛙可以分成两大类种群,武夷山、建阳、顺昌、将乐、明溪、永安、华安、德化和新罗聚为一类(西部群体),武夷山、政和、屏南、宁德、福鼎、永泰、德化、新罗和福安聚为另一类(东部群体),其中,武夷山、德化、新罗在两个大类均有分布。为了更快地基于12SrRNA基因区分两个群体,筛选了这两大类种群的12SrRNA基因的SNP位点,并利用竞争性等位基因特异性PCR(KASP,KompetitiveAllele-SpecificPCR)方法,开发了一种针对这两个群体的特异性SNP快速分型方法,结果显示所开发的KASP检测体系能准确区分福建省野生棘胸蛙的两个支系。该结果可为深入了解福建省内棘胸蛙的种群分化、有效鉴别不同地理种群,以及为种质资源保护和育种工作提供科学依据。
关键词:棘胸蛙;分子标记;SNP位点;KASP技术
中图分类号:S966.3 文献标志码:A 文章编号:0253−2301(2024)07−0010−07
DOI: 10.13651/j.cnki.fjnykj.2024.07.002
Analysis on Genetic Aiversity of Different Geographical Populations of Quasipaa spinosa in Fujian
Province Based on the Mitochondrial 12S Gene
WURui-qiong,ZHANGXue-ping,YANGLi-cheng,CHENYou-ling,HUANGZhen(College of Life Sciences, FujianiKq2cmobzTOlEtZSUODJlnjfDnOn02Na5QCIRysQ29Q= Normal University , Fuzhou, Fujian 350117, China)
Abstract: Quasipaa spinosaisalarge-scalefrogspecieswithauniquedistributioninFujianProvinceandislistedasaprotectedspeciesunderthe“ ThreeProtectedCategories” .ThedevelopmentofnewrapidmethodstoidentifydifferentgeographicalpopulationsofQuasipaa spinosainFujianProvinceisextremelyimportantfortheprotectionofthisspecies.Inthisstudy,theDNAwasextractedfromthewildQuasipaa spinosasamplescollectedfrom14regionsinFujianProvince,andthemitochondrial12SrRNAgenewasamplifiedbyusingPCR.Then,thefirst-generationsequencingwasconducted.Theevolutionarytreewasconstructedbasedontheobtained12SrRNAgenesequence.TheresultsshowedthatQuasipaaspinosainFujianProvincecouldbedividedintotwomajorpopulations:Wuyishan,Jianyang,Shunchang,Jiangle,Mingxi,Yong'an,Hua'an,DehuaandXinluowereclusteredintoonegroup(thewesternpopulation).Wuyishan,Zhenghe,Pingnan,Ningde,Fuding,Yongtai,Dehua,XinluoandFu'anwereclusteredintoanothergroup(theeasternpopulation). pq99zM26SE0sTay+mSwoR125iOnilgSY9YSH3JpVr2Y=;Amongthem,Wuyishan,DehuaandXinluoweredistributedinbothmajorcategories.Inordertodistinguishthetwopopulationsbasedonthe12SrRNAgenemorequickly,theSNPlociofthe12SrRNAgeneinthetwomajorpopulationswerescreened.AndarapidspecificSNPgenotypingmethodforthesetwopopulationswasdevelopedbasedonthecompetitiveallele-specificPCR(KASP,KompetitiveAllele-SpecificPCR)method.TheresultsshowedthatthedevelopedKASPdetectionsystemcouldaccuratelydistinguishthetwolineagesofwildQuasipaa spinosainFujianProvince.Theresultscouldprovideascientificbasisforthein-depthunderstandingofthepopulationdifferentiationofQuasipaa spinosainFujianProvince,andtheeffectiveidentificationofdifferentgeographicalpopulations,aswellastheprotectionandbreedingofgermplasmresources.
Key words: Quasipaa spinosa;MolecularMarkers;SNPloci;KASPtechnology
棘胸蛙Quasipaa spinosa属两栖纲Amphibia无尾目Anura叉舌蛙科Dicroglossidae棘胸蛙属Quasipaa,是我国南方地区特色的食药用蛙类。近年来,随着生态环境的恶化,棘胸蛙的栖息地遭受破坏[1],以及人为的滥捕现象加剧,这些因素共同导致福建省内部分地区的野生棘胸蛙资源受到严重破坏,种群数量日趋减少[2]。针对这一现象,如何有效监测省内的野生棘胸蛙的种质资源遗传多样性,并开展种质资源保护,成为当前亟待解决的一大问题。棘胸蛙属于两栖类,其扩散能力有限,容易受历史地质、气候事件等因素的影响,而产生遗传上的谱系地理分歧演化。国内许多学者开展过棘胸蛙的遗传谱系地理分化的研究[3],发现其种群存在支系分布现象,且不同支系间存在显著的遗传分化。目前,从全国范围来分析,国内分布的棘胸蛙存在3个支系,一支分布于中国东南部:浙江、安徽及福建部分地区;一支分布于中国中南部:湖南、江西、广东、广西及福建部分地区;一支分布于中国西南部:云南[3−6]。然而,目前关于福建省内野生棘胸蛙不同地理种群支系的遗传多样性报道较少,这在一定程度上阻碍了野生棘胸蛙保护工作的开展。因此,如何快速、准确地对福建省内的棘胸蛙地理种群支系进行判断,对于福建省棘胸蛙资源的保护具有重要的现实意义。
线粒体是真核生物细胞核外的一种半自主细胞器,是细胞进行有氧呼吸的主要场所,是动物体内唯一的核外遗传信息载体,具有分子量小、严格母系遗传、结构简单等特点,已被广泛用于研究物种的遗传多样性和群体间遗传结构的差异[7]。其中,线粒体12SrRNA基因是无尾两栖动物在遗传多样性研究中应用较多的分子标记。单核苷酸多态性(SingleNucleotidePolymorphism,SNP),是指基因组水平上由单核苷酸变异引起的序列多态性。基因组中在同一位置发生相同碱基突变的概率极低,因此SNP具有遗传稳定、数量极大、特异性强、便于批量检测等优点,被广泛用作主流的分子标记[8−9],并在遗传疾病诊断[10−11]、分子遗传学[12]以及动植物育种[12−14]中发挥着重要作用。本研究基于线粒体12SrRNA基因数据,分析了福建省野生棘胸蛙不同地理种群的遗传多样性,并根据不同支系的棘胸蛙在12SrRNA基因上的SNP差异,开发了竞争性等位基因特异性PCR(KompetitiveAlleleSpecificPCR,KASP)的分型方法,这有助于解析福建省内野生棘胸蛙不同种群间的遗传差异,也有助于建立棘胸蛙的遗传资源库,为未来的遗传育种、种质资源保护以及可持续利用提供基础数据。
1 材料与方法
1.1野生棘胸蛙的来源
本研究所用的野生棘胸蛙样本采集自福建省15个地区:武夷山市、建阳区、顺昌县、将乐县、明溪县、永安市、华安县、德化县、新罗区、政和县、屏南县、宁德市、福鼎市、永泰县、福安市。均取野生棘胸蛙的大腿内侧肌肉组织,用于DNA抽提。
1.2野生棘胸蛙DNA提取
剪取棘胸蛙大腿内侧肌肉组织,取适量的肌肉组织于EP管中,加入蛋白酶K,用涡旋振荡仪将其混匀。按照Quick-DNATMMiniprepPlusKit试剂盒(ZYMORESEARCH)说明书的步骤,提取棘胸蛙的DNA。提取好的DNA保存在−80℃冰箱中。
1.3棘胸蛙线粒体12SrRNA基因的PCR引物设计及反应体系
用于扩增棘胸蛙线粒体12srRNA基因的PCR引物序列见表1。PCR反应体系(20µL)组成:2×AccurateTaqMasterMix(艾科瑞生物有限公司)10µL,上下游引物各0.2µL,DNA模版1µL,ddH2O8.6µL。PCR扩增程序:94℃预变性5min;94℃变性30s,52℃退火30s,72℃延伸30s,循环35次;72℃再延伸7min,4℃冷却保存。PCR扩增所得产物经2%琼脂糖凝胶电泳,剩余PCR产物送往铂尚生物技术(上海)有限公司进行测序。
1.4线粒体12srRNA基因序列数据分析
PCR产物的测序序列全部通过NCBI官网进行同源性比对,从而确定所得的序列为棘胸蛙的线粒体12SrRNA基因序列。采用MEGA7.0软件对所采集的棘胸蛙线粒体12srRNA基因序列构建系统发生树,参数设置为邻接法(Neighborjoining,NJ),同时用自展检验法(BootstrapTest)获得系统进化树各分支的置信值,重复抽样1000次。构建进化树时以脆皮大头蛙Limnonectes fragilis和斑腿树蛙Polypedates megacephalus作为外群。
1.5KASP反应体系及扩增程序
采用ClusterW软件分析野生棘胸蛙在福建省内两大地理种群分支的线粒体12SrRNA基因序列,通过序列比对,找到两个地理种群分支中独有的SNP位点。进一步基于DNAMAN软件和引物设计软件Primer3针对SNP位点设计KASP特异性引物(见表2),引物设计完成后经铂尚生物技术(上海)有限公司合成。KASP引物混合体系的配置:12SrRNA-F1(100μmol·L−1)12µL,12SrRNA-F2(100μmol·L−1)12µL,12SrRNA-R(100μmol·L)30µL,ddH2O46µL,混合均匀。KASP反应体系(10µL):HiGeno2×ProbeMix(北京嘉程生物科技有限公司)5µL,KASP引物0.14µL,DNA模板1µL,ddH2O4µL。AQPTM基因分型系统的PCR扩增程序:95℃预变性10min;95℃变性20s,61℃退火40s,循环35次,每循环1次,温度降0.6℃;95℃变性20s,55℃退火/延伸40s,循环45次;4℃冷却保存。PCR扩增循环结束后,采用TaqManGenotyperSoftware对AQPTM基因分型数据进行分析。
2 结果与分析
2.1棘胸蛙样本的DNA提取结果
提取的棘胸蛙基因组DNA,采用1%琼脂糖凝胶电泳(图1)检测,条带明亮清晰,并且无明显拖带现象,可用于后续的基因PCR扩增。
2.2棘胸蛙线粒体12SrRNA基因的扩增
棘胸蛙线粒体12SrRNA基因片段的PCR扩增结果经2%琼脂糖凝胶电泳检测,图谱显示均获得明亮清晰的条带,12SrRNA扩增所获片段长度为400bp左右,见图2。
2.3基于线粒体12SrRNA基因序列分析福建省野生棘胸蛙的种群分化
以脆皮大头蛙和斑腿树蛙的线粒体12SrRNA基因对应序列为外群,对所筛得的福建野生棘胸蛙单倍型进行聚类分析。结果显示,以邻接法构建的系统进化树(图3)中,可以将福建省野生棘胸蛙分成两大支,武夷山(WY)、建阳(JY)、顺昌(SC)、将乐(JL)、明溪(MX)、永安(YA)、华安(HA)、德化(DH)和新罗(XL)聚为一支A1,武夷山(WY)、政和(ZH)、屏南(PN)、宁德(ND)、福鼎(FD)、永泰(YT)、德化(DH)、新罗(XL)和福安(FA)聚为一支B1。其中,武夷山、德化、新罗在两个大支均有分布。
2.4福建省内野生棘胸蛙线粒体12SrRNA基因序列上地区特异SNP筛选
将所有棘胸蛙的线粒体12SrRNA基因测序结果分别进行序列比对,对获得的分组进行组间序列比较,挑选出每组特有的SNP位点(图4)。
2.5基于线粒体12SrRNA基因序列上地区特异SNP开发KASP快速分型体系
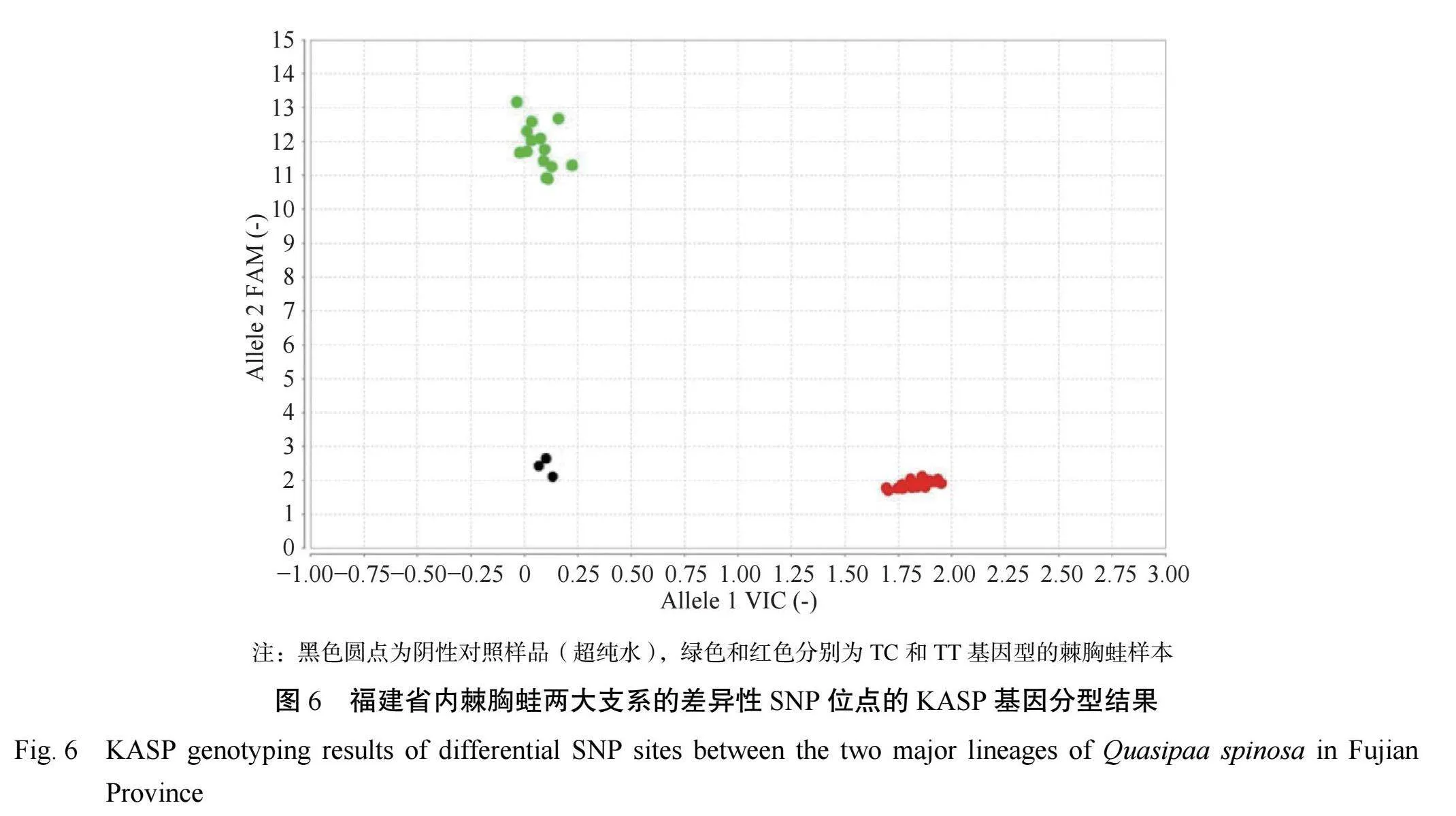
本研究基于线粒体12SrRNA基因数据分析,在12SrRNA上筛选出1个特异的SNP位点,根据该SNP位点(图5)设计1对KASP引物。KASP反应测试在ABIStepOnePlus实时荧光定量PCR仪基因分型平台上进行,KASP基因分型的实验结果见图6。图中绿色为TC基因型,对应于武夷山、建阳、顺昌、将乐、明溪、永安、华安、德化和新罗的地理种群;红色为TT基因型,包括武夷山、政和、屏南、宁德、福鼎、永泰、德化、新罗和福安的地理种群。对KASP标记分型结果进行统计,基因分型结果与基于线粒体12SrRNA基因片段对野生棘胸蛙的聚类结果一致,说明采用所设计的KASP分型引物,能正确区分福建省野生棘胸蛙的两大支系。对KASP标记分型结果进行统计,与预测结果一致,说明采用本研究所设计的KASP分型引物,能正确区分福建省野生棘胸蛙的两大支系。
3 讨论与结论
Che等[15]利用棘蛙属的核基因和线粒体基因进行系统发生树分析时,提出了一个假设,即棘蛙属这一物种内部可能隐藏着未被识别的隐种,他们的研究揭示了棘蛙属在遗传层面上的复杂性。随后,路庆芳[4]研究通过对13个棘胸蛙地理种群的线粒体DNA细胞色素b基因562bp序列的测定,发现棘胸蛙可以分为4个不同的支系,这4个支系分别与越南、中国西部、中国中部和中国东部的种群相对应。这些支系之间的地理分布互不重叠,且遗传距离较大,接近种间分化水平。Zheng等[16]采用了磁珠富集法筛选了15对多态性较高的微卫星引物。这些引物在来自两个种群的38只棘胸蛙个体中表现出了较高的多态性,显示出它们在棘胸蛙种群遗传结构研究中的潜在价值。郑荣泉[3]利用其中多态性较高的12对微卫星引物,进行了棘胸蛙的遗传结构分析。结果显示,棘胸蛙具有较高的遗传多样性,进一步证实了棘胸蛙种群的遗传多样性。聚类分析和系统发生树分析的结果与线粒体DNA数据基本一致,均支持棘胸蛙存在4个遗传分化单元,这些单元严格符合进化显著单元的条件。黄华[17]在测定国内的棘胸蛙及其近缘种的线粒体16SrRNA和12SrRNA基因序列后,也得出了类似的结论。他认为广泛分布在中国南部的棘胸蛙可能至少包含两个隐存种。这些隐存种在形成过程中可能从西南向东北方向扩散。这些研究为理解棘胸蛙的遗传结构、系统发生以及可能的隐种存在提供了宝贵的见解,揭示了这一物种在进化和遗传上的复杂性。
由于福建省多山,而棘胸蛙属于两栖类,其扩散能力有限,容易受历史地质因素的影响,而产生遗传上的谱系地理分歧演化。因此,福建省内野生的棘胸蛙种群也可能存在隐存种。本研究通过采集了福建省内多个地区的野生棘胸蛙样本,进行了遗传多样性研究。结果显示,基于棘胸蛙的12SrRNA基因片段所构建的NJ系统发生树可以把福建省野生棘胸蛙种群分为两大支系:一支包括武夷山、建阳、顺昌、将乐、明溪、永安、新罗、德化和华安的地理种群;另一支包括武夷山、政和、新罗、永泰、德化、屏南、福安、宁德和福鼎的地理种群。其中武夷山、德化、新罗样品在两个大支系中均有分布。这两个支系已经在遗传上产生了分化,因此可能是隐存种。通过对于地区的地貌特征查看,这两种福建省内野生棘胸蛙的隐存种的分布趋势可能与山脉形成有关。最后,为了对福建省的野生棘胸蛙进行两大支系的判断更加便捷和低成本,本研究基于福建省野生棘胸蛙两个支系在线粒体12SrRNA基因上的差异位点,开发竞争性等位基因特异性PCR(KASP)的分型方法。研究结果显示,采用本研究所设计的KASP分型引物,能正确区分福建省野生棘胸蛙的两个支系。与传统的PCR扩增测序方法相比,KASP标记利用特异荧光引物,对目标SNP位点进行精准的扩增,通过扫描荧光信号进行基因型分型,不需要进行电泳、照相和读带分析,具有便捷、快速的特点。本研究基于KASP所建立的方法具有操作简单、时间短、成本低的优点,可应用于大规模样品的检测。本研究成果有助于解析福建省内野生棘胸蛙不同种群间的遗传差异,也有助于建立棘胸蛙的遗传资源库,为未来的遗传育种、种质资源保护以及可持续利用提供基础数据。
参考文献:
[1]曾占壮,张新艳,王剑锋,等.福建省养殖棘胸蛙主要病害调查[J].渔业研究,2020,42(3):269−276.
[ 2] 王茂元,赖铭勇,黄洪贵,等.闽西山区养殖棘胸蛙两性异 形[J].渔业研究,2020,41(5):409−417.
[ 3] 郑荣泉.棘胸蛙体大小、热生理学变异及系统地理学研究[D]. 南京:南京师范大学,2009.
[ 4] 路庆芳.利用线粒体 DNA 分子标记探讨棘胸蛙种群遗传结 构[D].金华:浙江师范大学,2008.
[ 5] 叶容晖.棘胸蛙微卫星分子标记筛选及其种群遗传分析[D].金 华:浙江师范大学,2009.
[ 6] 叶书培.棘蛙属物种存在隐种和渐渗杂交:分子系统地理学证 据[D].金华:浙江师范大学,2013.
[ 7] 杨子萍,李大命,刘燕山,等.基于 Cytb 序列的太湖和洪泽湖翘 嘴 鲌 遗 传 多 样 性 和 遗 传 结 构 分 析 [ J] . 渔 业 研 究 , 2023, 45(1):1−7.
[ 8] FRANCKI M, DURSTEWITZ G, POLLEY A, et al. SNP discoverybyampliconsequencingandmultiplexSNPgenotypingin
[ 9] the allopolyploid species Brassica napus[ J] . Genome, 2010, 53(11):948−956. HE C, HOLME J, ANTHONY J. SNP Genotyping: The KASP Assay[J].Methods in Molecular Biology,2014,1145:75−86.
[ 10] LIC,LIY,XU J,et al. Disease-driven detection ofdifferential inherited SNP modules from SNP network[ J] . Gene, 2011, 489(2):119−129.
[ 11] LOOPV,NILSENG,NORDGARDSH,etal.AnalyzingCancer Samples with SNP Arrays[ J] . Methods in molecular biology (Clifton,NJ),2012,802:57−72.
[ 12] VANDERZANDES,ZHENGP,CAIL,etal.The cherry6+9K SNParray:acost-effectiveimprovementtothecherry6KSNParray forgeneticstudies[J].Scientific Reports,2020,10(1):7613.
[ 13] DRYWA A, POCWIERZ-KOTUS A, DOBOSZ S, et al. Identification of multiple diagnostic SNP loci for differentiation of three salmonid speciesusing SNP-arrays[ J] . Marine Genomics, 2014,15(1):5−6.
[ 14] POECKE R V, MACCAFERRI M, TANG J, et al. Sequencebased SNP genotyping in durum wheat[ J] . Plant Biotechnology Journal,2013,11(7):809−817.
[ 15] CHEJ,HUJS,ZHOUWW,eta1.PhylogenyoftheAsianspiny frog tribe Paini ( Family Dicroglossidae) sensu Dubois[ J] . Molecular Phylogenetics and Evolution,2009,50:59−73.
[ 16] ZHENG R Q, YE R, YU Y, et al. Fifteen polymorphic microsatellite markersforthe giant spiny frog,Paa spinosa[J]. Molecular Ecology Resources,2010,9(1):336−338.
[ 17] 黄华.利用线粒体 DNA 分子标记揭示棘胸蛙中的隐种多样性[D]. 金华:浙江师范大学,2012.
(责任编辑:柯文辉)
