金钗石斛AP2/ERF基因家族的鉴定和表达分析
2024-10-10于相丽刘雅银李勇慧周晓君


摘要:对金钗石斛AP2/ERF转录因子基因家族在全基因组上进行鉴定及生物信息学分析,为进一步阐明金钗石斛AP2/ERF转录因子的功能研究奠定理论基础。利用BLASTP比对金钗石斛蛋白,并通过在线网站NCBI CDD、InterPro和SMART进一步鉴定,共获得97个金钗石斛AP2/ERF基因,然后通过ProtParam分析金钗石斛AP2/ERF家族蛋白的理化性质、亲疏水性,使用MEGA11.0软件构建进化树,利用Map Gene Chrome、GSDS、MEME、PlantCARE在线工具分析AP2/ERF转录因子的基因定位、基因结构和保守基序,并预测基因上游的顺式作用元件,利用TBTools分析并可视化AP2/ERF基因在各部位的表达情况。结果表明,金钗石斛AP2/ERF转录因子家族的成员,大部分定位在细胞核中,相对分子质量介于2 726.88~71 682.81 u之间,氨基酸数量介于94~659个之间,等电点介于4.45~10.44之间。进化树分析显示,AP2/ERF家族成员分为AP2、RAV、ERF及DREB 4个亚家族。97条基因不均匀地分布在19条染色体上,各个亚族都有独特基序,AP2/ERF基因启动子上游区域光响应作用元件种类最多,数量也最多。大多数DnAP2s基因在根中的表达量最大,不同的居群DnAP2s基因表达有较大不同。鉴定得到97个金钗石斛AP2/ERF蛋白,DnAP2s基因在根茎叶花中表达具有差异性,地理分布导致不同居群的遗传多样性水平不同。
关键词:金钗石斛;AP2/ERF基因家族;生物信息学;表达分析
中图分类号:Q943.2;S567.23+9.01 文献标志码:A
文章编号:1002-1302(2024)17-0047-11
收稿日期:2023-08-11
基金支持:国家自然科学基金(编号:31870697)。
作者简介:于相丽(1975—),女,河南漯河人,硕士,讲师,主要从事植物生物技术和生物信息学研究。E-mail:yuxli2001@163.com。
转录因子(transcription factor,TF)是能够与真核基因启动子区域中顺式作用元件发生特异性相互作用的DNA结合蛋白,包含DNA结合区、核定位信号、蛋白质相互作用和转录调控等区域[1]。植物基因组中有很大一部分基因编码转录因子[2],如BBS[3]、NAC[4]、WRKY[5]、LIM[6]、bZIP[7]、MYB[8]等。AP2/ERF基因家族是植物所特有的一类转录因子,该类转录因子参与多种生物学过程,包括植物生长、花发育、果实发育、种子发育、损伤、病菌防御、高盐、干旱等环境胁迫响应等[1],已在簸箕柳(Salix suchowensis)[9]、欧李(Cerasus humilis)[10]、芝麻(Sesamum indicum L.)[11]、欧洲红豆杉(Taxus baccata L.)[12]、绿豆(Kigna radiata L.)[13]、罗布麻(Apocynum venetum L.)[14]、桑树(Morus balba L.)[15]、猕猴桃(Actinidia chinensi Planch)[16]等单双子叶植物中得到鉴定。
金钗石斛(Dendrobium nobile L.)为兰科石斛属草本植物,具有药用、食用和观赏价值。金钗石斛以茎入药,性寒、味苦、淡、微咸,具有增强免疫力、强阴益精、生津养胃、润肺止咳、抗肿瘤等功效[17]。其富含生物碱、多糖等活性物质,因此成为医疗、食品等领域的热门研究对象[18]。除具有较高的药用价值外,金钗石斛花色艳丽,形态优美,有非常高的观赏价值[19-20]。近年来,大量采集和生态破坏导致金钗石斛野生资源稀缺,已被国家列入濒危植物名录加以保护[17]。当前,关于金钗石斛AP2/ERF基因家族的鉴定及表达分析的研究未见报道,本研究在全基因组范围内鉴定金钗石斛AP2/ERF转录因子家族成员和表达模式,旨在为进一步探索金钗石斛AP2/ERF基因功能提供参考。
1 材料与方法
1.1 基因鉴定及序列分析
本试验的研究对象是金钗石斛AP2/ERF家族。于2023年2月到4月在洛阳师范学院生命科学学院植物组织培养实验室(215)进行软件操作及信息处理。拟南芥(Arabidopsis thaliana)AP2/ERF基因家族的蛋白质序列下载于数据库Tair(https://www.arabidopsis.org/index.jsp),金钗石斛基因组文件、蛋白质文件、cds文件、gff文件等下载于NCBI生物信息数据库(http://www.ncbi.nlm.nih.gov/),将拟南芥AP2/ERF蛋白质序列文件作为标准,利用TBtools[21]与金钗石斛蛋白质文件进行Blast比对,得到初次筛选的蛋白质文件。然后利用NCBI CDD(https://www.ncbi.nlm.nih.gov/cdd)、InterPro(http://www.ebi.ac.uk/interpro/)和SMART(https://smart.embl.de/)对初选蛋白质文件进行下一步鉴定。
利用ProtParam(https://web.expasy.org/protparam/)和WolF PSOERT(https://wolfpsort.hgc.jp)分别预测金钗石斛AP2/ERF蛋白质的氨基酸数目、相对分子数量、等电点数据及蛋白质位置,并列表记录。
1.2 系统进化分析
为进一步分析AP2/ERF家族成员的进化特点,利用 MEGA11.0[22]软件进行比对,将金钗石斛与拟南芥AP2/ERF蛋白序列以邻接法(neighbor-joining,NJ)(自展值1 000)构建系统发育进化树。
1.3 转录因子的基因定位
根据gff文件中基因的位置,利用Map Gene Chrome,将AP2/ERF基因定位至染色体上。
1.4 基因结构及保守序列分析
利用GSDS(http://gsds.gao-lab.org/)绘制基因结构图和MEME(http://meme-suite.org/meme/tools/meme)在线工具对金钗石斛基因蛋白序列上的保守基序进行预测。保守基序数量设置为20,其他参数为默认设置,并采用TBtools软件绘制保守基序图。
1.5 顺式作用元件分析
选取启动子上游2 000 bp序列,在线网站Plant CARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)分析金钗石斛AP2/ERF转录因子顺式作用元件,筛选整理后,绘制顺式作用元件图。
1.6 基因表达模式分析
1.6.1 组织特异性分析 从NCBI生物信息数据库获取已公开的金钗石斛不同部位(根、茎、叶、花)表达情况的数据(SRR1503682,SRR1503683,SRR1503684,SRR1503685,SRR1503686,SRR1503687,SRR1503688,SRR1503689,SRR1503691,SRR870475,SRR870484),TBtools 软件分析后取FPKM的 log2(FPKM+1) 值,TBtools工具包可视化Dn-AP2s基因的表达水平。
1.6.2 不同居群金钗石斛中的表达分析 为探讨不同居群金钗石斛AP2/ERF基因表达模式的差异,从NCBI生物信息数据库获取已公开的四川、贵州和云南等10个居群的金钗石斛表达情况数据(PRJNA747120),选取表达量较高的基因,对不同居群的金钗石斛表达量进行比较。TBtools软件分析后取FPKM的log2(FPKM+1)值,TBtools工具包可视化Dn-AP2s的表达水平。
2 结果与分析
2.1 金钗石斛AP2/ERF基因家族鉴定及序列分析
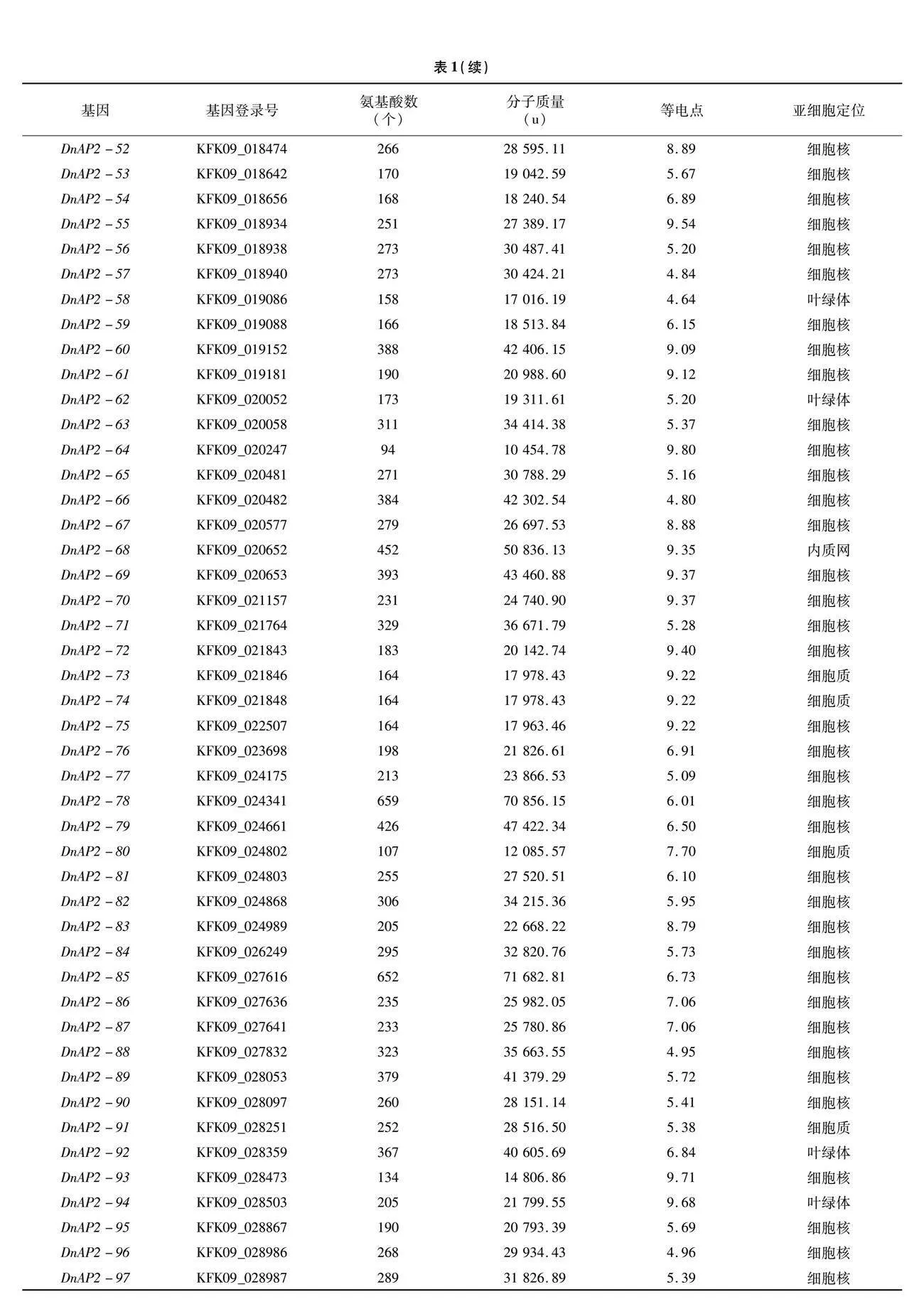
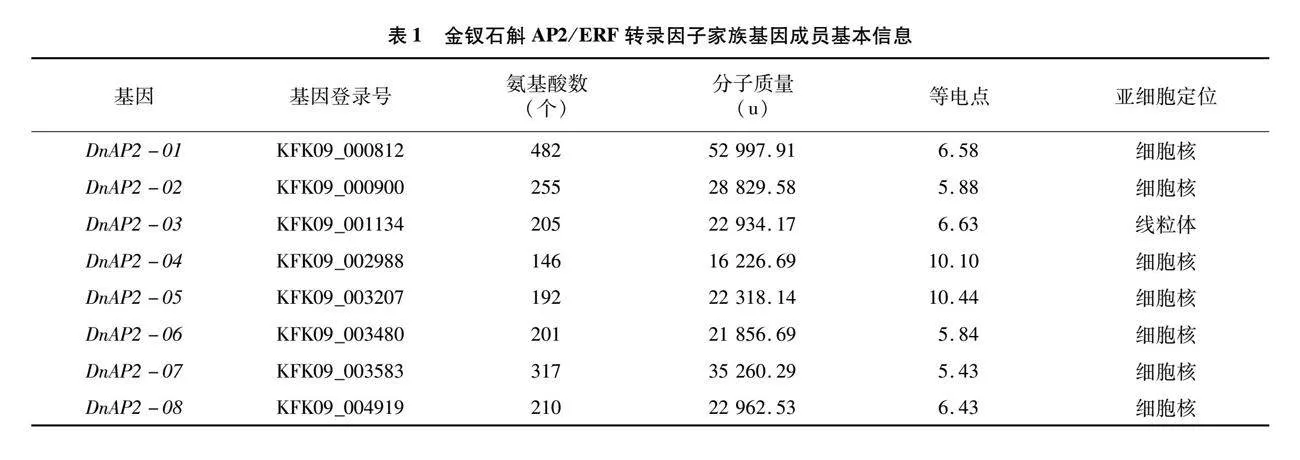
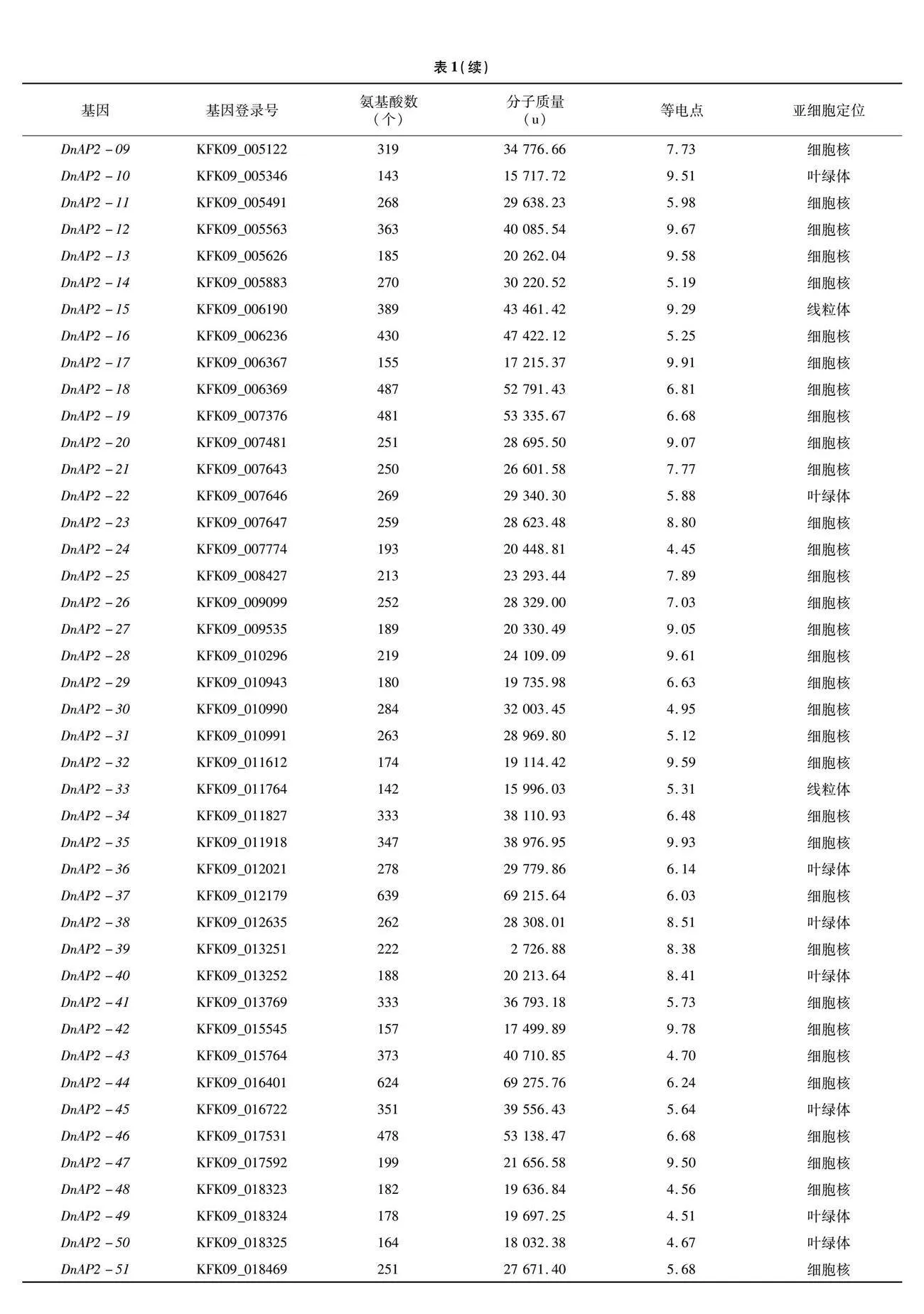
通过BLAST、NCBI CDD、InterPro和SMART鉴定,最终鉴定到97条AP2/ERF基因,按照基因ID从小到大的顺序,依次命名为DnAP2-01~DnAP2-97。从表1可以看出,DnAP2蛋白分子质量介于 2 726.88~71 682.81 u之间,氨基酸数量介于94~659个之间,等电点介于4.45~10.44之间。其中,56个蛋白的等电点小于7,偏酸性;其余41个蛋白的等电点大于7,偏碱性。除了少数定位于叶绿体(12个)、线粒体(3个)和内质网(1个)外,其余金钗石斛AP2/ERF基因家族成员都定位于在细胞核。
2.2 金钗石斛AP2/ERF基因家族系统进化分析
依据AP2/ERF结构域个数及其结构域同源性,拟南芥AP2/ERF转录因子可分为5个亚家族:AP2、ERF、DREB、RAV和Soloist。以拟南芥AP2/ERF转录因子为参照对象,对97个金钗石斛AP2/ERF转录因子进行分类,将其分为4个亚家族:AP2、ERF、DREB、RAV,缺少Soloist(图1)。金钗石斛97个DnAP2s中有48个成员属于ERF亚家族,29个成员属于DREB亚家族,15个成员属AP2亚家族,5个成员属于RAV亚家族。ERF亚家族作为最大的亚家族,可进一步分类为5个组:B1、B2、B3、B5和B6;DREB亚家族可分为5个组:A1、A2、A4、A5和A6。进化树中共有64对旁系同源基因,15对直系同源基因。其中金钗石斛同源基因26对,拟南芥同源基因38对。AP2、RAV亚家族分支非常明显,表明其在很早时就发生了分化,基因序列发生较大分化[16];而DREB和ERF亚家族分布和猕猴桃有所不同,说明与猕猴桃AP2/ERF家族进化途径有所不同。
2.3 金钗石斛AP2/ERF基因定位
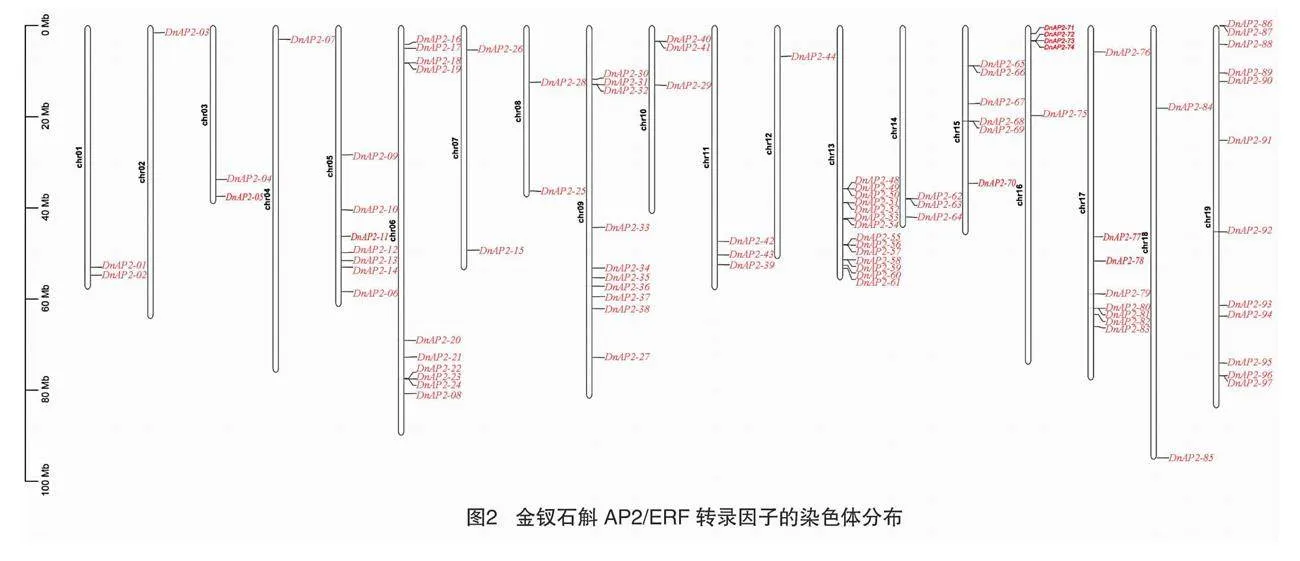
基因定位是指确定基因所属连锁群及基因在染色体上的分布位置,有助于了解基因的作用功能及其之间的相互作用关系[16]。对金钗石斛97个转录因子进行基因定位(图2)发现,在19条染色体上均有AP2/ERF基因的分布,但分布并不均匀。其中以chr13分布最多,为14条,chr4分布最少,仅为1条,chr6和chr9都为10条,chr19为12条。多条染色体上存在基因簇,这些基因簇由串联重复或片段重复构成,如DnAP2-48、DnAP2-49和 DnAP2-50,DnAP2-62和DnAP2-63。
2.4 金钗石斛AP2/ERF基因结构与保守序列分析
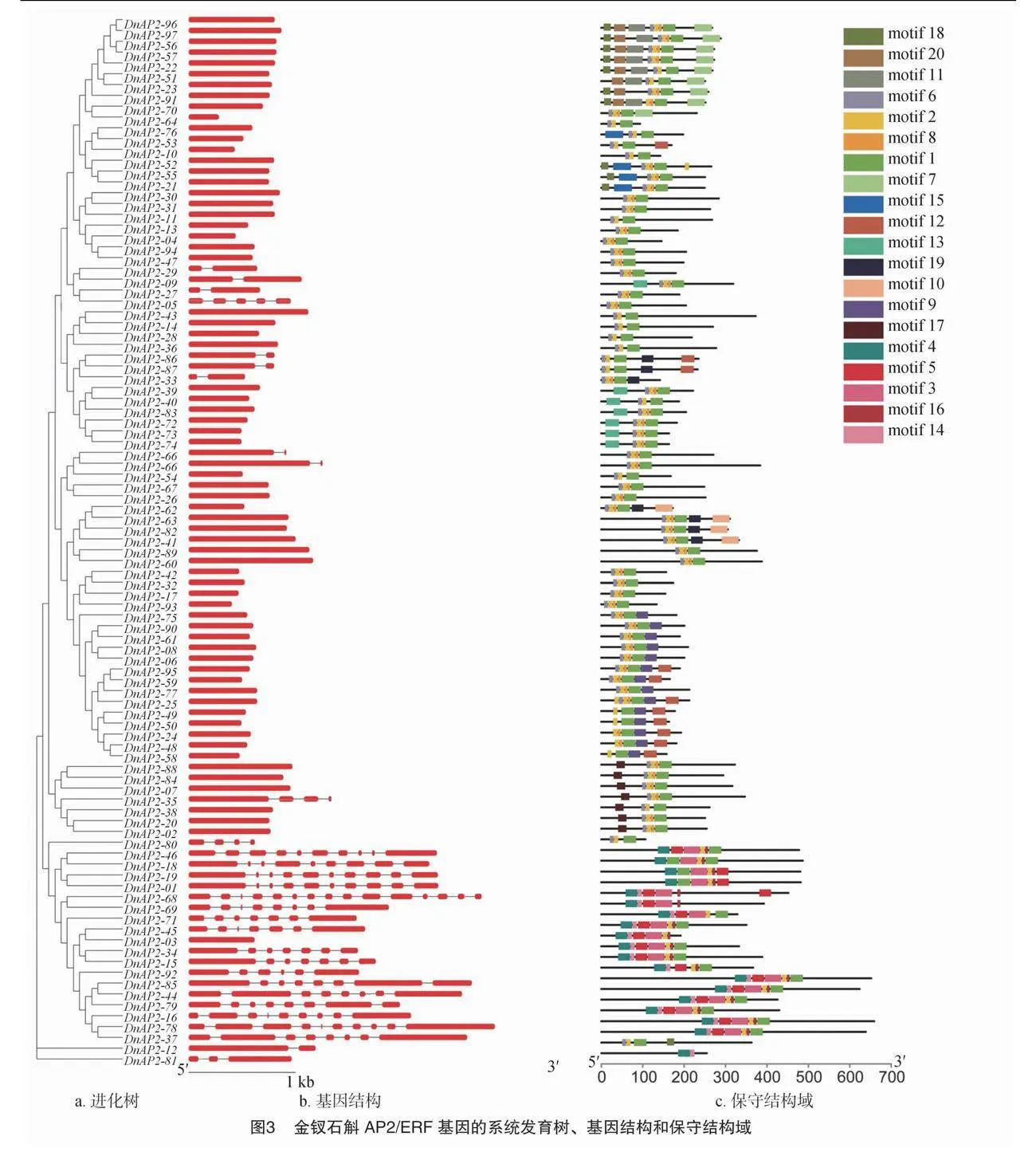
保守序列的分析能够揭示蛋白质的相似性、结构和功能间的关系,为进一步深入地研究生物学功能奠定基础。从金钗石斛AP2/ERF基因结构(图3-b)来看,ERF和DREB这2个亚家族大部分只有外显子,没有内含子。RAV亚家族和AP2亚家族含有大量内含子,如AP2-68,包含14个内含子,而且这2个亚家族的基因长度明显长于ERF亚家族和DREB亚家族,推测较高数量的内含子增加了基因长度,能够提高基因间的重组频率。进一步对金钗石斛AP2/ERF转录因子的保守基序进行分析,结果(图3-c)表明AP2/ERF基因家族均包含motif1、motif2这2个保守基序,而且每个亚家族都有其特有序列。ERF亚家族基本上都含有motif1、motif2和motif6,其中B3组一部分含有motif7、motif8、motif11、motif18和motif20,另外一部分含有motif15;B5组有motif17。DREB亚家族基本上都含有motif1、motif2和motif8,其中A1组和A4组都含有motif9和motif12,A2组和A5组都只有motif8,A6组含有motif10和motif19。RAV亚家族都含有motif1、motif2、motif3、motif4、motif5和motif16,但是DnAP2-12却含有motif1、motif2、motif6和motif18。AP2亚家族都有motif1、motif2、motif3、motif4、motif5、motif14和motif16。
2.5 金钗石斛AP2/ERF基因家族顺式作用元件分析
顺式作用元件是基因的重要组分,能够反映基因潜在的功能和调控途径。对金钗石斛AP2/ERF基因上游2 000 bp序列进行顺式作用元件分析发现,DnAP2基因含有38种主要顺式作用元件,分别参与植物光响应、激素响应、环境胁迫响应和生长发育等过程(图4),例如G-box(331)、TGACG-motif(303)、CGTCA-motif(302)、BOX4(292)、ABRE(295)、ARE(214)、CATA-box(92)。由图4可以看出,参与光响应的主要启动子类型占多数,数量也最多;参与激素应答和植物生长发育的主要启动子类型较多,分别为11和8类;主要参与环境胁迫种类相对较少,只有6类,但数量相对较多。
2.6 金钗石斛AP2/ERF基因家族基因表达模式分析
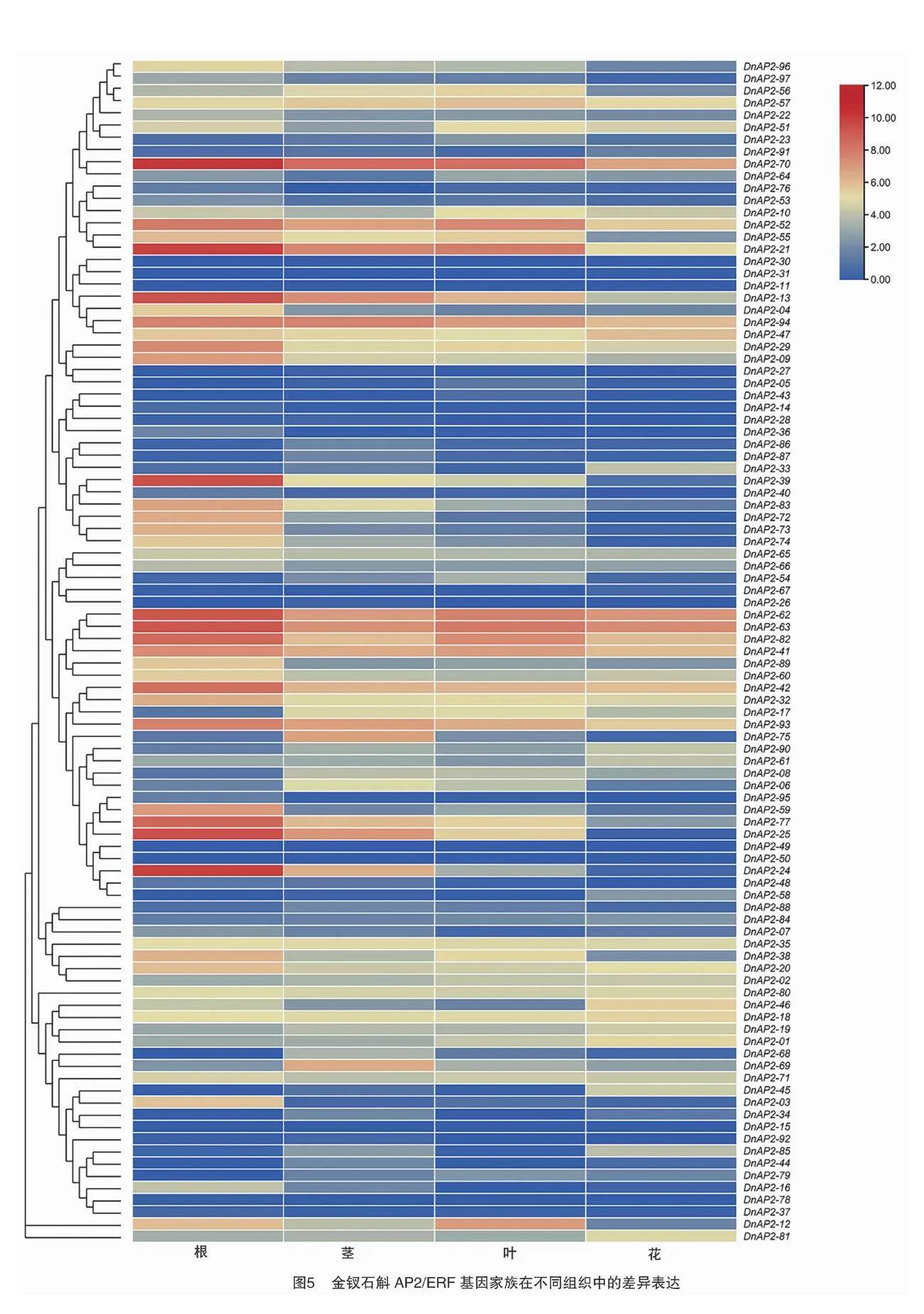
2.6.1 组织特异性分析 利用金钗石斛转录组数据分析DnAP2s在金钗石斛根、茎、叶和花中的表达量结果(图5)表明,DnAP2s基因在根茎叶花中表达具有差异性。DnAP2-11、DnAP2-15、DnAP2-30、DnAP2-49、DnAP2-50等基因在根茎叶花中均未表达,推测在正常条件下,这些基因的功能与组织特异性表达无关。表达量最高的基因DnAP2-13、DnAP2-21、DnAP2-24、DnAP2-25、DnAP2-39、DnAP2-62、DnAP2-63、 DnAP-70和DnAP2-77等均在根中。DnAP2-05、DnAP2-45、 DnAP2-58、DnAP2-68、DnAP2-79等基因中仅在根中不表达,但DnAP2-78仅在根中表达;DnAP2-76仅在茎中不表达,但DnAP2-26、DnAP2-27等基因只在茎中表达;DnAP2-16仅在叶中不表达;在花中不表达的基因较多,有DnAP2-14、DnAP2-29、DnAP2-40、DnAP2-48、DnAP2-72和DnAP2-95,但DnAP2-31只在花中表达,DREB亚家族的B3组在花中表达相对较高,说明B3组DnAP2s可能
参与开花过程。
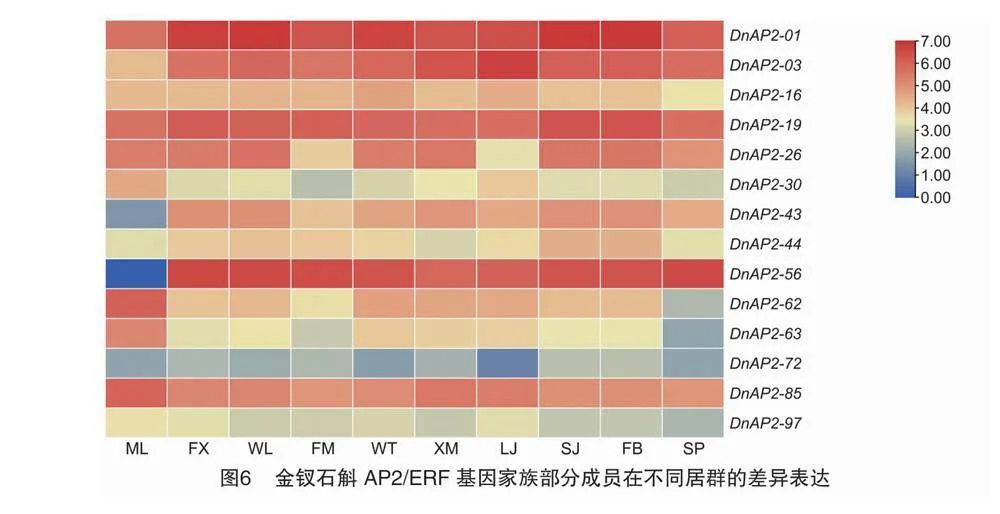
2.6.2 不同居群金钗石斛中的表达分析 为探讨不同居群Dn-AP2s基因表达模式的差异,对来自云南的1个居群(ML)、贵州的2个居群(FX和WL)和四川的7个居群(FM、WT、XM、LJ、SJ、FB、SP)的部分DnAP2基因表达量进行分析比较(图6)。结果显示,所有居群中,DnAP2-01和FDrEkkurac1Bya1KwTmIUvMwANqU6UWyvwaihdaeoxk=DnAP2-56表达量都很高,但DnAP2-56在ML居群中不表达。ML居群和其他居群相比,除了DnAP2-16、DnAP2-26、DnAP2-44、DnAP2-72和DnAP2-97表达和其他居群没有明显区别外,其余DnAP2成员表达量显著高于或低于其他居群;FX和WL这2个居群基因表达量无明显区别,DnAP2s表达更接近于四川居群;来自四川的居群FM的DnAP2-26、
DnAP2-30和DnAP2-62表达量和其他居群有明显区别,LJ居群的 DnAP2-26和DnAP2-72表达量较低,明显区别于其他5个居群,SP居群的DnAP2-16、DnAP2-62、DnAP2-63和DnAP2-72表达量明显低于其他居群,其他4个居群DnAP2s具有相似的表达量,无明显差异。
3 讨论与结论
AP2/ERF转录因子在高等植物的生长发育、应对外界刺激反应以及基因的调控等方面发挥重要的作用[23]。本研究从金钗石斛中共鉴定出97条AP2/ERF家族蛋白,目前不同植物中鉴定出AP2/ERF家族蛋白数目各不相同,绿豆(Vigna radiata L.)87条[13]、新疆野苹果(Malus sieversii)106条[24]、蓝莓(Vaccinium spp.) 160条[25]、油橄榄(Canarium oleosum) 110条[26]、欧洲山杨(Populus tremula) 210条[27],说明不同物种之间AP2/ERF家族蛋白条数存在差异。
对97条金钗石斛蛋白质的理化性质等方面进行研究分析,发现存在很大差异,最短蛋白质只有94个氨基酸,最长蛋白质有659个氨基酸;一部分蛋白质是酸性,一部分是碱性。这些差异在绿豆[13]、罗布麻(Apocynum venetum)[14]、桑(Morus alba L.)[15]、猕猴桃(Actinidia chinensi)[16]等植物中也存在不同,推测其可能决定了各转录因子执行着不同的生物学功能。通过motif蛋白保守基序研究发现,不同类型的AP2/ERF转录因子蛋白序列存在差异,但均包含motif1和motif2这2个保守结构域,这表明 AP2/ERF 转录因子在物种进化过程中高度保守。而每个亚家族各有自己特有的motif,可能是植物在进化过程中为了适应环境而产生了变异[25],使得不同亚家族中保守的氨基酸中存在较大的差异。
金钗石斛AP2/ERF转录因子大部分只含有外显子,并无内含子,在光皮桦[28]、小麦[29]、蓖麻[30]中也出现此现象。RAV亚家族含有内含子和外显子,AP2亚家族除了DnAP2-03无内含子以外,其余基因都含外显子和内含子。成员之间内含子长度、位置和数目存在差异,说明其家族在进化过程中发生了较强的分化,这可能也是导致其成员功能不同的原因[28]。
真核生物的转录过程主要在细胞核中发生,所以转录因子蛋白一般情况下也是定位在细胞核中,这符合其作为转录因子调控下游基因表达的特性[25],有时候转录因子也定位在叶绿体和线粒体中[31]。金钗石斛AP2/ERF转录因子家族蛋白大部分定位在细胞核、线粒体和叶绿体中,只有1个定位在内质网。原始小球藻[31]AP2/ERF转录因子蛋白定位在细胞核、细胞质和细胞外基质;蓝莓[25]和小麦[28]AP2/ERF转录因子蛋白除了定位在细胞质、细胞核、线粒体、叶绿体、质膜和细胞外基质。猕猴桃[16]、新疆野苹果[24]、阿拉伯岩荠[32]、白桦[33]、甘薯[34]等植物中均未发现AP2/ERF转录因子蛋白定位在内质网中。这说明不同植物AP2/ERF转录因子蛋白定位略有差异,可能与该种植物生活方式、生存环境及在AP2/ERF转录因子蛋白植物体内执行的作用和功能有关,具体作用仍需要进一步研究探讨。
金钗石斛AP2/ERF基因家族在不同组织中的表达存在很大差异,有的基因只在一种组织中表达,有的基因只在2种组织中表达,还有的基因只在3种组织中表达,更有部分基因在4种组织中均未表达,这表明DnAP2s表达具有组织特异性。ERF亚家族响应外界的非生物胁迫,拟南芥AtERF49在盐碱胁迫中有响应[35],野生大豆GsERF6在水稻中过表达可提高水稻耐盐[36],棉花GhERF5能响应枯萎病胁迫[37],光皮桦ERF和DREB对高温胁迫有明显响应[28]。金钗石斛ERF亚家族中的DnAP2-11和DnAP2-30在各个组织中均不表达,表明其可能在外界胁迫中发挥作用。
AP2/ERF基因能够调节叶片大小[38],植物枝条和侧根[39],促进水稻不定根发育[40],调控株高[41-42]和花序分生与小穗分生组织[43],参与萼片修饰[44]和唇瓣表皮发育[41],调节生菜种子形状[45],促进果实成熟[46]。AP2/ERF基因功能导致植物具有不同的形状。金钗石斛不同居群之间存在较高的遗传分化,可能是由于地理隔离、分散分布和自然居群之间缺乏交流,在长期进化过程中存在高度的遗传分化[47]。试验采集的样本居群分布在云南低纬度地区、贵州高原赤水河沿岸和四川盆地丘陵地区。如此广泛的地理分布导致不同居群的遗传多样性水平不同。这些群体之间的遗传分化程度不同可能与它们对不同环境的适应能力有关[47]。
参考文献:
[1]Zhang K,Jiang L,Wang X,et al. Transcriptome-wide analysis of AP2/ERF transcription factors involved in regulating taxol biosynthesis in Taxus×media[J]. Industrial Crops and Products,2021,171:113972.
[2]郭 慧,金司阳,刘 寒,等. 甘蓝AP2/ERF转录因子的克隆和生物信息学分析[J]. 中国药师,2017,20(1):6-10.
[3]何基泽,龙小琴,聂 聪,等. 铁皮石斛BBS基因家族生物信息学及表达分析[J]. 基因组学与应用生物学,2022,41(6):1329-1338.
[4]郭怡婷,赵文菊,任延靖,等. 菊芋NAC转录因子家族基因的鉴定及分析[J]. 生物技术通报,2023,39(6):217-232.
[5]水德聚,孙 继,熊自立,等. 番茄WRKY转录因子比较鉴定及细菌胁迫响应[J]. 福建农业学报,2023,38(3):281-293.
[6]陈奕博,杨万明,岳爱琴,等. 大豆LIM转录因子家族鉴定及盐胁迫响应分析[J]. 中国农业大学学报,2023,28(4):26-40.
[7]曹丽茹,马晨晨,庞芸芸,等. 玉米bZIP转录因子ZmbZIP26的分子特征、亚细胞定位及响应非生物胁迫的表达分析[J]. 华北农学报,2023,38(4):1-10.
[8]李 芳,唐秀华,张彦锋,等. 甘蓝型油菜R2/R3型MYB转录因子BnPAP2a介导花青素累积研究[J]. 西北农林科技大学学报(自然科学版),2023,51(7):35-44.
[9]田应明,王皆恒,沈方圆,等. 簸箕柳AP2/ERF家族基因的全基因组分析[J]. 四川大学学报(自然科学版),2019,56(5):983-988.
[10]张晓旭,郭彩珍,穆霄鹏,等. 欧李AP2基因家族的鉴定与表达分析[J]. 分子植物育种,2022,20(7):2162-2169.
[11]赵冰雪,聂功平,张小惠,等. 芝麻 AP2/ERF基因家族生物信息学鉴定与分析[J]. 应用技术学报,2022,22(4):399-412.
[12]杨艳芳,刘洪伟,邱德有. 欧洲红豆杉AP2/ERF转录因子基因序列分析[J]. 氨基酸和生物资源,2014,36(4):54-59.
[13]陈 珂,张 君,刘嘉斐,等. 绿豆AP2/ERF转录因子家族的生物信息学鉴定与特征分析[J]. 分子植物育种,2020,18(20):6605-6617.
[14]谢博勋,李国旗,解 盛,等. 罗布麻AP2/ERF转录因子全基因组鉴定及生物信息学分析[J]. 基因组学与应用生物学,2022,41(1):159-174.
[15]董亚茹,李茹霞,赵东晓,等. 7个桑树AP2/ERF转录因子的生物信息学及表达分析[J]. 山东农业科学,2022,54(1):7-13.
[16]邓 浪,沈兵琪,王连春,等. ‘红阳’猕猴桃全基因组AP2/EREBP转录因子生物信息学分析[J]. 果树学报,2017,34(7):790-805.
[17]张本厚,胡燕花,牛志韬,等. 基于表型性状的金钗石斛种质资源多样性评价[J]. 中国生物工程杂志,2022,42(11):5-17.
[18]罗 丹,张朝凤,林 萍,等. 金钗石斛化学成分的研究[J]. 中草药,2006,37(1):36-38.
[19]明兴加,赵纪峰,米本中,等. 基于形态学的金钗、金钗石斛名实续考[J]. 中国中药杂志,2018,43(11):2396-2401.
[20]余玉珠,陆艳柳,陈卫国,等. 12种石斛属植物不同栽培技术及观赏价值研究[J]. 安徽农业科学,2020,48(17):156-157,205.
[21]Chen C J,Chen H,Zhang F Y,et al. TBtools:an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant,2020,13(8):1194-1202.
[22]Kumar S,Nei M,Dudley J,et al. MEGA:a biologist-centric software for evolutionary analysis of DNA and protein sequences[J]. Briefings in Bioinformatics,2008,9(4):299-306.
[23]魏海超,刘 媛,豆明珠,等. 大豆AP2/ERF基因家族的分子进化分析[J]. 植物生理学报,2015,51(10):1706-1718.
[24]赵明奇,刘晓洁,梁玉青,等. 新疆野苹果AP2/ERF转录因子家族鉴定与响应腐烂病的表达分析[J]. 西北植物学报,2022,42(6):930-942.
[25]马 芮,李瑞雪,郑煜聆,等. 蓝莓AP2/ERF基因家族鉴定及其在休眠解除过程中的表达分析研究[J]. 浙江农业科学,2022,63(12):2854-2864.
[26]乔永刚,陈 亮,崔芬芬,等. 基于转录组金银花AP2基因家族的生物信息学及表达分析[J]. 核农学报,2019,33(9):1698-1706.
[27]赵金玲,姚文静,王升级,等. 杨树AP2/ERF转录因子家族生物信息学分析[J]. 东北林业大学学报,2015,43(10):21-29.
[28]黄奕孜,钱 旺,邱 姗,等. 光皮桦AP2/ERF基因家族鉴定与表达分析[J]. 浙江农林大学学报,2022,39(6):1183-1193.
[29]卞云迪,张 驰,王雪晴,等.小麦 AP2/ERF转录因子家族生物信息学分析[J]. 天津师范大学学报(自然科学版),2022,42(4):39-45.
[30]闫星伊,风 兰,韩雯毓,等. 蓖麻AP2/ERF转录因子家族的生物信息学分析[J]. 分子植物育种,2020,18(4):1088-1102.
[31]付 春,唐 易,江 纳,等. 原始小球藻AP2转录因子的生物信息学分析[J]. 广西植物,2020,40(12):1800-1815.
[32]付 春,刘晓伟,王 玲,等. 阿拉伯岩芥AP2基因家族的生物信息学分析[J]. 安徽农业科学,2020,48(8):114-123.
[33]张文慧,黄 勇,贺登美,等. 白桦AP2/ERF家族基因的生物信息学分析[J]. 西部林业科学,2020,49(6):112-117.
[34]阮先乐,张 杰,张福丽,等. 甘薯AP2基因家族的生物信息学分析[J]. 分子植物育种,2017,15(6):2066-2072.
[35]阮 航,多浩源,范文艳,等. AtERF49在拟南芥应答盐碱胁迫中的作用[J]. 生物技术通报,2023,39(1):150-156.
[36]才晓溪,胡冰霜,沈 阳,等. GsERF6基因过表达对水稻耐盐碱性的影响[J]. 作物学报,2023,49(2):561-569.
[37]赵曾强,郭文婷,张 析,等. 棉花抗枯萎病相关基因GhERF5-4D的克隆及功能分析[J]. 生物技术通报,2022,38(4):193-201.
[38]Marsch-Martinez N,Greco R,Becker J D,et al. BOLITA,an Arabidopsis AP2/ERF-like transcription factor that affects cell expansion and proliferation/differentiation pathways[J]. Plant Molecular Biology,2006,62(6):825-843.
[39]Nie J,Wen C,Xi L,et al. The AP2/ERF transcription factor CmERF053 of chrysanthemum positively regulates shoot branching,lateral root,and drought tolerance[J]. Plant Cell Reports,2018,37(7):1049-1060.
[40]Neogy A,Garg T,Kumar A,et al. Genome-wide transcript profiling reveals an auxin-responsive transcription factor,OsAP2/ERF-40,promoting rice adventitious root development[J]. Plant Cell Physiology,2019,60(10):2343-2355.
[41]Ma Z M,Wu T,Huang K,et al. A novel AP2/ERF transcription factor,OsRPH1,negatively regulates plant height in rice[J]. Frontiers in Plant Science,2020(11):709.
[42]Qi W W,Sun F,Wang Q J,et al. Rice ethylene-response AP2/ERF factor OsEATB restricts internode elongation by down-regulating a gibberellin biosynthetic gene[J]. Plant Physiology,2011,157(1):216-228.
[43]Chuck G,Meeley R,Hake S. Floral meristem initiation and meristem cell fate are regulated by the maize AP2 genes ids1 and sid1[J]. Development,2008,135(18):3013-3019.
[44]Lai P H,Huang L M,Pan Z J,et al. PeERF1,a SHINE-like transcription factor,is involved in nanoridge development on lip epidermis of Phalaenopsis flowers[J]. Frontiers in Plant Science,2020(10):1709.
[45]Luo C,Wang S L,Ning K,et al. The APETALA2 transcription factor LsAP2 regulates seed shape in lettuce[J]. Journal of Experimental Botany,2021,72(7):2463-2476.
[46]Zhang Z J,Zhang H W,Quan R D,et al. Transcriptional regulation of the ethylene response factor LeERF2 in the expression of ethylene biosynthesis genes controls ethylene production in tomato and tobacco[J]. Plant Physiology,2009,150(1):365-377.
[47]Ye M R,Liu W,Xue Q Y,et al. Phylogeography of endangered Dendrobium moniliforme in East Asia based on mitochondrial DNA sequence variations[J]. Biodiversity & Conservation,2017,26(7):1659-1674.
