玉米GRAS 基因家族的全基因组鉴定及生物信息学分析
2024-06-16吴占清陈威赵展许海良李豪远彭星星陈东旭张明月
吴占清 陈威 赵展 许海良 李豪远 彭星星 陈东旭 张明月
摘要:GRAS 基因家族是植物中广泛存在的一类转录因子,在植物生长发育、生物和非生物逆境胁迫、光信号和激素信号应答等多个过程中发挥重要作用。对玉米GRAS 基因家族成员的理化性质、染色体定位、系统发育、顺式作用元件等特征进行了分析。结果表明,在玉米全基因组中共鉴定出49个ZmGRAS 基因,不均匀地分布于1~10号染色体上,编码蛋白质理化性质差异较大,可能在不同的微环境下发挥作用。系统进化分析将GRAS蛋白分为8个亚家族,可能在调节自身生长发育、逆境应答等过程中具有重要作用。玉米GRAS 基因家族的启动子区含有激素应答、光响应、胁迫应答等多种顺式作用元件,推测其可能响应激素、胁迫等多种信号。共线性分析显示,具有共线性关系的基因可能是染色体片段复制的结果,且属于同一亚家族,具有相似的结构和功能。研究结果为进一步研究玉米GRAS 基因的功能和逆境胁迫响应机制提供了依据。
关键词:玉米;GRAS 基因家族;全基因组鉴定;生物信息学;分析
doi:10.13304/j.nykjdb.2023.0551
中图分类号:S513 文献标志码:A 文章编号:1008‐0864(2024)03‐0015‐11
GRAS转录因子家族广泛存在于植物中,其命名最初来自于3 个已确定的成员:GAI(gibberellic acid insensitive)、RGA(repressor repressorof GA1)和SCR(scarecrow)[1]。GRAS转录因子在植物生长发育和信号应答等过程中起重要作用,过表达SiGRAS40能够提高番茄对干旱和盐胁迫的耐受性[2];玉米ZmGRAS20 过表达能够引起淀粉含量下降[3];拟南芥BrLAS过表达显著增强干旱胁迫[4]。
典型的GRAS蛋白通常包含400~770个氨基酸,主要分布在细胞核内,其N端区域可变,C端区域高度保守。C端包含5个不同的序列基序:LHRI(leucine heptapeptide repeatⅠ)、VHIID、LHRⅡ(leucine heptapeptide repeat Ⅱ)、PFYRE 和SAW。保守的C端区域对于蛋白互作具有重要作用,而可变的N端区域主要参与植物发育过程中蛋白的特异性调节和分子识别[1,5‐6]。在早期研究中,根据拟南芥中GRAS蛋白的结构域位置分为7个亚家族:SCR、DELLA、Ls、SHR、HAM、PAT1 和SCL9[6]。GRAS蛋白的亚家族分类在不同物种中也有所不同,不同亚家族GRAS转录因子具有不同功能。Sun等[7]总结了不同物种GRAS转录因子10个亚家族所具有的主要生物学功能,其中,矮牵牛HAM亚家族的功能主要是维持茎生长;拟南芥SCR和SHR亚家族主要参与放射状根的形态发育和生长;水稻DLT具有参与油菜素内酯信号转导的作用;毛白杨SHR亚家族具有提高生长率的作用;LAS亚家族具有促进番茄腋生分生组织形成的作用;麝香百合LISCL亚家族参与调节小孢子形成过程中的转录;拟南芥SCL3亚家族在根细胞伸长过程中整合多个信号;拟南芥和短柄草DELLA亚家族的作用分别是调节茉莉酸信号和通过GA信号途径调节植物生长;拟南芥、大豆、马铃薯中的PAT1亚家族参与干旱、盐胁迫和激素应答等。目前,已经对大麦、甜瓜、绿豆、谷子等多个物种的GRAS 基因家族进行了全基因组鉴定和生物信息学分析,都表明GRAS 基因家族在植物的生长发育、信号转导、响应生物和非生物逆境胁迫等方面具有重要的作用。孔豆豆等[8]在大麦基因组中共鉴定出41条GRAS 基因序列,并发现部分基因在大麦发育阶段起重要作用。郑玲等[9]在甜瓜中鉴定出37个GRAS 基因,对GRAS 基因的基本信息、共线性等进行了全面分析,并发现GRAS 基因不同亚族共同参与调控甜瓜发育过程。张文慧等[10]在绿豆中共鉴定出58个GRAS 基因,通过对这些基因进行全基因组分析和非生物胁迫下的表达模式分析发现,绿豆GRAS 基因在叶片中能够不同程度的响应盐、干旱、碱和低温等非生物胁迫,且PAT1亚家族基因对绿豆响应逆境胁迫起重要调控作用。王智兰等[11]对谷子GRAS 转录因子家族进行全基因组鉴定后发现,谷子全基因组共包含52个GRAS 转录因子基因,各亚族基因具有明显的组织表达特异性。
玉米是我国重要的粮食、经济和饲料兼用作物,本研究利用最新的玉米基因组数据库获得GRAS 家族基因,从理化性质、系统进化关系、基因结构、保守结构域、启动子顺式作用元件、共线性等方面进行系统分析,为进一步探讨玉米GRAS基因的功能提供依据。
1 材料与方法
1.1 玉米GRAS 基因家族成员的全基因组鉴定与染色体定位
玉米基因组数据集(Zm-B73-REFERENCENAM-5.0)来源于EnsemblPlants 数据库(https://plants.ensembl.org/index.html)。从InterPro数据库(https://www.ebi.ac.uk/interpro/)下载GRAS蛋白结构域的隐马可夫模型,并利用hmmsearch扫描玉米蛋白质数据集。获得的GRAS 基因家族成员分别通过SMART 蛋白数据库(SMART: Main page(embl-heidelberg.de))和NCBI-CDD(Home-ConservedDomains-NCBI (nih.gov))网站进行确认,筛选在2个数据库中均包含的GRAS 家族成员并命名。利用ProtParam(https://web.expasy.org/protparam/)对候选GRAS 家族成员的蛋白质分子量(molecularweight,MW)、等电点(isoelectric point,pI)、氨基酸长度、带负电荷残基总数、带正电荷残基总数、不稳定指数、脂肪族氨基酸指数、平均疏水性(grandaverage of hydropathicity,GRAVY)等指标进行分析。利用MG2C_v2.1 在线软件(http://mg2c.iask.in/mg2c_v2.1/)对已获取的候选GRAS 基因进行染色体定位。
1.2 玉米GRAS 基因家族的系统进化树构建
从EnsemblPlants数据库(http://plants.ensembl.org/Arabidopsis_thaliana/Info/Index)分别下载拟南芥、水稻、普通小麦和番茄4个物种的GRAS氨基酸序列,利用MEGA7内置的 ClustalW,采用邻接法(Neighbor Joining, NJ)进行多序列比对,将自展法系数(Booststrap) 设置为1 000次重复,替代模型(substitution model)为p-distance,使用的数据子集为部分删除(partial deletion),位点覆盖率(sitecoverage cutoff)为50%,其他参数采用默认值,构建系统进化树,并利用ITOL软件(https://itol.embl.de/)进行展示。
1.3 玉米GRAS 基因家族编码蛋白保守基序与基因结构分析
利用MEME 5.5.0 在线工具(https://memesuite.org/meme/tools/meme)对玉米GRAS蛋白的保守基序进行分析,设置最大检索保守基序数量为10,其他参数默认。利用MEGA7.0对候选GRAS家族成员间的进化关系进行分析,并采用TBtools对候选GRAS家族成员的保守基序和基因结构进行可视化。
1.4 玉米GRAS 基因家族的顺式作用元件预测利用 TBtools 软件提取玉米候选GRAS 家族基因转录起始位点上游2 000 bp 的序列。利用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对已获得的玉米GRAS 基因家族成员启动子区的顺式作用元件进行预测分析,并通过TBtools软件对预测的结果进行可视化。
1.5 玉米GRAS 基因家族的共线性分析
利用TBtools 软件的Advanced Circos 功能对玉米基因组内的GRAS 基因进行共线性分析并可视化。
2 结果与分析
2.1 玉米GRAS 基因家族成员的理化性质和染色体分布
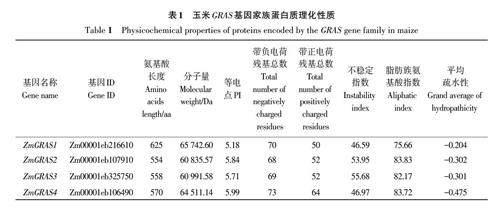
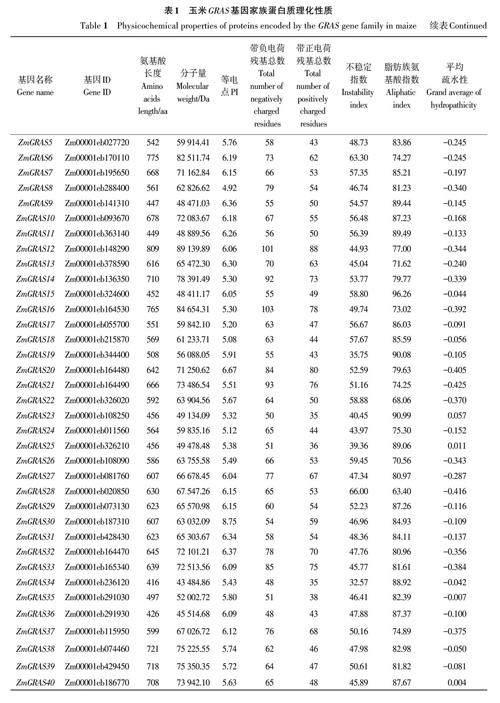
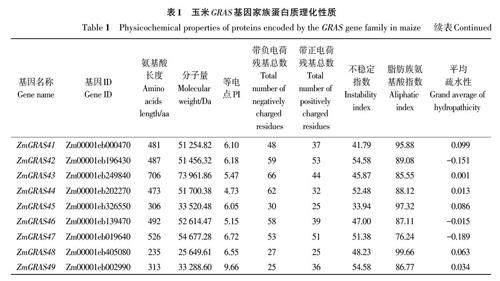
玉米全基因组共鉴定出49 个GRAS 家族成员,分别命名为ZmGRAS1~ZmGRAS49(表1),编码氨基酸长度在235~809 aa,平均长度568 aa;分子量变化范围为25 649.61~89 139.89 Da,平均61 335.45 Da;理论等电点为4.73~9.66;带负电荷残基总数为25~103,带正电荷残基总数为25~88;不稳定指数为32.57~66.00;脂肪族氨基酸指数为63.40~99.66;平均疏水性为-0.475~0.099,表明不同玉米GRAS蛋白理化性质差异较大,且可能在不同的微环境下发挥作用。
染色体定位结果(图1)显示,ZmGRAS 家族成员分布于1~10号染色体上,其中4号染色体上数目最多,有11个ZmGRAS 基因家族成员;2号染色体上数目为9 个;9 号染色体上数目最少,只有1个成员为ZmGRAS13;8号染色体上有2个成员,分别是ZmGRAS11 和ZmGRAS19,6号和10号染色体上均有3个成员;3号和5号染色体上均有4个,1号和7号染色体上分别有7个和5个成员。这些结果表明,玉米ZmGRAS 基因家族成员在染色体上呈不均匀分布,且在染色体两端分布的数量较多。
2.2 GRAS 家族成员在不同物种间的系统发育分析
为了探究玉米ZmGRASs的功能和进化关系,利用MEGA7及其内置的ClustalW对24个拟南芥AtGRASs、43 个水稻OsGRASs、27 个普通小麦TaGRASs、10 个番茄SlGRASs 和49 个玉米ZmGRASs氨基酸序列进行系统进化分析。结果(图2)表明,5个物种的GRAS蛋白可被分成8个亚家族,分别为LAS、DELLA、SCR、SCL3、PAT、HAM、SHR和LISCL,几乎每个物种都包含这些亚家族,表明不同物种亚家族成员的功能比较保守,LAS、DELLA 亚族中均含有3 个玉米ZmGRAS 成员,SCL3、SCR、SHR、PAT和LISCL亚族中分别含有4、5、7、8和9个ZmGRAS成员,HAM亚族中的数量最多,含有10个ZmGRAS成员。已有研究证明,不同亚家族具有调控植物根、茎、腋芽等的生长发育,参与赤霉素、光信号、油菜素内酯等信号转导通路,响应植物对干旱、盐胁迫等重要作用[7],推测玉米不同亚家族的ZmGRAS 蛋白可能在调节自身生长发育、逆境应答等过程中具有重要的作用。
2.3 玉米ZmGRAS 基因家族保守结构域和基因结构分析
利用MEGA7.0、MEME、TBtools软件分别对玉米ZmGRASs 编码蛋白的保守基序与基因结构进行分析(图3)。
蛋白保守基序结果显示,大多数成员都含有从Motif1到Motif10的10个Motifs,同一亚族具有相似的蛋白保守基序排列,而有些成员缺少1或多个,如在LISCL 亚家族中,ZmGRAS21 没有Motif1,ZmGRAS37 没有Motif8 和Motif10;在SHR亚家族中,ZmGRAS22、ZmGRAS26、ZmGRAS28均没有Motif10;在HAM 亚家族中,ZmGRAS40、ZmGRAS43、ZmGRAS38 和ZmGRAS39 也均没有Motif10,而ZmGRAS41 没有Motif10、Motif2 和Motif4,ZmGRAS49 只有Motif3、Motif8 和Motif5;ZmGRAS48 有Motif3、Motif5、Motif6、Motif9 和Motif10,存在于SCL3亚家族中。总的来看,同一亚家族中的各成员具有相似的保守域,不同亚族成员间的保守域数量和组成差异较大,而且个别亚族内不同ZmGRAS蛋白序列结构也存在差异,这可能与不同亚家族的基因存在不同的功能有关。
基因结构分析结果显示,49个ZmGRAS 家族成员的外显子数均只有1、2 和3 个,其中ZmGRAS7、ZmGRAS10、ZmGRAS16、ZmGRAS33、ZmGRAS42 和ZmGRAS45 均含有2 个外显子,ZmGRAS6 和ZmGRAS8 均含有3个外显子,而其他成员的外显子数均为1,说明玉米ZmGRAS 家族成员的基因结构整体比较简单,同一亚族内基因结构相似,结合保守结构域分析结果,同一亚家族的基因可能具有相似的功能。
2.4 玉米ZmGRASs 家族成员启动子顺式作用元件分析
为了进一步研究玉米ZmGRAS 家族成员启动子中可能的顺式作用元件及其作用,利用TBtools 软件获取玉米ZmGRASs 起始位点上游2 000 bp 的序列,并对其顺式作用元件进行分析,结果(图4)表明,玉米ZmGRASs 启动子区主要含有激素应答、光响应、胁迫应答等多种顺式作用元件,其中,所有成员中均含有光应答相关的响应元件;除了ZmGRAS5、ZmGRAS11、ZmGRAS14、ZmGRAS28、ZmGRAS45 外,其余成员中均含有茉莉酸甲酯应答相关的响应元件;ZmGRAS11~ZmGRAS14、ZmGRAS17、ZmGRAS18等28 个成员中含有生长素应答元件;除了ZmGRAS5、ZmGRAS17 外,所有成员中均含有脱落酸响应元件;ZmGRAS2、ZmGRAS4、ZmGRAS6、ZmGRAS7、ZmGRAS10 等26个成员中含有干旱诱导的应答元件;ZmGRAS1、ZmGRAS2、ZmGRAS4、ZmGRAS6、ZmGRAS7 等26个成员中含有赤霉素应答元件; ZmGRAS1、ZmGRAS2、ZmGRAS7、ZmGRAS9、ZmGRAS10 等27个成员中含有低温应答元件; ZmGRAS1、ZmGRAS2、ZmGRAS4、ZmGRAS5、ZmGRAS6 等41个成员中均含有防御和应激响应元件;说明玉米ZmGRAS基因家族可能响应激素、胁迫等多种信号。
2.5 玉米基因组内ZmGRASs 共线性分析
利用TBtools 对玉米基因组内的ZmGRAS 基因进行共线性分析(图5),共有19 对基因具有共线性关系,存在共线性的基因中有9 个位于2 号染色体上,11 个位于4 号染色体上,分别与这2条染色体上ZmGRAS 基因分布的数量一致,说明这2 条染色体上的ZmGRAS 基因可能是片段重复的扩增;部分ZmGRAS 基因与多个基因存在共线性关系,如位于1 号染色体的ZmGRAS28分别与2号染色体的ZmGRAS26 和7号染色体的ZmGRAS22 存在共线性,结合进化分析结果,这3 个基因均属于SHR 亚家族;位于10 号染色体的ZmGRAS39 分别与2 号染色体的ZmGRAS38、4 号染色体的ZmGRAS40 和5 号染色体的ZmGRAS43 存在共线性,它们均属于HAM 亚家族,说明具有共线性关系的基因可能是染色体片段复制的结果,且属于同一亚家族,具有相似的结构和功能。
3 讨论
不同物种中GRAS 基因数量差异较大,在大麦、甜瓜、绿豆、谷子、水稻和大豆中分别鉴定出41、37、58、52、57 和117 个GRAS 基因[8-13],本研究利用玉米最新基因组共鉴定出49个GRAS 基因。本研究中玉米GRAS 基因家族成员编码蛋白的氨基酸数量变化范围为235~809 aa,与大麦、甜瓜、绿豆、谷子等的氨基酸数量变化范围相似,均变化较大;等电点变化范围为4.73~9.66,其中47个小于7,2个大于7,偏酸性蛋白质多,该结果与甜瓜、谷子的基本一致。这些结果表明,不同物种GRAS基因家族成员的理化性质具有相似性。对玉米GRAS 基因家族成员的染色体定位显示49个成员在10条染色体上呈不均匀分布,与大麦、甜瓜等的研究结果一致。
对玉米49 个GRAS 成员的基因结构分析显示,除了14个成员有内含子外,其余35个成员均没有内含子,该结果与甜瓜中多数成员没有内含子一致。玉米49个GRAS 基因家族成员中,37个含有从Motif1 到Motif10 的10 个Motif,与甜瓜中37个CmGRAS 中仅有Motif5和Motif6的结果不一致,与绿豆中的蛋白保守基序结果也有较大差异[9‐10];玉米同一亚族具有相似的蛋白保守基序排列,与绿豆、谷子结果一致[10‐11],说明不同物种蛋白保守基序有差异,而玉米GRAS蛋白基序相对较保守。系统发育分析对于研究物种的进化关系具有十分重要的意义,分析基因家族的起源和进化关系有利于进一步研究其功能[14-18]。本研究对拟南芥、水稻、普通小麦、番茄、玉米5 个物种共153个GRAS 家族基因进行系统发育分析,发现玉米GRAS 基因在8个亚家族中呈不均匀分布,与甜瓜等物种类似[9]。
GRAS家族作为植物体内一类重要的转录因子,参与植物体内生长和发育的多个生物过程[5],包括赤霉素信号转导[9,19-21]、茎顶端分生组织(shoot apical meristem, SAM)形成[22]、雄性配子体发生[23]、光敏色素A信号转导[24]、生物和非生物抗逆性[3‐4,8,25]、胚乳细胞发育[26‐27] 等。甜瓜中的DELLA亚族通过赤霉素信号传导途径调控甜瓜生长[9],拟南芥的GAI和RGA负调控赤霉素信号转导[19-21];矮牵牛中的HAM介导来自外侧器官原基和茎前脉管系统的信号,该信号对于维持SAM是必要和特异的[22];百合花药中的LISCL参与小孢子发生过程中的转录调控[23];在柳枝稷中,PVGRAS17 和PVGRAS103 基因可分别用于耐干旱和耐盐育种[3],拟南芥中的PAT1亚族在光信号转导中有重要的调控作用[24],BrLAS 过表达的植株抗旱性显著增强[4],绿豆GRAS 基因能够响应干旱、碱和低温胁迫,且干旱胁迫和碱胁迫的表达模式存在相似性[10];在玉米中,过表达ZmGRAS11 导致胚乳细胞变大,从而增加籽粒大小和重量,与此相一致的是,ZmGRAS11 在胚乳充盈期正调控ZmEXPB12 的表达,而ZmEXPB12 是细胞扩增所必需的[26],O2-ZmGRAS11模块介导籽粒灌浆过程中胚乳的增效膨大[27]。在本研究中,顺式作用元件分析显示,玉米GRAS 基因家族启动子区主要含有激素、光、非生物逆境胁迫等响应元件,其中激素应答顺式作用元件主要包括茉莉酸甲酯、生长素、脱落酸、赤霉素、水杨酸等,非生物逆境胁迫应答主要是干旱、低温胁迫应答,该结果与绿豆和谷子中的GRAS 基因家族启动子区也包含茉莉酸甲酯、生长素、脱落酸激素应答元件一致,而绿豆中还含有乙烯响应元件,与甜瓜GRAS 基因家族启动子区也包含低温和干旱胁迫应答元件一致[9-11],推测玉米GRAS基因家族可能在激素、干旱、低温等生物和非生物逆境胁迫应答中起重要作用。此外,本研究在玉米基因组内发现19对GRAS 基因存在串联重复和片段复制,比甜瓜中的28对共线性基因数量少[9]。总的来说,对玉米GRAS基因的全基因组鉴定和生物信息学分析为进一步探究其功能以及响应生物和非生物逆境胁迫的分子机制奠定重要基础。
参考文献
[1] HOU S, ZHANG Q, CHEN J, et al .. Genome-wide identi fi
cation and analysis of the GRAS transcription factor gene
family in Theobroma cacao [J/OL]. Genes (Basel), 2022, 14(1):
57 [2023-09-03]. https://doi.org/10.3390/genes14010057.
[2] LIU Y, HUANG W, XIAN Z, et al .. Overexpression of
SIGRAS40 in tomato enhances tolerance to abiotic stresses and
influences auxin and gibberellin signaling [J/OL]. Front. Plant
Sci., 2017, 8:1659 [2023-09-03]. https://doi.org/10.3389/fpls.
2017.01659.
[3] WANG X, LI G, SUN Y, et al .. Genome-wide analysis and
characterization of GRAS family in switchgrass [J]. Bioengineered,
2021, 12(1):6096-6114.
[4] LI P, ZHANG B, SU T, et al .. BrLAS, a GRAS transcription
factor from Brassica rapa, is involved in drought stress
tolerance in transgenic arabidopsis [J/OL]. Front. Plant Sci., 2018,
9:1792 [2023-09-03]. https://doi.org/10.3389/fpls.2018.01792.
[5] LU X H, LIU W Q, XIANG C G, et al .. Genome-wide
characterization of GRAS family and their potential roles in
cold tolerance of cucumber (Cucumis sativus L.) [J/OL]. Int. J.
Mol. Sci., 2020, 21(11): 3857 [2023-09-03]. https://doi. org/
10.3390/ijms21113857.
[6] BOLLE C. The role of GRAS proteins in plant signal
transduction and development [J]. Planta, 2004, 218(5):
683-692.
[7] SUN X, JONES W T, RIKKERINK E H. GRAS proteins: the
versatile roles of intrinsically disordered proteins in plant
signalling [J]. Biochem. J. 2012, 442(1):1-12.
[8] 孔豆豆,毛成志,王蕾,等.大麦基因组GRAS 基因家族的全基
因组鉴定与表达分析 [J]. 分子植物育种, 2021, 19(1):22-33.
KONG D D, MAO C Z, WANG L, et al .. Genome-wide
identification and phylogenetic analysis of the GRAS gene
family in barley (Hordeum vulgare L.) [J]. Mol. Plant Breed.,
2021, 19(1):22-33.
[9] 郑玲,闫晓曼.甜瓜GRAS家族全基因组的鉴定与表达分析[J].
江苏农业科学, 2023, 51(11):53-59.
[10] 张文慧,何光鑫,王子鑫,等.绿豆GRAS 基因家族鉴定及其非
生物胁迫下的表达模式分析[J].农业生物技术学报, 2022,
30(5):861-872.
ZHANG W H, HE G X, WANG Z X, et al .. Identification of
GRAS gene family and its expression on pattern analysis under
abiotic stress in Vigna radiata [J]. J. Agric. Biotechnol., 2022,
30(5):861-872.
[11] 王智兰,韩康妮,杜晓芬,等.谷子GRAS转录因子家族的全基
因组鉴定、表达分析及标记开发 [J]. 核农学报, 2022, 36(9):
1723-1737.
WANG Z L, HAN K N, DU X F, et al .. Identification,
expression analysis and marker development of GRAS
transcription factor in foxtail millet [J]. Acta Agric. Nucl. Sin.,
2022, 36(9):1723-1737.
[12] TIAN C, WAN P, SUN S, et al .. Genome-wide analysis of the
GRAS gene family in rice and Arabidopsis [J]. Plant Mol. Biol.,
2004, 54(4):519-532.
[13] WANG T, YU T, FU J, et al .. Genome-wide analysis of the
GRAS gene family and functional identification of GmGRAS37
in drought and salt tolerance [J]. Front. Plant Sci., 2020, 11:
604690 [2023-09-03]. https://doi.org/10.3389/fpls.2020.604690.
[14] 郭栋,宋雅菲,张佳阔,等.玉米CCCH 基因家族鉴定及分析[J].
中国农业科技导报, 2019, 21(8):19-27.
GUO D, SONG Y F, ZHANG J K, et al .. Identification and
analysis of CCCH gene family in maize [J]. J. Agric. Sci.
Technol., 2019, 21(8):19-27.
[15] HUSON D H, BRYANT D. Application of phylogenetic
networks in evolutionary studies [J]. Mol. Biol. Evol., 2006, 23:
254-267.
[16] NAM J, MA H, NEI M. Antiquity and evolution of the
MADSbox gene family controlling flower development in plants [J].
Mol. Biol. Evol., 2003, 20(9):1435-1447.
[17] MOORE R C, PURUGGANAN M D. The evolutionary
dynamics of plant duplicate genes [J]. Curr. Opin. Plant Biol.,
2005, 8(2):122-128.
[18] TANG H, BOWERS J E, WANG X, et al .. Synteny and
collinearity in plant genomes [J]. Science, 2008, 320 (5875):
486-488.
[19] PENG J, CAROL P, RICHARDS D E, et al .. The Arabidopsis
GAI gene defines a signaling pathway that negatively regulates
gibberellin responses [J]. Genes Dev., 1997, 11:3194-3205.
[20] SILVERSTONE A L, CIAMPAGLIO C N, SUN T. The
Arabidopsis RGA gene encodes a transcriptional regulator
repressing the gibberellin signal transduction pathway [J].
Plant Cell, 1998, 10:155-169.
[21] IKEDA A, UEGUCHI-TANAKA M, SONODA Y, et al ..
Slender rice, a constitutive gibberellin response mutant.; is
caused by a null mutation of the SLR1 gene, an ortholog of the
height-regulating gene GAI/RGA/RHT/D8 [J]. Plant Cell, 2001,
13:999-1010.
[22] STUURMAN J, JAGGI F, KUHLEMEIER C. Shoot meristem
maintenance is controlled by a GRAS-gene mediated signal
from differentiating cells [J]. Genes Dev., 2002, 16:2213-2218.
[23] MOROHASHI K, MINAMI M, TAKASE H, et al .. Isolation
and characterization of a novel GRAS gene that regulates
meiosis-associated gene expression [J]. J. Biol. Chem., 2003,
278:20865-20873.
[24] BOLLE C, KONCZ C, CHUA N H. PAT1, a new member of the
GRAS family, is involved in phytochrome A signal transduction [J].
Genes Dev., 2000, 14:1269-1278.
[25] YUAN Y, FANG L, KARUNGO S K, et al .. Overexpression of
VaPAT1, a GRAS transcription factor from Vitis amurensis,
confers abiotic stress tolerance in Arabidopsis [J]. Plant Cell
Rep., 2016, 35:655-666.
[26] JI C, XU L N, LI Y J, et al .. The O2-ZmGRAS11
transcriptional regulatory network orchestrates the coordination
of endosperm cell expansion and grain filling in maize [J]. Mol.
Plant, 2022, 15(3):468-487.
[27] LI Y, MA S, ZHAO Q Q, et al .. ZmGRAS11, transactivated by
opaque2, positively regulates kernel size in maize [J]. J. Integr.
Plant Biol., 2021, 63(12):2031-2037.
(责任编辑:温小杰)
