基于ISSR标记分析连云港地区野生灵芝种质资源遗传多样性及亲缘关系
2024-04-24纪伟苏文英刘晓梅王一璞任立凯奚小艳陈克龙
纪伟,苏文英,刘晓梅,王一璞,任立凯,奚小艳,陈克龙
(1连云港市农业科学院,江苏连云港 222000;2青海师范大学,西宁 810000)
0 引言
灵芝(Ganodermalingzhi)在中国有着悠久的食药用历史,属于大型可食用真菌[1],近年来,灵芝产业在中国乡村振兴中发挥着重要的作用,灵芝产业的高质量发展也将有力助推“健康中国”战略的实施。研究表明,灵芝含有丰富的药理成分,主要包括灵芝多糖、多糖肽、三萜、甾醇、生物碱和核苷等[2-4],在镇痛、解毒、降血糖、抗辐射、提高免疫力、治疗哮喘和抗肿瘤等方面有着极高的药用价值[5-7]。灵芝还拥有极高的文化观赏价值[8],自古被视为吉祥、富贵之物,灵芝造型各异,可塑性强,在生长期通过控制光照方向、空气湿度等环境因素,可将之培育成外形美观的观赏植物[9]。
ISSR(Inter-Simple Sequence Repeats)标记技术最早创建于20世纪90年代,其原理是根据基因组中存在着广泛的微卫星序列,通过设计的通用引物对基因组进行PCR 扩增,从而检测基因组中微卫星序列的变异,该技术具有很好的稳定性和多态性,常被应用在生物种质资源、遗传多样性、居群遗传结构等方面的研究[10-12]。ISSR分子标记技术在食用菌的聚类分析方面已经得到了广泛应用,陈小敏等[13]发现野生灵芝菌株的ISSR电泳图谱具有明显的差异性,聚类分析显示出相似系数小于0.52时,供试菌株聚为一类,相似系数为0.54 时,聚类成两大类群,说明供试12 株灵芝菌株间表现出明显的遗传多样性;肖自添等[14]应用ISSR分子标记技术对24个灵芝菌株进行了遗传多样性研究,从43 个引物中选出10 个ISSR 引物扩增得到110 个扩增位点,大小分布在500~2000 bp,并利用NTSYS.PC2.0分析软件对这些供试菌株的遗传差异性进行聚类分析,研究结果表明相似水平在71%左右时24个菌株分为4 个组群。陆欢等[15]经非加权算数平均配对法(Unweighted pair-group method with arithmetic mean,UPGMA)聚类分析,24 个金顶侧耳菌株被分为7 个大类,并发现野生和栽培菌株聚类均比较集中。
目前灵芝的ISSR 遗传多样性分析主要集中在通过遗传相似系数对所研究区域灵芝进行分类,而有效等位基因数(Ne)、Nei's遗传多样性(He)和Shannon信息指数(I)等指标还未见有公开报道,连云港地区野生灵芝资源丰富[16],但研究甚少。本研究以连云港地区采集的15 株野生灵芝为研究材料,通过ISSR 分子标记技术进行遗传多样性分析,计算多态性位点、等位基因数、有效等位基因数(Ne)、Nei's 遗传多样性(He)和Shannon 信息指数(I)等指标,并根据ISSR 分子标记的遗传聚类图进行分类,确定连云港地区野生灵芝种质资源遗传多样性和亲缘关系,为灵芝资源保护和品种选育提供参考依据。
1 材料与方法
1.1 野生灵芝采集
本项目组于2022年6月至2022年9月在连云港市范围内采集获得野生灵芝标本,根据形态特征和生境特点选取具有代表性的标本15个,野生灵芝标本信息如表1所示。将采集的野生灵芝进行组织分离得到菌株,保存于连云港市农科院食用菌菌种库。

表1 野生灵芝标本信息
1.2 灵芝菌株基因组提取及ISSR-PCR扩增
采用真菌基因组DNA 快速提取试剂盒提取基因组DNA,试剂盒为生物工程(上海)股份有限公司生产,扩增条件为95℃预变性5 min,95℃变性30 s,退火温度根据各引物梯度试验筛选出的最佳退火温度(UBC807 为48℃、UBC809 为45℃、UBC811 为51℃、UBC836 为54℃、UBC840 为42℃、UBC841 为48℃、UBC842 为42℃、UBC880 为45℃、UBC881 为48℃和UBC888 为42℃),退火30 s,72℃延伸2 min,45 个循环,72℃延伸10 min。
1.3 统计与分析
采用Photoshop 软件处理图片,利用DPS 软件绘制遗传关系聚类图,采用Popgen 32软件计算遗传多样性指数,ISSR-PCR电泳图,以Marker 2000作为参考。
2 结果与分析
2.1 引物筛选及退火温度优化
选取10 条ISSR 引物,引物编号分别为UBC807、UBC809、UBC811、UBC836、UBC840、UBC841、UBC842、UBC880、UBC881和UBC888,委托生工生物工程(上海)股份有限公司合成。对上述合成的10 条ISSR 引物的退火温度开展梯度试验,经PCR 扩增,筛选出扩增条带清晰明亮对应的退火温度,即为最佳退火温度,如表2所示。筛选出多态性好的ISSR引物一条,为引物UBC888,如图1所示,电泳图清晰明亮。

表2 10条ISSR引物及退火温度
2.2 基于ISSR 分子标记的连云港野生灵芝遗传多样性分析
通过选取的10 条引物对15 株野生灵芝进行PCR扩增,共扩增出40条多态性条带,如表3所示,条带大小在250~2000 bp 之间;平均等位基因位点1.8 个,平均有效等位基因数1.2454,平均Nei's 遗传多样性0.1742,平均Shannon 信息指数0.2925;有效等位基因数(Ne)的变化范围为1.0000~1.6423,其中引物UBC807 和UBC842 对应的Ne最高,引物UBC841 对应的Ne最低;Nei's 遗传多样性(He)的变化范围为0.1031~0.3911,其中引物UBC807 和UBC842 对应的He最高,引物UBC841 和UBC881 对对应的He最低;Shannon 信息指数(I)的变化范围为0.2235~0.5799,其中引物UBC807 和UBC842 对应的I最高,引物UBC841对应的I最低。
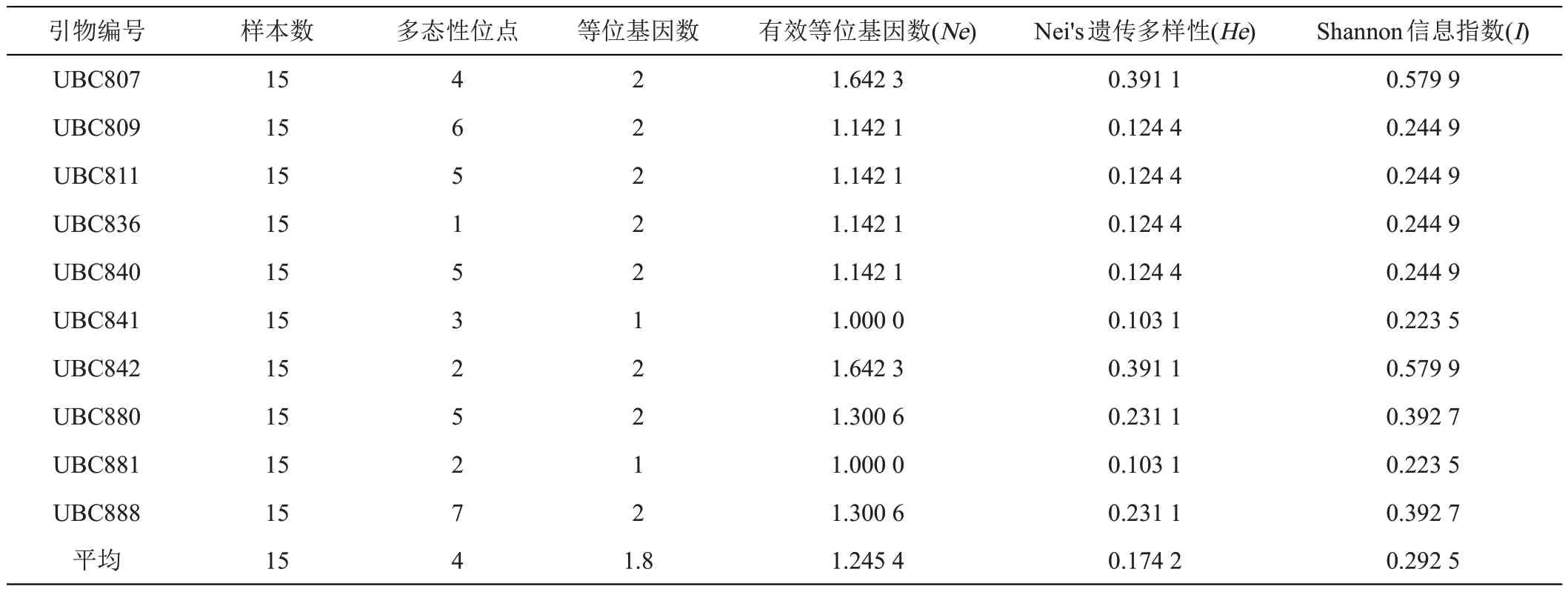
表3 基于10条ISSR引物标记的灵芝遗传多样性数据
2.3 连云港野生灵芝聚类分析
基于遗传关系聚类图,如图2 所示,相似系数为0.63时,将15株野生灵芝分为4组,第1组有3株灵芝,分别为灵芝GL01、GL10 和GL03,其中灵芝GL01 和GL10距离更近;第2组有4株灵芝,分别为灵芝GL02、GL06、GL11 和GL12,其中灵芝GL06、GL11 和GL12距离较近;第3 组有7 株灵芝,分别为灵芝GL04、GL05、GL15、GL13、GL14、GL07 和GL09,其中灵芝GL04、GL05 和GL15 距离较近,灵芝GL13 和GL14 距离较近,灵芝GL07和GL09距离较近;第4组有1株灵芝,为灵芝GL08。
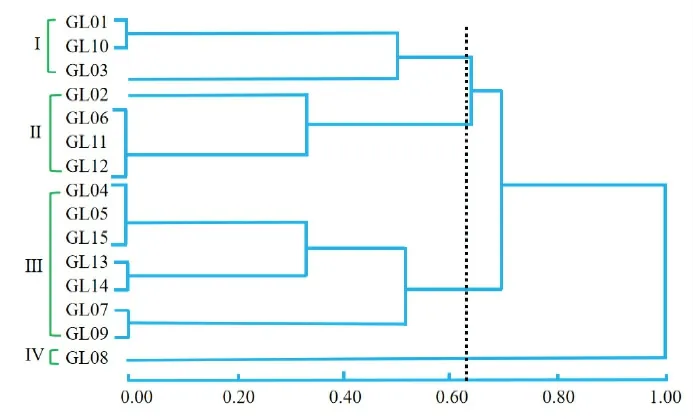
图2 基于ISSR分子标记的灵芝遗传关系聚类图
本研究采集的野生灵芝生境植被为麻栎、板栗等阔叶树种,如图3所示,其中GL02为白肉灵芝,虽然上述将灵芝GL02、GL06、GL11 和GL12 聚为一类,但灵芝GL02 与其他3 株灵芝距离更远,灵芝GL06、GL11和GL12 三个灵芝均处于子实体的幼嫩阶段,其子实体外观都比较规则,边缘均呈现淡黄色至黄色,符合形态学特征;灵芝GL04、GL05、GL15、GL13、GL14、GL07 和GL09,该组灵芝的特点非常明显,都具有菌柄,菌盖颜色均为红褐色,是典型的赤芝,上述聚类分析也分别将其聚为一组;GL08 为典型的树舌灵芝,上述将灵芝GL08单独聚为一组,符合形态学特征;上述聚类结果与形态学特征分类结果相近,说明上述聚类结果具有很高的可信度。

图3 野生灵芝子实体
2.4 连云港野生灵芝遗传相似系数分析
15株野生灵芝的遗传相似系数,如表4所示,相似系数在0.415~0.866 之间,平均相似系数为0.624,有7.62%的菌株间相似系数大于0.8,有19.05%的菌株间相似系数介于0.7~0.8之间,有73.33%的菌株间相似系数小于0.7,说明菌株间遗传差异较大,亲缘关系最近的为菌株GL13 和GL14,相似系数为0.866,亲缘关系最远的为菌株GL06和GL08,相似系数为0.415。
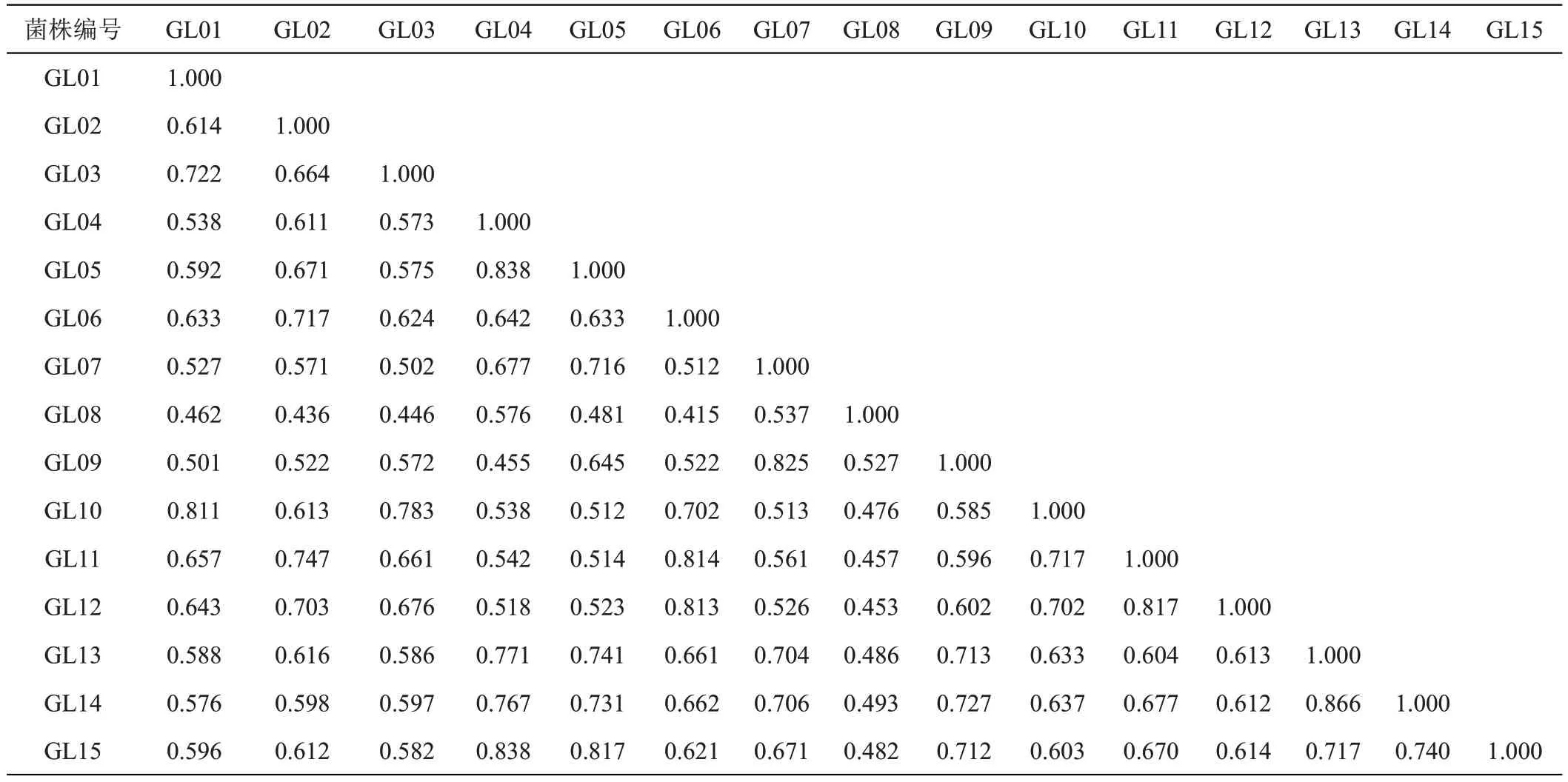
表4 灵芝的遗传相似系数
3 讨论
3.1 ISSR引物最佳退火温度
野生灵芝因所处的生境、温度、湿度和基质养分等自然因素差异的影响,其子实体外观形态差异较大,仅仅从外观形态对灵芝进行分类,并不能科学准确的反映其遗传距离[17]。ISSR 分子标记可以从DNA 分子水平上来检测基因组DNA的多态性,不受繁育阶段的气候环境因素影响,可快速、准确和稳定的将菌株进行分类[14]。本研究使用ISSR 分子标记技术对连云港地区15 株野生灵芝的遗传多样性进行分析。首先对筛选的10 条ISSR 引物最佳退火温度进行梯度筛选试验,在PCR试验中,引物的退火温度过低或过高都会影响试验结果的准确性,温度过低会导致特异性扩增条带增多,而温度过高,则会抑制引物与DNA的结合,扩增出的条带会出现弥散情况,本研究经梯度试验最终筛选到各引物的最佳退火温度,UBC807 为48℃、UBC809 为45℃、UBC811 为51℃、UBC836 为54℃、UBC840 为42℃、UBC841 为48℃、UBC842 为42℃、UBC880 为45℃、UBC881 为48℃和UBC888 为42℃,筛选出多态性好的引物一条,为UBC888。
3.2 野生灵芝之间遗传多样性和亲缘关系分析
本研究使用ISSR分子标记技术从15株野生灵芝中扩增出多态性条带40 条,片段大小在250~2000 bp之间,这与肖自添等[14]研究结果较为相近,平均等位基因位点1.8 个,平均有效等位基因数1.2454,平均Nei's遗传多样性0.1742,平均Shannon 信息指数0.2925,有效等位基因数(Ne)的变化范围为1.0000~1.6423,Nei's遗传多样性(He)的变化范围为0.1031~0.3911,Shannon信息指数(I)的变化范围为0.2235~0.5799,从分析结果看连云港地区采集的这15 株野生灵芝存在着一定的遗传差异,具有很好的遗传多样性。
根据ISSR 分子标记的遗传聚类图可有效进行分类,刘小英等[18]使用ISSR分子标记技术对20份龙眼材料进行分析,在遗传相似系数0.734 0时,可将20份龙眼材料分4 类;裴云霞等[19]使用ISSR 分子标记技术对枫香进行分析,当遗传相似系数为0.94时,可将7个群体分为4个大类。本研究的15株灵芝相似系数范围在0.415~0.866 之间,相似系数为0.63 时,可以将15 株野生灵芝分为四组,这与张彬彬等[20]对30株野生灵芝的分类结果相近,该分类和野生灵芝的外观形态有一定的对应关系,第1 组有3 株灵芝,分别为灵芝GL01、GL10 和GL03,该组灵芝中GL01 和GL03 均为子实体的幼嫩阶段,菌盖均呈红褐色,边缘肥厚呈淡黄色,都有轮纹,非常接近,而与GL10 外观差异较大,这可能因GL10受外界因素影响和生理阶段已经处于老化阶段所致;第2 组有4 株灵芝,分别为灵芝GL02、GL06、GL11 和GL12,其中灵芝GL06、GL11 和GL12 距离较近,GL02 为白肉灵芝,外观形态明显区别与其他3 株灵芝,这与聚类分析中GL02与其他3株灵芝遗传距离更远的结果对应,而GL06、GL11 和GL12 三个灵芝菌株均处于幼嫩发育阶段,其子实体外观都比较规则,边缘均呈现淡黄色至黄色;第3组有7株灵芝,分别为灵芝GL04、GL05、GL15、GL13、GL14、GL07 和GL09,该组灵芝的特点非常明显,都具有菌柄,菌盖颜色均为红褐色,是典型的赤芝;第4组有1株灵芝,为灵芝GL08,是典型的树舌灵芝。上述聚类结果与形态学特征分类结果基本一致,说明上述聚类结果具有很高的可信度。
4 结论
本研究采用ISSR 分子标记技术对连云港地区的15株野生灵芝进行分析,并构建聚类图谱。15株野生灵芝中扩增出多态性条带为40 条,片段大小在250~2000 bp之间,平均等位基因位点1.8个,平均有效等位基因数1.2454,平均Nei's 遗传多样性0.1742,平均Shannon信息指数0.2925,有效等位基因数(Ne)的变化范围为1.0000~1.6423,Nei's 遗传多样性(He)的变化范围为0.1031~0.3911,Shannon 信息指数(I)的变化范围为0.2235~0.5799,根据ISSR 分子标记的遗传聚类图,15株灵芝相似系数范围在0.415~0.866之间,相似系数为0.63时,可以将15株野生灵芝分为4组,聚类结果与形态学特征结果基本一致。
ISSR 分子标记用于遗传多样性分析有着操作简单、快速和成本低廉等优点,但在PCR 扩增时需要一定的时间摸索最适反应条件,对于显性遗传标记,将无法区分显性纯合基因型和杂合基因型[21],作者认为本研究仍需进一步深化,扩大样品量,选取更多类型的野生灵芝材料,并结合全基因组重测序进行分析,确保结果的准确性,更好为连云港地区灵芝资源分类、保护和种质创制提供优质材料。
