山西谷子地方品种微核心种质构建
2024-04-23侯森秦慧彬李萌王海岗穆志新
侯森 秦慧彬 李萌 王海岗 穆志新


摘要:构建山西谷子地方品种微核心种质,旨在为谷子目标性状精准鉴定与相关候选基因挖掘提供种质基础。在598 份山西谷子核心种质的基础上,选取抽穗期、株高、节数、颈长、叶长、叶宽、穗长、穗粗、茎粗、单穗质量、穗粒质量、码数、码粒数、千粒质量和蛋白质含量等15 个农艺、品质性状,使用QGAStation 软件进行微核心种质构建抽样分析、全基因组重测序技术去除近缘种质,以构建微核心种质。结果表明,山西谷子地方品种微核心种质共包含322 份种质资源。微核心种质与原始种质比较结果表明,各性状的均值与方差均没有表现出显著性差异,均值差异百分率与方差差异百分率均为0,极差符合率为98.36%,变异系数变化率为104.19%,遗传多样性指数变化率为94.81%。微核心种质较好地保留了原始种质的遗传变异,具有较高的变异度,同时具备较强的代表性。
关键词:谷子;地方品种;微核心种质;重测序技术;山西
中图分类号:S515 文献标识码:A 文章编号:1002?2481(2024)01?0019?08
谷子(Setaria italica)起源于我国,是人类最早驯化的农作物之一[1],是我国北方干旱、半干旱地区的重要杂粮作物,粮饲兼用,具有耐旱、耐贫瘠、适应性强等优点。山西及其周边地区是我国禾谷类杂粮的富集中心,其中山西谷子地方品种数居全国第一位,占全国的26.2%[2]。山西省南北横跨6 个纬度,谷子种植区包含春播早熟区、春播中熟区、春播晚熟区和夏播区,多样的生态类型孕育出丰富的谷子资源[3]。谷子具有较小的基因组,自花授粉且生育期较短,是十分理想的C4 模式作物[4-5]。随着谷子基因组测序工作的深入,通过关联分析深度挖掘谷子复杂性状相关候选基因的工作逐渐展开,群体构建合理性与性状鉴定精准度将直接影响关联分析的效力[6-7]。
对作物目标性状进行鉴定评价的第一步是构建合适的材料群体。核心种质是从原始种质中筛选出的种质群体,以较少种质数量最大限度保留原始种质群体的遗传多样性[8-9]。丰富的原始种质资源是筛选核心种质的前提。陆平[10]考察收集、引进谷子和高粱种质3 000 余份,编目入库谷子和高粱及稀有作物种质7 000 余份,编著《中国谷子品种志(1986—2010 年)》、《谷子种质资源描述规范和数据标准》等著作,丰富了谷子研究种质基础,明确了谷子性状鉴定标准。李国营等[11]从2 057 份谷子初级核心种质中随机选取439 份作为研究对象,比较了不同生态种植区对谷子生育酚含量的影响。温琪汾等[12]对山西省收集保存的5 000 余份谷子地方品种进行初步筛选,去除同名同种、异名同种等种质后保留其中3 761 份作为筛选种质并进行了田间抗旱鉴定,筛选出58 份抗旱且丰产的优异种质。王海岗等[13]依据农艺性状通过聚类后随机抽取结合多样性指数检测的方法,从5 600 余份山西谷子地方种质资源中筛选出638 份作为初选核心种质,初选核心种质与原始种质的极差符合率为87.4%,变异系数变化率为102.0%,能够较好的代表原始种质。完全随机的抽取可能会导致抽取种质的遗传多样性不佳,初级核心种质的群体大小对于发掘目标性状相关基因的关联分析工作仍显庞大,需要结合农艺性状进行筛选以提高筛选种质的多样性和代表性,并在不影响关联准确度的同时尽可能减少筛选种质的数量,以提高关联分析效率。
微核心种质是核心种质的代表性子集,代表原始种质遗传多样性的同时进一步精简资源数量[14-15]。烟草中以446 份烟草核心种质为基础,利用SSR 引物进行遗传多样性分析,通过简单比例法和对数比例法筛选构建了127 份烟草的微核心种质,并对微核心种质的株高等12 个植物学性状进行了遗传效应分析[16]。黍稷中对636 份核心种质进行遗传多样性分析后,利用次级聚类群体随机抽样的方法构建了含253 份黍稷的微核心种质,并对微核心种质的遗传多样性进行了评价[17]。大豆中利用微核心种质与地方品种杂交和回交,后代的抗倒伏、抗病等性状得到了有效改良[18]。蚕豆中对来自国内外的1 075 份初级核心种质进行了筛选,通过基于SSR 标记的遗传多样性分析获得包含129 份国内和63 份国外资源的微核心种质[19]。微核心种质对作物育种研究具有重要的意义,构建山西谷子地方品种微核心种质,旨在为谷子遗传育种改良、资源鉴定保护等工作提供一定理论支撑。
1 材料和方法
1.1 试验材料
598 份山西谷子地方品种核心种质收集自山西各地市(表1),数据来源于山西农业大学农业基因资源研究中心谷子种质资源数据库[20]。
1.2 试验方法
1.2.1 农艺性状调查
供试材料于2017—2019 年在山西农业大学东阳试验基地种植,采取普通大田管理措施。参照《谷子种质资源描述规范和数据标准》对抽穗期、株高、节数、颈长、叶长、叶宽、穗长、穗粗、茎粗、单穗质量、穗粒质量、码数、码粒数、千粒质量、蛋白质含量15 个性状进行调查测定[20]。
1.2.2 DNA 提取
供試材料生长至抽穗期时取倒二叶,液氮速冻后-80 oC 保存。采用CTAB 法提取DNA,通过酶标仪测定DNA 浓度后-20 oC 保存。对质检合格的DNA 进行全基因组重测序。
1.2.3 微核心种质构建
利用各性状数据,通过最短距离法进行聚类,通过多次聚类随机取样法对供试材料进行取样,构建微核心种质。
1.3 统计分析
使用EXCEL 2019 软件进行描述统计分析与遗传多样性指数计算。使用QGAStation 软件进行微核心种质构建抽样分析[21]。
2 结果与分析
2.1 谷子农艺与品质性状
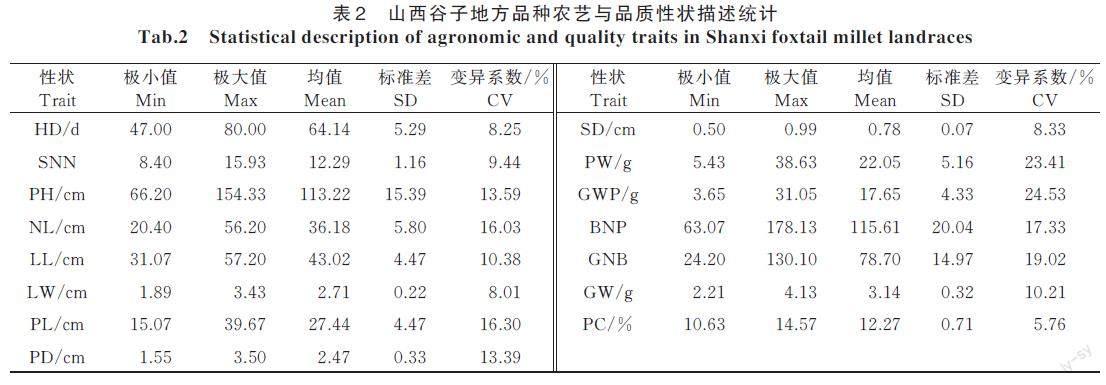
鉴定了抽穗期(HD)、株高(PH)、节数(SNN)、颈长(NL)、叶长(LL)、叶宽(LW)、穗长(PL)、穗粗(PD)、茎粗(SD)、单穗质量(PW)、穗粒质量(GWP)、码数(BNP)、码粒数(GNB)、千粒质量(GW)与成熟籽粒蛋白质(PC)含量15 个农艺与品质性状。其中,穗粒质量的变异系数最大(24.53%),蛋白质含量的变异系数最小(5.76%)(表2)。
2.2 微核心种质筛选
利用QGAStation 软件中的种质资源库构建功能,通过15 个农艺与品种性状对598 份原始种质进行取样,在抽样比率为65% 时,以多次聚类随机取样法共抽取388 份种质资源。依据谷子原始核心种质通过重测序数据获得的亲缘关系结果,对抽取种质中亲缘关系最近的两材料间随机剔除1 份,群体大小得到进一步压缩,最终获得包含322 份种质资源的山西谷子地方品种微核心种质(表3)。
微核心种质与原始种质之间,各性状的均值与方差在P=0.05 水平上均没有差异显著性。微核心种质与原始种质相比,均值差异百分率(MD)与方差差异百分率(VD)均为0,遗传多样性指数变化率(GR)为94.81%,表明微核心种质较好地保存了原始种质的遗传变异。极差符合率(CR)为98.36%,表明微核心种质在保留原始种质结构的同时具有较高的变异度。变异系数变化率(VR)为104.19%,表明微核心种质在具有较高变异度的同时具备较强的代表性(表4)。
3 结论与讨论
种质资源是农业的芯片,是作物育种的基础。发掘优异基因资源,是作物遗传改良与种质高效利用的重要途径。山西谷子种质资源丰富,构建微核心种质,在保存遗传多样性的同时尽量去除冗余,对谷子种质资源鉴定评价和基因资源挖掘有着重要意义。近年来,随着基因组测序研究的深入,各作物中广泛开展了基于全基因组关联分析(GWAS)的候选基因挖掘工作[22,6]。针对产量、耐逆性、抗病性等由多基因控制的复杂农艺性状,全基因组关联分析是挖掘相关候选基因的有力方法[23]。影响全基因组关联分析效力的主要因素有群体结构、性状鉴定准确性、分子标记密度和关联模型4 个方面[24]。群体结构既要保证种质资源丰富度和遗传多样性,同时还要兼顾群体大小,过于庞大的群体对前期目标性状鉴定和后期关联分析都会造成很大的压力,从原始种质中筛选出微核心种质进行研究是合理的解决途径。
原始种质资源的丰富程度决定着微核心种质的质量,原始种质遗传背景广泛、遗传多样性丰富有利于微核心种质特异性、代表性和遗传多样性的保持。胡兴雨等[25-26]对国家种质资源库中收集保存的8 000 余份黍稷种质资源的株高等11 个农艺性状进行了主成分分析和聚类分析,分为5 大群组并各有一定的形态学特征,依据地理来源将黍稷资源分为23 组,每组内在11 个农艺性状聚类的基础上按比例法取样,构建了包含780 资源的初选核心种质,为黍稷育种研究筛选了良好的研究种质。山西省于20 世纪50 年代进行了第1 次大规模的谷子种质资源征集,共征集到谷子地方品种和农家种2 600 余份,20 世纪80 年代进行了第2 次补充征集,共征集到各类谷子种质资源4 200 余份[27]。近年来,开展的第3 次全国农作物种质资源普查行动进一步丰富了山西省的谷子种质资源,多代种质资源人的薪火相传不懈努力,为作物育种研究打下了坚实的种质基础。
构建微核心种质通常对原始种质的身份信息与农艺性状等数据进行分析,依据种质类型、种质来源、生态区域和农艺性状等进行分组,聚类分析后在组内抽取核心样品,组建微核心种质。聚类方法有最短距离法、最长距离法、中间距离法、重心法、可变法、类平均法、离差平方和法等,取样方法有多次聚类随机取样法、多次聚类优先取样法、多次聚类偏离度取样法等[28]。采用最短距离法聚类,多次聚类随机取样法取样是核心种质构建的常用策略[29]。在前人构建辣椒核心种质库的研究中对多种方法进行比较,最短距离法聚类的极差符合率(CR)为100%,同时具有最高的变异系数变化率(VR),保留原始资源遗传结构的同时具有较高的变异度;多次聚类随机取样法取样的极差符合率(CR)为100%,均值差异百分率(MD)为0,方差差异百分率(VD)同样最小,较好地保留了原始资源的遗传变异[30]。另一项构建粳稻核心种质的研究中,则认为偏离度取样法优于优先取样法和随机取样法,聚类方法最好的是最短距离法和可变类平均法[31]。在具体分析中需将筛选出的种质与原始种质进行比较,通过均值差异百分率(MD)、方差差异百分率(VD)、极差符合率(CR)和变异系数变化率(VR)等指标评价筛选结果[32]。本研究中微核心种质的MD、VD、CR 和VR 分别为0、0、98.36% 和104.19%,其中均值差异百分率(MD)小于20%,变异系数变化率(VR)大于80%,能够较好地代表原始种质的遗传变异。
本研究对598 份山西谷子地方品种核心种质进一步筛选,利用抽穗期、株高、穗粒质量、蛋白质含量等15 个农艺与品质性状,通过多次聚类随机取样法进行取样,去除近缘种质后构建了包含322 份种质资源的山西谷子地方品种微核心种质,为谷子种质资源保护利用、基因资源挖掘以及遗传改良提供了种质基础。
参考文献:
[1] 贾冠清,刁现民. 谷子(Setaria italica(L.) P. Beauv.)作为功能基因组研究模式植物的发展现状及趋势[J]. 生命科学,2017,29(3):292-301.
JIA G Q,DIAO X M. Current status and perspectives of re?searches on foxtail millet(Setaria italica(L.) P. Beauv.):a po?tential model of plant functional genomics studies[J]. ChineseBulletin of Life Sciences,2017,29(3):292-301.
[2] 刘旭,黎裕,曹永生,等. 中国禾谷类作物种质资源地理分布及其富集中心研究[J]. 植物遗传资源学报,2009,10(1):1-8,171,169.
LIU X,LI Y,CAO Y S,et al. Geographic distribution andgermplasm-rich region of cereals in China[J]. Journal of PlantGenetic Resources,2009,10(1):1-8,171,169.
[3] 王殿瀛,郭桂兰,王节之,等. 中国谷子主产区谷子生态区划[J]. 华北农学报,1992,7(4):123-128.
WANG D Y,GUO G L,WANG J Z,et al. The ecological divi?sion of main production area of Chinese millet(Setaria italicabeauv) in China[J].Acta Agriculturae Boreali-Sinica,1992,7(4):123-128.
[4] PENGR H,ZHANGB H. Foxtail millet:a new model for C4plants[J]. Trends in Plant Science,2021,26(3):199-201.
[5] HE Q,TANG S,ZHI H,et al. A graph-based genome and pangenomevariation of the model plant Setaria[J]. Nature Genetics,2023,55:1232-1242.
[6] LIU X D,YANG Y,HOU S Y,et al. The integration ofgenome-wide association study and homology analysis to ex?plore the genomic regions and candidate genes for paniclerelatedtraits in foxtail millet[J]. International Journal of Molecu?lar Sciences,2022,23(23):14735.
[7] BANDYOPADHYAY T,SWARBRECK S M,JAISWAL V,et al. GWAS identifies genetic loci underlying nitrogen respon?siveness in the climate resilient C4 model Setaria italica(L.)[J].Journal of Advanced Research,2022,42:249-261.
[8] FRANKEL O,BROWN A. Plant genetic resources today:acritical appraisal[M]//HOLDEN J,WILLIAMS J. Crop Ge?netic Resources:Conservation & Evaluation. London:GeorgeAllan and Urwin Ltd,1984:249-257.
[9] BROWN A. The case for core collections[M]//BROWN A,FRANKEL O,MARSHALL R,et al. The Use of Plant Ge?netic Resources. Cambridge:Cambridge University Press,1989:136-156.
[10] 陆平. 谷子种质资源描述规范和数据标准[M]. 北京:中国农业出版社,2006.
LU P. Descriptors and data standard for foxtail millet(Setariaitalica(L.) beauv.)[M]. Beijing:China Agriculture Press,2006.
[11] 李国营,范志影,陆平,等. 谷子初级核心种质生育酚的组分及其评价[J]. 植物遗传资源学报,2009,10(3):378-384.
LI G Y,FAN Z Y,LU P,et al. Identification and evalution oftocopherol content in primary core-collection of foxtail millet[J]. Journal of Plant Genetic Resources,2009,10(3):378-384.
[12] 温琪汾,刘润堂,王纶,等. 山西省谷子品种资源的抗旱性和丰产性研究[J]. 山西农业大学学报(自然科学版),2004,24(3):224-226.
WEN Q F,LIU R T,WANG L,et al. Drought resistance andhigh yield of the foxtail millet germplasm in Shanxi Province[J].Journal of Shanxi Agricultural University,2004,24(3):224-226.
[13] 王海岗,温琪汾,乔治军,等. 山西谷子地方品种初选核心种质构建[J]. 农学学报,2019,9(4):26-31.
WANG H G,WEN Q F,QIAO Z J,et al. Core germplasmconstruction of foxtail millet Landrace in Shanxi[J]. Journal ofAgriculture,2019,9(4):26-31.
[14] UPADHYAYA H D,ORTIZ R. A mini core subset for cap?turing diversity and promoting utilization of chickpea genetic re?sources in crop improvement[J]. Theoretical and Applied Ge?netics,2001,102(8):1292-1298.
[15] 毛秀红,闫少波,葛磊,等. 毛白杨微核心种质构建方法的探讨[J]. 北京林业大学学报,2023,45(2):58-67.
MAO X H,YAN S B,GE L,et al. Study on constructionmethod of microcore germplasm of Populus tomentosa[J]. Jour?nal of Beijing Forestry University,2023,45(2):58-67.
[16] 张兴伟. 烟草微核心种质构建及相关性状数量遗传分析[D].北京:中国农业科学院,2013.
ZHANG X W. Construction and quantitative genetic analysisof tobacco mini core collection[D]. Beijing:Chinese Academyof Agricultural Sciences,2013.
[17] 连帅. 黍稷种质资源遗传多样性研究及微核心种质的构建[D]. 太谷:山西农业大学,2017.
LIAN S. Genetic diversity assessment of broomcorn millet(Panicum miliaceum L.) and construction of A mini core col?lection in china[D]. Taigu:Shanxi Agricultural University,2017.
[18] 邱丽娟,李英慧,关荣霞,等. 大豆核心种质和微核心种质的构建、验证与研究进展[J]. 作物学报,2009,35(4):571-579.
QIU L J,LI Y H,GUAN R X,et al. Establishment,represen?tative testing and research progress of soybean core collectionand mini core collection[J]. Acta Agronomica Sinica,2009,35(4):571-579.
[19] 姜俊烨. 蚕豆微核心种质构建及SSR 遗传连锁图谱加密[D].北京:中国农业科学院,2014.
JIANG J Y.Mini-core collection construction and genetic link?age map density enhancement of faba bean(Vicia faba L.)with SSR markers[D]. Beijing:Chinese Academy of Agricul?tural Sciences,2014.
[20] 王海岗,温琪汾,穆志新,等. 山西谷子核心资源群体结构及主要农艺性状关联分析[J]. 中国农业科学,2019,52(22):4088-4099.
WANG H G,WEN Q F,MU Z X,et al. Population structureand association analysis of main agronomic traits of Shanxi corecollection in foxtail millet[J]. Scientia Agricultura Sinica,2019,52(22):4088-4099.
[21] CHENG B,ZHUZ X,ZHANGF T,et al. Quantitative geneticanalysis station for the genetic analysis of complex traits[J].Chinese Science Bulletin,2012,57(21):2721-2726.
[22] LIU H J,YAN J B. Crop genome-wide association study:aharvest of biological relevance[J]. The Plant Journal:for Celland Molecular Biology,2019,97(1):8-18.
[23] HE J B,GAI J Y. Genome-wide association studies(GWAS)[J]. Methods in Molecular Biology,2023,2638:123-146.
[24] HUANG C,NIE X H,SHEN C,et al. Population structureand genetic basis of the agronomic traits of upland cotton inChina revealed by a genome-wide association study using highdensitySNPs[J]. Plant Biotechnology Journal,2017,15(11):1374-1386.
[25] 胡興雨,陆平,贺建波,等. 黍稷农艺性状的主成分分析与聚类分析[J]. 植物遗传资源学报,2008,9(4):492-496.
HU X Y,LU P,HE J B,et al. Principal components and clus?ter analysis of agronomic traits of proso millet(Panicum mili?aceum)[J]. Journal of Plant Genetic Resources,2008,9(4):492-496.
[26] 胡兴雨,王纶,张宗文,等. 中国黍稷核心种质的构建[J]. 中国农业科学,2008,41(11):3489-3502.
HU X Y,WANG L,ZHANG Z W,et al. Establishment ofbroomcorn millet core collection in China[J]. Scientia Agricul?tura Sinica,2008,41(11):3489-3502.
[27] 温琪汾,王纶,王星玉. 山西省谷子种质资源及抗旱种质的筛选利用[J]. 山西农业科学,2005,33(4):32-33.
WEN Q F,WANG L,WANG X Y. The foxtail millet germ?plasm resources and screening and utilization of drought resis?tance germplasm in Shanxi[J]. Journal of Shanxi AgriculturalSciences,2005,33(4):32-33.
[28] 栾明宝,陈建华,许英,等. 苎麻核心种质构建方法[J]. 作物学报,2010,36(12):2099-2106.
LUAN M B,CHEN J H,XU Y,et al. Method of establishingramie core collection[J]. Acta Agronomica Sinica,2010,36(12):2099-2106.
[29] 朱志翔. 遗传分析软件QGAStation 2.0 和GMDR-GPU 的开发[D]. 杭州:浙江大学,2012.
ZHU Z X. Development of genetic analysis software QGASta?tion 2.0 and GMDR-GPU[D]. Hangzhou:Zhejiang University,2012.
[30] 何建文,韩世玉. 基于SSR 标记不同距离聚类与抽样方法构建辣椒核心种质库[J]. 西南农业学报,2015,28(5):2199-2204.
HE J W,HAN S Y. Construction of core Capsicum germplasmbank based on different distance clustering and samplingmethod of SSR marker[J]. Southwest China Journal of Agricul?tural Sciences,2015,28(5):2199-2204.
[31] 马洪文,陈晓军,殷延勃,等. 利用基因型值构建宁夏粳稻核心种质的方法[J]. 种子,2012,31(5):43-49.
MA H W,CHEN X J,YIN Y B,et al. The establishment ofNingxia Japonica rice core collection by the value of genotype[J]. Seed,2012,31(5):43-49.
[32] 姚启伦,方平,杨克诚,等. 基于SSR 标记构建西南玉米地方品种核心种质的方法[J]. 湖南农业大学学报(自然科学版),2009,35(3):225-228.
YAO Q L,FANG P,YANG K C,et al. Methods of construct?ing a core collection of maize landraces in southwest Chinabased on SSR data[J]. Journal of Hunan Agricultural University(Natural Sciences),2009,35(3):225-228.
