基于环境DNA 的长江中华鲟分布特征探究
2024-02-16周权杜浩王洁邵芸闫振广
周权,杜浩,王洁,邵芸,闫振广*
1.环境基准与风险评估国家重点实验室,中国环境科学研究院
2.中国水产科学研究院长江水产研究所
中华鲟(Acipenser sinensis)是我国一级水生野生保护动物,属于鲟形目(Acipenseriformes)、鲟科(Acipenseridae)、鲟属(Acipenser)。中华鲟作为古老而珍稀的软骨硬磷鱼类,在生物进化、地球变迁等方面具有重要的学术研究价值,而且中华鲟的历史比现代长江还要悠久,可谓地质年代的活化石,有人将中华鲟比作“水中大熊猫”,是长江流域重要的鲟类旗舰物种[1]。中华鲟曾广泛分布于我国黄海北部海洋岛至海南省万宁市近海,甚至远至海外[2],但是近年来中华鲟资源量急剧下降,1988 年中华鲟被列为国家一级重点保护动物,并进入濒危物种名单[3],2010 年,中华鲟被世界自然保护联盟(International Union for Conservation of Nature,IUCN)发布的物种红色目录列为CR(极危)等级物种。中华鲟的濒危情况受自身种群质量、人类活动及环境变化等多方面的综合影响[4],人类活动和环境变化对中华鲟的胁迫更呈现出了多样性与复杂性[5]。中华鲟属于底栖性鱼类,有洄游习性,在淡水中出生,在海洋中成长发育至性成熟后,洄游到淡水河流中产卵,繁殖期后再返回海洋。研究发现,长江中华鲟种群接近性成熟的个体在6—8 月进入长江口进行溯河生殖洄游,于次年10—11 月到达长江上游和葛洲坝下产卵场[6]。在空间分布上,中华鲟在洄游过程中多分布于江水的中下层,沿主河槽迁移,偶尔有向上层或露出水面迁移的现象[7]。
监测中华鲟的方法多样,可以利用声呐、水下摄影、食卵鱼解剖等方法观测中华鲟的具体个体,还可以通过地理信息系统(GIS)和遥感系统(RS)调查中华鲟[8],也可以利用放流标记追踪技术、卫星标记技术研究放流的中华鲟分布情况[9]。目前中华鲟监测常用的方法为分子检测方法,包括基于核苷酸微卫星开发的聚合酶链反应(PCR)[10]、利用微卫星DNA 探索中华鲟繁殖行为[11],以及通过线粒体DNA 研究中华鲟遗传多样性[12]。但是这些方法都需要观测到中华鲟个体,甚至取得其组织样品,取样困难并且调查成本高昂。如何在流域尺度上开展中华鲟监测仍然是难题[13]。
长江大保护中关于禁渔的要求使得对长江鱼类进行无损监测更加迫切,近年来,环境DNA(eDNA)技术越来越多地被用于生物多样性研究[14]。eDNA检测是一种对生物体无损的环境友好型监测技术,eDNA 是指来自目标物种的生物外遗传物质,它允许在不直接观察或捕获整个生物体的情况下,通过生物体脱落的细胞和粪便等释放到环境中的DNA 检测到物种的存在或最近的存在[15],这些DNA 可能是来自各种来源的线粒体或核DNA,包括脱落的部分生物体组织、尿液、黏液、卵子或精子等,可以以细胞和细胞外形式存在[16]。因此eDNA可以实现对生物体的无创监测,还可以提高监测特异性和敏感性,这些特点都十分适合研究长江中华鲟。eDNA 技术于2008 年首次用于追踪美国牛蛙的分布,此后被广泛用于检测两栖动物和鱼类[17]。与传统的人工形态学调查方法相比,eDNA 方法具有省时省力等优势[18],既可以用于调查一定区域内的生物类群遗传信息[19],又可以对外来种或入侵种的扩散趋势进行监测,目前已经有研究尝试将eDNA 技术纳入水生态安全监测体系[20]。这些研究进一步表明了用eDNA技术调查和研究中华鲟的可行性。
目前,利用eDNA 对长江鱼类进行研究已有不少探索[21],其中已有学者利用eDNA 方法研究中华鲟的分布特征。如探讨宜昌—南京江段中华鲟eDNA 的季节性分布特征[22]、长江口特定区域内中华鲟的种群特征[23],以及宜昌江段中华鲟的月度变化特征[24]等。由于中华鲟洄游时大部分在中下层水域游动,不同水层中华鲟eDNA 的丰度可能因此有差异,目前有关在长江水体中分层采集中华鲟eDNA 的研究鲜有报道,在长江流域不同区域同时开展中华鲟eDNA 采样及丰度对比分析的研究也少见。笔者筛选评估了文献报道的中华鲟eDNA 引物,并用筛选后的特异性引物扩增了从中华鲟产卵场(宜昌江段)至长江入海口的4 个点位的中华鲟eDNA,分析了不同点位以及不同水层中检测到的中华鲟eDNA 的差异,以期为后续基于eDNA 开展中华鲟种群研究提供参考。
1 材料与方法
1.1 组织样品DNA 提取
中华鲟、长江江豚(Neophocaena asiaeorientalis)、达氏鲟(Acipenser dabryanus)的肌肉组织采集自中国水产科学研究院长江水产研究所,青鱼(Mylopharyngodon piceus)、草鱼(Ctenopharyngodon idella)、鲢(Hypophthalmichthys molitrix)和鳙(Aristichthys nobilis)购自市场。取保存完好的上述7 种动物的肌肉组织30 mg,采用TIANGEN 公司的海洋动物组织基因组DNA 提取试剂盒(离心柱型),依据使用说明书分别提取动物基因组DNA,主要步骤如下:56 ℃水浴条件下加入缓冲液溶解组织样品,溶解完全后加入后续缓冲液和无水乙醇处理,转移至吸附柱上加多种缓冲液离心,最后滴加洗脱缓冲液TE 洗脱DNA,将所得DNA 样品保存于−80 ℃备用,作为下一步特异性引物筛选的模板。
1.2 特异性引物筛选
以“中华鲟”“达氏鲟”“引物”“环境DNA”“eDNA”“基因”“扩增”等为关键词,检索中国知网(CNKI)和Web of Science 文献数据库,得到4 组中华鲟特异引物(表1),对这些引物进行扩增特异性验证及筛选,以供建立中华鲟eDNA 扩增方法参考。

表1 文献发表的中华鲟基因扩增引物Table 1 Literature published on Acipenser sinensis gene amplification primers
合成表1 中引物,以江豚、中华鲟、达氏鲟和青鱼、草鱼、鲢、鳙组织提取到的DNA 样品为模板,用不同的引物进行PCR 扩增,通过凝胶电泳比较分析PCR 产物特异性。使用2×Fast HiFi PCR MasterMix(GeneZe)进行PCR 扩增,25 µL 扩增体系包括稀释成50 ng/µL 的模板1 µL,每个引物各0.5 µL(10µmol/L),12.5 µL 的2×Fast HiFi PCR MasterMix。反应条件为94 ℃预变性5 min。热循环包括:94 ℃变性30 s;参照不同引物报道的扩增条件,选择对应的退火温度,引物1~4 分别为50、56、55、50 ℃,扩增30 s;72 ℃延伸1 min,共进行35 个循环;最终72℃延伸5 min。将PCR 产物用1.5%~2.0%的琼脂糖凝胶检测,根据所测PCR 产物的电泳图谱和回收到的样品浓度对这4 组引物进行特异性检测。
1.3 环境DNA 样品采集
2022 年9 月,选择长江流域中有中华鲟和长江江豚调查报道的4 个相关区域进行eDNA 样品的断面采样。采样断面包括长江中游葛洲坝下中华鲟产卵场附近的宜昌江段区域〔断面南侧采样点(111.293 7°E,30.681 1°N)、断面北侧采样点(111.295 5°E,30.683 3°N)〕,长江中游岳阳市洞庭湖湖口附近区域〔断面西侧采样点(113.102 1°E,29.404 2°N)、断面东侧采样点(113.105 4°E,29.399 7°N)〕,长江中下游分界九江市鄱阳湖湖口附近区域〔断面西侧采样点(116.217 8°E,29.745 8°N)、断面东侧采样点(116.221 6°E,29.744 7°N)〕,以及上海崇明岛和长兴岛之间靠近长江入海口附近区域〔断面南侧采样点(121.737 5°E,31.422 8°N)、断面北侧采样点(121.761 7°E,31.455 5°N)〕。4 个采样点的具体分布见图1。

图1 4 个采样区域特征和采样点位分布Fig.1 Characteristics of four sampling areas and distribution of sampling points
在每个采样断面分左右岸分别使用采水器采集水下0.5 m 作为表层水样和水下15 m 作为底层水样,在每个采样点采集1 份5 L 水样,采集器在使用前进行漂洗和消毒,采集人员佩戴一次性手套,取样后更换。共计采集16 个点位的eDNA 样品,具体点位编号和命名:Y1(宜昌北侧表层),Y2(宜昌北侧底层),Y3(宜昌南侧表层),Y4(宜昌南侧底层);D1(洞庭湖湖口东侧表层),D2(洞庭湖湖口东侧底层),D3(洞庭湖湖口西侧表层),D4(洞庭湖湖口西侧底层);P1(鄱阳湖湖口东侧表层),P2(鄱阳湖湖口东侧底层),P3(鄱阳湖湖口西侧表层),P4(鄱阳湖湖口西侧底层);S1(长江入海口北侧表层),S2(长江入海口北侧底层),S3(长江入海口南侧表层),S4(长江入海口南侧底层)。水样使用真空泵现场抽滤,将5 L 水样 的eDNA 抽滤至1 张0.45 µm 孔径的PES 滤膜(Pall Corporation)上,过滤同样体积的纯净水作为阴性对照。将滤膜装于5 mL 冻存管中,冷冻运输回实验室,储存于−80 ℃超低温冰箱,之后尽快进行DNA 提取。
1.4 环境DNA 提取和PCR 扩增
使用QIAGEN 公司的DNeasy® PowerWater®Kit 试剂盒提取滤膜DNA 样本,具体过程:将5 L 水样过滤得到的滤膜剪裁,加入裂解液后在涡旋振荡器上振荡,离心取上清液加入后续溶液处理,转移至离心柱上,离心柱用多种缓冲液洗涤后加入100 µL的洗脱液洗脱得到eDNA 样品,将提取的eDNA 样品置于−80 ℃冷冻保存。不同采样点的样品分开保存,避免交叉污染。
以4 个点位提取的eDNA 样品为模板,选取经组织样品筛选得到的特异性较高的引物,用相同条件扩增,同样取50 ng 的eDNA 样品作为模板,使用步骤2 中特异性引物的扩增体系和扩增条件进行PCR 扩增。PCR 产物使用TaKaRA MiniBEST Agarose Gel DNA Extraction Kit Ver.4.0(TaKaRa)胶回收试剂盒回收目的片段。
1.5 测序和数据分析
已有研究表明,根据中华鲟基因设计的引物可以很好地区分中华鲟和长江中的常见鱼类,但是会检测到一些亲缘较近的鲟鱼,可以通过测序将不同种类的鲟鱼区分出来[22]。本研究采用高通量测序的方法检测中华鲟的eDNA,有效避免长江中其他鲟类eDNA 的干扰。
将成功扩增的片段送至生工生物工程(上海)股份有限公司,连接测序接头建库后,通过2%琼脂糖凝胶电泳检测文库大小,使用Qubit3.0 荧光定量仪将所有样本按照1∶1 的等体积混合进行文库浓度测定。使用Illumina Miseq™平台(美国)进行高通量测序,测序原始数据文件经碱基识别(base calling)分析转化为原始测序序列(sequenced reads)。去除引物接头序列测序后,将得到的PE reads 根据overlap关系进行拼接,将成对reads 拼接成1 条序列,根据各样本barcode 序列和引物序列拼接后数据中分割出各样本数据,并校正序列方向,区分样本后,对序列质量进行质控、过滤,最后得到各样本测序结果。
操作分类单元(OTUs)是在系统发生学或群体遗传学研究中,为了便于分析人为给某一个分类单元(品系、属、种、分组等)设置的统一标志。从各样本测序结果中提取非重复序列,所有样本去冗余序列合并后去除没有重复的单序列;按照97%相似性对非重复序列进行OTUs 聚类,在聚类过程中去除嵌合体,最终得到OTUs 的代表序列。将所有优化序列比对至OTUs代表序列,选出与代表序列相似性在97%以上的序列,从而完成OTUs 聚类。利用BLAST(basic local alignment search tool)将序列与NCBI(National Center for Biotechnology Information)数据库(https://www.ncbi.nlm.nih.gov/)进行比对,筛选出序列的最佳比对结果,并对比对结果进行过滤,以相似度大于97%的序列注释OTUs 以用于后续分类,进一步验证引物的特异性,分析中华鲟eDNA 分布特征。
2 结果
2.1 引物特异性评估
通过琼脂糖凝胶电泳对不同引物的扩增产物进行比较,对比检测结果发现,表1 中的2 号引物的特异性最强,该引物的巢氏PCR 第1 轮扩增结果见图2。通过观察不同组织在100~600 bp 范围内的扩增产物可以发现,使用其外引物即可得到特异性的鲟类PCR 产物(第1~4 泳道),扩增条带大小与2 号引物第1 轮扩增产物310 bp[22]大小相吻合。由图2可知,中华鲟肌肉和肝脏组织扩增的PCR 产物条带(第1 泳道和第2 泳道)最清晰;达氏鲟的肌肉和肝脏组织也能被扩增出较为明显的产物条带(第3 泳道和第4 泳道),但不如中华鲟的特异性强;而2 号引物对于江豚和青鱼、草鱼、鲢、鳙组织的基因扩增条带很模糊,说明2 号引物适合于鲟类基因的扩增,特别对中华鲟基因的扩增效果良好。

图2 长江中华鲟引物特异性评估2 号引物凝胶电泳图谱Fig.2 Primer specificity evaluation gel electrophoresis pattern of primer No.2 of Chinese Sturgeon
2.2 环境DNA 提取、扩增和测序
将所得滤膜样品进行DNA 提取后,成功提取总计16 个点位的eDNA 样品。以提取的环境样品为模板,使用筛选得到的2 号引物,成功扩增回收纯化得到中华鲟的目的基因片段。样品测序的稀释曲线见图3。16 条曲线对应16 个采样点位编号,随测序得到的reads 数增加,各样品OTUs 种类数均到达平台期,判断本次测序深度适宜。
图3 不同点位eDNA 测序样品的Alpha 指数稀释性曲线Fig.3 Alpha exponential dilution curves of sequenced samples at different sampling points
2.3 eDNA 检测到的中华鲟分布情况
将测序得到的序列进行聚类得到OTUs 后,通过比对NCBI 数据库对序列进行注释,成功注释出4 种长江现有鲟类,分别是中华鲟、达氏鲟、杂交鲟(Acipenser_dabryanus×Acipenser_schrenckii)和史氏鲟(Acipenser schrenckii)。各点位获得的鲟类OTUs中所含的reads 数量见图4 和表2。
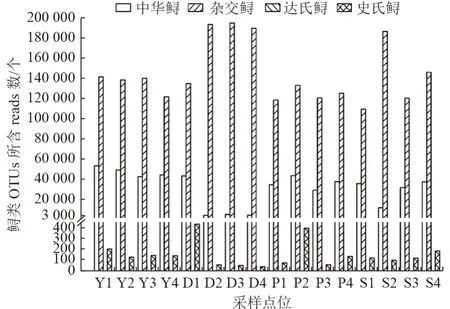
图4 各采样点位检测获得的鲟类OTUs 所含reads 数Fig.4 Number of reads contained in sturgeon OTUs detected at each site

表2 各点位检测到的鲟类OTUs 所含reads 数Table 2 Number of reads contained in sturgeon OTUs detected at each site 个
由表2 可以看出,在16 个点位中,达氏鲟仅在个别点位有少量检出,史氏鲟在各点位都有少量检出,各点位的杂交鲟reads 检出量最多。中华鲟reads 在各点位均有检出,其中宜昌江段断面最多,洞庭湖湖口断面最少,16 个点位中,Y1 点位水样中中华鲟reads 检出量最高,D2 点位水样的中华鲟reads 检出量最低。就4 个区域来说,宜昌江段断面的中华鲟reads 数量最多,每个样品平均检出46 767个;其次是鄱阳湖湖口断面,平均检出35 607 个;最少的是洞庭湖湖口断面,平均检出13 500 个。各区域相比,长江入海口断面相比洞庭湖口断面平均多111.07%;鄱阳湖湖口断面与长江入海口断面相比平均多24.96%;宜昌江段断面与鄱阳湖湖口断面相比平均多31.34%。最多的宜昌江段断面数量是最少的洞庭湖湖口断面数量的3.46 倍。
同时,对比表层和底层样品中测得的中华鲟eDNA 数据(图5)可知,洞庭湖湖口断面检出的中华鲟eDNA 表层比底层总计多5 倍以上,表层和底层的差异最为明显;宜昌江段断面两侧点位检出的中华鲟eDNA 表层与底层相差2.87%,2 种水深检出的eDNA 差异较小;鄱阳湖湖口断面表层比底层总体上少21.99%,是所有区域中唯一表层比底层检出中华鲟eDNA 少的区域;长江入海口断面表层比底层总计多了38.7%,表层中华鲟eDNA 的检出量较高。从4 个断面整体上看,表层水样中检出的中华鲟eDNA 比底层水样多了19.13%,其中,部分断面表层和底层水样间差异十分明显,而有的断面不同水层检出的eDNA 差异则较小。可以看出在大多数断面,不同水深条件下中华鲟eDNA 的检出量存在一定差别。

图5 不同点位表层和底层水样检出中华鲟eDNA 情况Fig.5 eDNA of Chinese Sturgeon detected in the surface and bottom water samples at different sampling points
3 讨论
3.1 eDNA 检测中华鲟分布的可行性及其优势
中华鲟的稳定有效监测是保护中华鲟的重要手段之一,在目前长江大保护[27]和10 年禁渔的背景下[28],要利用中华鲟组织样本对中华鲟的遗传多样性、分布情况或者种群特征进行研究,只能依靠偶然获得的少量个体样本[13],该方法可以得到的序列遗传信息较少,且样品来源不稳定。而使用eDNA 对中华鲟[29]进行无损且高效的检测是行之有效的方法。
利用eDNA 可以调查生物多样性[30]或者珍稀动物[31],eDNA 已经用于大范围检测鱼类种群分布[32],甚至用于研究大型水生动物鲸鲨的遗传多样性[33]。我国也有利用eDNA 调查长江上游珍稀鱼类[34]和长江中下游旗舰物种江豚的分布特征[35]等成功案例,前人研究为使用eDNA 监测中华鲟奠定了一定的基础[22,24]。本研究选取了从葛洲坝到入海口的长距离江段中的历史产卵场、两大通江湖泊湖口和长江入海口4 个典型区域的断面,分表、底两层进行取样,在时间上选取了临近中华鲟繁殖期之前,中华鲟向葛洲坝下产卵场移动这一中华鲟活动较为活跃的9 月进行采样,可以在一定程度上揭示繁殖期前的中华鲟种群分布情况。
3.2 eDNA 检测到的中华鲟分布情况
研究中,通过组织样品筛选得到了有较高特异性的中华鲟基因扩增引物,并成功从4 个采样点的表层和底层的环境样品中,扩增得到中华鲟的目的基因序列,表明筛选出的特异性引物能有效检测中华鲟的eDNA。除成功扩增出中华鲟目的基因序列外,测序还得到了之前报道存在于长江中的杂交鲟的大量eDNA 和少量的史氏鲟以及极少的达氏鲟eDNA,检测结果与长江历史调查中的鲟类物种一致[36]。测序结果显示杂交鲟在各点位eDNA 的检出量均明显高于中华鲟,表明作为外来物种的杂交鲟可能在长江中广泛存在。前人对长江鱼类的研究中也有不少中华鲟的观测记录,其中2017 年7—10 月底在长江口区域,皆检测到了中华鲟幼体[37];在对宜昌葛洲坝下游中华鲟产卵场的调查中,也曾长期监测到中华鲟在该水域的活动[38];对洞庭湖和鄱阳湖的鱼类调查研究中,也观测到中华鲟的出现[39-40]。这些报道均与本研究揭示的4 个区域检测到的中华鲟eDNA 相一致,表明了eDNA 技术在检测中华鲟分布特征方面的可行性。
本次调查时间为9 月,检出的中华鲟序列在宜昌江段丰度最高,推测是因为中华鲟即将进入繁殖期(11 月)[6],开始频繁出现在宜昌;鄱阳湖口中华鲟序列数高于入海口,可能是因为中华鲟游入长江产卵时间为6—8 月,调查时中华鲟已经大部分游过长江口。从调查点位表层和底层样品的检出结果对比可以发现,中华鲟eDNA 在表层水样和底层水样的检出有明显差别,这可能是因为中华鲟在洄游过程中,多分布于江水的中下层,偶尔移至上层[7],因此不同水深检出中华鲟eDNA 的差异性也值得关注。而在对于eDNA 采样的空间结构相关研究中也发现,在空间上更全面地采样可以揭示物种更加精细的分布模式[41]。建议在今后的长江中华鲟eDNA 调查中可采用混合或者立体采样,这样比单一空间位点的采样能够更加全面地采集到中华鲟的信息。
本文通过水生动物组织筛选得到特异性引物,从测序结果上看,其可能是适用于长江现有鲟类的通用引物,仅从产物电泳结果上看不易严格区分鲟类,但可以通过测序将长江中的鲟类区分开。后续研究可以通过继续开发新的特异引物,或者依靠长江鲟类分布的区域性特征和区域的历史调查结果,更加精确地区分引物,并通过高通量测序对长江鲟类进行准确识别。
4 结论
(1)从4 对中华鲟相关引物中筛选得到了有较高特异性的中华鲟基因扩增引物,从环境样品中检测出中华鲟eDNA 并成功测序验证。
(2)使用筛选得到的特异性引物共测得了包含中华鲟在内的4 种长江鲟类基因,通过测序很好地区分了4 种鲟类,其中杂交鲟的eDNA 检出量最高,中华鲟其次,达氏鲟的eDNA 检出量最低。作为长江外来物种的杂交鲟可能在长江中广泛存在,中华鲟eDNA 的检测结果也与之前报道的中华鲟的形态学观察较为吻合。
(3)中华鲟eDNA 在宜昌江段检出量最高,可能是因为中华鲟即将进入繁殖期,开始出现在宜昌河段准备繁殖。中华鲟eDNA 在表层水样和底层水样的检出量有一定差别,后续中华鲟eDNA 调查采用混合或者立体采样可能会比单一空间位点的采样能够得到更加丰富的eDNA 信息。还可以通过开发新的特异引物,或者与其他调查方法相结合的方式开展更全面的调查研究,以保护中华鲟。
