木姜叶柯SRAP-PCR反应体系优化、引物筛选及验证
2024-02-09罗超颖严武平汪玉玲王梦醒彭莎廖红程建峰





摘要:【目的】建立一套适用于木姜叶柯SRAP-PCR的最佳反应体系,筛选出应用于木姜叶柯种质资源遗传多样性分析的多态性引物,为后续木姜叶柯保护遗传学研究及其开发利用提供科学依据。【方法】采用单因素试验和正交试验相结合的方法,对SRAP-PCR扩增效果的主要影响因素(模板DNA用量、dNTPs浓度、Taq DNA聚合酶用量和引物浓度)进行优化。利用最佳反应体系筛选出适用于木姜叶柯的SRAP多态性引物。【结果】优化获得木姜叶柯SRAP-PCR最佳反应体系(20.00 μL):10×PCR Buffer 2.00 μL,Taq DNA聚合酶1.50 U,dNTPs 0.25 mmol/L,引物浓度0.600 μmol/L和模板DNA 30.00 ng。各因素对木姜叶柯PCR扩增效果影响程度排序:引物浓度gt;dNTPs浓度gt;Taq DNA聚合酶用量gt;模板DNA用量。应用优化后的SRAP-PCR最佳反应体系和筛选到的20对多态性引物对3个居群的24个木姜叶柯样本进行SRAP-PCR扩增,结果发现4对SRAP引物从3个野生木姜叶柯居群24个样本中共扩增出38个位点,每对引物平均扩增出9.5个位点,其中多态性位点共21个,多态性比率为55.26%。3个木姜叶柯居群的平均等位基因数(Na)、平均有效等位基因数(Ne)、Nei’s基因多样性(H')和Shannon’s信息指数(I)分别为1.55、1.30、0.18和0.27,总遗传多样性(Ht)、居群内遗传多样性(Hs)和遗传分化系数(Gst)分别为0.18、0.11和0.36,基因流(Nm)为0.90(lt;1.00),说明3个居群间存在一定程度的基因交流,但基因交流程度有限。【结论】木姜叶柯SRAP-PCR扩增效果对引物浓度最敏感。建立木姜叶柯的SRAP-PCR最佳反应体系和筛选到的SRAP引物扩增条带清晰稳定,多态性丰富,能较好地反映
出不同木姜叶柯个体和居群间的亲缘关系,可应用于木姜叶柯种质资源的遗传多样性和遗传结构分析。
关键词:木姜叶柯;SRAP-PCR;反应体系;优化;引物筛选;遗传多样性
中图分类号:S567.19文献标志码:A 文章编号:2095-1191(2024)11-3231-11
Optimization of SRAP-PCR reaction system, primer screeningand verification for Lithocarpus litseifolius
LUO Chao-ying1, YAN Wu-ping1, WANG Yu-ling1,2, WANG Meng-xing1, PENG Sha1,LIAO Hong1, CHENG Jian-feng1*
(1School of Agricultural Sciences, Jiangxi Agricultural University, Nanchang, Jiangxi 330045, China;2School of Breeding and Multiplication( Sanya Institute of Breeding and Multiplication), Hainan University, Sanya,Hainan 572025, China)
Abstract:【Objective】To establish an optimal reaction system applicable to SRAP-PCR of Lithocarpus litseifolius, and screen out polymorphic primers for the genetic diversity analysis of L. litseifolius germplasm resources, thereby pro‐viding scientific basis for the subsequent studies on conservation genetics and its development and utilization.【 Method】The combination of single-factor experiment and orthogonal experiment was adopted to optimize the main influencing fac‐tors( template DNA amount, dNTPs concentration,Taq DNA polymerase amount and primer concentration) affecting the amplification effect of SRAP-PCR. Using optimal reaction system, SRAP polymorphic primers suitable for L. litseifoliuswere screened.【 Result】The optimal reaction system for SRAP-PCR of L. litseifolius( 20.00 μL) was obtained as follows:10× PCR Buffer 2.00 μL,Taq DNA polymerase 1.50 U, dNTPs 0.25 mmol/L, primer concentration 0.600 μmol/L and tem‐plate DNA 30.00 ng. The degree of influence of each factor on the PCR amplification effect of L. litseifolius was ranked as follows:primer concentration gt; dNTPs concentration gt;Taq DNA polymerase amount gt; template DNA amount. Using the optimized SRAP-PCR optimal reaction system and the selected 20 pairs of polymorphic primers for SRAP-PCR amplifica‐tion of 24 samples from 3 populations of L. litseifolius, it was found that 4 pairs of SRAP primers amplified a total of 38 loci from the 24 samples of the 3 wild populations, with an average of 9.5 loci per pair of primers. Among them, 21 loci were polymorphic, with a polymorphic rate of 55.26%. The average number of alleles(Na), average effective number of alleles(Ne), Nei’s gene diversity(H'), and Shannon’s information index(I) of the 3 populations of L. litseifolius were 1.55, 1.30, 0.18 and 0.27 respectively. The total genetic diversity(Ht), within-population genetic diversity(Hs), and genetic differentiation coefficient(Gst) were 0.18, 0.11 and 0.36 respectively. The gene flow(Nm) was 0.90(lt;1.00), in‐dicating that there was a certain degree of gene exchange among the 3 populations, but the degree of gene exchange was limited. 【Conclusion】The amplification effect of SRAP-PCR of L. litseifolius is most sensitive to primer concentration. The established optimal reaction system of SRAP-PCR of L. litseifolius and the selected SRAP primers produce clear and stable amplification bands with rich polymorphism, which can better reflect the genetic relationships among different indi‐viduals and populations, and can be applied to the analysis of genetic diversity and genetic structure of L. litseifolius germ‐plasm resources.
Key words:Lithocarpus litseifolius; SRAP-PCR; reaction system; optimization; primer screening; genetic diversity
Foundation items:National University Students Innovation and Entrepreneurship Training Plan Project(20201041 0005X); Science and Technology Research Project of Jiangxi Department of Education(GJJ20295, GJJ200445); Doc‐toral Research Start-up Foundation Project of Jiangxi Agricultural University(9232309477)
0 引言
【研究意义】木姜叶柯(Lithocarpus litseifolius)俗称甜茶,素有“树上虫草”“辐射的克星”“长寿茶”的美誉(彭勇等,2020),是壳斗科柯属高大乔木(中国科学院中国植物志编辑委员会,1997),广泛分布于我国长江以南地区,以江西与福建交界的武夷山脉分布最广(林协全等,2023)。木姜叶柯富含多种生物活性物质且安全无毒,兼具药、茶、糖3种功能(万蓝婷, 2022),但目前在开发利用上基本依靠野生资源,过度采挖导致木姜叶柯野生资源逐年减少,发掘优良种质资源,对缓解木姜叶柯供需矛盾问题已势在必行。评估遗传变异对于保护和开发该物种的遗传资源至关重要。SRAP分子标记技术是通过独特的双引物设计扩增开放阅读框的特定区域,多态性取决于不同个体及物种的内含子、启动子与间隔区长度不同,具有操作简单、高共显性、多态性高、在基因组中分布均匀等优点,被广泛应用于植物的遗传多样性分析和遗传图谱构建等研究(Yan et al.,2019;毛立彦等,2023;孙阳阳等,2023;丁海麦等,2024)。因此,探究木姜叶柯SRAP-PCR最佳反应体系,并应用于木姜叶柯遗传多样性和遗传结构分析,对木姜叶柯种质资源保护与利用具有重要意义。【前人研究进展】目前木姜叶柯的研究主要集中在对其药用功能成分提取和测定及药理功能分析等方面(彭勇等,2020),有关种质资源的研究报道较少,仅Cheng等(2016)利用11个SSR分子标记分析我国6个省(区)共11个木姜叶柯种群的遗传变异和种群结构。由于木姜叶柯种质资源相关研究报道较少,导致育种工作停滞不前,阻碍了木姜叶柯潜在药用价值的研究与开发。木姜叶柯叶片表型遗传变异丰富,不同地区间叶片的主要活性成分含量也存在丰富的变异(万蓝婷,2022;赵鹏霞等,2023)。因此,探究木姜叶柯野生种群遗传多样性和遗传结构是其种质创新和遗传改良的必由之路,也是其野生资源有效保护的重要手段。由于SRAP分子标记技术是采用独特的双引物设计扩增开放阅读框的特定区域,能全面体现不同个体间的多态性,已广泛应用在栝楼(何庆元等,2023)、黄精(李亚萍等,2023)和金线莲(黄锦春等,2023)等药用植物的遗传多样性分析。SRAP-PCR反应体系中各因子浓度或用量均会影响其扩增结果,且不同物种适宜的反应体系也不尽相同(王燕等,2007;严武平等,2018;孙秀秀等,2021;赵琦琦等,2022)。梁芳瑜等(2021)采用单因素结合正交试验优化了宽叶缬草SRAP-PCR反应体系,结果发现各因素对宽叶缬草SRAP-PCR扩增结果的影响程度排序为Taq Mixgt;Taq DNA聚合酶gt;Mg2+gt;DNA模板。胡小虎等(2022)采用单因素分析法建立独脚金的SRAP-PCR最佳反应体系,结果发现dNTPs和Taq DNA聚合酶对扩增效率的影响最大。【本研究切入点】建立一套适用于木姜叶柯的SRAP-PCR最佳反应体系是基于SRAP分子标记进行木姜叶柯种质资源遗传多样性和遗传结构分析的前提。但目前尚未见有关木姜叶柯SRAP-PCR反应体系优化及验证的相关研究报道。【拟解决的关键问题】采用单因素试验结合正交试验的方法,对影响PCR扩增效果的主要因素(模板DNA、dNTPs、Taq DNA聚合酶和引物)进行优化,建立一套适用于木姜叶柯的SRAP-PCR最佳反应体系,并应用该体系筛选适用于木姜叶柯的SRAP多态性引物,为木姜叶柯野生资源保护和遗传育种提供技术支撑。
1 材料与方法
1. 1 试验材料
供试木姜叶柯资源种质收集于江西吉安市和萍乡市,经江西农业大学程建峰教授鉴定为木姜叶柯,材料居群编号:FGS、HKC和MYX(表1)。植物基因组DNA提取试剂盒(DE-06111)购自成都福际生物技术有限公司;DL2000 DNA Marker、Taq DNA聚合酶(Mg2+ plus Buffer)和dNTP Mix均购自南京诺唯赞生物科技股份有限公司。主要仪器设备:TG-16E离心机(博科控股集团有限公司)、GS-521T一体式凝胶成像系统(上海骋克仪器有限公司)、DYY-6D型电泳仪(北京市六一仪器厂)、A300基因扩增仪(杭州朗基科学仪器有限公司)、NanoDrop 2000超微量分光光度计(ThermoFisher Scientific公司)。
1. 2 试验方法
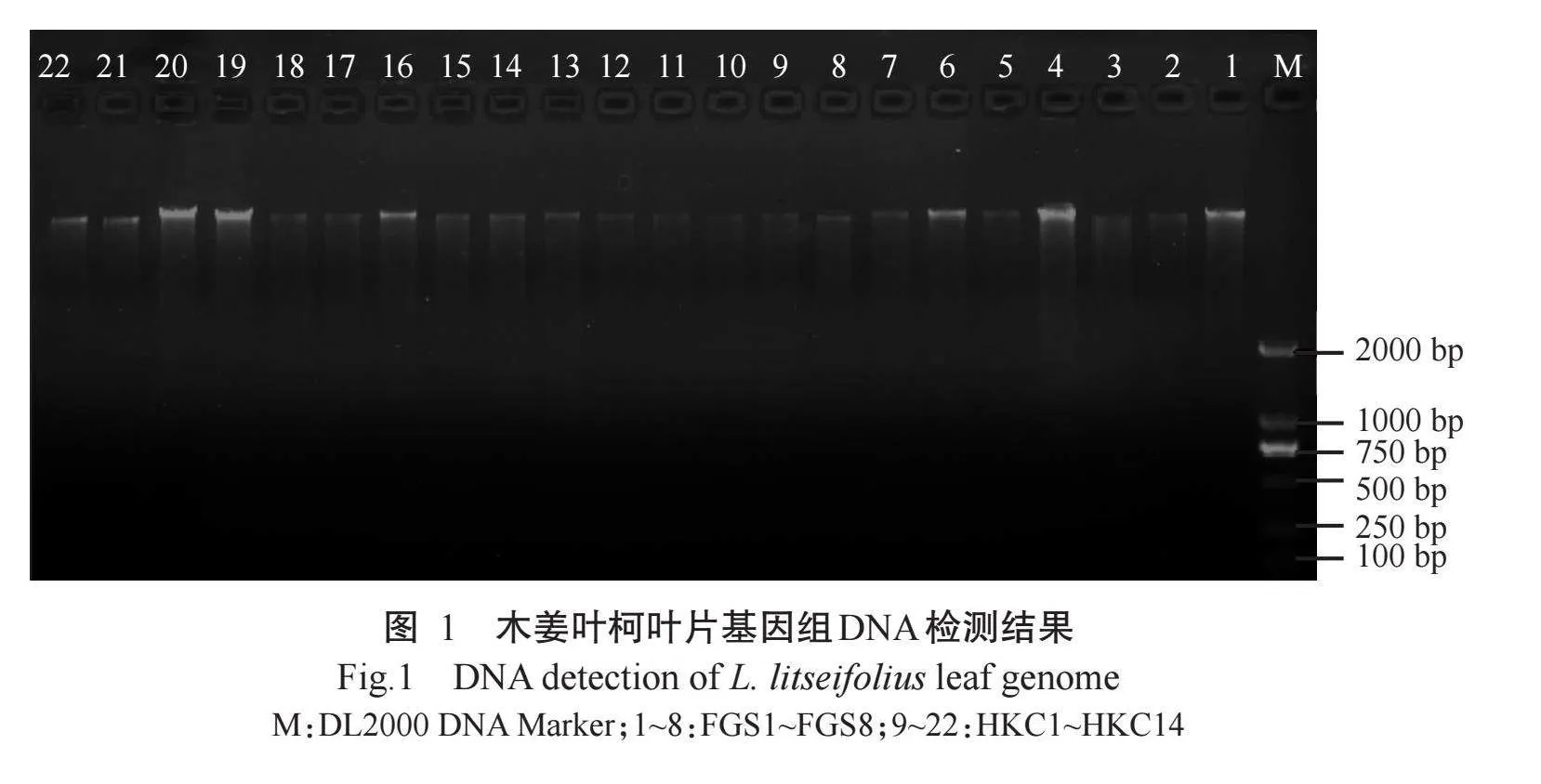
1. 2. 1 木姜叶柯基因组DNA提取 从3个居群中选取24株健康无病虫害的植株,摘取其健康嫩叶,液氮速冻带回实验室。采用植物基因组DNA提取试剂盒说明提取嫩叶基因组DNA,采用1.0%琼脂糖凝胶电泳检测其完整性,使用NanoDrop 2000超微量分光光度计评估DNA纯度和浓度。经检测合格的DNA样品置于-20 ℃冰箱保存备用。
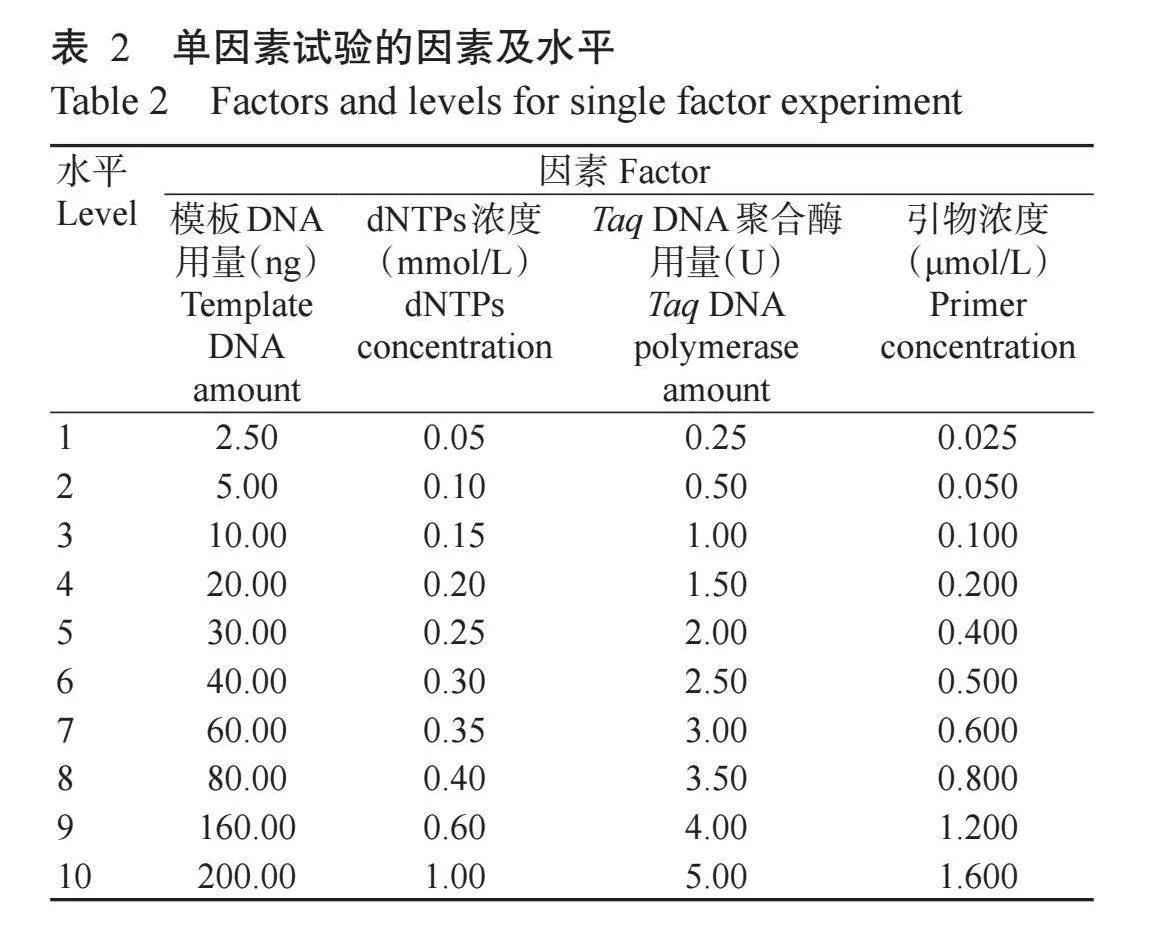
1. 2. 2 单因素试验设计 参考严武平等(2018)的方法设计单因素试验。基于实验室现有的PCR反应体系和扩增程序进行单因素试验。反应体系20.00 μL:10×PCR Buffer 2.00 μL,Taq DNA聚合酶(含Mg2+)1.50 U,dNTPs 0.20 mmol/L,引物浓度0.400 μmol/L,模板DNA 20.00 ng,ddH2O补足至20.00 μL。扩增程序:94 ℃预变性5 min;94 ℃ 1 min,35 ℃ 1 min,72 ℃ 1 min,进行5个循环;94 ℃ 1 min,退火温度(据各引物设定)1 min,72 ℃ 1 min,进行35个循环;72 ℃延伸10 min,PCR产物4 ℃保存。按照以上扩增程序和反应体系对Li和Quiros(2001)公布的96对SRAP引物进行初筛,选择扩增条带清晰且多态性良好的引物组合ME1-EM8进行反应体系优化。设计10个梯度的模板DNA用量、引物浓度、dNTPs浓度和Taq DNA聚合酶用量,开展单因素试验(表2)。采用直观法确定各因素在木姜叶柯SRAP-PCR反应体系中的适宜浓度范围。
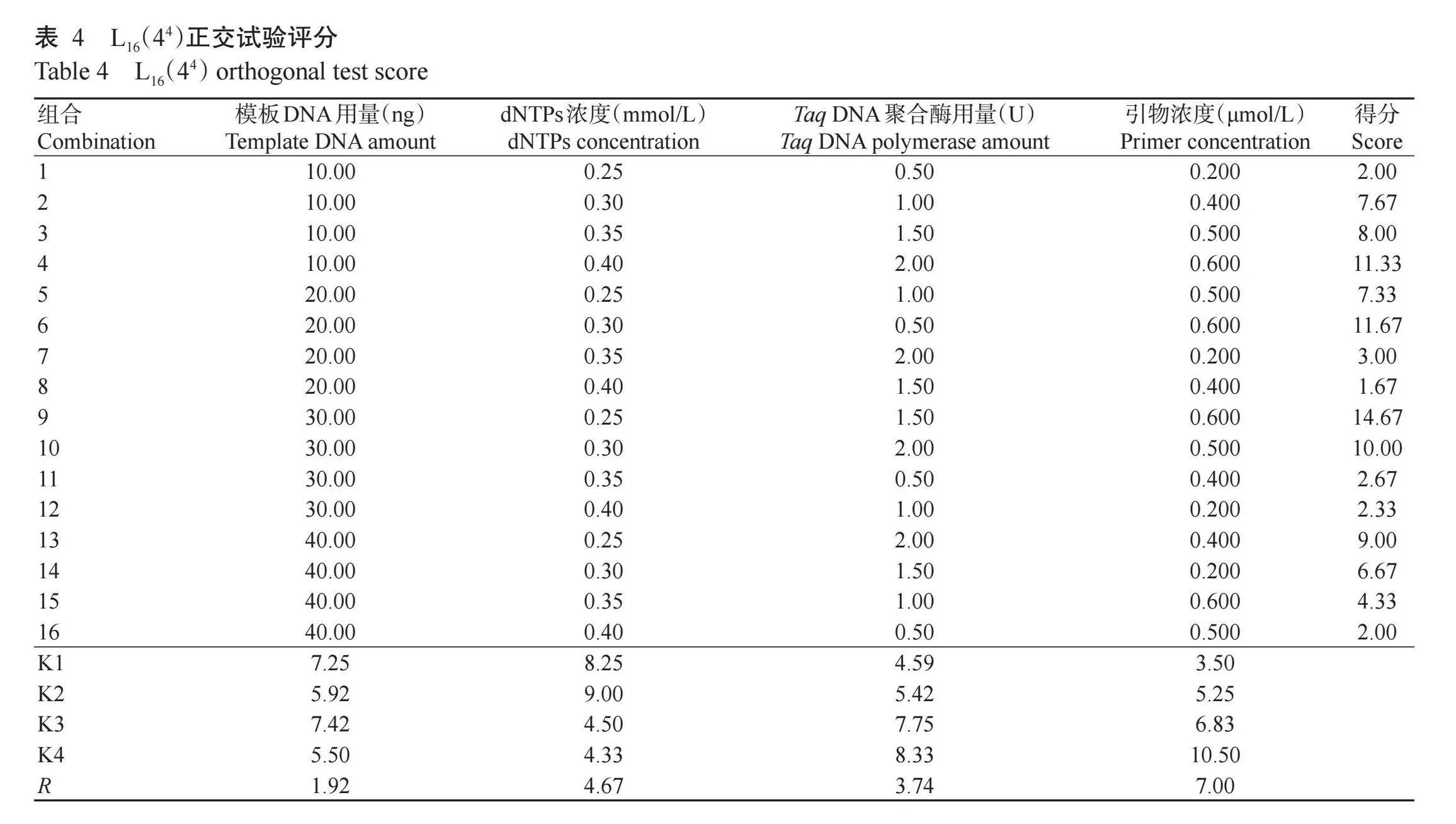
1. 2. 3 正交试验设计 基于单因素试验筛出的各因素适宜浓度范围,设计正交试验L16
(44),每个组合设3次重复。参考张尚文等(2023)的直观分析法进行评分,以扩增条带数目、扩增背景、条带清晰度和亮度等作为评分标准,满分为16分,评分越高,表示扩增效果越好。K表示某因素不同水平下的评分平均值,反映各水平的扩增效果;R表示同一因素不同处理水平评分平均值的极差,反映各因素对扩增结果的影响程度,根据评分结果确定木姜叶柯SRAP-PCR最佳反应体系,取3次重复得分的平均值。
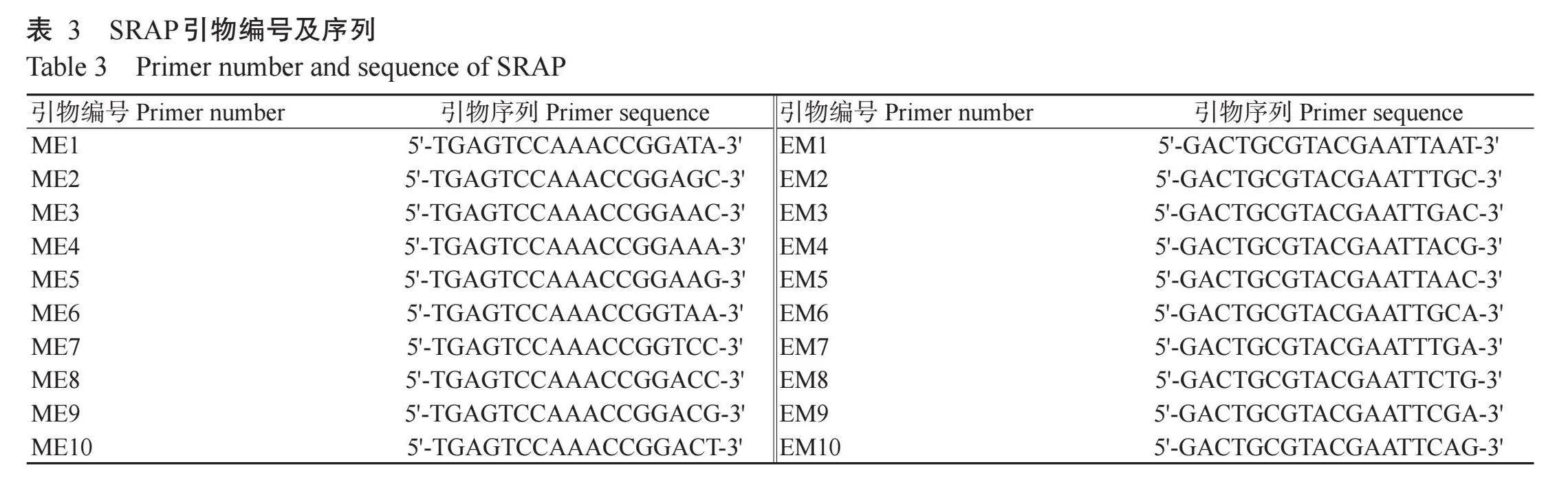
1. 2. 4 木姜叶柯SRAP多态性引物筛选 参照Li和Quiros(2001)公布的SRAP引物序列信息(表3),委托北京擎科生物科技股份有限公司合成。以FGS居群的1个样本基因组DNA为模板,利用建立的SRAP-PCR最佳反应体系对96对SRAP引物组合进行筛选,选择条带清晰度高、背景明亮、条带数量多的引物组合作为木姜叶柯SRAP多态性分析的候选引物。
1. 2. 5 木姜叶柯SRAP-PCR最佳反应体系及多态性引物稳定性验证 选择3个木姜叶柯居群(FGS、HKC和MYX)共24个样本的基因组DNA为模板,基于建立的最佳反应体系和4个候选引物(ME7-EM1、ME7-EM2、ME8-EM4和ME8-EM8)进行SRAP-PCR扩增,以验证优化后反应体系的稳定性和多态性引物的有效性。PCR产物用1.0%琼脂糖凝胶电泳检测,使用凝胶成像系统自带软件进行读带。将电泳图谱上清晰可见且可重复的条带标记为“1”,同一位点无条带标记为“0”,生成0,1矩阵。使用Pop‐gene version 1.32分析生成的0,1矩阵,以估算供试居群的遗传多样性参数。使用MEGA version 6.06的非加权配对平均法(UPGMA)计算遗传距离,并以此进行聚类分析。
2 结果与分析
2. 1 木姜叶柯基因组DNA提取及质量检测
24份木姜叶柯叶片基因组DNA提取结果如图1所示。提取的基因组DNA条带清晰,有轻微拖带和无弥散现象,经NanoDrop 2000超微量分光光度计检测OD260 nm/OD280 nm为1.80~2.00,表明提取的基因组DNA质量较好,可用于后续试验。
2. 2 模板DNA用量对木姜叶柯PCR扩增效果的影响
由图2可知,10个模板DNA用量梯度中,多数浓度均能产生扩增条带,但扩增条带在数量和清晰度等方面存在差异,当模板DNA用量低于10.00 ng,扩增条带数量不稳定且条带偏模糊,可能是模板DNA用量过少导致扩增产量低;当模板DNA用量为10.00~80.00 ng,扩增条带清晰且带形基本一致;而当模板DNA用量大于80.00 ng,可能是DNA样本中残留的某些抑制物含量增多,从而抑制了PCR扩增反应,导致条带数减少。基于经济节约的目的,在20.00 μL反应体系中选择10.00~40.00 ng的模板DNA用量进行后续正交试验。
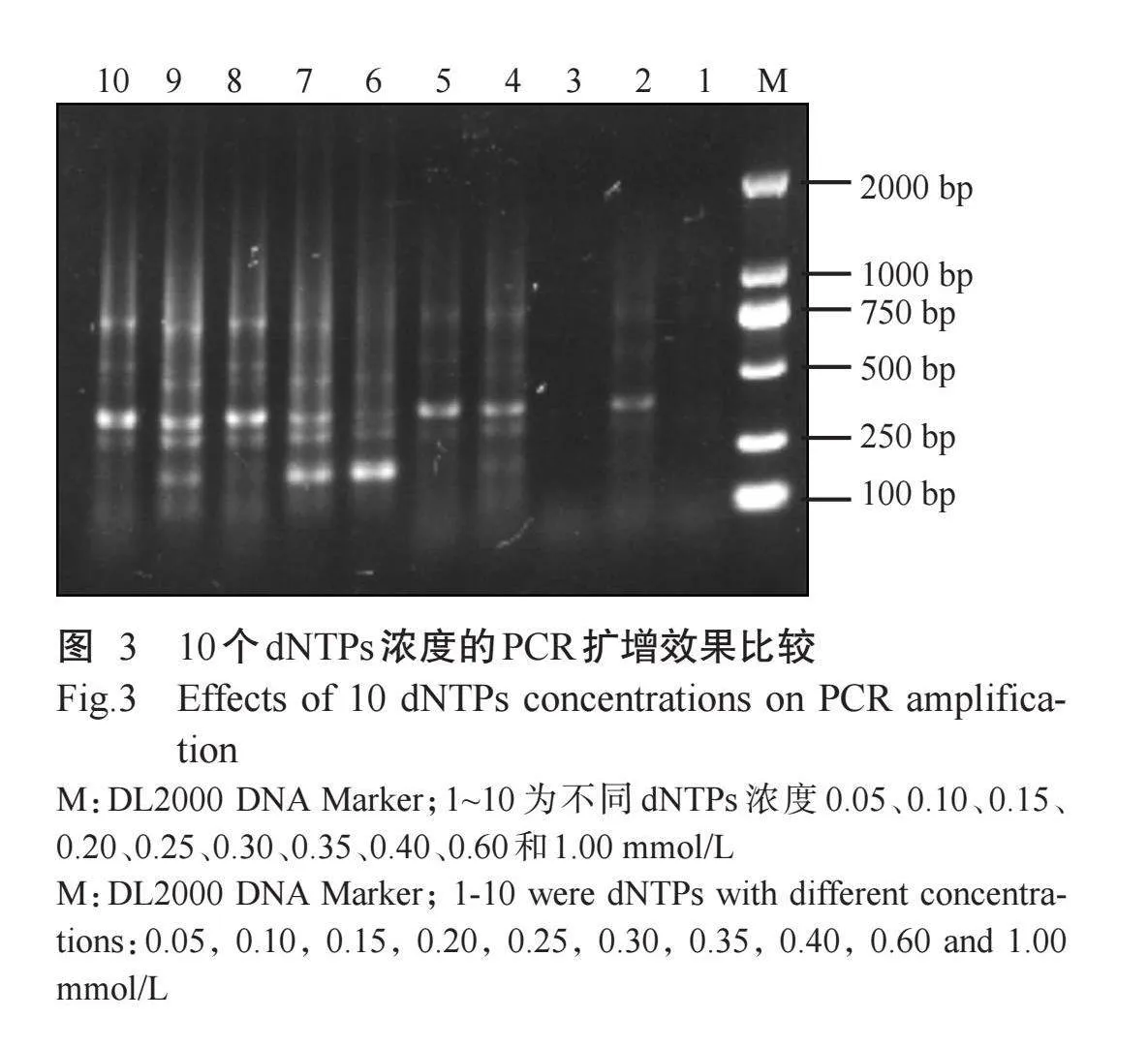
2. 3 dNTPs浓度对木姜叶柯PCR扩增效果的影响
由图3可知,当dNTPs浓度为0.05~0.20 mmol/L,扩增条带不清晰,可能是参与合成的原材料不足,导致无法充分合成产物;当dNTPs浓度为0.25~0.40 mmol/L时,扩增条带清晰且丰富;而dNTPs浓度大于0.60 mmol/L时,扩增条带又开始模糊并减少。综合评估,选择dNTPs浓度范围为0.25~0.40 mmol/L进行后续正交试验。
2. 4 Taq DNA聚合酶用量对PCR扩增效果的影响
由图4可知,当Taq DNA聚合酶用量低于0.50 U,扩增条带不完整;随着Taq DNA聚合酶用量的不断增加,扩增条带开始逐渐清晰且稳定;当Taq DNA聚合酶用量超过3.00 U后,扩增条带又开始模糊并减少。基于经济节约的目的,选择0.50~2.00 U的Taq DNA聚合酶用量进行后续正交试验。
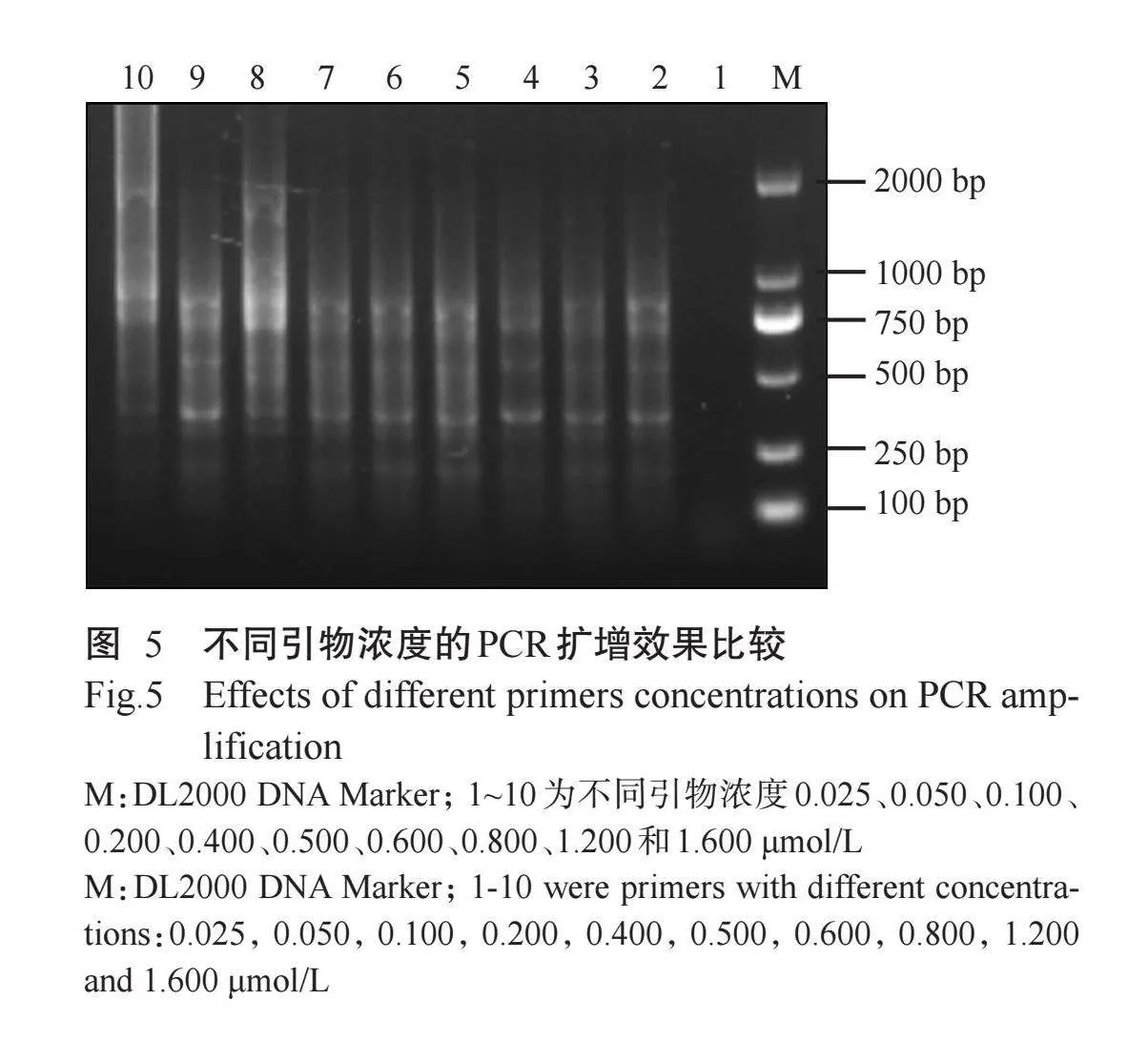
2. 5 引物浓度对PCR扩增效果的影响
由图5可知,当引物浓度低于0.20 μmol/L时,PCR扩增效果差,扩增条带少且模糊;随着引物浓度的升高,扩增条带清晰且带形趋于稳定;当引物浓度超过0.600 μmol/L时,引起了错配和非特异性扩增。因此,选择0.200~0.600 μmol/L引物浓度进行后续的正交试验。
2. 6 正交试验结果
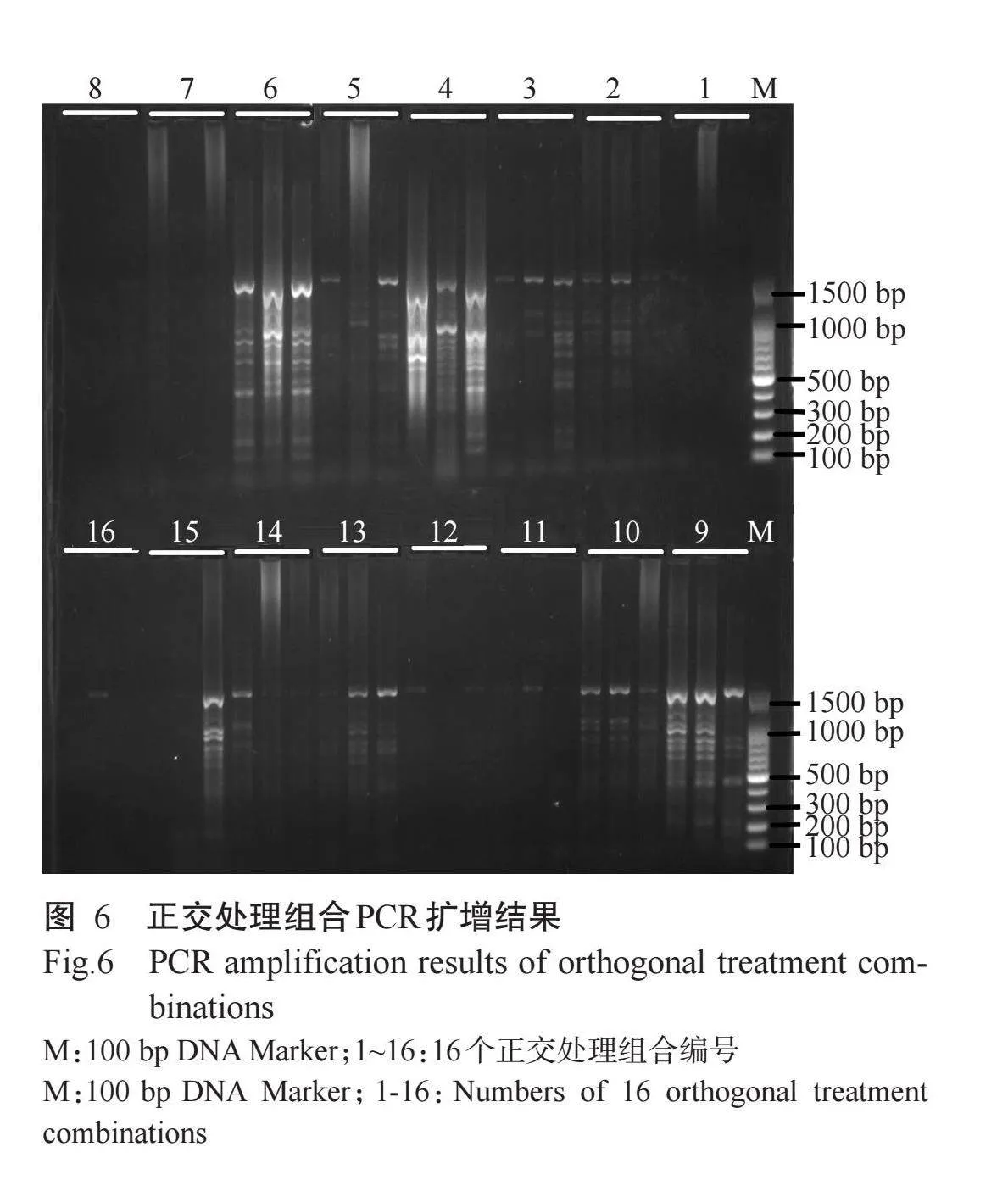
根据上述单因素试验确定的各因素适宜浓度和用量范围,选用L1(644)正交表设计正交试验,结果如图6和表4所示。虽选择的浓度均是较适宜的范围,但不同组合所产生的扩增条带质量差异显著。如组合1、组合7、组合10和组合13均未出现明显扩增条带,表明扩增效果差;而组合3、组合4、组合5和组合8的扩增条带清晰且带形一致,扩增效果最好。按照直观分析法,将条带亮度、数量、清晰度及背景混杂程度等作为评分标准,对正交试验设计的各个水平扩增效果进行评估打分,可得出组合9(20.00 μL反应体系:10×PCR Buffer 2.00 μL,Taq DNA聚合酶1.50 U,dNTPs 0.25 mmol/L,引物0.600 μmol/L,模板DNA 30.00 ng)的扩增效果最好,扩增产物清晰稳定、条带丰富。由R值可知,各因素对木姜叶柯PCR扩增效果影响程度排序:引物浓度gt;dNTPs浓度gt;Taq DNA聚合酶用量gt;模板DNA用量。由此可见,引物浓度变化对木姜叶柯SRAP-PCR反应最敏感。
2. 7 木姜叶柯SRAP多态性引物筛选
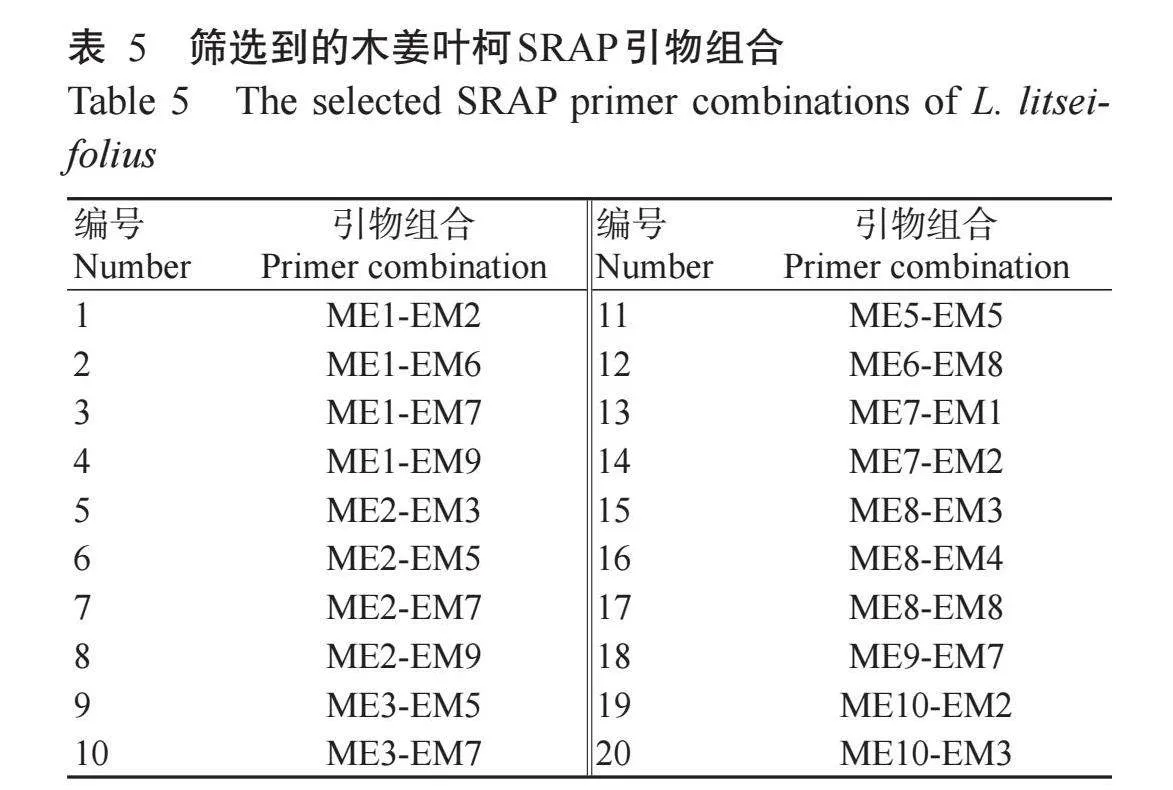
基于优化后的最佳反应体系,以1个FGS居群样本的基因组DNA为模板,对96对SRAP引物进行筛选(图7),以背景清晰度和条带数量为筛选指标,获得20对条带清晰、多态性丰富的引物组合(表5)。
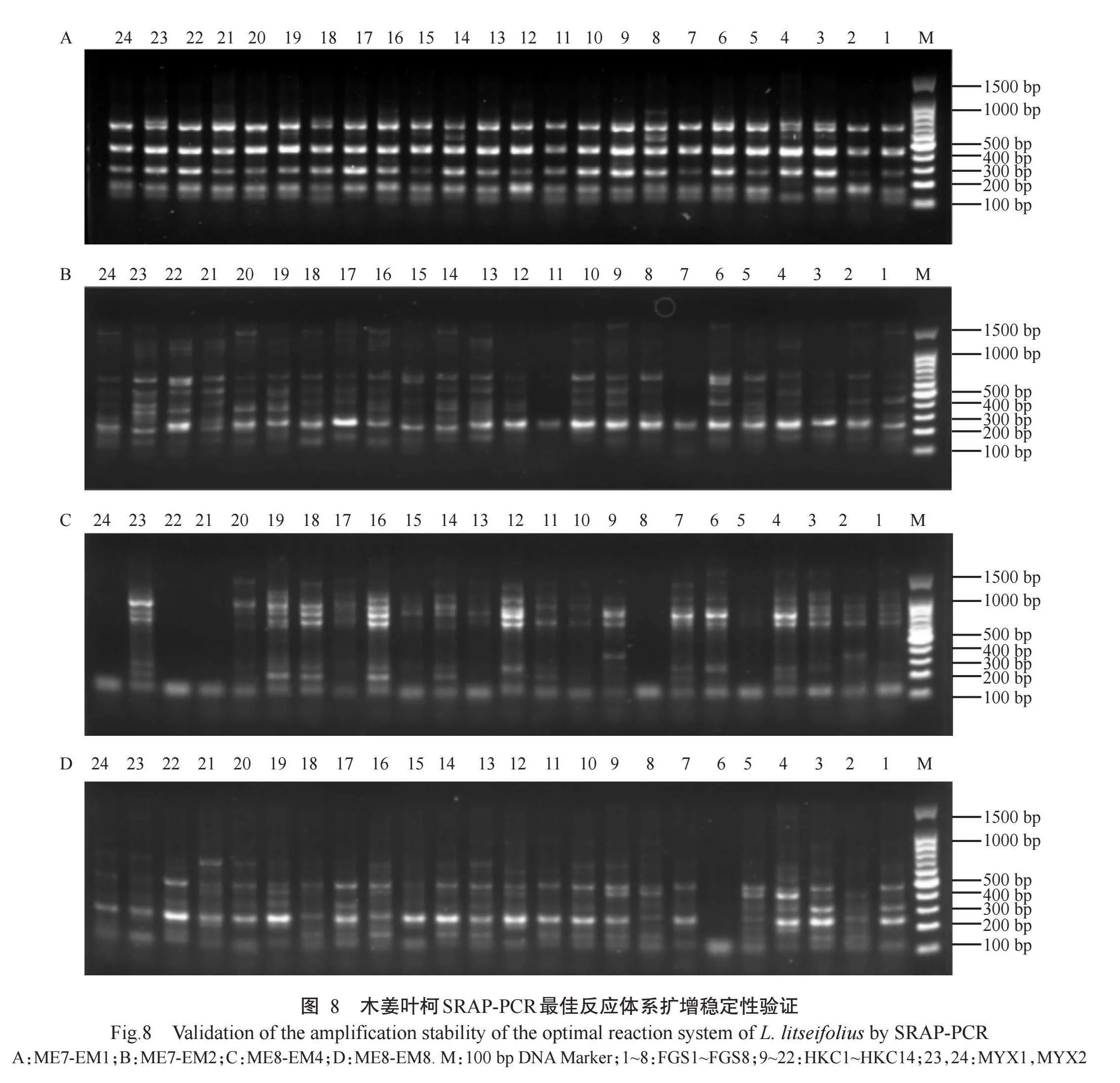
2. 8 木姜叶柯SRAP-PCR最佳反应体系稳定性验证
以地理位置相隔远和生境差异大的3个野生木姜叶柯居群(FGS、HKC和MYX)共24个样本的基因组DNA为模板,利用上述SRAP-PCR最佳反应体系及筛选的4对多态性引物(ME7-EM1、ME7-EM2、ME8-EM4和ME8-EM8)进行SRAP-PCR扩增,部分结果如图8所示。扩增的条带清晰、背景明亮、多态性丰富,表明优化后SRAP-PCR最佳反应体系能稳定扩增木姜叶柯。
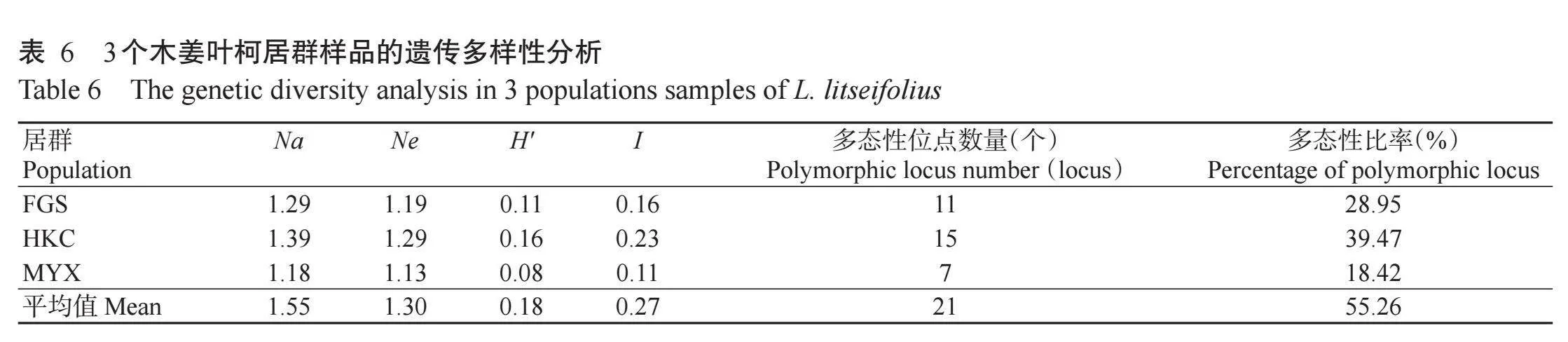
4对SRAP引物从3个野生木姜叶柯居群24个样本中共扩增出38个位点,每对引物平均扩增出9.5个位点,其中多态性位点共21个,多态性比率为55.26%。由表6可知,3个木姜叶柯居群的平均等位基因数(Na)、平均有效等位基因数(Ne)、Nei’s基因多样性(H')和Shannon’s信息指数(I)分别为1.55、1.30、0.18和0.27。基于遗传多样性参数计算出3个居群的总遗传多样性(Ht)和居群内遗传多样性(Hs),再以此计算出不同居群间的遗传分化系数(Gst)和基因流(Nm),结果显示,3个居群的Ht为0.18,Hs为0.11,Gst为0.36,表明在总的遗传变异中,居群间的遗传变异占36.00%,居群内的遗传变异占64.00%,表明在居群内和居群间均存在遗传分化,主要遗传变异存在于居群内;Nm为0.90(lt;1.00),表明供试居群间虽存在一定的基因交流,但基因交流程度有限。利用UPGMA方法,基于遗传距离对3个居群共24个样本进行聚类分析,在遗传距离为0.11时,能将24个样本分为三大类(图9),聚类结果与地理距离不相关。可见,构建的SRAP-PCR最佳反应体系和筛选到的多态性引物能较好地体现不同居群木姜叶柯的遗传多样性和遗传结构。
2 讨论
木姜叶柯是一种药食兼用的天然植物资源,应用广泛,加强木姜叶柯种质资源收集保护和评价是加快其育种产业化和开发利用步伐的前提(Chenget al., 2016)。SRAP分子标记是双引物特异扩增,具有多态性高和操作简单等优点(Yan et al., 2019),已被广泛应用在植物种质资源鉴定和遗传多样性分析等方面(姜武等,2023;王洁等,2023)。不同物种影响SRAP-PCR反应体系扩增效果的主要因素并不相同。陈兰艳等(2019)研究表明,模板DNA用量和引物浓度对郁金香SRAP-PCR扩增效率影响最大。谢秋霞等(2023)研究发现,Taq DNA聚合酶用量对雷公笋SRAP-PCR扩增效果影响最小,引物浓度和dNTPs浓度对雷公笋SRAP-PCR扩增效果影响最大。但本研究发现,一定范围内Taq DNA聚合酶和模板DNA用量的变化对木姜叶柯SRAP-PCR扩增效果的影响较小,但引物浓度对木姜叶柯SRAP-PCR 扩增效果影响最大,其次是dNTPs浓度,与海南油茶(戚华沙等,2020)和青蒿(石紫薇等,2020)的研究结果一致。引物是PCR扩增的关键,PCR产物的特异性取决于引物与模板DNA互补的程度。引物浓度低会降低SRAP-PCR反应体系的扩增效率,浓度过高则会引起错配和非特异性扩增,并易形成引物二聚体。
目前大多数分子标记PCR反应体系优化试验主要采取单因素试验或正交试验确定最佳反应体系,但单一试验方法无法综合考虑各因素间的相互作用(赵宏宇等,2008;严武平等,2018)。例如,单因素试验虽能迅速地确定PCR反应体系各组分的最适浓度范围,但忽略了PCR反应体系中各组分之间相互作用的影响。正交试验虽具有分散可比和齐整均衡等特点,可弥补各因素之间的相互影响,但往往选取的各组分浓度范围并非最优,导致得不到最佳扩增效果。本研究先使用单因素试验确定反应体系各组分的适宜浓度范围,再结合正交试验,建立了一套木姜叶柯SRAP-PCR最佳反应体系(20.00 μL):10×PCR Buffer 2.00 μL,Taq DNA聚合酶1.50 U,dNTPs 0.25 mmol/L,引物浓度0.600 μmol/L和模板DNA 30.00 ng。最后,通过3个不同木姜叶柯居群的24个样本对SRAP-PCR最佳反应体系和多态性引物进行稳定性验证,结果表明,优化后的最佳反应体系和筛选到的SRAP引物能很好地体现木姜叶柯种质资源间的遗传关系和物种的遗传多样性,可应用于后续木姜叶柯种质资源遗传多样性分析。
3 结论
本研究通过单因素试验结合正交试验的方法,优化并建立了一套适用于木姜叶柯SRAP-PCR的最佳反应体系(20.00 μL):10×PCR Buffer 2.00 μL,Taq DNA聚合酶1.50 U,dNTPs 0.25 mmol/L,引物浓度0.600 μmol/L和模板DNA 30.00 ng,引物浓度对该反应体系扩增效果影响最大。基于优化后的反应体系,筛选得到20对高多态性SRAP引物,为木姜叶柯种质资源的开发利用及种质创新提供技术支撑。
参考文献((References)):
陈兰艳,马秀花,唐楠,侯志强,巨秀婷. 2019. 郁金香SRAP-PCR体系建立与优化[J]. 分子植物育种,17(20):6711-6717. [Chen L Y,Ma X H,Tang N,Hou Z Q,Ju X T. 2019. Establishment and optimization of SRAP-PCR sys‐tem for tulip[J]. Molecular Plant Breeding,17(20):6711-6717.]doi:10.13271/j.mpb.017.006711.
丁海麦,白羽,周红兵,石松利. 2024. 不同居群蒙古扁桃的遗传多样性SRAP分析[J]. 分子植物育种,22(8):2583-2589.[ Ding H M,Bai Y,Zhou H B,Shi S L. 2024. SRAP analysis of genetic diversity in different populations of Amygdalus mongolica[J]. Molecular Plant Breeding,22(8):2583-2589.]doi:10.13271/j.mpb.022.002583.
何庆元,刘丽,孙武,朱军军,惠雪,朱浩波,徐荣华. 2023. 栝楼SRAP引物筛选及遗传多样性分析[J]. 中草药,54(20):6813-6818.[ He Q Y,Liu L,Sun W,Zhu J J,Hui X,Zhu H B,Xu R H. 2023. Primer screening of SRAP reac‐tion system and analysis on genetic diversity of Trichosan‐thes[J]. Chinese Traditional and Herbal Drugs,54(20):6813-6818.]doi:10.7501/j.issn.0253-2670.2023.20.024.
胡小虎,陈柯欣,郭艺鹏,刘秦,耿晓珊,方振名,卢恩科,周碧莹,姚贤秋,黄龙娣,刘强. 2022. 独脚金SRAP-PCR反应体系的优化及引物筛选[J]. 分子植物育种,20(22):7534-7539.[ Hu X H,Chen K X,Guo Y P,Liu Q,Geng X S,Fang Z M,Lu E K,Zhou B Y,Yao X Q,Huang L D,Liu Q. 2022. Optimization of SRAP-PCR reaction systems and primers screening for Striga asiatica[J]. Molecular Plant Breeding,20(22):7534-7539.]doi:10.13271/j.mpb. 020.007534.
黄锦春,万思琦,陈扬,李丽红,张自力,朱建军,吴梅,邢丙聪,邵清松,陆晨飞. 2023. 利用ISSR与SRAP分子标记分析金线莲种质资源遗传多样性[J]. 浙江农林大学学报,40(1):22-29.[ Huang J C,Wan S Q,Chen Y,Li L H,Zhang Z L,Zhu J J,Wu M,Xing B C,Shao Q S,Lu C F. 2023. Genetic diversity of Anoectochilus roxburghii based on ISSR and SRAP molecular markers[J]. Journal of Zhe‐jiang Aamp;F University,40(1):22-29.]doi:10.11833/j.issn. 2095-0756.20220473.
姜武,李亚萍,陈家栋,陶正明. 2022. 基于ISSR和SRAP分子标记的黄精种质遗传多样性研究[J]. 中草药,53(21):6865-6873. [Jiang W,Li Y P,Chen J D,Tao Z M. 2022. Genetic diversity analysis of Polygonati Rhizoma based on ISSR and SRAP molecular markers[J]. Chinese Traditional and Herbal Drugs,53(21):6865-6873.]doi:10.7501/j.issn. 0253-2670.2022.21.025.
李亚萍,戴惠明,姜武,陈家栋,段晓婧,陶正明. 2023. 基于SRAP标记的不同产区黄精的遗传多样性[J].浙江农林大学学报,40(3):658-664.[ Li Y P,Dai H M,Jiang W,Chen J D,Duan X J,Tao Z M. 2023. Genetic diversity of Polygonatum spp. from different production areas based on SRAP markers[J]. Journal of Zhejiang Aamp;F University,40(3):658-664.]doi:10.11833/j.issn.2095-0756.20220436.
梁芳瑜,何茂秋,徐昌艳,杨小生,杨娟,罗忠圣,覃容贵. 2021. 宽叶缬草SRAP-PCR反应体系的优化及引物筛选[J]. 中国实验方剂学杂志,27(23):163-171.[ Liang F Y,He M Q,Xu C Y,Yang X S,Yang J,Luo Z S,Qin R G. 2021. Optimization of SRAP-PCR system for Valeriana officinalis var. latifolia and primer screening[J]. Chinese Journal of Experimental Traditional Medical Formulae,27(23):163-171.]doi:10.13422/j.cnki.syfjx.20211412.
林协全,汪其双,覃兴化,黄思琦,严洪,邹双全,邹小兴. 2023. 甜茶“木姜叶柯”本草考证[J]. 福建农林大学学报(自然科学版),52(6):785-792. [Lin X Q,Wang Q S,Qin X H,Huang S Q,Yan H,Zou S Q,Zou X X. 2023. Herbalogical study on sweet tea Lithocarpus litseifolius(Hance) Chun[J]. Journal of Fujian Agriculture and Fore-stry University( Natural Science Edition),52(6):785-792.]doi:10.13323/j.cnki.j.fafu(nat.sci.).2023.06.009.
毛立彦,龙凌云,黄秋伟,丁丽琼,李慧敏,池昭锦,唐毓玮,苏群,农晓慧,朱天龙. 2023. 基于SRAP分子标记的147份睡莲属植物遗传多样性分析[J]. 南方农业学报,54(2):454-466.[ Mao L Y,Long L Y,Huang Q W,Ding L Q,Li H M,Chi Z J,Tang Y W,Su Q,Nong X H,Zhu T L. 2023. Genetic diversity analysis of 147 Nymphaea Linn. plants based on SRAP molecular marke[r J]. Journal of Southern Agriculture,54(2):454-466.]doi:10.3969/j.issn.2095-1191.2023.02.014.
彭勇,周君,黄宇潮,曹晓平. 2020. 基于文献计量的木姜叶柯国内研究现状分析[J]. 江西科学,38(4):607-610.[ Peng Y,Zhou J,Huang Y C,Cao X P. 2020. A bibliometric analysis on research of Lithocarpus litseifolius[J]. Jiangxi Science,38(4):607-610.]doi:10.13990/j.issn1001-3679. 2020.04.030.
戚华沙,陈加利,杨立荣,孙秀秀,郑道君,于靖. 2020. 海南油茶SRAP-PCR体系优化及有效引物筛选[J].分子植物育种,18(10):3273-3281.[ Qi H S,Chen J L,Yang L R,Sun X X,Zheng D J,Yu J. 2020. Optimization of SRAP-PCR system and screening of effective primers for tea-oil Camellia resources of Hainan island[J]. Molecular Plant Breeding,18(10):3273-3281.]doi:10.13271/j.mpb.018. 003273.
石紫薇,梁妍,姜婷婷,梁剑平,贾宁,辛志君,陆锡宏,周翔,李雪虎. 2020. 正交设计优化青蒿SRAP-PCR反应体系及引物筛选[J]. 分子植物育种,18(10):3303-3310.[ Shi Z W,Liang Y,Jiang T T,Liang J P,Jia N,Xin Z J,Lu X H,Zhou X,Li X H. 2020. Optimization of SRAP-PCR sys‐tem on Artemisia annua L. using orthogonal design and selection of primers[J]. Molecular Plant Breeding,18(10):3303-3310.]doi:10.13271/j.mpb.018.003303.
孙秀秀,陈加利,杨立荣,云勇,戚华沙,郑道君. 2021. 文昌锥SRAP-PCR体系优化及有效引物筛选[J].西南林业大学学报(自然科学),41(1):39-46.[ Sun X X,Chen J L,Yang L R,Yun Y,Qi H S,Zheng D J. 2021. Optimization of SRAP-PCR system and screening of effective primers for Castanopsis wenchangensis[J]. Journal of Southwest Forestry University (Natural Sciences),41(1):39-46.]doi:10.11929/j.swfu.201909020.
孙阳阳,王晓英,董丽娟,吕新民,张继亮. 2023. 基于SRAP分子标记红仁核桃自然杂交F1代的遗传多样性分析[J]. 北方园艺,(20):28-33.[ Sun Y Y,Wang X Y,Dong L J,Lü X M,Zhang J L. 2023. Genetic diversity analysis of natural hybrid F1 generation of red-kernel walnut based on SRAP molecular labeling[J]. Northern Horticulture,(20):28-33.]doi:10.11937/bfyy.20230641.
万蓝婷. 2022. 江西省木姜叶柯枝叶主要活性成分的生境差异及形成机制[D]. 南昌:江西农业大学. [Wan L T. 2022. Habitat differences and formation mechanism of the main active components in branches and leaves of Litho‐carpus litseifolius(Hance) Chun in Jiangxi Province[D]. Nanchang:Jiangxi Agricultural University.]doi:10.27177/d.cnki.gjxnu.2022.000132.
王洁,朱锡彭,王腾斐,朱建军,李文俊,邢丙聪,郑颖. 2023. 白及种质资源遗传多样性分析[J]. 浙江农林大学学报,40(2):321-329.[ Wang J,Zhu X P,Wang T F,Zhu J J,Li W J,Xing B C,Zheng Y. 2023. Genetic diversity analysis of Bletilla striata germplasm by ISSR and SRAP markers[J]. Journal of Zhejiang Aamp;F University,40(2):321-329.]doi:10.11833/j.issn.2095-0756.20210690.
王燕,龚义勤,赵统敏,刘广,郁樊敏,叶海龙,柳李旺. 2007. 番茄SRAP-PCR体系优化与品种分子鉴定[J]. 南京农业大学学报,30(1):23-29.[ Wang Y,Gong Y Q,Zhao T M,Liu G,Yu F M,Ye H L,Liu L W. 2007. Optimization of SRAP-PCR system and cultivar molecular identification in tomato(Lycopersicon esculentum L.)[J]. Journal of Nan‐jing Agricultural University,30(1):23-29.]
谢秋霞,孙秀秀,王春梅,夏腾飞,郑道君,陈业光,陈加利,冯依欣,任家乐,王健. 2023. 雷公笋SRAP-PCR体系建立及有效引物筛选[J]. 分子植物育种,21(17):5701-5708. [Xie Q X,Sun X X,Wang C M,Xia T F,Zheng D J,Chen Y G,Chen J L,Feng Y X,Ren J L,Wang J. 2023. Estab‐lishment of SRAP-PCR system and selection of effective primer for Costus speciosu[sJ]. Molecular Plant Breeding,21(17):5701-5708.]doi:10.13271/j.mpb.021.005701.
严武平,李娟玲,吴淑敏,覃少昌,杨祥飞,王钰. 2018. 鹧鸪茶SRAP-PCR反应体系优化技术[J]. 分子植物育种,16(10):3252-3258. [Yan W P,Li J L,Wu S M,Qin S C,Yang X F,Wang Y. 2018. Optimization techniques for SRAP-PCR reaction system in Mallotus oblongifolius(Miq.) Muell. Arg[. J]. Molecular Plant Breeding,16(10):3252-3258.]doi:10.13271/j.mpb.016.003252.
张尚文,杨天为,黄诗宇,李婷,高曼熔,何龙飞,张向军. 2023. 广西壮瑶药瘤果紫玉盘SCoT-PCR反应体系建立及引物筛选[J]. 南方农业学报,54(6):1646-1655.[ Zhang S W,Yang T W,Huang S Y,Li T,Gao M R,He L F,Zhang X J. 2023. Establishment of SCoT-PCR reaction system and primer screening of Guangxi Zhuang-Yao medicine Uvaria kweichowensis[J]. Journal of Southern Agriculture,54(6):1646-1655.]doi:10.3969/j.issn.2095-1191.2023.06.006.
赵宏宇,蔡禄,赵秀娟,王晶妍. 2008. 应用正交实验法优化DNA重复序列的PCR扩增体系[J]. 内蒙古科技大学学报,27(4):346-350.[ Zhao H Y,Cai L,Zhao X J,Wang J Y. 2008. Optimization on PCR reaction system of DNA repeat sequence by orthogonal experiment[J]. Joumal of Inner Mongolia University of Science and Technology,27(4):346-350.]doi:10.16559/j.cnki.2095-2295.2008.04.001.
赵鹏霞,杨旭,杨志玲,田朝霞,羊奕珣. 2023. 基于腊叶标本分析的木姜叶柯表型性状变异及地理分化研究[J]. 江西农业大学学报,45(2):285-297.[ Zhao P X,Yang X,Yang Z L,Tian Z X,Yang Y X. 2023. Phenotypic variation and geographical differentiation of Lithocarpus litseifolius based on herbarium-specimen analysis[J]. Acta Agricul‐turae Universitatis Jiangxiensis,45(2):285-297.]doi:10. 13836/j.jjau.2023029.
赵琦琦,郭玉静,于梦斐,王颖,高文伟,张斌. 2022. 油莎豆SRAP-PCR体系优化及遗传多样性分析[J]. 山东农业科学,54(8):31-38.[ Zhao Q Q,Guo Y J,Yu M F,Wang Y,Gao W W,Zhang B. 2022. Optimization of SRAP-PCR system and genetic diversity analysis of Cyperus esculentus[J]. Shandong Agricultural Sciences,54(8):31-38.]doi:10.14083/j.issn.1001-4942.2022.08.005.
中国科学院中国植物志编辑委员会. 1977. 中国植物志(第 22 卷)[M]. 北京:科学出版社.[ Editorial Committee of Flora of China. 1997. Flora of China( Volume 22)[M]. Bei‐jing:Science Press.]
Cheng J,Lyu L S,Shen Y B,Li K X,Liu Z H,Wang W X,Xie L. 2016. Population structure and genetic diversity of Lithocarpus lifeifolius(Fagaceae) assessed using microsa-tellite markers[J]. Nordic Journal of Botany,34:752-760. doi:10.1111/njb.01111.
Li G,Quiros C F. 2001. Sequence-related amplified polymor‐phism(SRAP),a new marker system based on a simple PCR reaction:Its application to mapping and gene tagging in Brassica[J]. Theoretical and applied genetic,103(2):455-461. doi:10.1007/s001220100570.
Yan W P,Li J L,Zheng D J,Friedman C,Wang H F. 2019. Analysis of genetic population structure and diversity in Mallotus oblongifolius using ISSR and SRAP markers[J]. PeerJ,7:e7173. doi:10.7717/peerj.7173.
(责任编辑陈燕)
