PTBP1通过调控FGFR2的可变剪接促进肝癌的发展
2024-01-24陈昱颖张倩桂梦会冯岚曹鹏博周钢桥
陈昱颖,张倩,桂梦会,冯岚,曹鹏博,周钢桥,
研究报告
PTBP1通过调控的可变剪接促进肝癌的发展
陈昱颖1,2,张倩3,桂梦会4,冯岚2,曹鹏博2,周钢桥1,2,4
1. 南华大学衡阳医学院,衡阳 421001 2. 军事科学院军事医学研究院辐射医学研究所,医学蛋白质组全国重点实验室,国家蛋白质科学中心,北京 100850 3. 四川大学华西医院,转化医学国家重大科技基础设施,成都 610000 4. 南京医科大学公共卫生学院,南京 211166
肝细胞癌(hepatocellular carcinoma,HCC)是原发性肝癌的主要类型,是一种早期无明显症状、易发生转移、存活率低的恶性肿瘤。多聚嘧啶区结合蛋白1 (polypyrimidine tract binding protein 1,PTBP1)是一种重要的RNA结合蛋白,可诱导促癌剪接事件的发生。虽然PTBP1在肝癌细胞中的促癌功能已被证实,但是其介导的促癌可变剪接事件及作用机制尚未得到完全解析。本文利用免疫共沉淀联合质谱分析发现与PTBP1结合的蛋白复合体显著富集于编码成纤维细胞生长因子受体2 (fibroblast growth factor receptor 2,FGFR2)的基因可变剪接调控过程。通过RNA免疫共沉淀和定量PCR实验,证实PTBP1可显著下调肝癌细胞中异构体的水平,上调异构体的水平,促进向的异构体转换。随后,通过CCK-8、transwell和平板克隆实验,在肝癌细胞系HepG2和Huh7中评价了和的肿瘤生物学功能。结果显示发挥抑癌功能,而发挥促癌功能。机制研究证实,向异构体的转换显著促进肝癌细胞的上皮-间充质转化(epithelial-mesenchymal transformation,EMT)及FGFR下游ERK和AKT信号通路的活化。本研究揭示了PTBP1促进肝癌进展的分子调控机制,为肝癌的防治提供了新的理论依据。
肝细胞癌;PTBP1;可变剪接;FGFR2;上皮-间充质转化
肝细胞癌(hepatocellular carcinoma,HCC;以下简称肝癌)占原发性肝癌(primary liver cancer,PLC)的90%,也是全世界第三大致死性恶性肿瘤[1]。在中国,肝癌在所有癌症中发病率位居第五,死亡率位居第二[2]。由于缺少适合的药物靶点,大多数晚期肝癌患者经诊治后的预后依然不佳。因此,对肝癌发生发展分子机制方面的研究,将为寻找特异性药物靶点及研发新的治疗策略提供重要理论依据。
多聚嘧啶区结合蛋白1 (polypyrimidine tract- binding protein 1,PTBP1)是分子量大小为59 kDa的RNA结合蛋白。之前有研究表明PTBP1可以调节mRNA代谢,包括调节可变剪接、多聚腺苷酸化、亚细胞定位和mRNA稳定性[3~5]。已有研究证实PTBP1作为癌基因,促进包括肝癌在内的多种肿瘤的恶性进展[6~9]。
人成纤维细胞生长因子受体2 (fibroblast growth factor receptor 2,FGFR2)是跨膜酪氨酸激酶受体家族成员之一。基因的前体mRNA通过选择性剪接可以产生两种互斥剪接的异构体:包含8号外显子且跳跃9外显子的;包含9号外显子且跳跃8外显子的。有研究表明,的两种异构体具有不同功能,具有上皮样功能,高表达的细胞排列紧密,呈典型的上皮样细胞形态;具有间充质样功能,高表达的细胞呈梭形,表现出典型的间充质样形态[10~12]。已有研究表明,异构体的转化通常与细胞上皮-间质转化(epithelial-mesenchymal transition,EMT)有关[13~15]。EMT是上皮细胞转变为间充质细胞表型的一个过程,主要表现为细胞连接与细胞黏附丧失并且转变成具有迁移能力的间充质细胞。EMT发生时,细胞失去极性、由紧密连接转变为间隙连接、迁移能力增强,而这些都是肿瘤细胞的特征。并且多项研究表明,EMT是多种癌症进展的恶性表型[16]。
虽然PTBP1在肝癌细胞中的促癌功能已被证实,但是其介导的促癌可变剪接事件及作用机制尚未得到完全解析。本文主要探究了剪接因子PTBP1促进肝癌进展的分子调控机制,旨在为肝癌的防治提供新的理论依据。
1 材料与方法
1.1 细胞培养
肝癌细胞系(HepG2、MHCC97H和Huh7)均来源于本实验室的细胞库。将离心好的细胞加入1 mL含有10% FBS的培养基,吹打混匀后加入装有8 mL 10% FBS培养基的培养皿,轻轻晃动培养皿混匀细胞后,将培养皿放入37℃、5%CO2的细胞培养箱中进行培养。此后每天观察细胞状态,细胞贴壁且生长状态良好可进行后续实验,当细胞面积达到80%~90%时进行细胞传代。
1.2 质粒构建及转染
用于瞬时过表达质粒的pCMV-Myc载体pCMV-Myc-购自重庆英茂盛业生物技术有限公司;用于瞬时过表达质粒的pcDNA3.1- 2×Flag载体Flag-及Flag-购自重庆英茂盛业生物科技有限公司。
将适量培养皿细胞加入完全培养基颠倒混匀后接种至一个6孔板,待6~8 h细胞贴壁且密度为30%~50%时进行转染:转染时取两个4 mL无菌离心管,一管加入500 μL双无(无双抗、无血清)DMEM和2 μg目的质粒,另一管中加入500 μL双无DMEM和4 μL lipofectamineTM2000,两管各自吹打均匀后室温孵育5 min,然后将两管混合,吹打均匀后室温孵育20 min,最后向6孔板每孔中加入1 mL转染混合体系,待6~8 h后更换为正常培养基继续培养24~48 h后进行后续实验。
1.3 siRNA转染
本研究所使用的小干扰RNA(small interfering RNA,siRNA)及其配套转染试剂(riboFECTTM CP Transfection Kit)均购自广州锐博生物公司。序列如下:
si-1:5′-GCACAGUGUUGAAGAUCAU-3′;
si-2:5′-GCGUGAAGAUCCUGUUCAA-3′;
si-1:5′-AAGUGCUGGCUCUGUUCAA-3′;
si-2:5′-UAUGUAAGGUCUCCAAUUA-3′;
si-1:5′-GGACAAAGAGAUUGAGGUU-3′;
si-2:5′-CUAUUGGGAUAUCCUUUCA-3′;
siCtrl:5′-UUCUCCGAACGUGUCACGU-3′。
将适量培养皿细胞加入完全培养基颠倒混匀后接种至一个6孔板,待6~8 h细胞贴壁且密度为30%~50%时进行转染:将转染试剂和siRNA混合孵育20 min后,将转染复合物加入装有细胞的培养皿中,在37℃、5%CO2培养箱中培养48 h。
1.4 迁移和侵袭实验
取对数生长期细胞并使用不含血清的DMEM饥饿12 h后,取30 μL细胞悬液进行细胞计数并使用无血清DMEM将细胞浓度稀释为3×105个/mL。取稀释后的细胞悬液200 μL加入迁移上室中,下室加入700 μL 20% FBS的DMEM。24~30 h后取出小室,放入含有多聚甲醛的1%结晶紫溶液中固定20 min后取出小室,冲洗去除结晶紫及小室内未转移的细胞。最后晾干小室,使用显微镜进行拍照并统计细胞数量。侵袭步骤基本同迁移步骤,不同之处在于侵袭所铺细胞数量为迁移细胞数量的两倍,培养箱内培养48~55 h后进行小室固定、染色、拍照和统计细胞数量。
1.5 细胞生长实验
CCK-8:取适量细胞进行细胞计数并稀释,按3000个细胞/孔接种于96孔板,每组3个重复孔,待6~8 h细胞贴壁后吸掉旧培养基并加入100 μL含有10%的CCK-8试剂的双无DMEM培养基孵育1 h。使用酶标仪(16039400,Tecan Sunrise,瑞士)测量每孔在450 nm处的吸光度值,记为第0天的数据,此后每天同一时间连续测量,直到得到连续6天的生长数据。
平板克隆:取适量细胞进行细胞计数并稀释,按3000个细胞/孔接种于6孔板,待细胞贴壁后,每隔3天更换一次培养基。待培养两周后,吸掉旧培养基,加入1 mL含有多聚甲醛的1%结晶紫溶液中固定20 min,冲洗孔板并晾干,使用高灵敏化学发光成像系统(ChemiDoc™ XRS,Bio-Rad,美国)进行拍照,并统计克隆数量。
1.6 RNA提取及RNA反转录
使用细胞总RNA提取试剂盒(深圳达科为生物技术股份有限公司)提取细胞总RNA,按说明书步骤进行,最后收集RNA使用分光光度计(DS-11,DENOVIX,美国)检测RNA浓度。
使用反转录试剂盒(RR037A,TaKaRa,日本)进行反转录,取500 ng RNA按试剂盒说明书步骤进行实验,使用梯度PCR仪(T100,Bio-Rad,美国)设置好反转录反应条件:37℃持续30 min,85℃持续5 s,最后保持4℃,反转录完成后将cDNA产物放入-20℃保存。
1.7 实时荧光定量PCR (quantitative real-time polymerase chain reaction,qRT-PCR)
在NCBI官网中输入基因名称,选择物种“human”,获取基因的编码序列(coding sequence,CDS)后,采用Primer 3网站(https://primer3.ut.ee/)设计qRT-PCR引物序列。引物由华大基因合成,序列如下:
PTBP1-F:5′-CCAAGTTCGGCACAGTGTTG-3′;
PTBP1-R:5′-TATACCAGGTGCACCGAAGG-3′;
FGFR2-IIIb-F:5′-AGGTTCTCAAGCACTCGGGGA-3′;
FGFR2-IIIb-R:5′-TTCCAGGCGCTTGCTGTTTTGG-3′;
FGFR2-IIIc-F:5′-TGAGGACGCTGGGGAATATAC-3′;
FGFR2-IIIc-R:5′-TAGTCTGGGGAAGCTGTAATCT-3′;
CDH1-F:5′-GGTGTATACAGCCTCCCACG-3′;
CDH1-R:5′-GCTGGACCGAGAGAGTTTCC-3′;
KRT19-F:5′-CTTCAGTCCGGCTGGTGAAC-3′;
KRT19-R:5′-TGGAGATGCAGATCGAAGGC-3′;
CDH2-F:5′-ACTTCTGCTGACTCCTTCACT-3′;
CDH2-R:5′-GATGTGCATGAAGGACAGCC-3′;
VIM-F:5′-AAGGTCAAGACGTGCCAGAG-3′;
VIM-R:5′-CGGGAGAAATTGCAGGAGGA-3′。
将反转录完成的cDNA样品稀释10倍后取2.8 μL作为反应的模板,配置qRT-PCR引物(F+R)为2 μmol/L,使用SYBR®FAST荧光定量PCR试剂盒(KK4601,Merck,美国)及荧光定量PCR仪(VIIA7,Applied Biosystems,美国)进行反应。避光配置10 μL qRT-PCR反应体系加入384孔板中,每组3个复孔,以基因作为对照,采用ΔΔCt法进行数据归一化。
1.8 细胞内总蛋白提取
用胰蛋白酶消化细胞3 min后,用等体积10%FBS培养基中和胰蛋白酶,转移至离心管中按720 r/min离心5 min后用PBS清洗细胞两次,再次离心弃上清。在细胞沉淀中加入适量RIPA裂解液,同时按照1∶1000加入蛋白酶抑制剂、磷酸酶抑制剂,重悬细胞后将细胞冰浴裂解30 min。最后离心管中加入适量的上样缓冲液,煮样10 min后12,000 r/min离心10 min,收集上清液于-80℃保存。
1.9 蛋白质免疫印迹(Western blot,WB)
制备8%聚丙烯酰胺凝胶上蛋白样、电泳、电转、5%脱脂牛奶内封闭1.5 h、4 ℃孵育一抗过夜、TBST溶液(Tris-HCI缓冲盐溶液+Tween)洗膜3次,二抗室温孵育1.5 h、TBST溶液洗膜3次,最后使用化学发光成像仪(5200,美国Tanon公司)进行显影,拍照保存。
PTBP1抗体(1∶1000,32-4800)购于美国Thermo公司;GAPDH抗体(1∶2000,CW0266)购于康为世纪公司;鼠源/兔源二抗(1∶2000,CW0102S/CW0103S)购于康为世纪公司;FGFR2抗体(1∶1000,23328S)、p-AKT抗体(1∶1000,9611S)、AKT抗体(1∶1000,4691S)、ERK1/2抗体(1∶2000,4696S)、p-ERK1/2抗体(1∶2000,4370S)、E-Cadherin抗体(1∶500,3195S)、N-Cadherin抗体(1∶500,13116S)、Vimentin抗体(1∶1000,5741S)均购于美国CST公司。
1.10 RNA免疫共沉淀(RNA immunoprecipitation,RIP)
利用Magna RIP™ RNA-Binding protein Immunoprecipitation Kit试剂盒(17-700,Merck,德国)按以下步骤进行RIP实验:在无核糖核酸酶的条件下,向细胞中加入RIP裂解液吹打混匀后裂解细胞。然后将PTBP1抗体(1∶200,32-4800,Thermo,美国)偶联到磁珠上,涡旋振荡后室温孵育30 min,将抗体-磁珠复合物与细胞的裂解物混合,4℃孵育3 h以上。用RIP Wash Buffer洗涤上述混合物数次,在最后一次清洗时,吸取少量磁珠进行质检,检测抗体-磁珠复合物是否与相应的靶蛋白结合。最后降解混合物中的蛋白质,纯化RNA后qRT-PCR进行RNA定量检测和数据分析。
1.11 免疫共沉淀联合质谱分析(immunoprecipitation-mass spectrometry,IP-MS)
首先通过裂解细胞收集蛋白样品,接下来使用磁珠与诱饵蛋白PTBP1偶联得到沉淀蛋白,然后加入可以和目的抗体PTBP1 (1∶100,32-4800,Thermo,美国)结合的protein A/G,形成目的蛋白-诱饵蛋白-诱饵蛋白抗体-proteinA/G复合物,待纯化此复合物后,采用SDS-PAGE分离蛋白并进行考马斯亮蓝染色。最后回收切割好的凝胶用质谱分析进行蛋白质鉴定,最后通过质谱检测和文库检索,分析质谱鉴定数据,从中筛选出可能与PTBP1相互作用的蛋白质进行后续验证。
1.12 统计学分析
从TSVdb数据库(http://www.tsvdb.com/plot.html)下载了HCC癌组织和癌旁组织外显子测序的原始数据、临床数据和生存数据。从TSVdb肝细胞癌(TSVdb-LIHC)数据集中剔除了FGFR2两种异构体外显子表达量水平为0的样本,剩下样本包括305个肝癌组织和50个癌旁组织,采用秩和检验计算肝癌患者不同分组间异构体表达的差异。基于341个肝细胞癌病例(生存期>30天)建立了风险预测模型。使用survival包绘制Kaplan-Meier生存曲线,使用秩和检验分析两组患者生存率差异。使用Cox比例风险回归模型进行单变量生存分析,计算风险比和值。<0.05被认为有统计学意义。将TCGA-LIHC数据集来源的PTBP1基因表达与FGFR2不同异构体表达绘制相关性散点图,采用Pearson方法计算相关性系数。所有实验数据均以平均值±标准差来表示,组间差异采用Student′s检验确定显著性,<0.05代表有统计学意义。数据可视化及统计分析采用GraphPad Prism 9.3完成。
2 结果与分析
2.1 PTBP1在肝癌细胞中参与调控mRNA的可变剪接
已有研究报道PTBP1可以增强肝癌细胞的恶性表型[17,18],然而其作为RNA代谢调控的重要蛋白,其发挥促癌功能的分子作用机理仍未得到系统解析。为了探究PTBP1在肝癌细胞中的作用机制,本研究在肝癌细胞HepG2中采用免疫沉淀联合质谱分析,鉴定到上百个与PTBP1相互作用的候选蛋白质。通过功能富集分析,本研究发现这些蛋白主要富集于mRNA可变剪接通路,特别是的可变剪接(包括PTBP1、HNRNPA1、POLR2A、POLR2B、RBFOX2、TIA1和TIAL1)(表1)。因此,本研究推测在肝癌细胞中,PTBP1可能通过调控的可变剪接发挥促癌功能。
2.2 PTBP1促进FGFR2-IIIb向FGFR2-IIIc异构体转换
已有研究发现前体mRNA通过可变剪接可以产生两种异构体:含8号外显子不含9号外显子的和含9号外显子不含8号外显子的[19]。基于以上背景,本研究推测PTBP1在肝癌细胞中可能影响FGFR2两种异构体和的表达水平。为了验证上述假设,本研究首先通过RIP实验验证PTBP1与前体mRNA的结合能力。结果显示,在HepG2中,PTBP1抗体可以显著富集到前体mRNA (图1A)。其次,在HepG2和MHCC97H细胞中采用siRNA干扰的表达水平,通过qRT-PCR来检测两种互斥异构体和的相对表达水平变化。结果显示,和对照组相比,敲低后,的表达水平显著升高,而的表达水平显著降低(图1B)。除此之外,本研究构建了过表达的质粒,在HepG2和Huh7细胞中转染质粒后,的表达降低,而的表达升高(图1C)。以上结果证明,PTBP1在肝癌细胞中可以调控的可变剪接,促进向异构体转化。
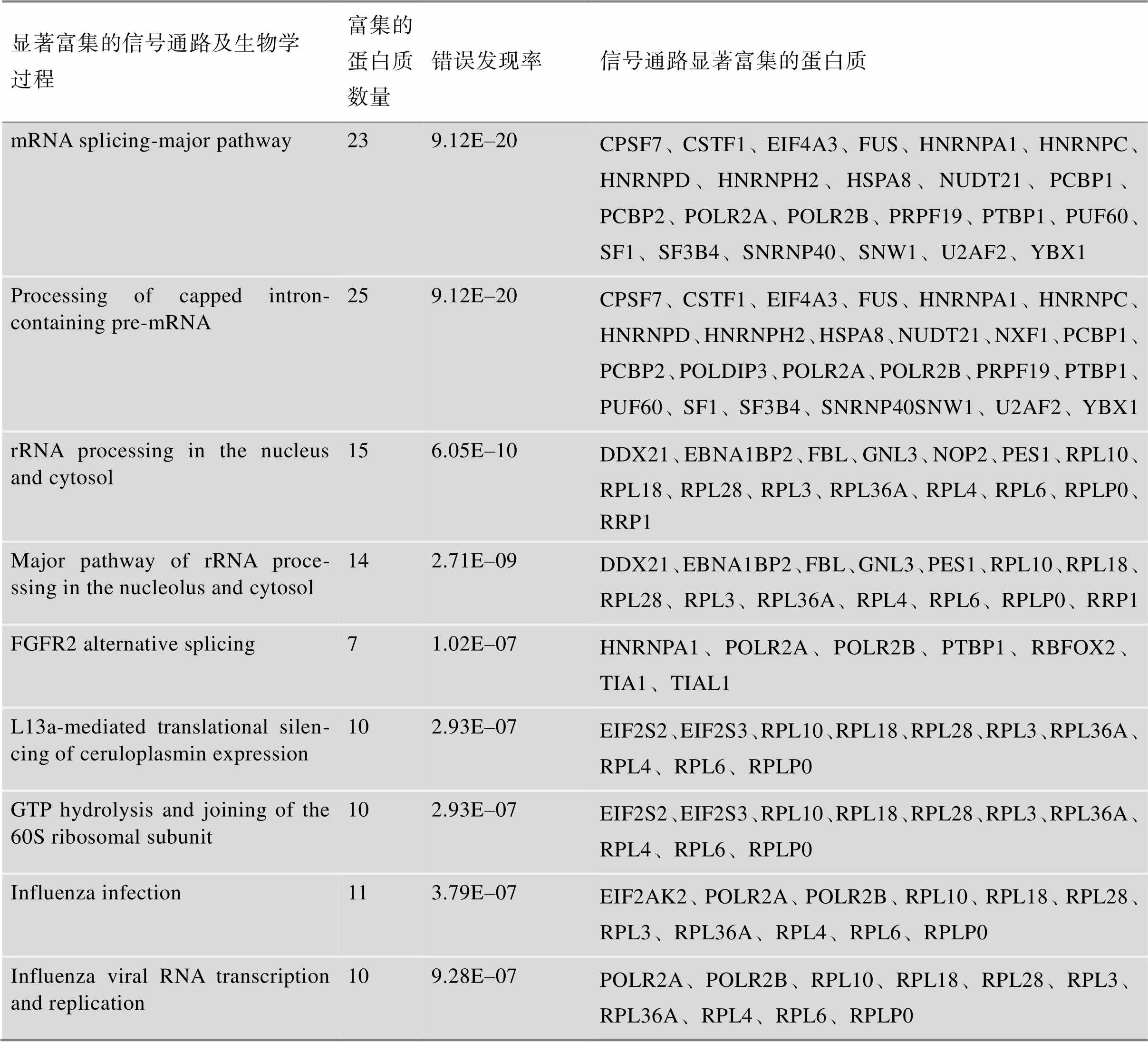
表1 与PTBP1相互作用的蛋白质所参与的信号通路及生物学过程
错误发现率:false discovery rate, FDR。
2.3 FGFR2-IIIb抑制HCC细胞的迁移、侵袭能力
本研究进一步探究了两个异构体在肝癌细胞中的肿瘤生物学功能。首先,用siRNA在HepG2和MHCC97H中敲低了的表达(图2A)。CCK-8和平板克隆形成实验均显示敲低对HepG2和MHCC97H细胞的生长和平板克隆形成能力无显著影响(图2,B和C)。然而,敲低后,HepG2和MHCC97H细胞的迁移和侵袭能力显著增加(图2D)。
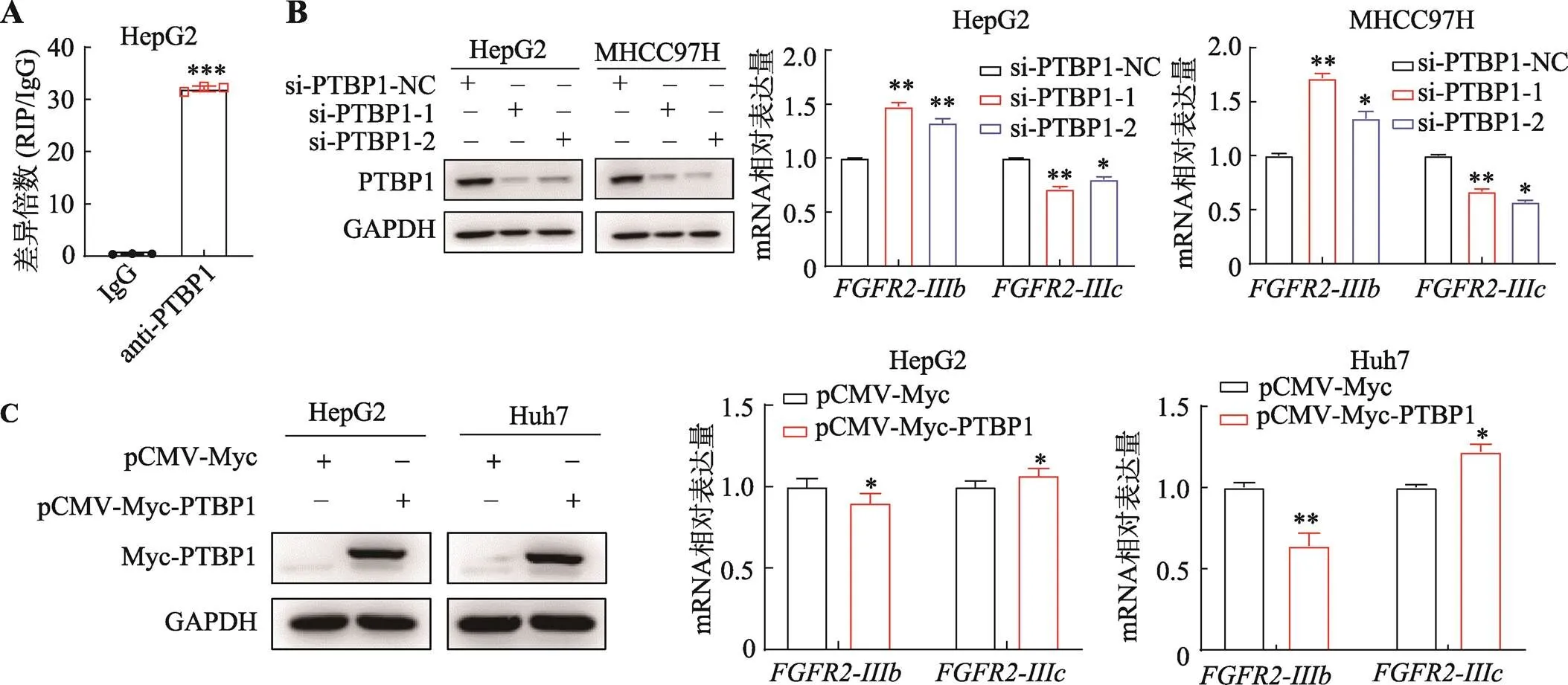
图1 PTBP1调控FGFR2的可变剪接
A:在HepG2中进行RIP实验证实PTBP1蛋白与前体mRNA存在相互作用;B:在两种肝癌细胞系(HepG2和MHCC97H)中敲低后通过qRT-PCR检测和的表达;C:在两种肝癌细胞系(HepG2和Huh7)中过表达后通过qRT-PCR检测和的表达。所有数据均以平均值±标准差来表示,每组实验设置3个重复孔。组间差异采用Student′s检验确定显著性;*表示<0.05,**表示<0.01。
当在HepG2和Huh7细胞中瞬时转染质粒后,这两种肝癌细胞的生长和克隆形成能力均无显著变化(图2,F~G);然而,过表达显著减弱了肝癌细胞的迁移和侵袭能力(图2H)。以上结果表明,可以显著抑制肝癌细胞的迁移和侵袭能力,提示在肝癌细胞中发挥抑癌功能。此外,已有研究报道发现在肝癌细胞系中过表达可以显著抑制裸鼠皮下移植瘤的恶性进展[20],这与本团队的研究结果一致提示在肝癌细胞中发挥抑癌功能。
2.4 FGFR2-IIIc促进HCC细胞的生长、迁移、侵袭能力
接下来,本研究进一步探究了异构体在肝癌细胞中的肿瘤生物学功能。本研究首先通过siRNA干扰了的表达(图3A)以研究它对肝癌细胞恶性表型的影响。CCK-8实验显示敲低显著抑制肝癌细胞HepG2和MHCC97H的生长(图3B)。通过平板克隆形成实验,本研究发现敲低同样可以显著抑制这两种肝癌细胞的克隆形成能力(图3C)。通过迁移侵袭实验,本研究发现在这两种肝癌细胞中敲低后,细胞的迁移和侵袭能力显著降低(图3D)。
除此之外,本研究在HepG2和Huh7细胞中转染质粒(图3E)以证实上述发现。本研究发现过表达后,两种细胞的生长能力、克隆形成能力、迁移和侵袭能力显著增加(图3,F~H)。以上结果表明,与的抑癌功能相反,在肝癌细胞中通过促进细胞迁移和侵袭能力发挥促癌功能。
2.5 FGFR2-IIIb/IIIc异构体转化促进肝癌细胞EMT
有研究发现,当肿瘤细胞发生EMT后,细胞的迁移和侵袭能力大大增加[21]。当细胞由上皮形态向间质形态转变的过程中,通常伴随着上皮细胞标志蛋白E钙粘蛋白(E-cadherin CDH1)、角蛋白(keratin,KER)等表达水平的降低以及间充质细胞标志蛋白N钙粘蛋白(N-cadherin,CDH2)及波形蛋白(vimentin,VIM)等表达水平的增加[22]。已有研究发现在子宫内膜癌细胞、食管鳞状细胞癌细胞中,的异常活化可以显著促进EMT的发生[23,24]。为了验证的异构体转换是否也参与调控肝癌细胞EMT的发生,本研究通过qRT-PCR实验和WB实验在HepG2和MHCC97H细胞中评价了两种异构体对上皮细胞标记分子(和)以及间充质细胞标记分子(和)表达水平的影响。结果显示,与对照组相比,敲低显著降低和的mRNA表达水平,并且显著降低CDH1的蛋白表达水平,而显著上调的mRNA表达水平和蛋白水平(图4,A和B);与之相反的是,当敲低后,和的mRNA表达水平显著升高,并且CDH1的蛋白表达水平显著升高,而和的mRNA表达水平和蛋白水平显著降低(图4,C和D)。为了证实上述结果,本研究进一步在HepG2和Huh7细胞中分别过表达和,qRT-PCR实验和WB实验均显示与分别敲低两者相反的结果(图4,E~H)。
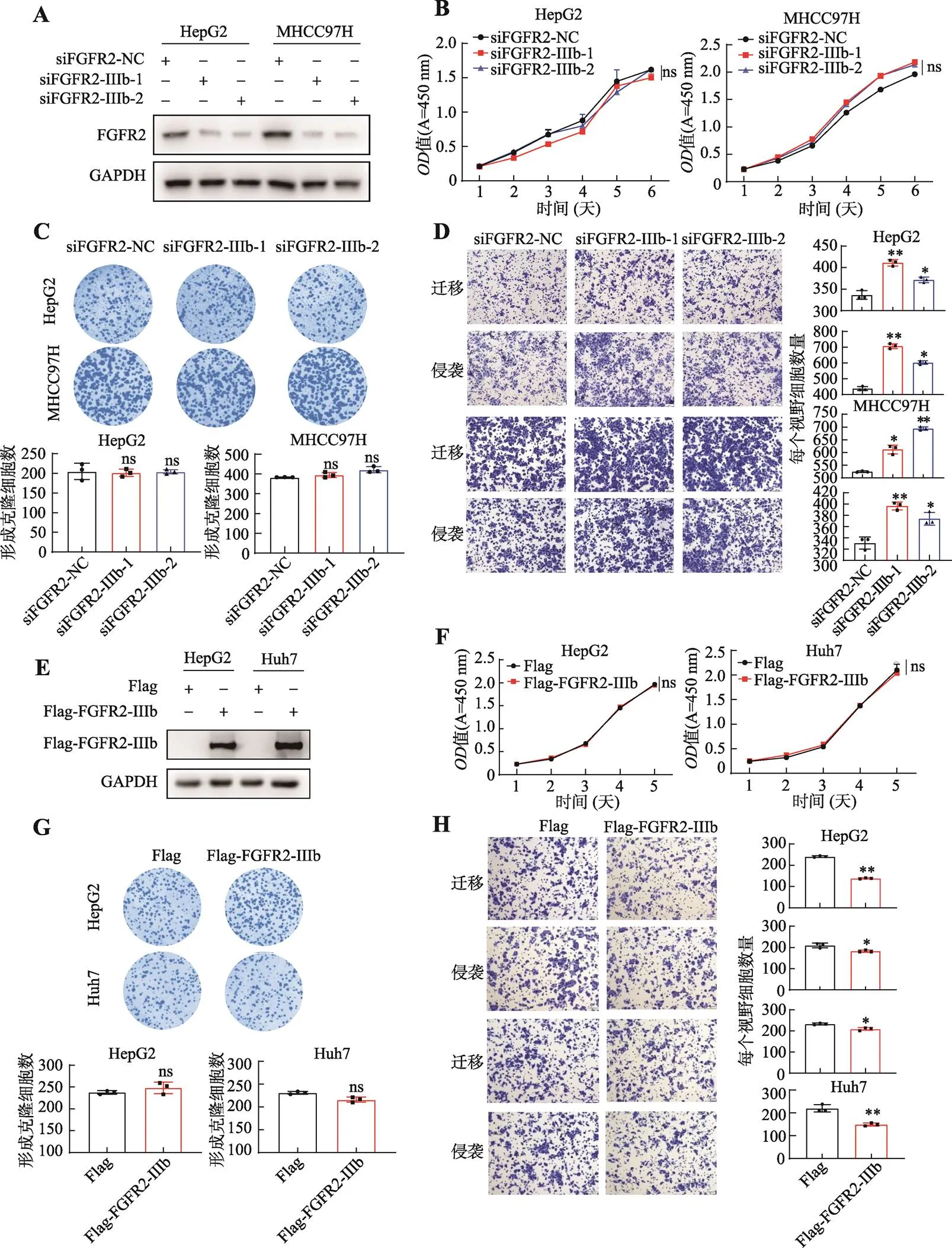
图2 FGFR2-IIIb抑制肝癌细胞的迁移和侵袭
A:在HepG2和MHCC97H细胞中敲低后通过Western blot实验检测其表达水平;B:通过CCK-8实验检测敲低对HepG2和MHCC97H细胞生长能力的影响;C:通过平板克隆形成实验检测敲低对HepG2和MHCC97H细胞克隆形成能力的影响;D:通过transwell实验检测敲低对HepG2和MHCC97H细胞迁移和侵袭能力的影响;E:在HepG2和Huh7细胞中过表达后通过Western blot实验检测其表达水平;F:通过CCK-8实验检测过表达对HepG2和Huh7细胞生长能力的影响;G:通过克隆形成实验检测过表达对HepG2和Huh7细胞克隆形成能力的影响;H:通过transwell实验检测过表达对HepG2和Huh7细胞迁移和侵袭能力的影响。所有数据均以平均值±标准差来表示,每组实验设置3个重复孔。组间差异采用Student′s检验确定显著性;ns表示>0.05,*表示<0.05,**表示<0.01,***表示<0.001。
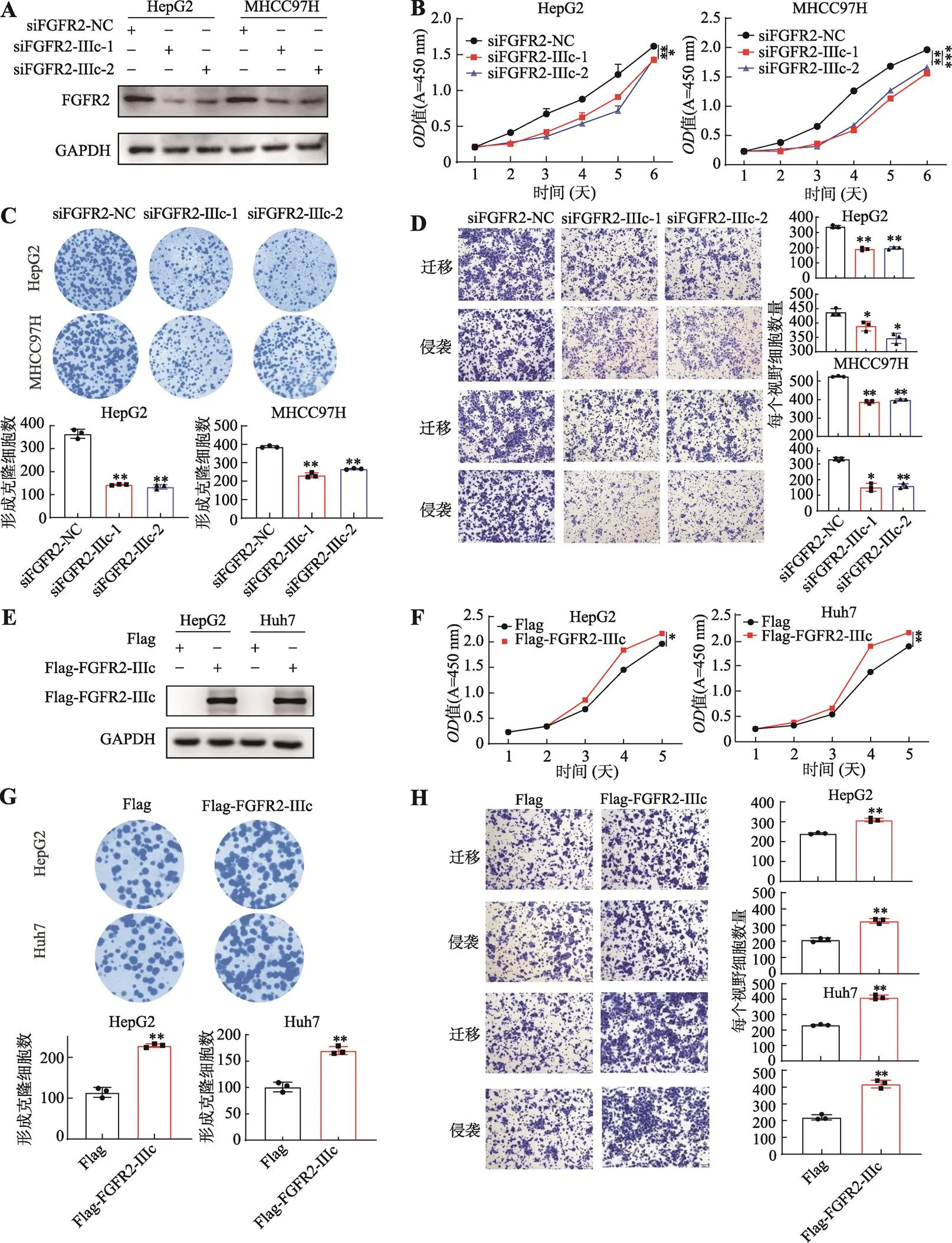
图3 FGFR2-IIIc促进肝癌细胞的生长、迁移和侵袭
A:在HepG2和MHCC97H细胞中敲低后通过Western blot实验检测其表达水平;B:通过CCK-8实验检测敲低对HepG2和MHCC97H细胞生长能力的影响;C:通过平板克隆形成实验检测敲低对HepG2和MHCC97H细胞克隆形成能力的影响;D:通过transwell实验检测敲低对HepG2和MHCC97H细胞迁移、侵袭能力的影响;E:在HepG2和Huh7细胞中过表达后通过Western blot实验检测其表达水平;F:通过CCK-8实验检测过表达对HepG2和Huh7细胞生长能力的影响;G:通过平板克隆形成实验检测过表达对HepG2和Huh7细胞克隆形成能力的影响;H:通过transwell实验检测过表达对HepG2和Huh7细胞迁移和侵袭能力的影响。所有数据均以平均值±标准差来表示,每组实验设置3个重复孔。组间差异采用Student′s检验确定显著性;*表示<0.05,**表示<0.01,***表示<0.001。

图4 FGFR2-IIIb/IIIc异构体转化对肝癌细胞中EMT标志分子表达水平的影响
A:通过qRT-PCR实验检测HepG2和MHCC97H细胞中敲低对上皮细胞标记分子(和)以及间充质细胞标记分子(和)表达水平的影响;B:通过Western blot实验检测HepG2和MHCC97H细胞中敲低对上皮细胞标记分子CDH1以及间充质细胞标记分子(CDH2和VIM)表达水平的影响;C:通过qRT-PCR实验检测HepG2和MHCC97H细胞中敲低对EMT标志分子表达水平的影响;D:通过Western blot实验检测HepG2和MHCC97H细胞中敲低对EMT标志分子表达水平的影响;E:通过qRT-PCR检测HepG2和Huh7细胞中过表达对EMT标志分子表达水平的影响;F:通过Western blot检测HepG2和Huh7细胞中过表达对EMT标志分子表达水平的影响;G:通过qRT-PCR检测HepG2和Huh7细胞中过表达对EMT标志分子表达水平的影响;H:通过Western blot检测HepG2和Huh7细胞中过表达对EMT标志分子表达水平的影响。所有数据均以平均值±标准差来表示,每组实验设置3个重复孔。组间差异采用Student′s检验确定显著性;ns表示>0.05,*表示<0.05,**表示<0.01,***表示<0.001。
以上结果表明,向的异构体转换可以促进肝癌细胞EMT的发生。
2.6 FGFR2-IIIb/IIIc异构体转化激活AKT和ERK通路
是酪氨酸激酶受体之一,其胞内的下游信号通路主要是MEK/ERK通路[25,26]和PI3K/AKT通路[27]。因此,本研究接下来尝试探究/异构体转化对MEK/ERK及PI3K/AKT通路活性的影响。本研究首先在生长因子FGF2刺激下的HepG2和MHCC97H细胞中分别敲低和,并采用WB实验检测MEK/ERK及PI3K/AKT两个通路活性的变化。结果显示,敲低可显著减弱肝癌细胞中磷酸化ERK1/ 2(p-ERK1/2)和AKT(p-AKT)的水平;相反,敲低可显著促进p-ERK1/2和p-AKT的活性(图5A)。相反地,当本研究在HepG2和Huh7细胞中分别过表达后,p-ERK1/2和p-AKT水平显著降低;然而,过表达后,p-ERK1/2和p-AKT水平显著升高(图5B)。以上结果表明,在肝癌细胞中/异构体转换可激活下游的MEK/ERK和PI3K/AKT信号通路,从而发挥促癌功能。
2.7 FGFR2-IIIc 高表达与肝癌患者的不良预后显著相关
为了探究不同异构体的表达水平的临床相关性,本研究从TSVdb数据库获得了TCGA肝癌癌组织和癌旁组织中FGFR2不同异构体表达水平及患者的生存信息。首先,分析了和异构体比例在癌和癌旁组织中的差异。结果显示,异构体比例在癌组织中显著下调(图6A),而异构体比例在癌组织中显著上调(图6C),证实在肝癌组织中发生了显著的异构体转化。然后,探究了和异构体比例与肝癌患者生存期之间的相关性。结果显示,异构体高表达的肝癌患者生存期与低表达的患者生存期无明显差异(图6B),而异构体高表达的肝癌患者生存期显著短于低表达的患者(图6D)。本研究进一步探究了表达与不同异构体表达水平的相关性。结果显示与前期机制实验结果一致:的表达水平与异构体的表达水平显著正相关,而与异构体的表达水平不存在相关性(图6,E和F)。以上结果表明,在肝癌中发生PTBP1介导的异构体转化,且可以作为肝癌患者预后的候选生物标志物。

图5 FGFR2-IIIb和IIIc异构体对MEK/ERK和PI3K/AKT信号通路活性的影响
A:通过Western blot实验检测敲低或对HepG2和MHCC97H细胞中MEK/ERK及PI3K/AKT通路活性的影响;B:通过Western blot实验检测过表达或对HepG2和Huh7细胞中MEK/ERK及PI3K/AKT通路活性的影响。将细胞提前饥饿24 h后使用FGF2(100 ng/mL)刺激10 min,提取蛋白用于Western blot实验。
A:在TCGA肝癌组织和癌旁组织中的表达水平,采用秩和检验计算组间差异;B:高表达及低表达(以中值分组)的肝癌患者的Kaplan-Meier生存曲线图;C:在TCGA肝癌组织和癌旁组织中的表达水平,采用秩和检验计算组间差异;D:c高表达及低表达(以中值分组)高表达及低表达(以中值分组)的肝癌患者的Kaplan-Meier生存曲线图。使用survival包绘制Kaplan-Meier生存曲线,采用Cox比例风险回归模型进行单变量生存分析,计算HR和值,<0.05被认为有统计学意义。E:TCGA-LIHC数据集中mRNA(TPM:transcript per million)表达水平与mRNA(TPM)表达水平的相关性散点图;F:TCGA-LIHC数据集中mRNA(TPM)表达水平与mRNA(TPM)表达水平的相关性散点图。采用Pearson方法计算相关性系数,<0.05被认为有统计学意义。
本研究首先通过免疫共沉淀联合质谱分析发现与PTBP1结合的蛋白复合体显著富集于FGFR2的可变剪接调控过程,然后本研究通过qRT-PCR和RIP实验证实了PTBP1参与调控该剪接事件,可导致肝癌中的异构体转换,即的表达水平降低,而的表达水平升高。进一步,本研究采用功能实验证实了抑制肝癌细胞的迁移和侵袭,而促进肝癌细胞的生长、迁移和侵袭。最后,通过免疫印迹实验,本研究证实了肝癌细胞中的异构体转换显著激活EMT及MEK/ERK和PI3K/AKT信号通路(图7)。综上所述,本项研究部分揭示了剪接因子PTBP1在肝癌细胞中发挥促癌功能的分子作用机理,为肝癌的防治提供了新的理论依据。
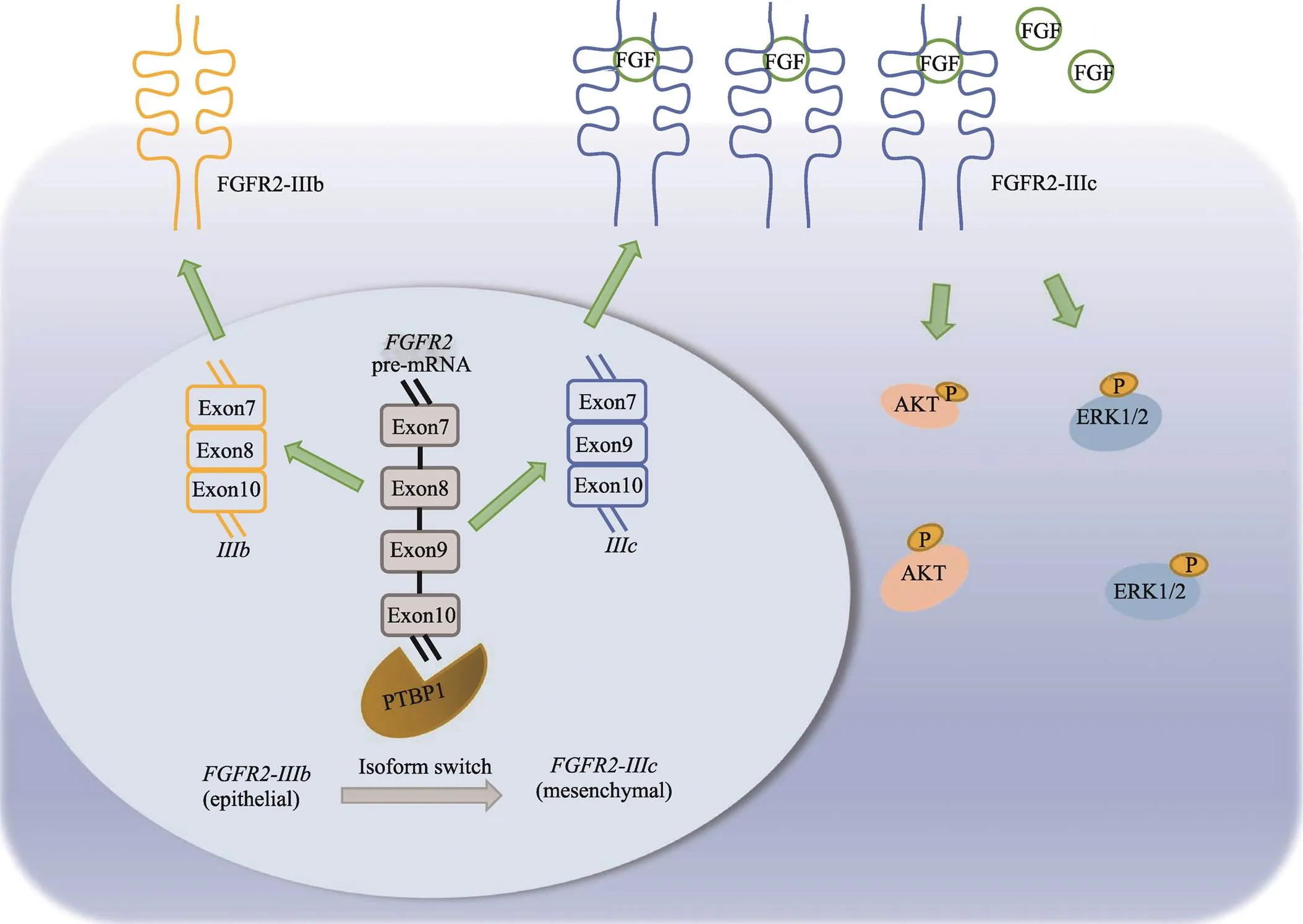
图7 PTBP1在肝癌细胞中发挥促癌功能的机制示意图
3 讨论
肝癌是一种高侵袭性和低存活率的恶性肿瘤,其发生发展是一个复杂的过程,基因表达失调和异常的分子信号转导会促进其恶性进展[28]。然而,肝癌的分子致病机制目前尚未得到完全解析。之前有研究证实了PTBP1通过促进肝癌细胞EMT通路进而促进肝癌细胞迁移和侵袭[29],但是其介导的促癌可变剪接事件及作用机制尚未完全得到解析。本研究的目的是通过一系列功能实验验证了PTBP1可增强/异构体转换,促进了肝癌细胞的EMT及下游ERK和AKT信号通路的活化,从而促进肝癌的发展。因此,本项研究揭示了剪接因子PTBP1促进肝癌进展的分子调控机制,为肝癌的防治提供了新的理论依据。
可变剪接又称为选择性剪接,是指mRNA前体经剪接后产生不同的mRNA,最终翻译成不同的蛋白质[30]。选择性剪接的蛋白产物可能具有不同的结构或互斥的功能,这可导致不同的异构体在同一细胞类型中具有不同的表达水平[31]。之前有研究发现PTBP1可通过调控和的可变剪接导致膀胱癌的恶性进展[32,33],并且PTBP1可通过调控的可变剪接促进胰腺癌细胞对临床药物吉西他滨的敏感性。PTBP1还可与剪接因子SRSF3形成RBP复合体调控基因的可变剪接从而调控结肠癌的发展[34],PTBP1也可与PSF形成复合体共同调节前体mRNA的可变剪接过程[35]。此外,有许多研究发现剪接因子ESRP1/2、FAST、T1A1可参与调控的可变剪接[36~39],并且RNA顺式作用元件ISE/ISS-3和ISAR也可调节的可变剪接[39,40],PTBP1可与ISS1结合共同调节的可变剪接。基于以上研究背景,本研究通过免疫共沉淀联合质谱分析发现与PTBP1结合的蛋白复合体显著富集于的可变剪接调控过程,并且通过qRT-PCR证实了PTBP1可使转变为c。本研究提示PTBP1可能与其他剪接因子或RNA结合蛋白形成复合体共同调控的可变剪接。
已有大量研究发现异构体与癌症的发生和发展密切相关。之前的研究发现在宫颈癌、食道癌和胰腺癌中的表达水平很高[41~43],特别是在胰腺癌和肺癌中,的表达水平与不良预后显著相关[44,45]。但有趣的是,在唾液腺肿瘤、前列腺癌和膀胱癌中诱导过表达可导致肿瘤细胞生长减少[46~48]。这些研究表明可能在不同癌症中发挥不同的肿瘤生物学功能。是上皮细胞的主要异构体,而异构体在细胞过渡到间质状态时上调表达[49,50],当组织发生EMT时,往往伴随向的转换[51]。之前有研究发现在前列腺癌和透明细胞肾细胞癌中发生/异构体转换[52,53],并且在肾细胞癌和胃癌中,的高表达与较高的肿瘤分级和较差的预后相关[36,53]。而本研究发现的两种异构体在肝癌细胞中发挥相反的功能:促进肝癌细胞的生长、迁移和侵袭,而抑制肝癌细胞的迁移和侵袭。并且本研究也证实了肝癌细胞中的异构体转换导致EMT的部分证据:转换促进间质标志基因的表达,而减少上皮标志基因和的表达。
有临床研究证实可作为肝癌治疗的靶点,酪氨酸激酶抑制剂Brivanib可抑制的表达从而肿瘤细胞生长[54]。本研究发现了剪接因子PTBP1可调控异构体的转换,但PTBP1的表达水平能否作为FGFR抑制剂在肝癌临床治疗指导用药中的标志物还有待进一步研究。总之,本研究揭示了剪接因子PTBP1促进肝癌进展的分子调控机制,为肝癌的防治提供了新的理论依据。
[1] Sung H, Ferlay J, Siegel RL, Laversanne M, Soerjomataram I, Jemal A, Bray F. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries., 2021, 71(3): 209–249.
[2] Qiu HB, Cao SM, Xu RH. Cancer incidence, mortality, and burden in China: a time-trend analysis and comparison with the United States and United Kingdom based on the global epidemiological data released in 2020., 2021, 41(10): 1037–1048.
[3] Steyerberg EW, Vergouwe Y. Towards better clinical prediction models: seven steps for development and an ABCD for validation., 2014, 35(29): 1925–1931.
[4] Guan Z, Cheng W, Huang D, Wei A. High MYBL2 expression and transcription regulatory activity is associated with poor overall survival in patients with hepatocellular carcinoma., 2018, 66(1): 27–32.
[5] Tang R, Wu JC, Zheng LM, Li ZR, Zhou KL, Zhang ZS, Xu DF, Chen C. Long noncoding RNA RUSC1-AS-N indicates poor prognosis and increases cell viability in hepatocellular carcinoma., 2018, 22(2): 388–396.
[6] Cheung HC, Hai T, Zhu W, Baggerly KA, Tsavachidis S, Krahe R, Cote GJ. Splicing factors PTBP1 and PTBP2 promote proliferation and migration of glioma cell lines., 2009, 132(Pt 8): 2277–2288.
[7] Takahashi H, Nishimura J, Kagawa Y, Kano Y, Takahashi Y, Wu X, Hiraki M, Hamabe A, Konno M, Haraguchi N, Takemasa I, Mizushima T, Ishii M, Mimori K, Ishii H, Doki Y, Mori M, Yamamoto H. Significance of polypyrimidine tract-binding protein 1 expression in colorectal cancer., 2015, 14(7): 1705–1716.
[8] Ferrarese R, Harsh GR 4th, Yadav AK, Bug E, Maticzka D, Reichardt W, Dombrowski SM, Miller TE, Masilamani AP, Dai FP, Kim H, Hadler M, Scholtens DM, Yu ILY, Beck J, Srinivasasainagendra V, Costa F, Baxan N, Pfeifer D, von Elverfeldt D, Backofen R, Weyerbrock A, Duarte CW, He XL, Prinz M, Chandler JP, Vogel H, Chakravarti A, Rich JN, Carro MS, Bredel M. Lineage-specific splicing of a brain-enriched alternative exon promotes glioblastoma progression., 2014, 124(7): 2861–2876.
[9] Kang H, Heo S, Shin JJ, Ji E, Tak H, Ahn S, Lee KJ, Lee EK, Kim W. A miR-194/PTBP1/CCND3 axis regulates tumor growth in human hepatocellular carcinoma., 2019, 249(3): 395–408.
[10] Wu DG, He XT, Wang WJ, Hu XT, Wang KF, Wang MH. Long noncoding RNA SNHG12 induces proliferation, migration, epithelial-mesenchymal transition, and stemness of esophageal squamous cell carcinoma cells via post-transcriptional regulation of BMI1 and CTNNB1., 2020, 14(9): 2332–2351.
[11] Li T, Jin XZ, Dong JR, Deng HB. Long noncoding RNA ARSR is associated with a poor prognosis in patients with colorectal cancer., 2020, 22(10): e3241.
[12] Wei L, Wang XW, Lv LY, Liu JB, Xing HX, Song YM, Xie MY, Lei TS, Zhang NS, Yang M. The emerging role of microRNAs and long noncoding RNAs in drug resistance of hepatocellular carcinoma., 2019, 18(1): 147.
[13] Gao G, Dai C, Yu X, Yin XB, Zhou F. Long noncoding RNA LINC00324 exerts protumorigenic effects on liver cancer stem cells by upregulating fas ligand via PU box binding protein., 2020, 34(4): 5800–5817.
[14] Khadirnaikar S, Chatterjee A, Kumar P, Shukla S. A greedy algorithm-based stem cell lncRNA signature identifies a novel subgroup of lung adenocarcinoma patients with poor pognosis., 2020, 10: 1203.
[15] Lu GM, Rong YX, Liang ZJ, Hunag DL, Ma YF, Luo ZZ, Wu FX, Liu XH, Liu Y, Mo S, Qi ZQ, Li HM. Multiomics global landscape of stemness-related gene clusters in adipose-derived mesenchymal stem cells., 2020, 11(1): 310.
[16] Sun LK, Wang L, Chen TX, Shi Y, Yao BW, Liu ZK, Wang YF, Li Q, Liu RK, Niu YS, Tu KS, Liu QG. LncRNA RUNX1-IT1 which is downregulated by hypoxia-driven histone deacetylase 3 represses proliferation and cancer stem-like properties in hepatocellular carcinoma cells., 2020, 11(2): 95.
[17] Shen LH, Lei SJ, Zhang B, Li SG, Huang LY, Czachor A, Breitzig M, Gao YM, Huang MY, Mo XM, Zheng Q, Sun HX, Wang F. Skipping of exon 10 in Axl pre-mRNA regulated by PTBP1 mediates invasion and metastasis process of liver cancer cells., 2020, 10(13): 5719–5735.
[18] He ZT, Ni QH, Li XC, Zhao MY, Mo QG, Duo YS. PTBP1 promotes hepatocellular carcinoma progression by regulating the skipping of exon 9 in NUMB pre-mRNA., 2023, 9(6): e17387.
[19] Guo JC, Yang YJ, Zheng JF, Zhang JQ, Guo M, Yang X, Jiang XL, Xiang L, Li Y, Ping H, Zhuo L. Silencing of long noncoding RNA HOXA11-AS inhibits the Wnt signaling pathway via the upregulation of HOXA11 and thereby inhibits the proliferation, invasion, and self-renewal of hepatocellular carcinoma stem cells., 2019, 51(11): 1–20.
[20] Amann T, Bataille F, Spruss T, Dettmer K, Wild P, Liedtke C, Mühlbauer M, Kiefer P, Oefner PJ, Trautwein C, Bosserhoff AK, Hellerbrand C. Reduced expression of fibroblast growth factor receptor 2IIIb in hepatocellular carcinoma induces a more aggressive growth., 2010, 176(3): 1433–1442.
[21] Yang H, Jiang Z, Wang S, Zhao YB, Song XM, Xiao YF, Yang SM. Long non-coding small nucleolar RNA host genes in digestive cancers., 2019, 8(18): 7693–7704.
[22] Zhu QY, Yang HJ, Cheng P, Han Q. Bioinformatic analysis of the prognostic value of the lncRNAs encoding snoRNAs in hepatocellular carcinoma., 2019, 45(2): 244–252.
[23] Adamczyk-Gruszka O, Horecka-Lewitowicz A, Gruszka J, Wawszczak-Kasza M, Strzelecka A, Lewitowicz P. FGFR-2 and epithelial-mesenchymal transition in endometrial cancer., 2022, 11(18): 5416.
[24] Maehara O, Suda G, Natsuizaka M, Shigesawa T, Kanbe G, Kimura M, Sugiyama M, Mizokami M, Nakai M, Sho T, Morikawa K, Ogawa K, Ohashi S, Kagawa S, Kinugasa H, Naganuma S, Okubo N, Ohnishi S, Takeda H, Sakamoto N. FGFR2 maintains cancer cell differentiation via AKT signaling in esophageal squamous cell carcinoma., 2021, 22(5–6): 372–380.
[25] Czaplinska D, Turczyk L, Grudowska A, Mieszkowska M, Lipinska AD, Skladanowski AC, Zaczek AJ, Romanska HM, Sadej R. Phosphorylation of RSK2 at Tyr529 by FGFR2-p38 enhances human mammary epithelial cells migration., 2014, 1843(11): 2461–2470.
[26] Cai SY, Yang YM, Jia BH, Wu ZH, Zhang JG, Shen JX, Qiu GX. Transcriptome-wide sequencing reveals molecules and pathways involved in neurofibromatosis type I combined with spinal deformities., 2020, 45(9): E489–E498.
[27] Chaffee BR, Hoang TV, Leonard MR, Bruney DG, Wagner BD, Dowd JR, Leone G, Ostrowski MC, Robinson ML. FGFR and PTEN signaling interact during lens development to regulate cell survival., 2016, 410(2): 150–163.
[28] Nagaraju GP., Dariya B, Kasa P, Peela S, El-Rayes BF. Epigenetics in hepatocellular carcinoma., 2022, 86(Pt 3): 622–632.
[29] Yang Y, Ren PW, Liu XF, Sun XY, Zhang CF, Du XJ, Xing BC. PPP1R26 drives hepatocellular carcinoma progression by controlling glycolysis and epithelial-mesenchymal transition., 2022, 41(1): 101.
[30] Zhao JX, Wang F, Xu ZR,Fan YM. The epigenetic effect on pre-mRNA alternative splicing., 2014, 36(3): 248–255.赵金璇, 王芳,徐峥嵘, 范怡梅. 表观遗传调控pre-mRNA的选择性剪接. 遗传, 2014, 36(3): 248–255.
[31] Liao RG, Jung J, Tchaicha J, Wilkerson MD, Sivachenko A, Beauchamp EM, Liu QS, Pugh TJ, Pedamallu CS, Hayes DN, Gray NS, Getz G, Wong KK, Haddad RI, Meyerson M, Hammerman PS. Inhibitor-sensitive FGFR2 and FGFR3 mutations in lung squamous cell carcinoma., 2013, 73(16): 5195–5205.
[32] Taniguchi K, Sakai M, Sugito N, Kumazaki M, Shinohara H, Yamada N, Nakayama T, Ueda H, Nakagawa Y, Ito Y, Futamura M, Uno B, Otsuki Y, Yoshida K, Uchiyama K, Akao Y. PTBP1-associated microRNA-1 and -133b suppress the Warburg effect in colorectal tumors., 2016, 7(14): 18940–18952.
[33] Xie RH, Chen X, Chen ZY, Huang M, Dong W, Gu P, Zhang JT, Zhou QH, Dong W, Han JL, Wang XS, Li H, Huang J, Lin TX. Polypyrimidine tract binding protein 1 promotes lymphatic metastasis and proliferation of bladder cancer via alternative splicing of MEIS2 and PKM., 2019, 449: 31–44.
[34] Kuranaga Y, Sugito N, Shinohara H, Tsujino T, Taniguchi K, Komura K, Ito Y, Soga T, Akao Y. SRSF3, a splicer of the PKM gene, regulates cell growth and maintenance of cancer-specific energy metabolism in colon cancer cells., 2018, 19(10): 3012.
[35] Miao H, Wu F, Li Y, Qin CY, Zhao YY, Xie MF, Dai HY, Yao H, Cai HY, Wang QH, Song X, Li L. MALAT1 modulates alternative splicing by cooperating with the splicing factors PTBP1 and PSF., 2022, 8(51): eabq7289.
[36] Teles SP, Oliveira P, Ferreira M, Carvalho J, Ferreira P, Oliveira C. Integrated analysis of structural variation and RNA expression of FGFR2 and its splicing modulator ESRP1 highlight the ESRP1(amp)-FGFR2(norm)-FGFR2- IIIc(high) axis in diffuse gastric cancer., 2019, 12(1): 70.
[37] Warzecha CC, Shen SH, Xing Y, Carstens RP. The epithelial splicing factors ESRP1 and ESRP2 positively and negatively regulate diverse types of alternative splicing events., 2009, 6(5): 546–562.
[38] Jones RB, Carstens RP, Luo Y, McKeehan WL. 5′- and 3′-terminal nucleotides in the FGFR2 ISAR splicing element core have overlapping roles in exon IIIb activation and exon IIIc repression., 2001, 29(17): 3557–3565.
[39] Simarro M, Mauger D, Rhee K, Pujana MA, Kedersha NL, Yamasaki S, Cusick ME, Vidal M, Garcia-Blanco MA, Anderson P. Fas-activated serine/threonine phosphoprotein (FAST) is a regulator of alternative splicing., 2007, 104(27): 11370–11375.
[40] Hovhannisyan RH, Warzecha CC, Carstens RP. Characterization of sequences and mechanisms through which ISE/ISS-3 regulates FGFR2 splicing., 2006, 34(1): 373–385.
[41] Yoshino M, Ishiwata T, Watanabe M, Matsunobu T, Komine O, Ono Y, Yamamoto T, Fujii T, Matsumoto K, Tokunaga A, Naito Z. Expression and roles of keratinocyte growth factor and its receptor in esophageal cancer cells., 2007, 31(4): 721–728.
[42] Ishiwata T, Friess H, Büchler MW, Lopez ME, Korc M. Characterization of keratinocyte growth factor and receptor expression in human pancreatic cancer., 1998, 153(1): 213–222.
[43] Kurban G, Ishiwata T, Kudo M, Yokoyama M, Sugisaki Y, Naito Z. Expression of keratinocyte growth factor receptor (KGFR/FGFR2 IIIb) in human uterine cervical cancer., 2004, 11(5): 987–991.
[44] Yamayoshi T, Nagayasu T, Matsumoto K, Abo T, Hishikawa Y, Koji T. Expression of keratinocyte growth factor/fibroblast growth factor-7 and its receptor in human lung cancer: correlation with tumour proliferative activity and patient prognosis., 2004, 204(1): 110–118.
[45] Cho K, Ishiwata T, Uchida E, Nakazawa N, Korc M, Naito Z, Tajiri T. Enhanced expression of keratinocyte growth factor and its receptor correlates with venous invasion in pancreatic cancer., 2007, 170(6): 1964–1974.
[46] Zhang Y, Wang H, Toratani S, Sato JD, Kan M, McKeehan WL, Okamoto T. Growth inhibition by keratinocyte growth factor receptor of human salivary adenocarcinoma cells through induction of differentiation and apoptosis., 2001, 98(20): 11336–11340.
[47] Matsubara A, Kan M, Feng S, McKeehan WL. Inhibition of growth of malignant rat prostate tumor cells by restoration of fibroblast growth factor receptor 2., 1998, 58(7): 1509–1514.
[48] Ricol D, Cappellen D, El Marjou A, Gil-Diez-de-Medina S, Girault JM, Yoshida T, Ferry G, Tucker G, Poupon MF, Chopin D, Thiery JP, Radvanyi F. Tumour suppressive properties of fibroblast growth factor receptor 2-IIIb in human bladder cancer., 1999, 18(51): 7234–7243.
[49] Thiery JP, Sleeman JP. Complex networks orchestrate epithelial-mesenchymal transitions., 2006, 7(2): 131–142.
[50] Warzecha CC, Carstens RP. Complex changes in alternative pre-mRNA splicing play a central role in the epithelial-to-mesenchymal transition (EMT)., 2012, 22(5–6): 417–427.
[51] Mittal V. Epithelial mesenchymal transition in tumor metastasis., 2018, 13: 395–412.
[52] Carstens RP, Eaton JV, Krigman HR, Walther PJ, Garcia-Blanco MA. Alternative splicing of fibroblast growth factor receptor 2 (FGF-R2) in human prostate cancer., 1997, 15(25): 3059–3065.
[53] Zhao Q, Caballero OL, Davis ID, Jonasch E, Tamboli P, Yung WK, Weinstein JN, Kenna Shaw for TCGA research network, Strausberg RL, Yao J. Tumor-specific isoform switch of the fibroblast growth factor receptor 2 underlies the mesenchymal and malignant phenotypes of clear cell renal cell carcinomas., 2013, 19(9): 2460–2472.
[54] Huynh H, Ngo VC, Fargnoli J, Ayers M, Soo KC, Koong HN, Thng CH, Ong HS, Chung A, Chow P, Pollock P, Byron S, Tran E. Brivanib alaninate, a dual inhibitor of vascular endothelial growth factor receptor and fibroblast growth factor receptor tyrosine kinases, induces growth inhibition in mouse models of human hepatocellular carcinoma., 2008, 14(19): 6146–6153.
PTBP1 promotes the progression of hepatocellular carcinoma by enhancing the oncogenic splicing switch of
Yuying Chen1,2, Qian Zhang3, Menghui Gui4, Lan Feng2, Pengbo Cao2, Gangqiao Zhou1,2,4
Hepatocellular carcinoma (HCC) is the most common type of primary liver cancer accounting for 90% of cases. It is a highly invasive and deadly cancer with a gradual onset. Polypyrimidine tract-binding protein 1 (PTBP1) is an important RNA-binding protein involved in RNA metabolism and has been linked to oncogenic splicing events. While the oncogenic role of PTBP1 in HCC cells has been established, the exact mechanism of action remains unclear. This study aimed to investigate the functional connection between PTBP1 and dysregulated splicing events in HCC. Through immunoprecipitation-mass spectrometry analyses, we discovered that the proteins bound to PTBP1 were significantly enriched in the complex responsible for the alternative splicing of(fibroblast growth factor receptor 2). Further RNA immunoprecipitation and quantitative PCR assays confirmed that PTBP1 down-regulated theisoform levels and up-regulated theisoform levels in HCC cells, leading to a switch fromtoisoforms. Subsequent functional evaluations using CCK-8, transwell, and plate clone formation assays in HCC cell lines HepG2 and Huh7 demonstrated thatexhibited tumor-suppressive effects, whiledisplayed tumor-promoting effects. In conclusion, this study provides insights into the PTBP1-mediated alternative splicing mechanism in HCC progression, offering a new theoretical basis for the prevention and treatment of this malignancy. Mechanistically, the isoform switch fromtopromoted epithelial-mesenchymal transformation (EMT) of HCC cells and activated the FGFR cascades ERK and AKT pathways.
hepatocellular carcinoma; PTBP1; alternative splicing; FGFR2; epithelial-mesenchymal
2023-08-23;
2023-11-08;
2023-12-05
国家自然科学基金项目(编号:82002573, 82172707)和国家重点研发计划项目(编号:2017YFA0504301)资助[Supported by the National Natural Science Foundation of China (Nos. 82002573, 82172707) and the National Key Research and Development Program of China (No. 2017YFA0504301)]
陈昱颖,硕士研究生,专业方向:基础医学。E-mail: 529897537@qq.com
张倩,硕士研究生,专业方向:生物医学。E-mail: 15111174004@163.com
陈昱颖和张倩并列第一作者。
周钢桥,博士,研究员,研究方向:医学遗传学与基因组学。E-mail: zhougq114@126.com
10.16288/j.yczz.23-224
(责任编委: 黄俊)
