鸡快慢羽基因分子检测新方法探究
2024-01-13郭屹凡熊林威张苗爽李竞一
郭屹凡,熊林威,张苗爽,李竞一
(华中农业大学,湖北武汉 430070)
家鸡的快慢羽基因是一对位于Z 染色体上的等位基因,由于其伴性遗传的特点,可以用于商品鸡出雏时鉴别雌雄,避免通过翻肛的方式来进行性别鉴定。翻肛不仅耗时费力,还需要一定的技术要求和经验,同时还会对幼雏造成应激[1],而快慢羽鉴别雌雄技术难度较低,所以被广泛使用。对于家禽育种工作来说,在慢羽纯系的培育过程中,需要使用测交来区分慢羽纯合子与杂合子公鸡,因此,寻找合适的分子检测手段来区分慢羽纯合与杂合,不仅可以代替繁琐的测交过程,也可以节约育种的时间与成本,对家禽行业具有重要意义。
2008 年,Elferink 等[2]研究表明,K*K 基因为Z 染色体上的一大段串联重复,重复由PRLR 和SPEF2 基因之间的基因间区、两个基因的部分重复以及其融合基因(dSPEF2-dPRLR)构成,白少川等[3]也发现了PRLR 和dPRLR 分别与dSPEF2和SPEF2 的5’ 末端以“头碰头” 方式连接。
Elferink 等[2]最先设计了两种引物用于判断慢羽杂合与纯合基因型,即通过定量检测基因组上融合基因dSPEF2/dPRLR 拷贝数的差异,使用荧光定量PCR 的方法,根据拷贝数差异判断纯合型与杂合型。然而此方法虽然理论上能用来判断慢羽的纯合型与杂合型,但并不能保证定量检测出来的拷贝数都为0 或1,往往会出现0.5 等数据难以判断其基因型,即检出率不高。罗成龙等[4]利用SNP 数据库中的137 个SNP 在欣华鸡的Z染色体上进行了关联研究,其中两个SNP(Gal-Gal6,chrZ:10238838bp 和chrZ:11247500bp)与羽毛表型显著相关,但并未在其他我国地方鸡群体中验证其关联性。2014 年,韩文朋等[5]发明了一种快慢羽基因型鉴别方法,对重复片段下游约390kb 处一个SNP 位点(GalGal6,chrZ:11139133bp)设计了一对引物进行PCR,使用Taq Ⅰ内切酶对扩增产物进行酶切,通过检测酶切后的片段长度由此判断基因型,但该方法也未在我国地方鸡群体中验证其适用性。总的来说,现在的快慢羽检测方法均有一些缺点,因此,本研究旨在利用公开数据库中全基因组数据开发新的能在全球各品系中准确区分快慢羽基因型的检测方法。
在本研究中,笔者从全基因组数据中寻找出与K*K 或K*N 连锁程度较高的分子遗传标记,对筛选出的关联SNP 利用KASP 法在我国地方鸡群体中检测其基因型与快慢羽的关联性,预期开发对快慢羽基因型进行检测的新方法,为快慢羽的标记辅助选择提供技术支持。
1 材料与方法
1.1 试验材料
试验选择湖北孝感安陆市欣华生态畜牧有限公司的496 只芦花母鸡和湖北荆门京山市神地农业科贸有限公司的179 只黑羽公鸡,根据这些公鸡与黑羽母鸡随机交配的结果,通过后代雏鸡的性别(翻肛鉴别)以及快慢羽表型对其快慢羽基因型进行反推。
1.2 样品采集
试验使用1.5mL 含柠檬酸钠抗凝剂的离心管进行血样收集,每只鸡使用采血针在翅中采集血液200μL 与15μL 4%浓度的柠檬酸钠充分混匀后放入冰盒中保存至实验室。使用DNeasy Blood and Tissue 试剂盒(QIAGEN)提取所需样本的DNA。
1.3 PCR 基因分型
以提取的DNA 为模板,将配制好的反应体系(表1)放入PCR 仪中进行反应,预变性94℃5min,第一轮循环14 次(变性94℃15s,退火68℃15s,且每次循环温度降低1℃,延伸72℃30s),第二轮循环25 次(变性94℃15s,退 火55℃15s,延 伸72℃30s),终延伸72℃5min。体系所用引物见表2。每批次的检测均附加慢羽对照和快羽对照以及阴性对照(空白对照)。反应完成后使用1.5%琼脂糖凝胶电泳观察成像。
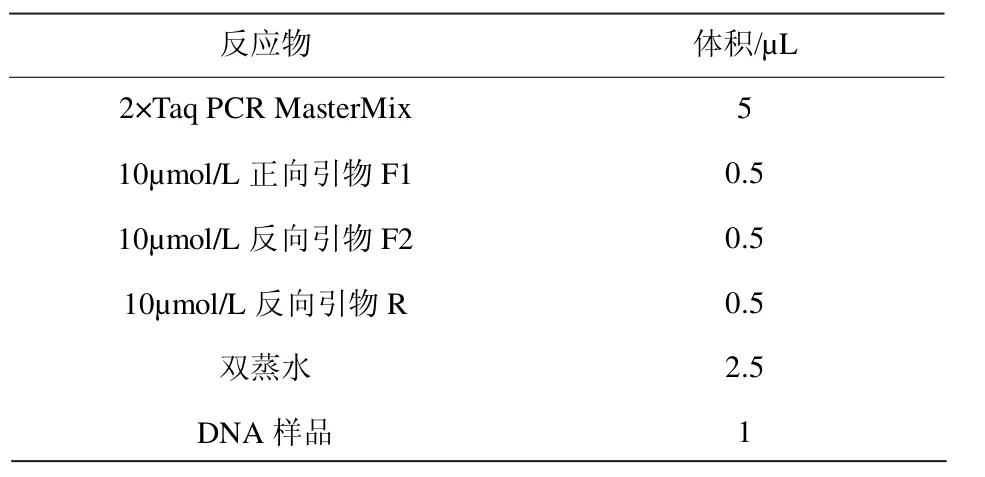
表1 10μL PCR 体系
1.4 数据分析
1.4.1 生物信息学分析
对本实验室前期采集自保种场的20 只洪山鸡和19 只郧阳大鸡血样所提取的DNA 进行基因组重测序,并从NCBI 的SRA 数据库中下载了1136 个家鸡个体的基因组重测序数据,所有1175 个测序反应均使用Illumina 平台。对测序结果利用BWA[6](比对至GalGal6 参考基因组)、SAMtools[7]、GATK[8]、Lumpy[9]、IGV、VCFtools等软件进行生物信息学分析。
使用vcftools 工具合并gatk(SNP)与lumpy(SV)的结果文件,分为W 染色体上的区间(4989932~5160560)和Z 染色体上的区间(10111794~11300060),其中分析的Z 染色体范围为快慢羽基因上下游共1.2Mb 区间。
1.4.2 W 与Z 染色体测序深度比值及性别分析
通过gatk 结果文件判断其在上述对应区间的测序深度,公鸡在W 上的区段理论测序深度为0,母鸡W 和Z 测序深度比值理论为1∶1。根据测序深度结果,将W 染色体的测序深度与W 和Z 染色体测序深度之和的比值作为判定依据。如果值小于0.01,判定为公鸡;值在0.4~0.6 判定为母鸡,删除比值位于上述区间外的个体。
1.4.3 快慢羽关联分子标记的筛选
将上一步得到的结果文件中的纯合突变型赋值为1,杂合子赋值为0.5,纯合野生型赋值为0,无法判断的个体用 “.” 表示。然后使用以下公式计算得到每个SNP 的absAFdif(等位基因频率差异的绝对值)值=(“快羽母鸡+快羽公鸡*2” 的基因频率-“慢羽母鸡+慢羽公鸡*2” 的基因频率)。上述公式中,“快羽母鸡” “快羽公鸡” “慢羽母鸡” “慢羽公鸡” 被代入对应样本赋值(1,0.5 或0)进行求和作为分子;样本总数(同样是公鸡乘以2)减去基因型无法判断的个体数作为分母;两者相除得到基因频率;最后通过两个基因频率的相减并取绝对值得到absAFdif。之所以将公鸡乘以2 是因为公鸡的Z 染色体数量为母鸡的2 倍。每个SNP 位点的absAFdif 为一个0~1 的值,值越接近1 则该分子标记与K 基因关联度越高。
1.4.4 lumpy 基因分型结果的校正
因为lumpy 软件无法准确区分公鸡的慢羽纯合与杂合,因此,我们使用igv 查看了172 个个体的原始bam 文件,以获得快慢羽重复片段上下游连接区域的read 数量,K*K 纯合子个体的read比对到参考基因组上时,来自重复片段中间接头的read 跟重复片段上游接头和下游接头的read 三者的理论比例是1∶1∶1,所以中间read 与上下游read 的比例应为1∶2。对于杂合子来说这个比例为1∶4。当比值在1∶2.5~1∶3.5 时无法准确判断其基因型,将其排除。
1.4.5 测序深度质控
在上述lumpy 基因分型结果校正过程中,我们发现51 个lumpy 结果显示为快羽公鸡中有8个样本含有重复片段,即lumpy 判断的结果存在误差,我们怀疑这些误差可能与测序深度较低有关,于是我们去除了测序深度低于5 的个体。
1.5 KASP 基因分型
根据LGC Genomics 公司开发的KASP 基因分型技术[10],按表3 配制好体系后置于CFX384实时定量PCR 仪中进行反应,预变性94℃15min,第一轮循环9 次(变性94℃20s,退火延伸61℃60s,且每次循环退火温度降低1℃),第二轮循环25 次(变性94℃20s,退火延伸55℃60s),终延伸72℃60s,然后仪器记录荧光量,每个样本设置两个重复。根据预实验的结果筛选出来的KASP 引物见表4。

表4 针对两个候选快慢羽关联SNP 的KASP基因分型引物序列
2 结果与分析
2.1 基因分型结果
结合179 只公鸡的血样提取DNA 后进行PCR 基因分型得到的凝胶电泳成像结果以及后代反推的基因型结果,得到互相吻合的慢羽纯合32个,慢羽杂合89 个,快羽31 个,共142 个。496 只母鸡的血样提取DNA 后进行PCR 基因分型得到凝胶电泳成像结果,其中慢羽189 个,快羽274 个,无结果33 个。
2.2 全基因组数据分析
共选用1175 个个体,排除216 个个体(9 个无法判断性别的个体,185 个测序深度低于阈值的个体以及26 个无法判断基因型的个体)后,剩余357 个快羽母鸡个体,414 个慢羽母鸡个体,80 个快羽公鸡个体,107 个慢羽公鸡个体,在重复片段上游500kb 到重复片段下游500kb 的区间内筛选出的12079 个分子标记。
在获得基因分型结果后我们没有对全基因组数据进行filter,目的是保证关联程度较高的突变不会因为质控被筛除掉,但由图1 可知,虽然排名前三个体(灰色部分)的absAFdif 值更高,但其错误率高达98%以上,即几乎所有样本都无法获得该位点的基因型,所以我们将它们排除掉,选择了图1 亮黄色部分absAFdif 值的8 个SNP 作为最初候选标记。在本研究的地方鸡群体中对这些候选标记进行预实验后,最终确定了2 个SNP位点作为候选分子标记(图1 亮黄色部分中absAFdif 值排名第1 和第3,均位于K*K 重复片段的内部,以下简称为SNP1 与SNP3)。根据预试验结果,由于这两个SNP 在母鸡群体中没有多态性,所以后续研究仅针对公鸡样本。
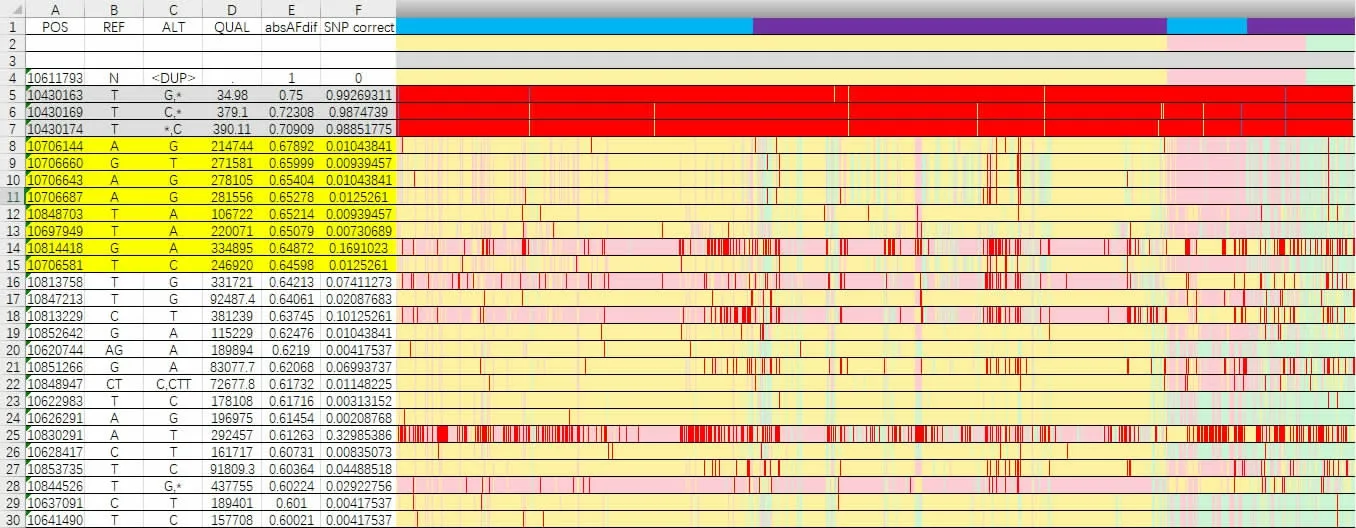
图1 959 个个体重测序数据的Z 染色体基因型分析结果与分析
2.3 KASP 基因分型结果
由图2 结果可知,SNP1 位点(chrZ:10706144bp)的KASP 结果相较于SNP3 位点(chrZ:10706643bp),其区域分离更为明显,检出率更高,重复性更好,然后我们将其中部分样本进行校准后得到结果见图2b。
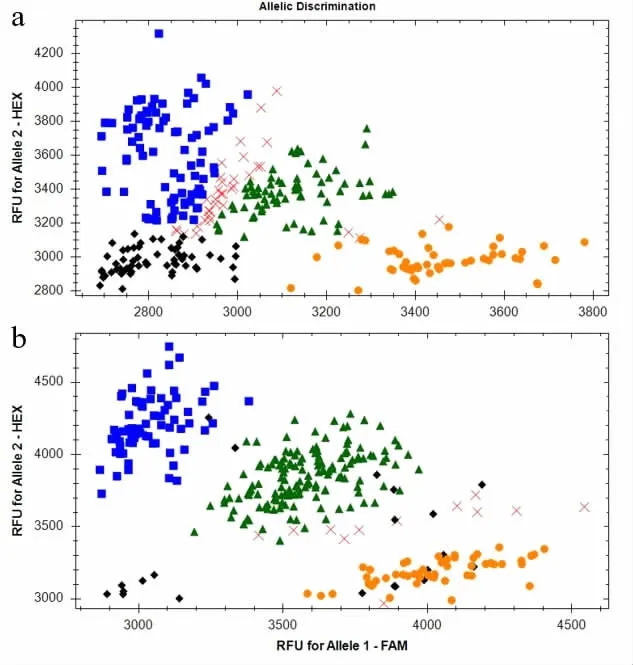
图2 针对两个候选快慢羽关联SNP 的KASP基因分型结果
2.4 KASP 基因分型结果准确性的验证
此次KASP 共检测了142 个个体,筛除无法判定(undifined)个体和无结果(no call)个体,获得了确定基因型结果的有129 个,检出率为91%。其中结果与已知快慢羽基因型相匹配的个体有100 个(77.52%),即利用SNP1。根据测交结果把142 个公鸡个体分为3 类,其中18 个快羽纯合个体有15 个吻合,其余3 个的KASP 结果为杂合子,准确率83.3%;其中30 个慢羽纯合个体有21 个吻合,其余2 个的KASP 结果为快羽纯合子、7 个为杂合子,准确率为70%;其中81 个杂合子个体有64 个吻合,其余6 个的KASP 结果为快羽纯合子、11 个为慢羽纯合子,准确率为79%。
3 讨论
我们从1.19Mb 的区间中找到了12079 个突变,由于结合了gatk 和lumpy 软件的结果,且没有使用任何针对突变的质控手段,所以理论上应该包含该区间内所有可能的突变(在一千多个世界主流的蛋鸡、肉鸡、地方鸡、观赏鸡、红原鸡范围内)。而且为了提高结果的可信度,将性别判断、快慢羽基因型判断可靠度不高以及测序深度不高的个体尽量排除。但结果显示,关联度最高的突变仅为0.68(即SNP1,位于重复片段内部),笔者尝试从进化的角度解释慢羽突变与周围突变关联度不高的原因,上述所有突变可被分为两类,一是位于重复片段外部,二是位于重复片段内部。前者可能在K*K 突变形成之初与K*K紧密关联,但由于K*K 在全球范围内的广泛应用,通过大量重组事件的积累,以至于我们检测不到紧密关联的突变。而重复片段内部的突变可能也会受重组的影响,即K*K 其中某一个拷贝与K*N 之间的重组,从而使K*K 的两个拷贝之间存在多态性,关联程度降低,如ev21 插入仅存在于其中一个拷贝[11];也可能在K*K 突变形成之初,这两个拷贝就存在着多态性,即K*K 的串联重复片段来自于非同源的片段插入而不是同一拷贝的重复,一个相关的线索来自于转座子的插入可能引起基因组的重复[12],这意味着可能是ev21 的插入引起的K*K 重复片段,而不是反过来。如果K*K 内部发生过重组,则可以对两个拷贝内部的突变分别进行分析,以找到重组发生的位置,有助于我们筛选可用的分子标记。但在二代测序的数据中难以区分重复片段内的突变来自于两个拷贝的哪一个,长读长测序技术可能解决该问题。还有一种可能,K*K 突变是多来源的,该突变并不一定只发生了一次,这样所有K*K 等位基因之间一开始就充斥着多态性。
距离慢羽基因被第一次报道已经超过了100年[13],恐难以再对K*K 形成之初的情景进行还原。但本研究结果至少说明了在K*K 重复片段的内部和周围,在全球各种快慢羽品系中都与之紧密关联的分子标记是不存在的,下一步需要在更小范围的样本中搜索可用的分子标记,如我国的黄羽肉鸡、优质蛋鸡等。如本研究在2 个不同品种的我国地方鸡群体(179 只公鸡与496 只母鸡)验证了数个SNP,其与快慢羽的关联程度都不是太高。但不排除其实在各自群体中存在连锁程度很高的突变,只能通过对该品种内(而不是中外各个鸡种)进行基因组层面的筛选才能发现。本研究计算了罗成龙等[4]和韩文朋等[5]在各自实验群体中检测出来快慢羽关联SNP 在本研究中959 个全基因组测序样本中的absAFdif 值,分别为0.086774(galGal6,chrZ:10238838bp),0.294473(galGal6,chrZ:11247500bp)和0.336827(gal-Gal6,chrZ:11139133bp),虽然这些SNP 的absAFdif 值不高,但这些研究利用上述SNP 在各自群体中检测快慢羽基因型的结果都较为准确,意味着需要在亲缘关系较近的群体内部搜索关联程度高的突变。从进化角度讲可能性是很高的,因为同一个品种内的所有K*K 等位基因的共同祖先可能并不是那么久远,也就是说重组事件发生的相对较少,如可能是数十年前由某个国外品种导入了慢羽基因,因为国外鸡种对我国本地鸡种基因渗入的事件常有发生[14]。
本研究的KASP 结果显示,利用SNP1 检测公鸡快慢羽基因型的准确率为77.52%,而具体来看,对快羽纯合个体检测的准确率较高,而对慢羽杂合和慢羽纯合的检测准确率略低。后者正是实际育种工作中需要的准确率,因此,该SNP 尚不能满足实际生产需要。但本研究所采用的KASP 基因分型新方法确实具有耗时短、通量大、检出率高等优点。
总的来说,本研究通过前沿的基因组与生物信息技术,以及充分利用共享资源数据、新兴检测技术来服务育种实践思路具有广阔的前景,也响应了习总书记对 “解决农业 ‘卡脖子’ 问题”的号召,并加快实施农业生物育种科技项目,早日实现重要农产品的种源自主可控。
4 结论
本研究对1175 个样本进行生物信息学分析,获得超过1.2 万个突变位点,并从中筛选出与重复片段关联程度最高的8 个SNP 位点,进一步通过预试验确定了2 个检测效果最理想的SNP 位点后,通过142 羽公鸡样本得到1 个效果最好的分子标记SNP 位点(位于Z 染色体10706144bp处)。与这些公鸡的测交结果相比,基因型吻合的个体比率为77.5%。该准确率距离实际育种工作中的要求还有一定差距,我们将继续改进分子标记的筛选范围,从而寻找到品种特异的更合适的分子标记。
