基于高密度单核苷酸多态性的共祖远亲缘关系预测算法准确性研究*
2024-01-03管珊珊魏以梁赵雯婷丽赵李彩霞
刘 京 李 晶 杨 澜 管珊珊 魏以梁 赵雯婷 江 丽赵 东 李彩霞**
(1)中国政法大学,证据科学教育部重点实验室,北京 100088;2)公安部鉴定中心,法医遗传学公安部重点实验室,现场物证溯源技术国家工程实验室,北京 100038;3)江苏师范大学生命科学学院,江苏省系统发育与比较基因组学重点实验室,徐州 221116)
短串联重复序列(short tandem repeat,STR)是司法鉴定领域进行亲缘关系鉴定的主要遗传标记,但其只能对亲子[1]、同胞[2]、祖孙[3]等2 级以内的近亲缘进行关系鉴定。单核苷酸多态性(single nucleotide polymorphism,SNP)位点具有分布广泛、突变率低等特点,随着全基因组测序(whole genome sequencing,WGS)、高密度SNP基因芯片等检测技术的发展成熟,利用高密度SNP数据预测远至7~9级亲缘关系成为近年来法医遗传学领域研究热点[4-10],该技术即为法医SNP系谱推断技术。2018 年美国警方首次使用法医SNP 系谱推断技术搜索到“金州杀手”的远亲[11],然后构建系谱树,进而找到凶手成功破获了42 年前的冷案,该技术被《科学》杂志(Science)评为当年十大科学突破之一。此后,美国警方利用该技术为数百起冷案积案提供关键线索。一项基于美国白人的研究表明[4],建立约占人口2%的SNP 数据库,即可为约99%的人口找到至少一名3 代表/堂亲(即7级亲缘)。研究和实践表明[12-14],法医SNP系谱推断与传统STR 技术相结合已成为法医DNA 服务冷案积案侦查的新模式。
目前基于高密度SNP 数据进行远亲缘关系推断的方法众多,包括:似然比算法、共享等位基因比 例(identical by state, ⅠBS) 算 法、 共 祖(identity-by-descent,ⅠBD)片段算法等[15]。似然比算法需提前给出一对个体之间具体关系类别的备择假设和两者无亲缘的原假设,再根据亲缘关系的两个互斥假设,观察个体间的遗传标记数据的条件概率,通过比较得到两假设条件概率的似然比。ⅠBS 算法[16-17]通过评估样本间每个SNP 等位基因频率,计算基因组中共享等位基因比例程度确定亲缘关系等级。以上两种算法适合预测4级以内亲缘关系,5 级以上的亲缘关系预测准确性显著降低[16]。ⅠBD片段算法通过检测ⅠBD,即来自一个共同祖先的相同DNA 片段长度和数量,判断亲缘关系远近。由于减数分裂时,父母双方的DNA 会发生断裂和重组,亲缘关系越远意味着传递代数(重组)越多,个体间共享的ⅠBD片段就越短,ⅠBD长度使用厘摩(cM)衡量。该算法适于预测7 级左右的亲缘关系,某些情况下可预测高达12 级的亲缘关系[18]。
ⅠBD 片段算法是目前法医系谱推断最常用算法。但在中国法医遗传学应用实践中,存在如下问题:a.国外研究大多基于模拟亲缘关系对和欧美人群亲缘关系对[18],欠缺适合中国人群真实亲缘关系的参数优化、准确性评估等系统性研究;b.需对高密度SNP 数据进行格式转换、同源染色体分型等处理,分析流程繁琐,普通法医工作者难以完成。本项目组在国内首次研究构建了适合法医应用的ⅠBD亲缘关系级预测的自动分析算法流程,实现了大量样本两两个体之间亲缘关系的批量计算,并为多起命案积案侦破提供了关键线索[12-13]。本文详述了该算法流程的构建和优化研究,并基于中国5个汉族大家系样本的真实亲缘关系对进行了ⅠBD片段算法预测准确性评估。本文研究成果将为中国法医SNP 系谱推断技术的研究和应用提供数据支撑和软件支持。
1 材料与方法
1.1 样本来源
采集本研究团队5 名成员的汉族家系253 人份样本,总共5 560 对亲缘关系(图1),包括一级(1st)、二级(2nd)、三级(3rd)、四级(4th)、五级(5th)、六级(6th)、七级(7th)、八级(8th)、九级(9th)、大于九级(10~14 级,>9th)亲缘关系,以及26 318对无亲缘关系。所有样本在采集前均签署知情同意书,本研究通过了公安部鉴定中心伦理委员会审查(编号:2021-006)。
1.2 DNA提取与检测
使用MagAttract M48 DNA Manual 试剂盒(Qiagen 公司,德国)提取DNA,使用NanoDrop 2000c 分光光度计(Thermo Scientific 公司,美国)进行DNA 定量。使用WeGene V2 基因芯片(安澜智能公司,中国)进行SNP 检测(DNA 模板量均大于500 ng,芯片位点检出率均大于98.5%),获得约70万SNP位点分型数据。
1.3 数据分析
本研究形成的ⅠBD片段算法集成到项目组前期开发的DNA 系谱推断系统DGA v1.0[19]进行亲缘关系预测。
1.4 亲缘关系推断算法参数优化
1.4.1 预测准确性评估指标
为了评估ⅠBD片段算法在中国真实家系亲缘关系对预测准确性, 本文使用绝对准确率(accuracy,AC)、置信区间准确率(confidence interval accuracy, CⅠA)、 假 阴 性 率 (false negative,FN)、假阳性率(false positive,FP)、预测可信度(prediction credibility,PC)作为评估指标。AC 是指某亲缘等级的调查亲缘关系对应的所有关系对中,预测结果同样是此等级的关系对所占的比例;CⅠA是指某亲缘等级的调查亲缘关系对应的所有关系对中,预测结果是此等级或此等级±1级的关系对所占的比例[16];FN是指某亲缘等级的调查亲缘关系对应的所有关系对中,预测结果是“无关”的关系对所占的比例;FP是调查亲缘关系为“无关”(大于14级)的所有关系对中,预测结果是“有关”(1~9 级)的关系对所占的比例;PC是指某亲缘等级的预测亲缘关系对应的所有关系对中,调查亲缘关系为“有关”的关系对所占的比例。
1.4.2 ⅠBD片段长度阈值优化
为了评估不同匹配片段最低检出长度阈值对预测准确性的影响,设置了0、3、6、9、12、15、20 cM 7 个不同的ⅠBD 片段长度阈值,评估不同最低检出ⅠBD片段长度阈值的预测准确性。
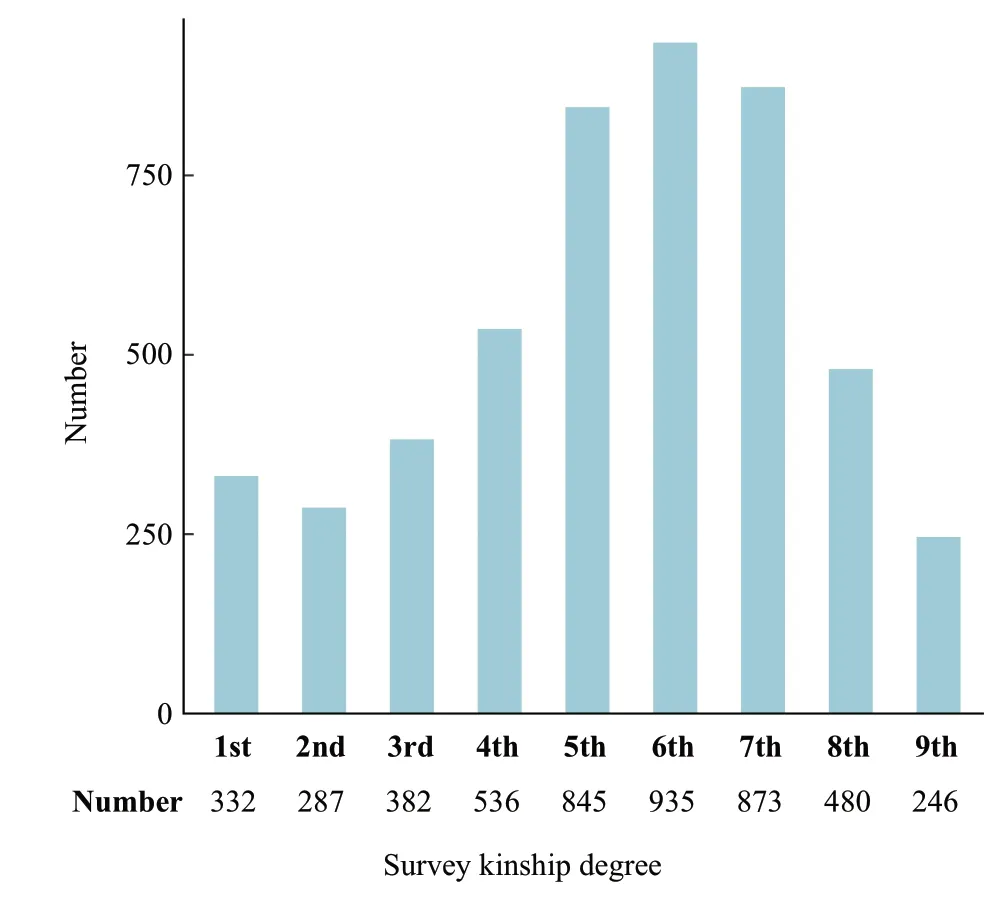
Fig.1 The distribution of survey kinship degree
1.4.3 支持向量机(SVM)优化
为了提高亲缘关系预测等级的准确性,本研究使用支持向量机(support vector machine,SVM)方法对ⅠBD片段算法中基于ⅠBD片段数量和长度预测亲缘关系等级进行优化,将预测有亲缘关系ⅠBD片段总长度和平均长度的最小值设为阈值。本研究根据阈值设置3 个集合(图2):集合1,预测为有亲缘关系的关系对;集合2,有ⅠBD片段结果的关系对中ⅠBD片段总长度和平均长度均小于该阈值的关系对;集合3,有ⅠBD 片段结果的关系对中ⅠBD片段总长度和平均长度均大于该阈值但被预测为无关的关系对,将集合1 和集合2 合并作为训练集,将集合3作为测试集,通过训练集对测试集重新进行分类,以降低较远(6级及以上)亲缘关系预测的FN。
1.5 不同数量SNP预测评估
由于法医物证受时间、环境等影响,经常会发生DNA 降解,检出的SNP 位点会随之减少。为了模拟降解DNA 对该系谱推断算法预测效能影响,本文对位点进行随机的梯度下降筛选,将筛选的位点组合进行两次亲缘关系预测的平均结果与原始数据结果进行比较,评估不同密度SNP 位点组合对预测准确性的影响。
2 结 果
2.1 基于IBD片段算法的亲缘关系分析流程
本文研究的ⅠBD 片段算法包括如下分析流程:过滤SNP 数据中的冗余信息,筛选位点,对筛选后数据进行格式转换,同源染色体分离,查找和合并各染色体上ⅠBD 匹配片段,基于ⅠBD 片段长度、数量等值预测个体间亲缘关系等级,在算法研制过程中进行ⅠBD 片段长度阈值、SVM 亲缘等级预测等优化(图3)。其中同源染色体分离选取了千人基因组中国人群作为参考数据集,使用隐马尔可夫(HMM)算法将待分析数据父源和母源染色体分离;ⅠBD片段长度计算时参考了HapMap计划网站中SNP 物理距离(bp)与遗传距离(cM)之间的关系进行厘摩值转换。基于以上分析流程,本研究使用Python 编写了基于ⅠBD 片段算法进行亲缘关系分析的pipeline,实现了数据预处理自动化、两两个体亲缘关系计算批量化等功能。
2.2 亲缘关系推断参数及SVM优化结果
2.2.1 亲缘关系推断算法结果
使用上述ⅠBD片段算法对253份高密度SNP数据进行亲缘关系计算,将所有个体间预测的1~9级亲缘关系等级与实际调查的亲缘关系进行比较并计算准确性评估指标(表1)。表1可以看出,前5级有较高的准确率,平均CⅠA 为99.14%,FN 为0。随着亲缘关系等级的增加,准确率也随之降低,6级开始出现假阴性,8 级及以上假阴性明显增加。1~7级亲缘的预测可信度较高,平均值为99.75%。
2.2.2 ⅠBD片段长度阈值研究结果
使用软件计算两两个体ⅠBD片段长度时,需设置最低检出ⅠBD片段长度阈值。为评估该参数对预测准确性影响,本文设置了0(即无最低检出ⅠBD片段长度限制)、3(默认参数)、6、9、12、15、20 cM 七个不同阈值长度阈值,计算在253 份样本中预测准确性变化情况。图4a 展示了不同ⅠBD 片段长度阈值下AC变化情况;图4b展示了不同ⅠBD片段长度阈值下CⅠA 变化情况;图4c 展示了不同ⅠBD片段长度阈值下PC变化情况;图4d展示了不同ⅠBD片段长度阈值下FN变化情况;图4e展示了不同ⅠBD 片段长度阈值下FN 平均值和FP 变化情况。对比显示0~9 cM ⅠBD 片段阈值参数对预测准确性影响不大,当ⅠBD 片段阈值大于9 cM,CⅠA、FP 均有一定程度降低,而PC、FN 有一定程度增加。
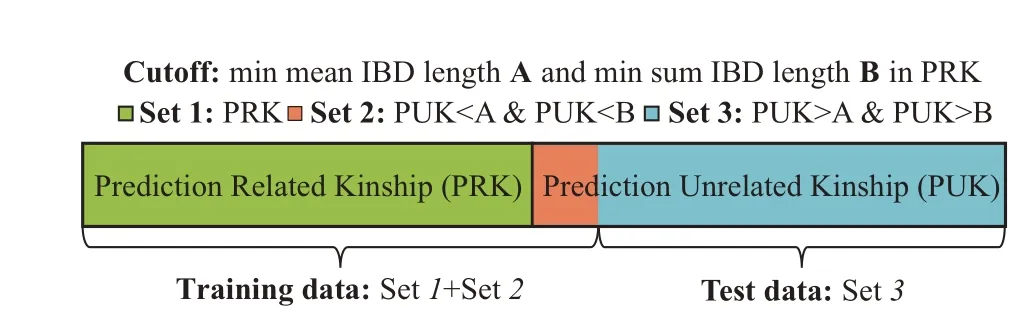
Fig.2 The SVM training and test data set

Table 1 Accuracy statistics of IBD algorithm in predicting kinship degree of 253 samples
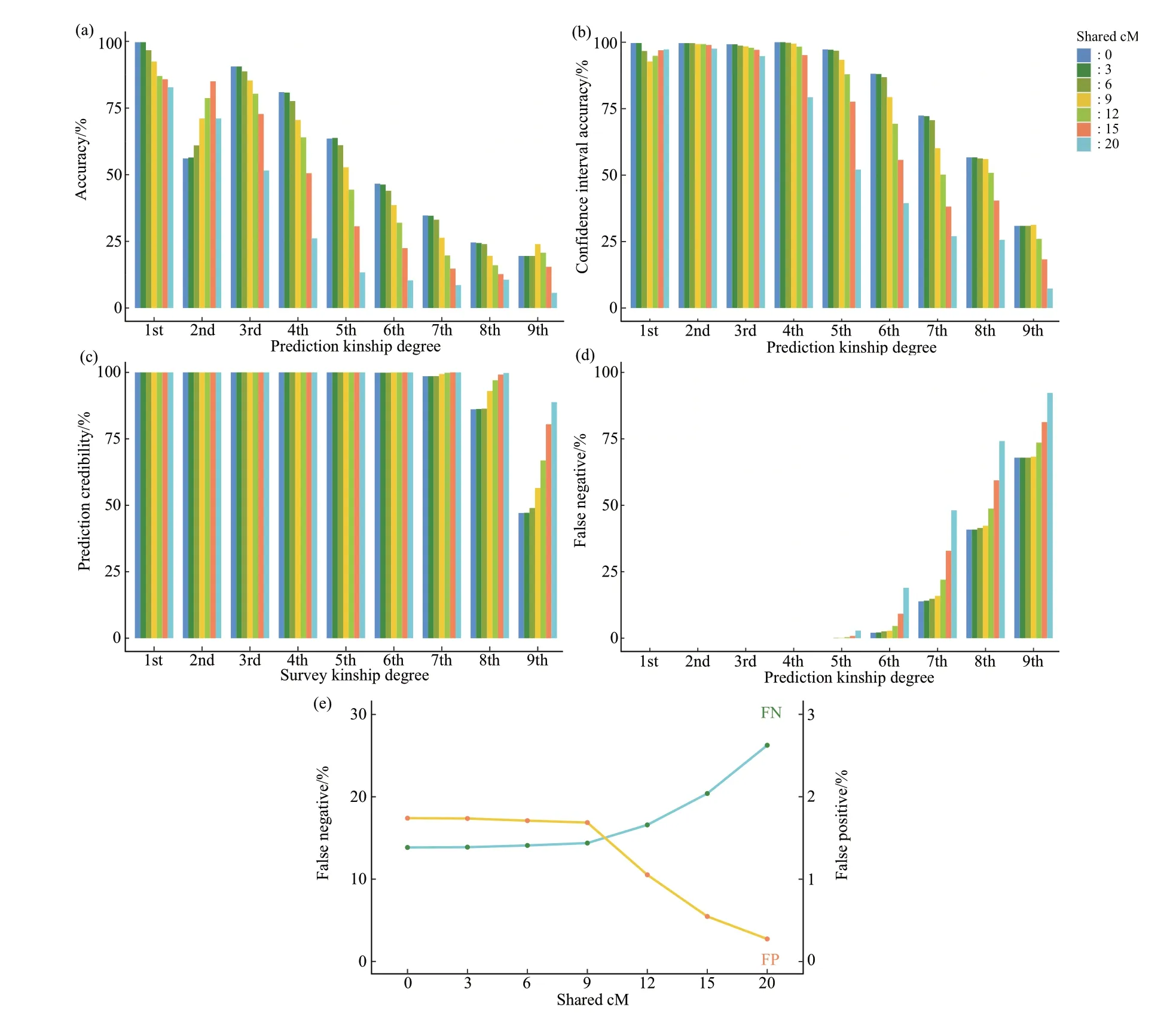
Fig.4 Accuracy evaluation of different IBD fragments threshold
2.2.3 SVM优化模型结果
表1 结果显示6 级及以上亲缘关系预测时会出现假阴性。为提高算法预测能力,降低FN,本研究使用SVM 方法对基于ⅠBD 片段数量和长度预测亲缘关系等级的过程进行优化,将所有个体间预测的1~9级亲缘关系等级与实际调查的亲缘关系进行比较并计算准确性评估指标(表2)。优化后前5级平均CⅠA 为99.16%,6 级FN 由优化前的2.14%降为0.43%,7 级FN 由优化前的14.09%降为7.10%。为进一步评估SVM 优化后预测性能,绝对准确率等评估指标与2.2.1常规流程进行横向对比(图5),结果显示SVM 优化后提高了远亲缘(6~9 级)关系等级CⅠA,降低了其FN。
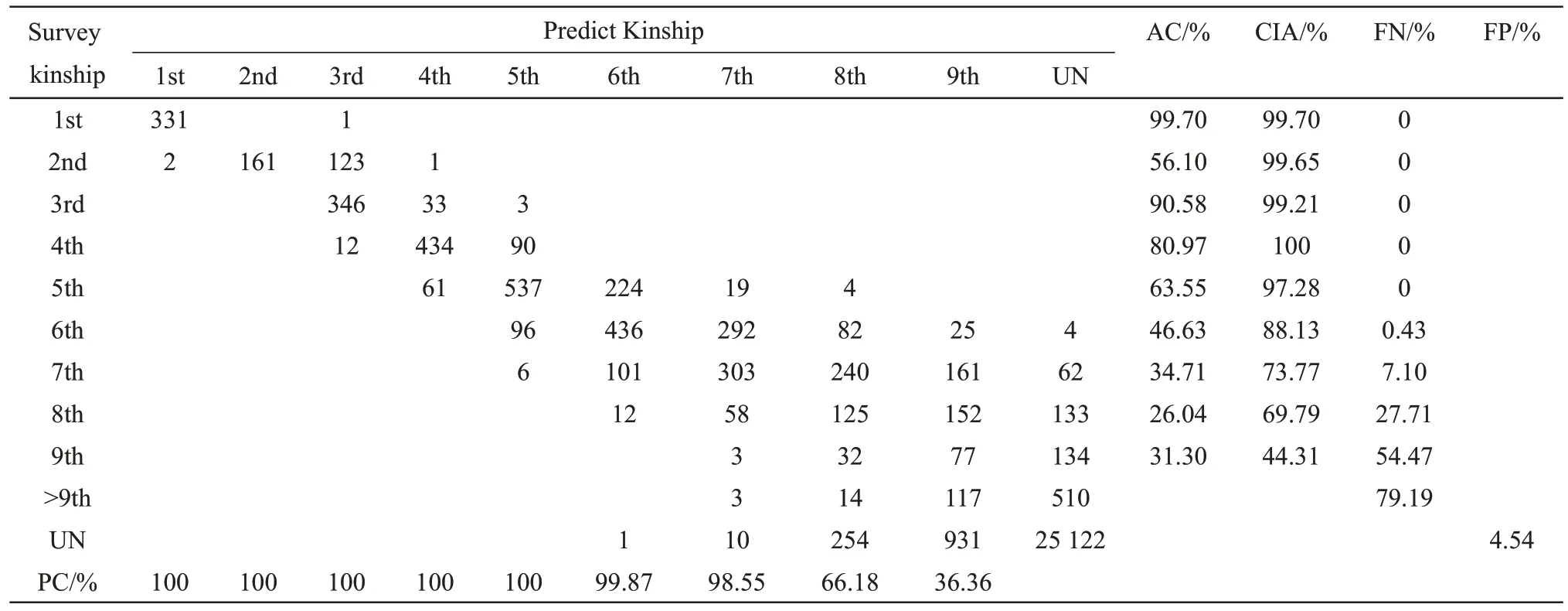
Table 2 Accuracy statistics of SVM IBD algorithm in predicting kinship degree of 253 samples
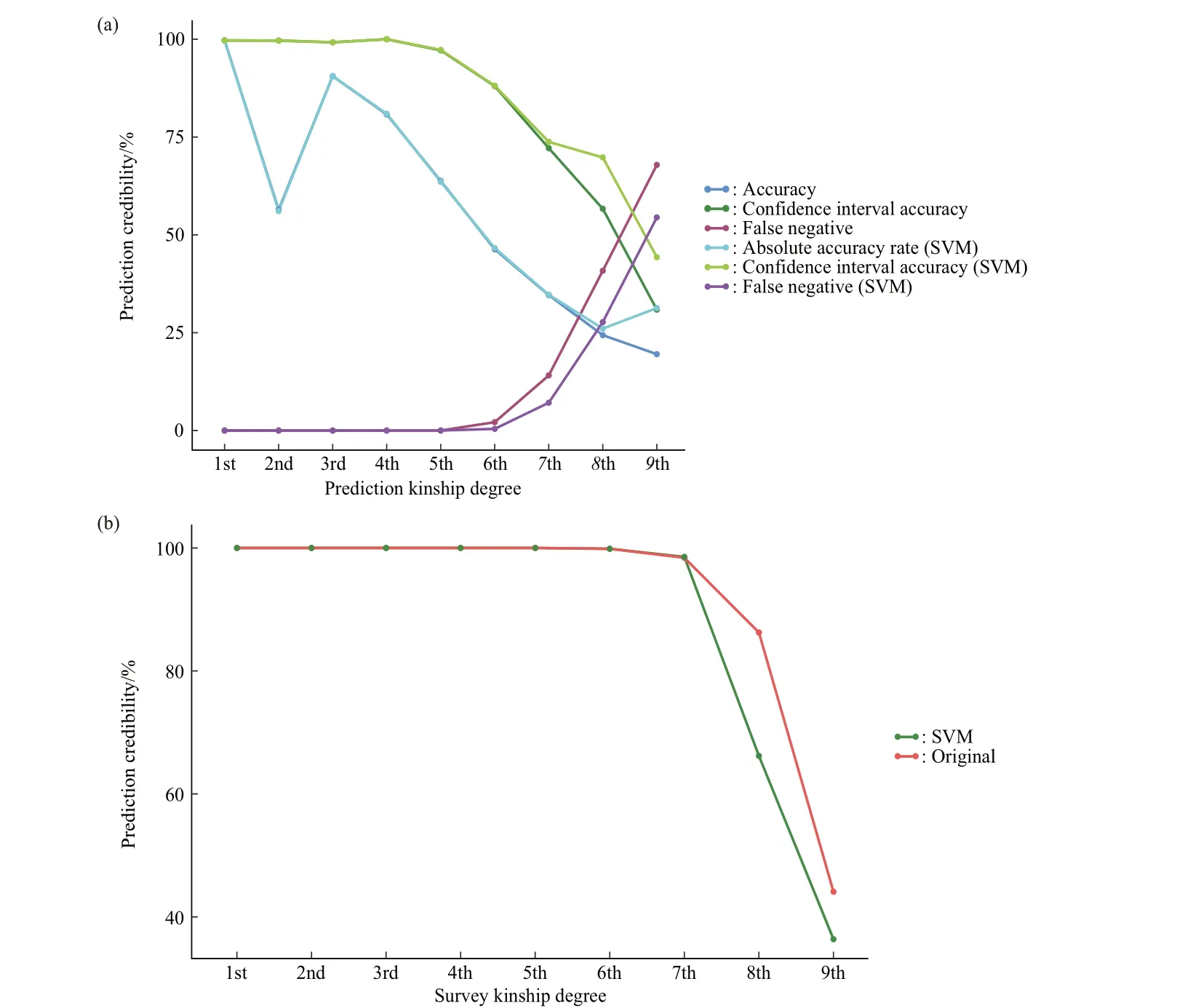
Fig.5 Accuracy evaluation before and after SVM optimization
2.3 真实亲缘关系IBD片段长度分布
通过研究构建的算法流程,本文分析253份样本所有真实亲缘关系对的ⅠBD 片段长度分布情况。图6 显示了253 份样本之间真实亲缘关系等级的ⅠBD片段长度分布图,可以看出1~4级关系能明显分离开来,5级以上亲缘关系ⅠBD片段长度分布出现重叠,亲缘关系越远长度分布的重叠越多。
2.4 不同SNP数量预测准确性
生物检材受时间和环境等因素影响,DNA 会发生降解,SNP位点检出数也会随之降低。因此本文通过随机筛选不同数量的位点组合,模拟低质量样本的预测结果。从253份样本数据的所有SNP位点中,随机筛选65万~10万、每组递减5万位点共12组,每组位点个数随机取2次,使用优化后ⅠBD片段算法预测亲缘关系,计算AC、CⅠA 等评估指标的2次平均值。不同数量位点预测准确性趋势如图7 所示。图7a 展示了不同SNP 数量AC 变化情况;图7b 展示了不同SNP 数量CⅠA 变化情况;图7c展示了不同SNP数量PC变化情况;图7d展示了不同SNP数量FN变化情况。结果显示,随着SNP数量的降低,各预测准确性评估指标会有一定程度的下降(FN 和FP 是随着SNP 数量的降低而略升高),SNP 位点数下降对于超过5 级的亲缘关系预测能力影响更明显。
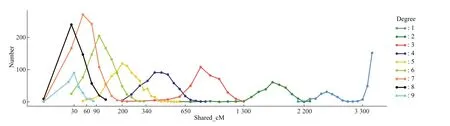
Fig.6 IBD fragment length statistics for each actual kinship degree of 253 samples
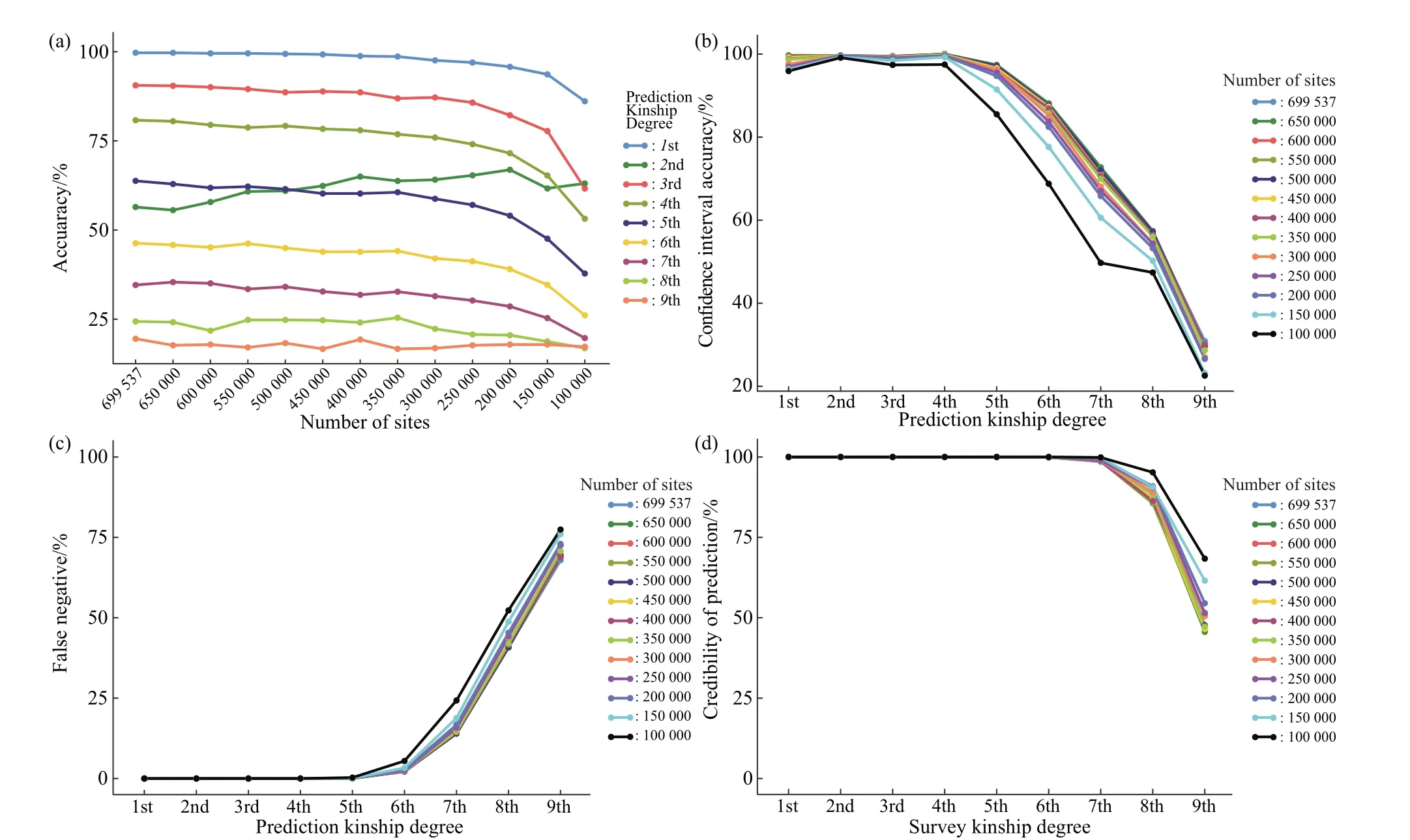
Fig.7 Accuracy evaluation of different SNP number
3 讨 论
在群体遗传学和法医遗传学应用研究中,使用高密度SNP 数据进行亲缘关系预测受到越来越多的关注。项目组先前构建了基于ⅠBS算法的分析流程,并评估了其在中国人群真实亲缘中预测准确性[16],结果显示,该算法在4 级以内亲缘关系有较高预测准确性,5级及更远亲缘关系预测准确性下降明显。ⅠBD片段算法可以准确预测1~7级亲缘关系,在法医SNP 系谱推断中有广泛应用。但国内缺乏ⅠBD片段算法分析流程、针对中国人群的算法优化以及真实亲缘关系预测准确性等系统研究。本文搭建了基于ⅠBD片段算法预测亲缘关系的全流程,实现了输入数据格式预处理、批量化计算两两个体间ⅠBD片段长度和亲缘关系等级等功能,并通过软件进行了分析流程的集成自动化。使用该分析流程对253份样本两两间1~9级亲缘关系进行预测(表1),结果显示ⅠBD 片段算法可以准确预测1~7级亲缘关系,平均CⅠA为94.49%。
为进一步提升ⅠBD片段算法预测准确性,本文进行了最低检出ⅠBD 片段阈值和SVM 等优化。不同最低检出ⅠBD 片段长度阈值结果(图4)显示,0~9 cM 的ⅠBD 片段阈值参数对预测准确性影响不大,大于9 cM时预测准确性会有一定程度的降低,分析其原因可能为:本流程所使用预测亲缘等级算法[18]根据两个个体间共享ⅠBD片段的数量、长度和位置,使用原假设(两个体不相关)与备择假设(两个体共享有共同祖先)进行概率比较。原假设中需要考虑群体中所有共享片段长度的均值,即群体中随机无关个体共享ⅠBD片段长度,故较短ⅠBD片段在该算法中会被当作群体背景噪音。Kling等[5]研究表明,最小ⅠBD 片段阈值最好选取在3~8 cM之间;De Vries等[20]研究表明,设置1~7 cM最小ⅠBD 片段阈值,在1 000 对1~5 级模拟亲缘关系中预测准确率基本无变化,以上研究结论与本文基本一致。本文还观察到亲缘关系越远,受不同ⅠBD 片段长度阈值影响越大(图4a),分析其原因可能为:由于基因重组的随机性,亲缘关系越远,两两个体间共祖片段越短,故受最低检出阈值影响就越大。结合本文研究结果,为在实战中尽可能找到多的亲缘关系,在后续分析中采用的阈值为3 cM。使用SVM优化ⅠBD片段算法后(图5),1~5级亲缘关系预测的AC、CⅠA等评估指标均无显著变化(ANOVA 方差分析,P=0.98),6 级FN 从2.1%降低至0.4%,7 级FN 从14.1%降低至7.1%,7~9级CⅠA也有一定升高,1~7级预测亲缘的PC经SVM 优化后依然保持较高准确率,平均PC 为99.77%。综上所述,原ⅠBD 片段算法经SVM 优化后在远亲缘关系的预测能力方面提升显著。本文对优化后的ⅠBD 片段算法预测结果进一步探究发现,7 级 的FN 为7.1%,8 级、9 级FN 急 剧 上 升 至27.71%、54.47%。Greytak等[6]研究表明,由于基因重组的随机性,大约有10%的3 代表亲(3rd cousin,即7 级)和50%的4 代表亲(4th cousin,即9级)没有可检测到的ⅠBD片段长度,与本文观察到的FN结果基本一致。原因可能在于全基因组SNP芯片检测的位点数量有限,在全基因组层面位点之间分布距离较大,导致部分远亲缘关系对的ⅠBD 片段检测不到;Al-Khudahair 等[21]使用WGS数据的探索研究表明,若SNP位点分布密度增加,有可能提高8 级以及更远亲缘的预测能力。5 级及以下FN为0%,6级以上出现假阴性,7级的FN为7.1%。故系谱推断实践应用时:5级及以下家系只需检测一个样本;6、7级家系至少检测两个样本,这样目标样本与至少一个样本确证可检出亲缘关系的概率可达到99.99%和99.50%。在为侦查提供线索过程中我们发现,更加关注的CⅠA对于1~7级亲缘均超70%,但是2级亲缘关系预测的AC却较低,大量2 级亲缘被预测到3 级。分析其原因可能是,本流程所使用开源的预测亲缘等级算法构建和验证均基于欧美人群[18],在其他人群中的AC会有所降低。Williams 等[22]研究发现,该算法在非洲辛巴族家系人群中,2 级亲缘AC 仅为67%;Ramstetter等[23]研究也发现,在墨西哥家系人群中,大量2级亲缘关系被预测为3级亲缘关系。在不同ⅠBD片段阈值AC结果中,2级亲缘AC值出现了与其他等级关系对不同的“先增后降”趋势,其原因也有可能与人群特异性有关。后续,需要增加中国人群2级和3级关系对数量,继续优化算法模型,提升中国人群中2级亲缘预测的AC。
基于253份样本真实亲缘关系的ⅠBD片段长度分布(图6)显示,1~4 级关系ⅠBD 长度分布区分显著,5 级以上亲缘关系ⅠBD 片段分布会出现重叠,亲缘关系越远分布重叠越多。分析其原因为:亲代向子代传递遗传物质时会发生基因片段的断裂与重组,亲缘关系越远,ⅠBD片段长度会缩短且具有一定随机性。通过将美国Ancestry 公司基于24 362份欧美样本模拟亲缘关系厘摩长度分布与本文中国汉族人群真实亲缘关系厘摩长度分布比较发现,1~7级厘摩分布范围的趋势大致相仿,但中国汉族人群每一级亲缘关系对ⅠBD片段分布范围的上下限数值与之有所不同。表明不同人群的祖先群体规模、婚配模式等的差异,会影响不同级别亲缘关系对的ⅠBD片段长度分布范围。下一步,我们将通过模拟家系和真实家系结合的方式,增加亲缘关系对数量,进一步验证和优化本研究针对汉族人群研究获得的ⅠBD片段分布情况和预测算法,并研究该体系在蒙古族等其他人群中的适应性。
本研究还进一步通过随机筛选位点数,模拟低质量样本的系谱推断预测结果。结果显示,预测准确性随位点数量的减少而降低,当位点数少于20 万位点时准确性下降较明显,但准确性依然处于较高水平,而且位点数量对1~3级近亲缘关系影响更小。但是,我们模拟随机位点数量减少时没有考虑低检出率SNP 芯片数据的分型错误率问题。De Vries 等[20]研究表明,当SNP 芯片的检出率降低时,SNP 位点分型的准确率会下降,而SNP 分型错误,会导致ⅠBD片段识别提前结束,造成ⅠBD片段丢失,并最终降低ⅠBD 片段算法预测准确率,后续将增加该指标进行模拟数据测试,并使用真实的低质量检材进行系统的测试。此外,高深度WGS 技术可生成同一位置大量短读序列片段(reads),从而确保检出SNP 位点分型的准确性,针对微量DNA 有可能获得比SNP 芯片更加准确的分型结果。
4 结 论
本研究构建了基于高密度SNP 数据的ⅠBD 片段算法分析流程并进行了优化,基于253份中国人群样本的真实亲缘关系评估了算法预测准确性。研究结果表明,该算法可实现1~7 级亲缘关系的预测。该方法可辅助物证鉴定工作,为冷案积案等疑难案件侦破提供重要科技支撑。
猜你喜欢
杂志排行
生物化学与生物物理进展的其它文章
- 光泵磁强计双轴探测听觉诱发脑磁信号的初步探索*
- Optically Pumped Magnetometer Lights up The Era of Vector Detection for Magnetoencephalography:an Experimental Evidence
- Prediction of m6A Methylation Sites in Mammalian Tissues Based on a Double-layer BiGRU Network*
- 人乳寡糖的结构及其分离分析*
- TRPM7生理病理学功能及其小分子调节剂的发现*
- GSDMs家族蛋白介导细胞焦亡在抗肿瘤免疫中的作用*