基于数据库探索HLA-DPB1基因在泛癌中的潜在价值
2023-12-31王司颖闫瑞玲管保章张伟
王司颖, 闫瑞玲, 管保章, 张伟*
(暨南大学 附属第一医院 1.乳腺外科; 2.妇产科; 3.肾脏内科, 广东 广州 510630)
肿瘤的发生发展是一个复杂的过程,涉及癌细胞的增殖、转移及免疫逃逸等环节。肿瘤免疫治疗通过重启肿瘤免疫循环,恢复机体抗肿瘤免疫反应从而抑制和杀灭肿瘤细胞,被认为是唯一有望攻克肿瘤的治疗方法,是当前肿瘤领域的研究热点[1-2]。人类白细胞抗原-DPB1(HLA-DPB1)是免疫系统重要的组成部分,其作为跨膜糖蛋白可与外源性肽类分子结合,介导经典型MHC-Ⅱ类分子抗原提呈途径,激活CD4+T细胞,促进T细胞增殖和细胞毒性反应[3-4]。HLA-DPB1不仅存在于免疫细胞表面,还存在于肿瘤细胞表面[5]。已有研究发现HLA-DPB1与乳腺癌、恶性黑色素瘤、宫颈癌等肿瘤的发生发展存在功能联系[6-8]。然而,目前尚缺少HLA-DPB1在泛癌中的研究,HLA-DPB1对肿瘤患者预后的影响及其潜在调控机制的研究更少。本研究利用多个组学数据库系统性分析了HLA-DPB1在泛癌组织中的差异性表达、基因突变图谱及其与免疫浸润的相关性,初步探讨了其与肿瘤患者预后及肿瘤微环境的相关性,为后续HLA-DPB1在肿瘤领域中的研究提供了参考。
1 材料与方法
1.1 泛癌数据采集
从UCSC Xena (https://xenabrowser.net/datapages/)获得经Toil流程统一处理的TCGA_GTEx数据集,其中包括TCGA样本和正常组织样本(Genotype-Tissue Expression,GTEx)的基因表达矩阵[9],同时从癌症基因组图谱(TCGA, https://portal.gdc.cancer.gov/)下载33种肿瘤的表达谱数据和临床数据。
1.2 HLA-DPB1在泛癌中的表达分析
在TCGA_GTEx样本、TCGA样本和TCGA配对样本中,分别比较了正常组织和肿瘤组织中HLA-DPB1的mRNA表达。在UALCAN数据库(http://ualcan.path.uab.edu/)中使用临床蛋白质组学肿瘤分析联盟(clinical proteomic tumor analysis consortium,CPTAC)板块分析HLA-DPB1在正常和肿瘤组织的蛋白质表达差异。此外,在人类蛋白质图谱(human protein atlas,HPA)数据库中检索获得HLA-DPB1在正常和肿瘤组织中的免疫组织化学染色图像。
1.3 HLA-DPB1在泛癌中的预后分析
分析TCGA数据库中HLA-DPB1表达在泛癌中预后评估的价值。根据每种肿瘤中HLA-DPB1表达量的中位数将患者分成高、低两组,使用“survfit”函数分析两组的预后差异,绘制Kaplan-Meier曲线观察HLA-DPB1的表达水平对每个肿瘤总生存期(overall survival,OS)的影响。此外使用 “coxph”函数建立Cox模型,分析HLA-DPB1在33 种肿瘤中的表达与肿瘤预后的相关性,计算风险比(hazard ratio,HR) 。
1.4 遗传突变分析
使用cBioportal 数据库 (https://www.cbioportal.org/)“Quick Search”模块选择 “TCGA PanCancer Atlas Studies”,输入HLA-DPB1进行泛癌基因突变类型汇总。通过“Mutation”模块整合了泛癌样本的突变数据,获得HLA-DPB1功能结构域图上具体的突变位点信息,进行可视化分析。此外在“Query”板块中分别选择某一类型的肿瘤,在 “Enter Genes”一栏输入HLA-DPB1,同时使用“Comparsion/Survival”模块观察各个肿瘤中HLA-DPB1突变对肿瘤预后的影响。
1.5 免疫浸润分析
分析TCGA数据库中HLA-DPB1的表达与泛癌中免疫学特征的相关性。分别使用ESTIMATE方法计算各个肿瘤样本中的基质评分(stromalscore),免疫评分(immunescore)和ESTIMATE 评分,采用Timer方法评估各个肿瘤样本中6种免疫浸润细胞评分(B 细胞、CD4+T 细胞、CD8+T 细胞、中性粒细胞、巨噬细胞和树突状细胞),使用“corr.test”函数计算了各个肿瘤中基因与免疫评分以及免疫细胞浸润评分的相关性。此外借助Timer 2.0(TIMER 2.0,http://timer.cistrome.org/)分析了HLA-DPB1的表达与8个免疫检查点相关基因(SIGLEC15,IDO1,CD274,HAVCR2,PDCD1,CTLA4,LAG3和PDCD1LG2)的相关性。
1.6 功能富集分析
使用GEPIA2(http://gepia2.cancer-pku.cn/)选择“Similar Genes Detection”板块输入“HLA-DPB1”,选中TCGA数据集中的所有肿瘤,提取具有类似HLA-DPB1表达模式的前100个基因。这100 个HLA-DPB1相关基因被用于 STRING 网站(https://cn.string-db.org/)上生成蛋白质-蛋白质相互作用(PPI)网络,所需的最低交互阈值设置为0.4。基于这100个基因使用R软件包clusterProfiler[4.4.4]进行基因本体论(gene ontology,GO)分析及京都基因与基因组数据库(kyoto encyclopedia of genes and genomes,KEGG)分析,探索HLA-DPB1相关基因在肿瘤患者中的潜在分子机制和信号转导途径。同时使用GSEA软件(4.3.2版)选择预后与HLA-DPB1表达相关的肿瘤表达矩阵文件,以HLA-DPB1表达值中位数为界将样本分成高表达组(≥50%)和低表达组(<50%),选择c2.cp.all.v2022.1.Hs.symbols.gmt [All Canonical Pathways],采用默认加权富集法,观察HLA-DPB1高、低表达组间的差异途径。随机组合设定为1 000次。以P<0.05且FDR<0.25为差异有统计学意义。
1.7 单细胞测序数据分析
CancerSEA(http://biocc.hrbmu.edu.cn/CancerSEA/home.jsp)是一个专门用于单细胞测序的数据库,可以在单细胞水平上提供肿瘤细胞的不同功能状态[10]。根据CancerSEA的单细胞测序数据,分析了HLA-DPB1表达与不同肿瘤的生物学行为的相关性。T-SNE图是一种降维可视化技术,可以将高维的表达谱数据转换成二维或三维图形,以便更好地观察细胞之间的相似性和差异性。单细胞T-SNE图在CancerSEA网站获得的。
1.8 统计学分析
所有统计学分析均使用R软件包(v4.0.2),Wilcoxon秩和检验分析HLA-DPB1在不同肿瘤组织和对应正常组织中的表达水平;单因素Cox回归分析计算HLA-DPB1在肿瘤中的表达和患者生存预后的相关性;Kaplan-Meier曲线和log-rank检验比用来比较HLA-DPB1的表达水平高低与肿瘤总生存期的差异;以P<0.05为差异有统计学意义。在Spearman相关性分析中,当P<0.05,|R|>0.8被认为具有强相关性。
2 结果
2.1 HLA-DPB1在恶性肿瘤组织中表达异常
箱式图显示HLA-DPB1在TCGA_GTEx、TCGA及配对TCGA样本中的表达情况,如图1A显示,与正常组织相比,在乳腺浸润癌(BRCA)、宫颈鳞癌和腺癌(CESC)、胆管癌(CHOL)、结肠癌(COAD)、弥漫性大B细胞淋巴瘤(DLBC)、食管癌(ESCA)、多形性胶质母细胞瘤(GBM)、肾透明细胞癌(KIRC)、肾乳头状细胞癌(KIRP)、急性髓细胞样白血病(LAML)、脑低级别胶质瘤(LGG)、肝细胞肝癌(LIHC)、肺腺癌(LUAD)、卵巢浆液性囊腺癌(OV)、胰腺癌(PAAD)、前列腺癌(PRAD)、直肠腺癌(READ)、皮肤黑色素瘤(SKCM)、胃癌(STAD)、睾丸癌(TGCT)、甲状腺癌(THCA)、胸腺癌(THYM)中HLA-DPB1高表达(P<0.05),在肾上腺皮质癌(ACC)、膀胱尿路上皮癌(BLCA)、肺鳞癌(LUSC)、子宫肉瘤(UCS)中HLA-DPB1低表达(P<0.05)。在图1B、C中同样观察到HLA-DPB1在BRCA、KIRC、KIRP、STAD表达上调,而在LUSC中表达下调。
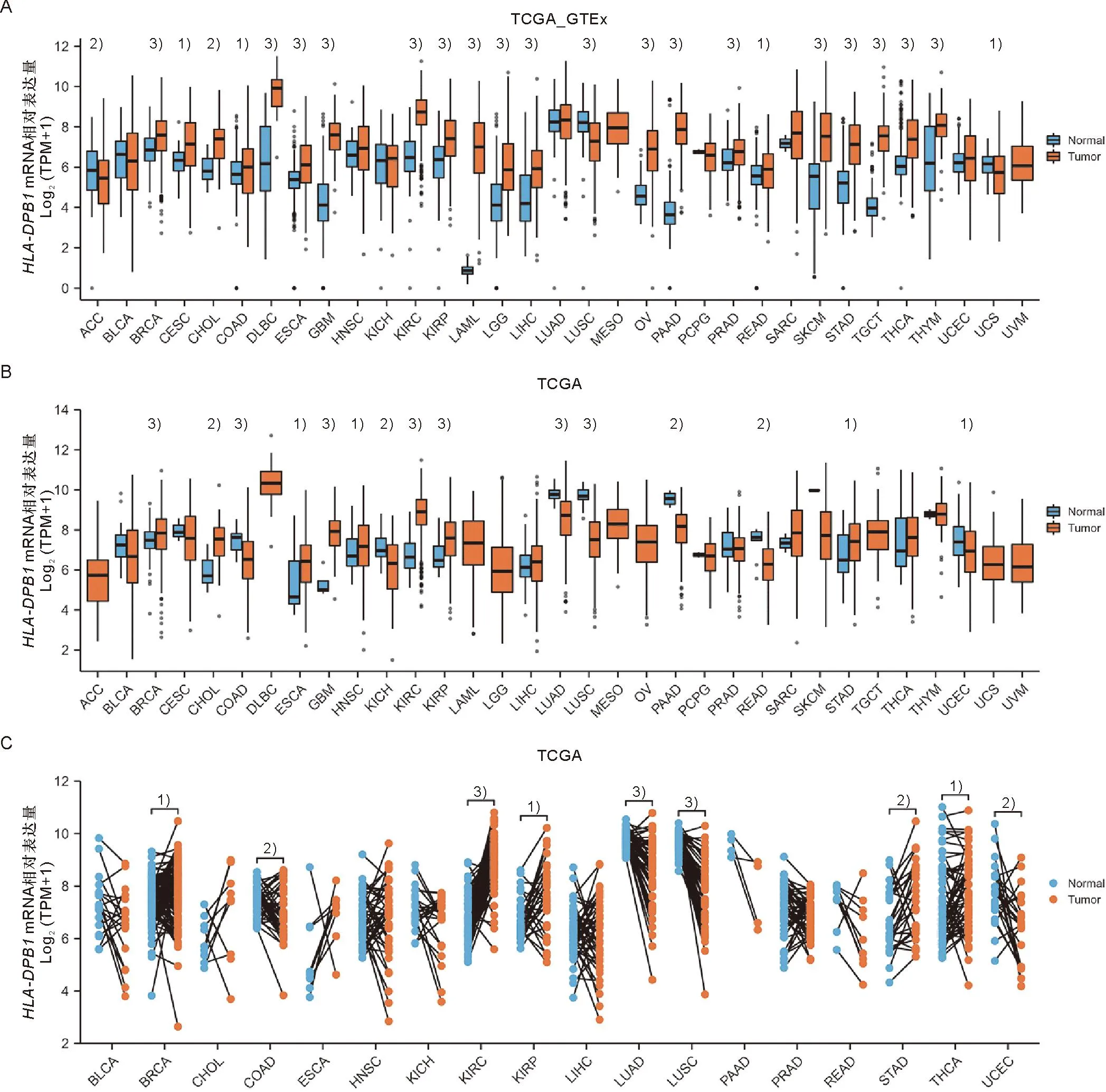
1)P<0.05, 2)P<0.01, 3)P<0.001A: TCGA_GTEx数据库中33 种肿瘤组织和正常组织中HLA-DPB1 mRNA的相对表达水平; B: TCGA 数据库中 33 种肿瘤组织和正常组织中HLA-DPB1 mRNA的相对表达水平; C: TCGA 数据库中18个肿瘤配对样本中HLA-DPB1 mRNA的相对表达水平。A: The mRNA expression of HLA-DPB1 in 33 tumors in TCGA_GTEx samples; B: The mRNA expression of HLA-DPB1 in 33 tumors in TCGA database; C: The mRNA expression of HLA-DPB1 in paired samples of 18 tumors in TCGA database.图1 HLA-DPB1 mRNA在肿瘤组织和正常组织中的表达分析Figure 1 The mRNA expression of HLA-DPB1 in normal and tumor tissues
在蛋白水平上进一步比较了HLA-DPB1在恶性肿瘤及其对应正常组织中的表达差异。结果提示在KIRC、子宫内膜癌(UCEC)、PAAD、头颈部鳞癌(HNSC)、GBM中HLA-DPB1蛋白表达上调(图2A)。HPA 数据库免疫组化染色数据表明在BRCA、OV、PAAD、STAD、SKCM、LIHC中HLA-DPB1蛋白表达上调(图2B)。

A:HLA-DPB1在正常和肿瘤组织中的标准化蛋白表达密度;B:从HPA中提取的正常和肿瘤组织中HLA-DPB1的IHC图像。A:Standardized expression density of HLA-DPB1 in human tumor tissues;B:The IHC images of HLA-DPB1 in normal and tumor tissues extracted from the HPA.图2 HLA-DPB1蛋白在肿瘤组织和正常组织中的表达分析Figure 2 The protein expression of HLA-DPB1 in normal and tumor tissues
综上,HLA-DPB1在大多数恶性肿瘤组织中存在异常表达且表达水平存在一定的组织特异性。其中,HLA-DPB1在BRCA、PAAD、STAD、KIRC、GBM中的表达(mRNA及蛋白水平)显著高于其对应正常组织(P<0.05)。
2.2 HLA-DPB1的差异性表达与肿瘤患者预后密切相关
本研究将患者的OS作为预后的衡量指标,进行了HLA-DPB1表达水平与OS的相关性分析。K-M生存曲线显示,HLA-DPB1在CESC(P=0.03)、KIRC(P=1.8×10-3)、LUAD(P=1.0×10-4)、UCEC(P=0.02)、SARC(P=4.5×10-3)、SKCM(P=5.6×10-7)中高表达的患者比低表达的预后更好,在LAML(P=3.1×10-3)、LGG(P=3.4×10-7)、THYM(P=9.2×10-3)、葡萄膜黑色素瘤(UM)(P=0.02)患者中HLA-DPB1高表达预后不良(图3A)。
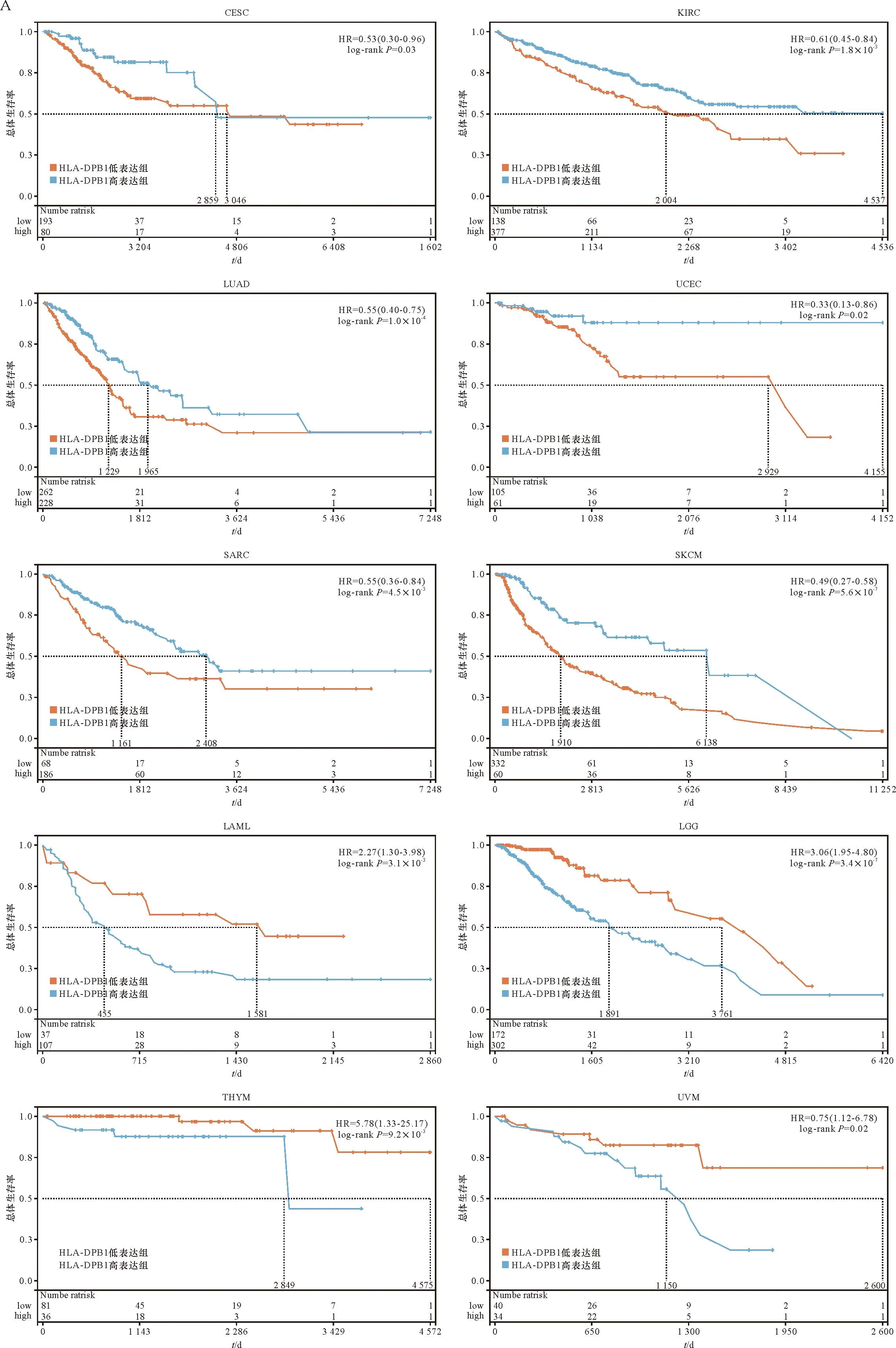
A: HLA-DPB1高/低表达对总生存的影响; B: HLA-DPB1表达与33个恶性肿瘤中OS相关性的森林图。A: Overall survival (OS) of patients with high and low HLA-DPB1 expression; B: HLA-DPB1 related to OS in pan-cancer were exhibited by a forest plot.图3 HLA-DPB1表达与泛癌预后的相关性Figure 3 Correlation between HLA-DPB1 expression and pan-cancer prognosis
HLA-DPB1在4种肿瘤中HR>1且P<0.05,分别为LAML(HR=1.18,P=5.4×10-3),LGG(HR=1.38,P=2.7×10-8)、THYM(HR=2.81,P=0.01)、UVM(HR=1.5,P=7.5×10-3),表明HLA-DPB1高表达是此类肿瘤的危险因素。HLA-DPB1在5种肿瘤中HR<1且P<0.05,分别为CESC(HR=0.81,P=6.0×10-3)、LUAD(HR=0.87,P=6.3×10-3)、肉瘤(SARC)(HR=0.89,P=0.03)、SKCM(HR=0.81,P=8.5×10-8)、KIRC(HR=0.85,P=8.2×10-3),表明HLA-DPB1高表达是此类肿瘤的保护因素(图3B)。
2.3 HLA-DPB1在泛癌中基因突变情况
利用癌症基因组学cBioPortal中TCGA泛癌数据分析了不同肿瘤中HLA-DPB1的遗传变异情况及基因突变与患者预后的相关性。结果显示,HLA-DPB1基因变异率排名前5的肿瘤分别是DLBC(4.17%)、CHOL(2.78%)、OV(2.57%)、葡萄膜黑色素瘤(UM)(2.57%)、SKCM(2.48%)。其中CHOL、OV、UM以扩增为主要类型(图4A)。进一步分析显示HLA-DPB1的错义突变是遗传改变的主要类型,HLA-DPB1蛋白序列的第160号氨基酸由谷氨酰胺(Q)突变为丝氨酸(S)并产生新的阅读框架,终止于第160号密码子下游35号密码子处(图4B)。此外,本研究分析了HLA-DPB1基因突变对肿瘤患者预后的影响。结果显示,携带HLA-DPB1基因突变的皮肤肿瘤患者与野生型患者相比预后更差,而在其他肿瘤中未观察到这一现象(图4C)。

A:HLA-DPB1在不同肿瘤中的突变情况;B:HLA-DPB1在每个蛋白结构域上具体的突变图谱;C: HLA-DPB1突变/未突变对皮肤肿瘤预后的影响。A: HLA-DPB1 mutations in different tumors; B: Specific mutation map of HLA-DPB1 on each protein structural domain; C: Prognostic impact of HLA-DPB1 mutated/unmutated in skin tumors.图4 HLA-DPB1在泛癌中的遗传变异情况Figure 4 Genetic variation of HLA-DPB1 in pan-cancer
2.4 HLA-DPB1表达水平与肿瘤免疫微环境密切相关
分析HLA-DPB1的表达与肿瘤微环境的相关性,图5A结果显示,在33种肿瘤中,HLA-DPB1表达量与基质评分最相关的3种肿瘤是LGG(r=0.78)、READ(r=0.86)和PAAD(r=0.78);与免疫评分最相关的3种肿瘤是COAD(r=0.87)、HNSC(r=0.86)和THCA(r=0.87);与ESTIMATE评分最相关的3种肿瘤是LUSC(r=0.84)、READ(r=0.90)和PAAD(r=0.86)。HLA-DPB1表达水平与肿瘤免疫细胞浸润的相关性分析显示, HLA-DPB1的表达水平与B细胞、CD4+T细胞、CD8+T细胞、树突状细胞、巨噬细胞和中性粒细胞的水平呈显著正相关(图5B)。HLA-DPB1与免疫检查点之间的相关性分析显示, HLA-DPB1的表达水平与CD274、CTLA4、HAVCR2、LAG3、PDCD1、PDCD1LG2、TIGIT的表达水平呈显著正相关 (图5C)。以上研究结果提示HLA-DPB1表达与恶性肿瘤微环境的调控密切相关,是具有重要价值的潜在泛癌免疫学标志物。
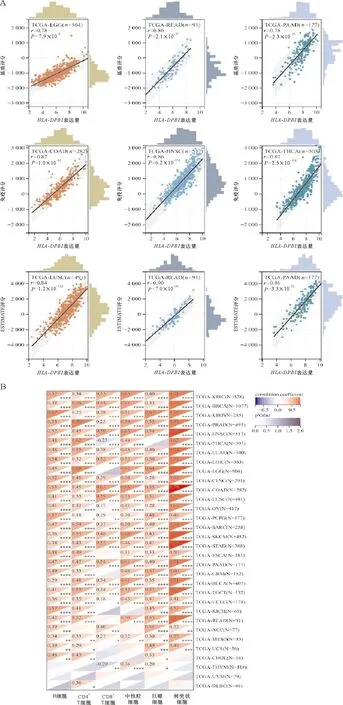
*P<0.05,**P<0.01,***P<0.001,****P<0.0001.A:HLA-DPB1表达与基质评分,免疫评分以及ESTIMATEScore的关系;B:泛癌中HLA-DPB1基因表达与免疫细胞浸润水平的相关性;C:泛癌中HLA-DPB1表达与免疫检查点基因表达的相关性。A:The relationship between HLA-DPB1 expression and StromalScore, ImmuneScore ESTIMATEScore;B. Relationship between HLA-DPB1 gene expression and immune cell infiltration in pan-cancer;C.Relationship between HLA-DPB1 expression and immune checkpoint gene expression in pan-caner.图5 HLA-DPB1对恶性肿瘤免疫微环境的影响Figure 5 Effect of HLA-DPB1 on tumor immune microenvironment
2.5 HLA-DPB1及相关基因通路富集分析
对与HLA-DPB1密切相关的前100个基因进行分析并绘制蛋白质互作网络图(图6A),同时对这100个基因进行了GO富集分析,包括生物学过程(biological process,BP)、分子功能(molecularfunction,MF)、细胞组成(celular component,CC)和KEGG富集分析,根据P值选取各组排名前10的条目绘制成图。KEGG富集分析发现,HLA-DPB1相关基因主要富集在免疫激活相关通路和自身免疫性疾病信号通路上(图6B)。GO富集分析发现,HLA-DPB1相关基因集主要富集在“质膜外侧”、 “MHC蛋白复合物”、“ 内吞性囊泡膜”等细胞组件,参与 “免疫受体活性”、“酰胺结合” 、 “肽结合”等分子功能,涉及 “白细胞介导的免疫”、“ 免疫球蛋白超家族构建的体细胞重组的适应性免疫应答”、“ 淋巴细胞介导的免疫”、“ 白细胞细胞间黏附”的生物学过程(图6C)。

A: 100个HLA-DPB1相关基因的蛋白质互作网络图;B: 基于100个HLA-DPB1相关基因的KEGG途径分析;C:基于100个HLA- DPB1相关基因的GO富集分析(BP、CC和MF); D:HLA-DPB1高、低表达组在CESC、KIRC、LUAD、UCEC、SARC、SKCM、LAML、LGG、THYM、UVM中的GSEA富集分析。A: 100 HLA-DPB1 related genes were used to generate a PPI network on the STRING website; B:KEGG pathways analysis based on 100 HLA-DPB1 related genes; C: GO enrichment analysis based on 100 HLA-DPB1 related genes (BP, CC, and MF); D: GSEA analysis between HLA-DPB1 high expression and low expression groups in CESC,KIRC,LUAD,UCEC,SARC,SKCM,LAML,LGG,THYM,UVM respectively.图6 HLA-DPB1相关基因的功能富集分析Figure 6 Functional enrichment analysis of HLA-DPB1 related genes
为了进一步阐明HLA-DPB1在恶性肿瘤中的生物学功能,采用GSEA软件分析了HLA-DPB1高表达组和低表达组差异富集的信号通路。结果显示,HLA-DPB1高表达组主要富集于FcεRI介导Ca2+动员、FcεRI介导MAPK激活、 FcεRI介导NF-κB通路激活、FCGR3调控白介素10合成及JAK-STAT信号等通路。HLA-DPB1低表达组在各个肿瘤中的富集通路不尽相同,在UVM、SKCM中主要富集于核糖体通路,而在UCEC、THYM中主要富集于C3类代谢型谷氨酸信息素受体通路上(图6D)。
2.6 HLA-DPB1在不同肿瘤细胞中的功能相关性分析
单细胞转录组测序是分析候选分子潜在功能的关键技术。Cancer SEA数据库分析显示,HLA-DPB1在大多数癌中的表达与细胞分化、炎症呈正相关;在视网膜母细胞瘤(RB)中与血管生成呈正相关;在急性骨髓性白血病(AML)中与细胞增殖、炎症等多个肿瘤生物行为呈正相关(图7A)。在UM中HLA-DPB1表达几乎与所有肿瘤生物行为呈高度负相关,如DNA损伤、DNA修复反应、细胞凋亡、侵袭和转移(图7B)。图7C所展示的T-SNE图显示了HLA-DPB1在RB、UM和AML 3种癌症类型中,单个细胞的HLA-DPB1表达谱提示不同癌症中存在表达HLA-DPB1的特定细胞亚群,其可能在免疫反应或肿瘤发展中起着特定的作用,这对于深入了解HLA-DPB1在泛癌中的功能和作用机制非常重要。
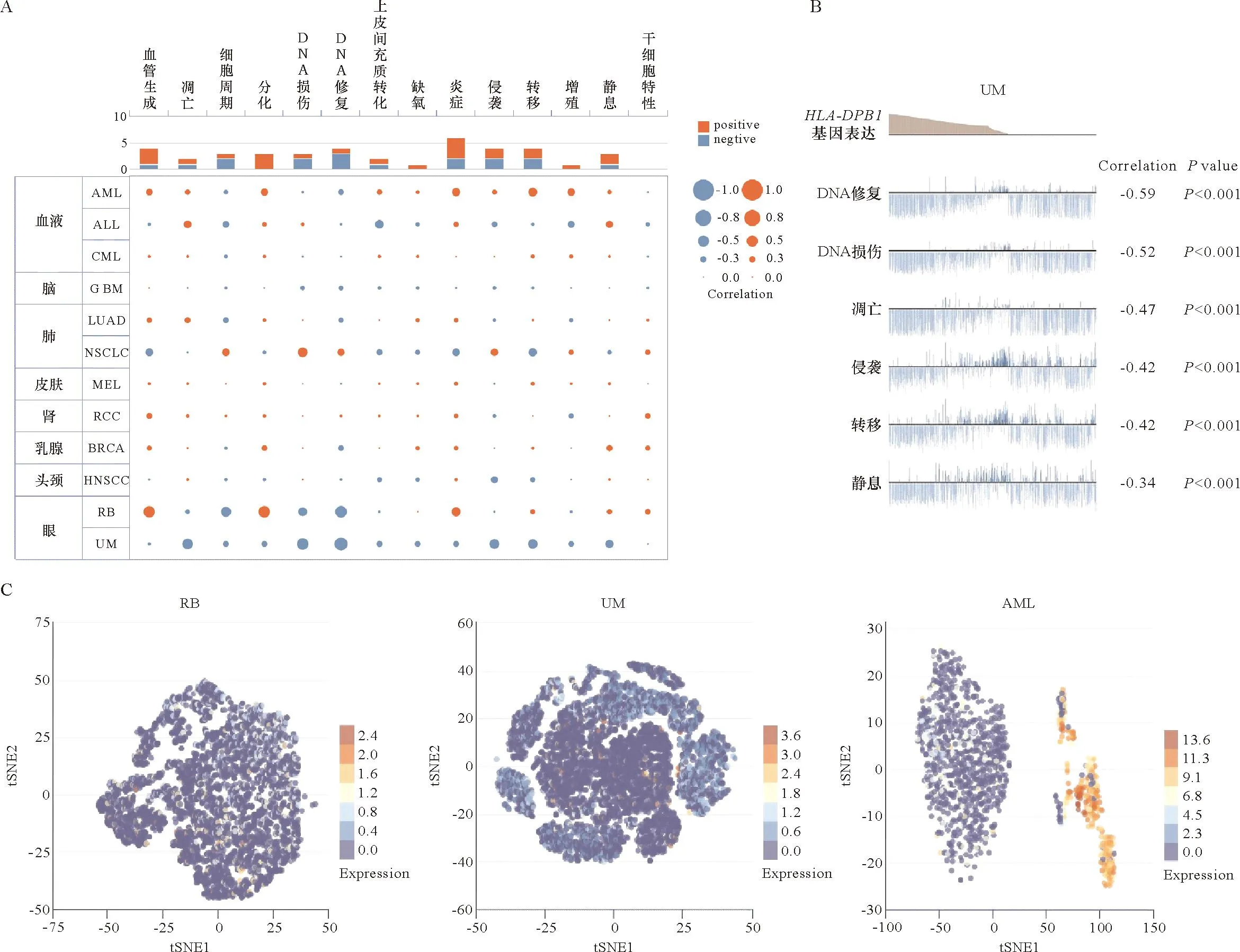
A: 热图显示HLA-DPB1表达与不同肿瘤功能状态的相关性; B: UM中HLA-DPB1表达与不同功能状态之间的关系; C: T-SNE图显示RB、UM和AML的单个单元格中HLA-DPB1表达谱。A: Heatmap showed the correlation between HLA-DPB1 expression and different tumor functional status based on CancerSEA database; B: Correlation between HLA-DPB1 expression and functional states in UM; C: HLA-DPB1 expression profiles were shown at single cells from RB, UM and AML by T-SNE diagram.图7 HLA-DPB1在单细胞水平上的表达与肿瘤功能状态的相关性Figure 7 Correlation between HLA-DPB1 expression at the single-cell level and functional status
3 讨论
目前癌症基因组学的研究已进入泛癌时代,泛癌研究将帮助人类了解基因在恶性肿瘤中的表达及作用,便于发现恶性肿瘤的潜在治疗靶点[11]。MHC-Ⅱ类分子包括HLA-DR、HLA-DQ和HLA-DP,参与机体抗肿瘤免疫过程[12-13]。在人类6号染色体HLA-DP区,包含编码α链的DPA1和DPA2以及编码β链的DPB1和DPB2。其中DPA1和DPB2为功能基因,可编码蛋白。近年来HLA-DPB1基因被证明与多种自身免疫性疾病密切相关[14-16]。HLA-DPB1的表达会影响过敏原免疫治疗的效果[14]。探讨HLA-DPB1的表达与恶性肿瘤的关系和作用机制,有助于理解HLA-DPB1在肿瘤发生发展中所扮演的角色,为肿瘤预后的预测和免疫治疗提供新思路、新途径。
本研究利用公共数据库中大量肿瘤患者基因组学信息,系统比较了HLA-DPB1在多种恶性肿瘤组织及对应正常组织中的差异性表达,并分析了其表达与患者预后的相关性。研究结果提示HLA-DPB1在大部分实体肿瘤组织中表达升高,并与CESC、UCEC、LUAD、SARC、SKCM、KIRC的良好预后相关。既往研究发现在IFN-γ,TNF等细胞因子诱导下,肿瘤细胞和癌旁细胞可发生MHC-Ⅱ类基因的异常表达[17]。MHC-Ⅱ类分子在肿瘤中的表达减少预示肿瘤恶性度高、预后差[18-19]。Leite等[20]报道了HLA-DPB1在小儿肾上腺皮质肿瘤中的低表达与不良预后显著相关;Lyu等[6]发现HLA-DPB1在乳腺癌中表达升高与良好预后相关。上述研究结果说明HLA-DPB1可能成为预测肿瘤预后的新标志物。基因突变与肿瘤的发生、进展密切相关[21],本研究发现HLA-DPB1突变广泛存在于多种肿瘤中,并且可能涉及氨基酸序列改变以及严重的移码突变,生存分析发现HLA-DPB1突变与皮肤肿瘤患者不良预后密切相关。
研究发现MHC-Ⅱ类分子在黑色素瘤细胞中异常表达可吸引肿瘤特异性CD4+T细胞,引发TNF主导的局部炎症反应[7]。Souri等[22]在高风险UM中发现LAG-3表达增加伴随着MHC-Ⅱ类分子的表达上调[22]。McCaw等[23]基于鼠类乳腺肿瘤细胞的研究,发现表达MHC-Ⅱ类分子的肿瘤细胞促进了CD4+T细胞的局部激活,间接促进了CD8+T细胞活化和扩增,与抗CTLA4 联合使用可促进肿瘤消退。本研究分析了TCGA肿瘤患者中HLA-DPB1的表达分别与免疫细胞浸润和免疫检查位点的相关性,结果提示,HLA-DPB1高表达的肿瘤患者表现出更高的免疫评分,与肿瘤浸润性免疫细胞和部分免疫检查点呈显著正相关。因此,HLA-DPB1是肿瘤免疫应答中的关键基因。HLA-DPB1的表达可增加肿瘤免疫微环境的免疫原性,促进淋巴细胞的渗透。通过诱导HLA-DPB1表达的策略可能会增强肿瘤患者对免疫治疗的反应,为肿瘤治疗提供一个新的治疗靶点。
目前,关于MHC-Ⅱ类分子介导肿瘤细胞信号传导通路的研究主要集中在HLA-DR。Aoudjit 等[24]发现HLA-DR在黑色素瘤细胞中的异常表达可激活MAPK/ERK通路、抑制Fas 介导的细胞凋亡。最近Costantini等[25]研究发现,HLA-DR在黑色素瘤细胞中的异常表达介导的信号传导增加了整合素、黏附受体、PD-L1和多种信号转导蛋白(FAK、AKT和STAT3)的表达,促进黑色素瘤细胞增殖和迁移。有研究报道称MHC-Ⅱ类分子表达的上调必须经过JAK/STAT 信号的激活[26],这与本研究在GSEA通路富集分析中观察到JAK-STAT通路富集在HLA-DPB1肿瘤高表达组中的结果一致。另外,本研究还观察到HLA-DPB1高表达组富集于FcεRI介导的Ca2+动员、MAPK激活、NF-κB通路上。结合Cancer SEA分析发现肿瘤细胞中HLA-DPB1的表达与炎症和细胞分化呈正相关。过去的研究证实HLA-DR在肿瘤细胞的表达与其分化程度正相关[27-30]。以上说明HLA-DPB1与HLA-DR功能上相似,均参与肿瘤的免疫反应过程及细胞分化、炎症、血管生成等。另有研究发现用IFN-γ处理黑色素瘤细胞后,HLA-DR、DQ和DP的表达上调程度有所不同[31]。未来需要进一步研究HLA-DR、DQ和DP在肿瘤组织中的差异性表达,以及单独分析这些不同表达在肿瘤发生发展中的作用,以寻找最为合适的治疗靶点。
本研究采用不同数据库对HLA-DPB1在泛癌中的作用进行了生物信息学分析,尚存在一定的局限性,缺乏体内外实验验证和进一步的机制研究。同时,HLA-DPB1在mRNA和蛋白质水平上的差异性表达存在不一致,一方面可能因为存在转录后调控,从转录到蛋白翻译还存在复杂的调节过程;另一方面,由于蛋白表达数据库中的样本数量较小,可能造成统计学的偏差,需要进一步获得临床病理数据进行验证。
综上所述,本研究对泛癌HLA-DPB1的生物信息学分析表明,HLA-DPB1表达水平与肿瘤患者的临床预后及免疫浸润高度相关,其可能通过 FcεRI介导Ca2+动员、MAPK激活及 NF-κB激活参与肿瘤的免疫、炎症反应过程,因此本研究可为HLA-DPB1作为新的肿瘤预后标志物和免疫治疗的潜在靶点提供了理论支持。
作者贡献声明
王司颖:数据统计分析、撰写论文;闫瑞玲:收集数据并修改论文;管保章:指导数据分析;张伟:提出研究思路和框架,修改论文。
利益冲突声明
本研究未受到企业、公司等第三方资助,不存在潜在利益冲突。
