一心维纳斯蝴蝶兰花香物质合成相关基因RT-qPCR内参基因筛选
2023-12-22章杨婷张燕萍黄静妍王文君周育真
章杨婷,张燕萍,黄静妍,王文君,童 妍,赵 凯,周育真*
一心维纳斯蝴蝶兰花香物质合成相关基因RT-qPCR内参基因筛选
章杨婷1,张燕萍1,黄静妍1,王文君1,童 妍1,赵 凯2,周育真1*
1. 福建农林大学风景园林与艺术学院/兰科植物保护与利用国家林业和草原局重点实验室,福建福州 350002;2. 福建师范大学生命科学学院,福建福州 350117
一心维纳斯蝴蝶兰(I-Hsin Venus)花型优雅、花期持久、花香馥郁,是优良的香花品种,具有较高的园林观赏价值和经济价值。为筛选适用于一心维纳斯蝴蝶兰花香物质合成相关基因表达分析的内参基因,本研究以一心维纳斯蝴蝶兰不同花期(中蕾期、初开期、盛开前期、盛开中期、盛开后期、衰败期)的转录组数据为基础,选取了、、、、、、和共8个管家基因作为候选内参基因,利用实时荧光定量PCR(RT-qPCR)技术检测候选内参基因在花朵不同发育时期的表达情况,并利用geNorm、NormFinder、BestKeeper三个内参基因稳定性分析软件对候选内参基因进行分析。综合分析结果表明,的稳定性最高,可作为一心维纳斯蝴蝶兰相关基因表达分析的最适内参基因。
一心维纳斯蝴蝶兰;内参基因;实时荧光定量PCR;表达稳定性
实时荧光定量PCR技术(quantitative real-time PCR, RT-qPCR)是指在PCR反应体系中添加荧光基团(探针或者染料),在PCR扩增进程中借助荧光信号的积累进行实时监测[1]。RT-qPCR目前已普遍应用于分子生物学研究和医学研究等领域[2]。因RT-qPCR的特异性强、无污染性和准确性高,其被广泛应用于基因表达验证方面的研究[3]。但是,RNA的质量或试验操作的偏差等因素会影响最终结果的精准度[4]。使用内参基因对结果进行校对并纠正,可以大大降低目标基因与数据表达水平之间的差异,从而提高试验的准确性。
内参基因通常选用管家基因,这类基因以稳定表达著称,但目前并没有哪个内参基因能确保在所有情况下都能稳定表达[5]。理想的内参基因在不同条件不同组织中均会稳定表达,而试验研究中内参基因的恒定表达仅仅是在一定的试验环境下和一定的组织中才会实现[6]。在特定试验条件下或给特定组织或品种进行RT-qPCR表达稳定分析前,应该选择合适的内参基因。目前常用的内参基因主要包括肌动蛋白基因()、18S核糖体RNA()、3-磷酸甘油醛脱氢酶基因()、转录延伸因子基因()、多聚泛素酶基因()、α微管和β微管蛋白基因(、)以及亲环蛋白基因()等。
蝴蝶兰(spp.)因花朵形态像蝴蝶而得名,又称蝶兰,其花色艳丽,花姿娇俏,花期长,广泛分布于中国、泰国、菲律宾、马来西亚等地,属热带亚热带气生兰,被称为“兰花皇后”。蝴蝶兰的原生种数量少,市场上广泛栽培与应用的品种部分是杂交种,在花卉市场上颇受欢迎[7]。随着市场的不断变化和消费者需求的多样化,品种的不断创新及改良是必要手段,蝴蝶兰香花品种的培育是目前重要的育种目标之一。一心维纳斯蝴蝶兰(I-Hsin Venus)是以金龙蝴蝶兰(Dragon’s Gold)和I-Hsin Viola Tria为亲本杂交选育而成的香花品种,具有花香浓郁、花色绚丽多彩、花型轻盈、花期持久等特性,是研究蝴蝶兰花香的理想材料。前期研究表明,一心维纳斯蝴蝶兰随着花朵开放,花香物质逐渐增加而产生花香,其香气成分主要为萜类物质,且成分复杂。目前还需对其花香合成与释放相关的分子机制进行更深入地挖掘。
本研究基于蝴蝶兰一心维纳斯品种的花朵转录组数据,筛选出、、、、、、和这8个候选内参基因,分别对其进行引物设计,并通过RT-qPCR得到在蝴蝶兰6个不同花期中的表达量。利用软件评估内参基因的稳定性,筛选出理想的内参基因。为后期开展一心维纳斯蝴蝶兰花香物质合成和释放相关功能基因研究提供参考。
1 材料与方法
1.1 材料
本研究以福建农林大学森林兰苑温室大棚中的蝴蝶兰一心维纳斯品种的花朵为材料,于2021年3—6月花朵集中开放时期的10:00—14:00采集,采集包括中蕾期(ZL)、初开期(CK,0 d)、盛开前期(S7,7 d)、盛开中期(S15,15 d)、盛开后期(S30,30 d)、衰败期(SB,45 d及以上)6个时期,设置3个生物学重复。采集后的样品迅速放入液氮中,迅速转移至‒80 ℃冰箱里备用。
1.2 方法
1.2.1 总RNA提取与cDNA合成 使用RNA提取试剂盒(Plant RNA Kit, OMEGA)提取一心维纳斯蝴蝶兰不同花期的总RNA。使用1%的琼脂糖凝胶电泳检测提取RNA的完整性,超微量紫外分光光度计检测RNA的浓度和纯度。使用反转录试剂盒(PrimeScriptTMRT reagent Kit with gDNA Eraser试剂盒,TaKaRa)将检测合格的样品RNA反转录成cDNA,将得到的cDNA样品放于-20 ℃冰箱备用。
1.2.2 候选内参基因筛选 根据已测序完成的一心维纳斯蝴蝶兰不同花期的转录组数据(未发表),FPKM值为92.61以上并且各基因样品表达量较为一致的情况下[8],通过网站(http://orchidbase.itps.ncku.edu.tw/)[9]查找小兰屿蝴蝶兰()基因组数据进入NCBI的Blast P程序进行同源基因比对,初步筛选出8个内参基因(表1)。
1.2.3 RT-qPCR 通过Primer 3(https://primer3.ut. ee/)在线网站和DNAMAN 9.0软件,基于已知的内参基因的核苷酸序列来设计引物(表2),引物由福州白鲸生物科技有限公司合成。使用TB Green Premix ExⅡ试剂盒(TaKaRa公司)进行RT-qPCR,反应体系为:10 μL TB Green Premix ExⅡ、正反引物各0.8 μL、0.4 μL ROX Reference DyeⅡ、6 μL灭菌水、2 μL cDNA溶液,反应程序为:95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火延伸30 s,40个循环。溶解曲线温度设置为60~95 ℃,升温速度为0.2 ℃/s。每个基因每个时期3次技术重复,得出的数据计算△Ct值后使用2‒△△Ct公式运算。
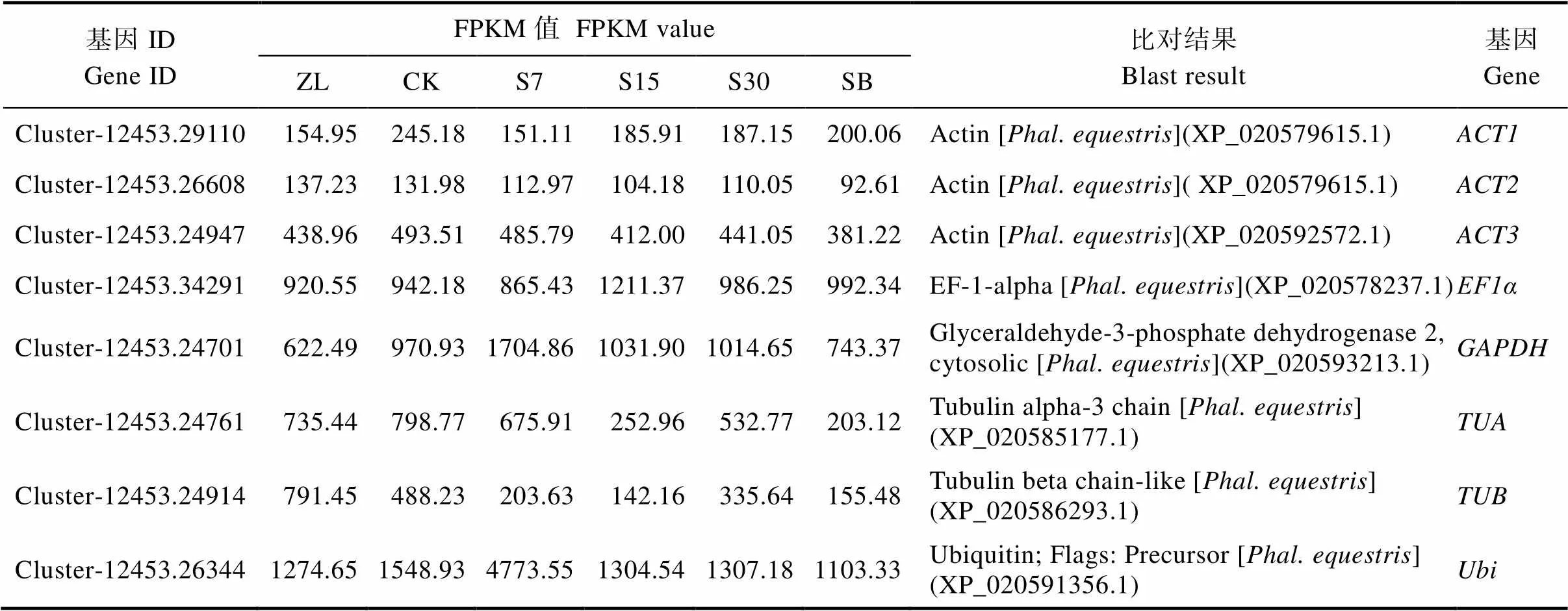
表1 根据FPKM值及Blast P比对筛选到的8个候选内参基因

表2 候选内参基因的引物序列
1.3 数据处理
将一心维纳斯蝴蝶兰6个花期的cDNA等量混合,设置5个浓度梯度(5‒1、5‒2、5‒3、5‒4、5‒5),每个浓度梯度设置3个重复,对样品进行RT-qPCR。以获得的5个梯度下各个内参基因的Ct值为纵坐标,稀释倍数的对数值为横坐标制作标准曲线,并利用公式计算各候选内参基因的扩增效率()[10],=(10‒1/k‒1)´100%。
参考当前已有的试验中对内参基因稳定性的分析方法[11],通过geNorm、NormFinder和BestKeeper三个分析软件对各候选内参基因在一心维纳斯蝴蝶兰6个不同花期中的稳定性进行分析,综合3个软件得出的稳定性排名,筛选出最适的内参基因。
2 结果与分析
2.1 内参基因RT-qPCR分析
RT-qPCR分析得出的结果如图1所示,溶解曲线均仅出现单一峰,表明8个内参基因均未出现引物二聚体。引物二聚体会产生荧光信号,干扰到目的基因的定量[12],无引物二聚体,说明引物的特异性较好,满足RT-qPCR下一步试验的条件。
以一心维纳斯蝴蝶兰不同发育阶段的cDNA为模板进行RT-qPCR扩增,根据标准曲线得到的斜率()、相关系数(2),计算引物扩增效率()。结果表明(表3),和扩增效率分别为121.95%和112.23%,高于110%。其余的6个候选内参基因的扩增效率均在90%~110%之间,且8个候选内参基因相关系数(²)在0.9826~ 0.9998之间,表明特异性及扩增效率良好,满足RT-qPCR操作。
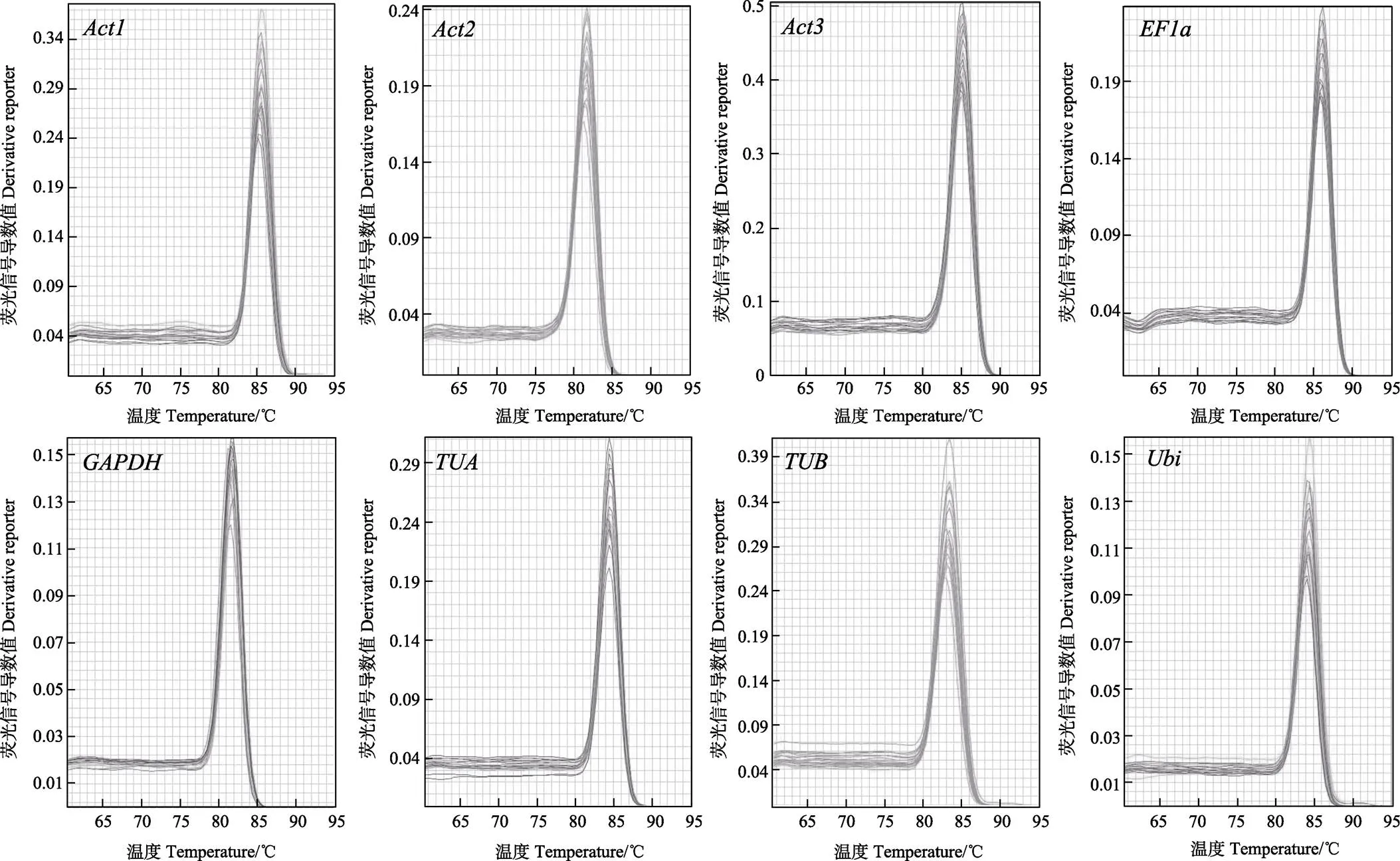
图1 候选内参基因溶解曲线
表3 8种引物的相关系数与扩增效率

Tab.3 Amplification efficiency and correlation of eight candidate genes
候选内参基因的Ct值越大,基因的表达量越小;反之,候选内参基因的Ct值越小,基因的表达量越大,且同一个候选内参基因Ct值的波动越大,说明该基因的表达稳定性越小。对8个内参基因在各样本中的表达水平(Ct值)进行分析(表4),在一心维纳斯蝴蝶兰6个花期中,各候选内参基因在不同样本中的Ct平均值为20.789~ 27.735。其中的平均Ct值最低,其表达量最高;的平均Ct值最高,其表达水平最低;、、、、、、、的平均表达水平依次升高。的Ct值变化幅度最大,Ct差值为3.594;波动范围最小,Ct差值为1.981。

表4 8个候选基因在6个样本中的Ct值
2.2 候选基因表达稳定性分析
2.2.1 geNorm软件分析 通过geNorm软件计算候选内参基因的表达稳定值(表5),值与稳定性呈负相关,<1.5时被认为是理想内参。在本研究中,8个候选内参基因在不同时期花序中的值均小于0.7,表现出较高的稳定性。在不同时期花序中,和2的值最低(0.207),稳定性最好,其次是(0.257),而相对来说,表达最不稳定(值最高,为0.663)。但由于<1.5时就能被认为是理想内参,则8个内参基因均有较为良好的稳定性,表达稳定性由大到小排序为=>>>>>>。

表5 geNorm分析候选基因的表达稳定性
2.2.2 NormFinder软件分析 使用NormFinder软件计算出的表达稳定值越小,说明该基因越稳定,反之,值越大,该基因稳定性就越差。如表6所示,在不同时期花序中,8个内参基因的稳定性依次为>>>>>>>,其中,表达稳定性最高,表达稳定性最低。

表6 NormFinder软件分析候选基因的表达稳定性
2.2.3 BestKeeper软件分析 使用BestKeeper软件计算Ct值的标准偏差(stardand deviaton, SD)及变异系数(coefficient of variation, CV)并比较各值的大小,分析判断该内参基因的表达稳定性。SD和CV越小,稳定性越好。由表7可知,除了之外的内参基因的SD值均小于1,表达稳定性依次为>>>>>>,其中稳定性最好,稳定性较差。的SD值大于1,说明稳定性较差,相对来说,不适合作为内参基因。

表7 BestKeeper软件分析候选基因的表达稳定性
2.2.4 综合评价 上述3个分析结果表明,3个软件的结果存在相似之处,但同时也存在差异。geNorm软件分析结果表明,和稳定性最高;NormFinder软件分析结果表明,稳定性最高;BestKeeper软件分析结果表明,稳定性最高。因此,对3个软件分析的结果进行几何平均值计算并排名(表8),排名越高,几何平均值越小,该基因表达越稳定。在一心维纳斯蝴蝶兰花朵不同发育时期中,的表达最稳定,是一心维纳斯蝴蝶兰花朵不同发育时期中最理想的内参基因,其次是;表达最不稳定的是,不合适用作内参基因。
表8 Excel综合分析候选基因的表达稳定性排名
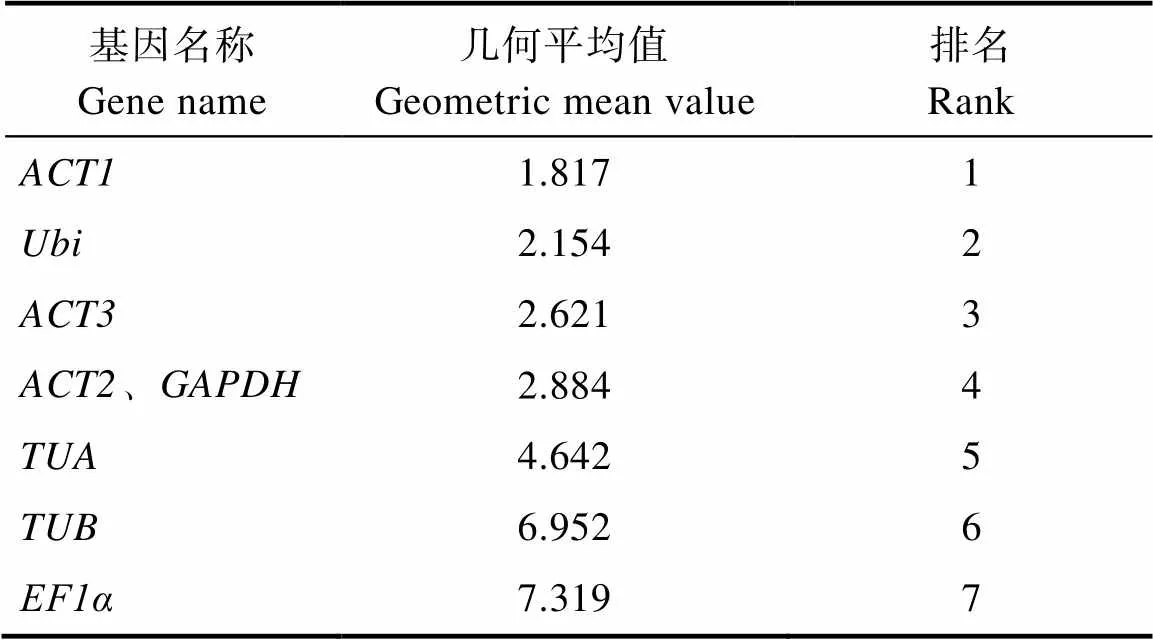
Tab.8 Expression stability of candidate genes was ranked by Excel comprehensive analysis
3 讨论
RT-qPCR技术是被广泛应用的基因表达分析的主要技术手段,但其结果的准确性需依靠试验所选出的对特定物种表达稳定性较高的内参基因,未经试验证实而使用其他试验中发表的内参基因可能会导致结果出现偏差[13]。因此,在进行目的基因表达水平研究时,首先需要筛选在特定条件下合适的内参基因。目前,多种观赏花卉的内参筛选试验均有报道,如石蒜()[14]、萱草()[10]、茉莉花()[15]等,大辣椒蝴蝶兰(Hybrid, Big Chili)在低温胁迫条件下的内参基因研究已有报道[16],但还未见蝴蝶兰花香释放过程中相关内参基因的筛选研究。
本研究通过3种软件分析了8个候选内参基因在一心维纳斯蝴蝶兰花朵不同发育时期中的稳定性情况。3种分析软件得出的结果有所不同,但表达比较稳定和最不稳定的候选内参基因基本相同。geNorm软件和NormFinder软件得出的结果较为相似,BestKeeper软件得出的结果相差较大,可能是BestKeeper软件分析时直接使用Ct值这一原始数据,导致与geNorm和NormFinder软件比较差异性相对大。该分析结果在其他内参基因筛选的报道中也常有出现,如曹映辉等[17]在建兰()花香物质合成相关基因内参基因筛选中,geNorm和NormFinder软件得出的结果一致性较高,均表明与表达较为稳定,而BestKeeper却不同;齐香玉等[15]在茉莉()不同组织(含花朵)中内参基因的筛选试验中表明,在BestKeeper中的稳定性最好,但是其在geNorm和NormFinder中的稳定性却较差,反之和在BestKeeper中的稳定性不好,但其在geNorm和NormFinder中的稳定性较好。因此,需要对3个软件的最后结果进行几何平均数计算并分析排名,降低误差,以提高结果的准确性,最后根据综合分析得到最理想的内参基因。
基因参与真核生物细胞的许多重要生命活动,如细胞形状的维持、胞质环流、细胞运动、细胞分裂、细胞分化、细胞内的物质运输、极性键成以及信号转导等[18]。在理想状态下,无论是细胞类型或发育阶段不同,还是处理方法不同,一个理想的内参基因都能稳定表达。由于基因在个体各个生长阶段的大多数组织中持续表达,变化小,序列高度保守,基因常作为内参基因使用[19]。一心维纳斯蝴蝶兰花朵不同发育时期的理想候选内参基因是。在其他物种的内参基因筛选报道中,也有被选为理想的内参基因的情况。李晗等[20]选择羽衣甘蓝(var.)不同发育时期的柱头作为材料通过qRT-PCR对7个候选内参基因进行筛选,筛选出基因为最合适的内参基因。在马铃薯()中也能稳定表达[21]。在甘草干旱条件下[22]、板蓝根ABA处理条件下及冬油菜低温胁迫条件下的表达均最为稳定[23-24]。
基因在一心维纳斯蝴蝶兰花朵不同时期中表达的稳定性最差,而仇志浪等[25]在甜樱桃()花芽不同时期的内参基因筛选中得出的最佳内参基因是;NICOT等[26]在马铃薯植株不同胁迫条件下的内参基因筛选中得出的最稳定基因也是。说明并不存在理想的内参基因适用于所有植物,在不同植物不同条件下,需要根据实际情况筛选合适的内参基因。
在聚宝红玫瑰蝴蝶兰(cultivar, Jiuhbao Red Rose)内参基因的研究中,基于geNorm和NormFinder分析,在蝴蝶兰的大多数测试样本中表现出高度稳定性,表明是蝴蝶兰qRT-PCR分析的合适参考基因[27]。CHUANG等[28]在研究贝丽娜蝴蝶兰()花香物质合成相关功能基因时,选用作为内参基因。本研究则着重筛选出蝴蝶兰花朵花香释放时期所适用的内参基因,为今后蝴蝶兰花香物质合成相关基因功能及分子机制的研究提供更有针对性的参考依据。
4 结论
本研究利用qRT-PCR技术,通过3个分析软件(geNorm、NormFinder、BestKeeper)来分析评估一心维纳斯蝴蝶兰的8个候选内参基因(,,,,,,,)在不同花期中的稳定性,经过综合分析表明,内参基因稳定性排序为>>>=>>>,得出为最为理想的内参基因,次之。在一心维纳斯蝴蝶兰花香物质合成与释放的研究过程中,对相对基因表达量进行分析时,可用基因作为内参基因进行校正。
[1] 纪冬, 辛绍杰. 实时荧光定量PCR的发展和数据分析[J]. 生物技术通讯, 2009, 20(4): 598-600. JI D, XIN S J. Development and data analysis of real-time fluorescent quantitative PCR[J]. Letters in Biotechnology, 2009, 20(4): 598-600. (in Chinese)
[2] 欧阳松应, 杨冬, 欧阳红生, 马鹤雯. 实时荧光定量PCR技术及其应用[J]. 生命的化学, 2004, 24(1): 74-76. OUYANG S Y, YANG D, OUYANG H S, MA H W. Real-time quantitative PCR technology and application[J]. Chemistry of Life, 2004, 24(1): 74-76. (in Chinese)
[3] DERVEAUX S, VANDESOMPELE J, HELLEMANS J. How to do successful gene expression analysis using real-time PCR[J]. Methods, 2010, 50: 227-230.
[4] XU Y Y, LI H, LI X G, LIN J, WANG Z H, YANG Q S, CHANG Y H. Systematic selection and validation of appropriate reference genes for gene expression studies by quantitative real-time PCR in pear[J]. Acta Physiologiae Plantarum, 2015, 37(2): 40.
[5] 袁伟, 万红建, 杨悦俭. 植物实时荧光定量PCR内参基因的特点及选择[J]. 植物学报, 2012, 47(4): 427-436. YUAN W, WAN H J, YANG Y J. Characterization and selection of reference genes for real-time quantitative RT-PCR of plants[J]. Chinese Bulletin of Botany, 2012, 47(4): 427-436. (in Chinese)
[6] 张艳君, 朱志峰, 陆融, 徐琼, 石琳熙, 简序, 刘俊燕, 姚智. 基因表达转录分析中内参基因的选择[J]. 生物化学与生物物理进展, 2007(5): 546-550. ZHANG Y J, ZHU Z F, LU R, XU Q, SHI L X, JIAN X, LIU J Y, YAO Z. Selection of control genes in transcription analysis of gene expression[J]. Progress in Biochemistry and Biophysics, 2007(5): 546-550. (in Chinese)
[7] 张元国. 蝴蝶兰组织培养快繁技术的优化[D]. 北京: 中国农业大学, 2005. ZHANG Y G. Improvement ofrapid multiplication in butterfly orchids[D]. Beijing: China Agricultural University, 2005. (in Chinese)
[8] 马璐琳, 崔光芬, 王祥宁, 贾文杰, 段青, 杜文文, 王继华, 陈发棣. 西南鸢尾花色变异实时定量PCR内参基因的筛选与验证[J]. 核农学报, 2019, 33(9): 1707-1716. MA L L, CUI G F, WANG X N, JIA W J, DUAN Q, DU W W, WANG J H, CHEN F D. Selection and validation of reference genes for quantitative real-time PCR analysis induring flower color variation[J]. Journal of Nuclear Agricultural Science, 2019, 33(9): 1707-1716. (in Chinese)
[9] CAI J, LIU X, KEVIN V, SEBASTIAN P, TSAI W C, LIU K W, CHEN L J, HE Y, XU Q, CHAO B, ZHENG Z J, SUN F M, LIU W Q, HSIAO Y Y, PAN Z J, HSU C C, YANG Y P, HSU Y C, CHUANG Y C, ANNE D, DUFAYARD J F, XU X, WANG J Y, WANG J, XIAO X J, ZHAO X M, DU R, ZHANG G Q, WANG M N, SU Y Y, XIE G C, LIU G H, LI L Q, HUANG L Q, LUO Y B, CHEN H H, PEER Y V, LIU Z J. The genome sequence of the orchid[J]. Nature Genetics, 2015, 47: 65-72.
[10] 梁锦, 刘海婷, 钟荣, 李昊, 尹冬梅, 刘翔, 秦巧平, 张志国, 段可, 倪迪安. 萱草不同器官实时荧光定量PCR内参基因的筛选[J]. 植物生理学报, 2020, 56(9): 1891-1898. LIANG J, LIU H T, ZHONG R, LI H, YIN D M, LIU X, QIN Q P, ZHANG Z G, DUAN K, NI D A. Screening of reference genes for quantative real-time PCR in different organs of[J]. Plant Physiology Journal, 2020, 56(9): 1891-1898. (in Chinese)
[11] 吴建阳, 何冰, 杜玉洁, 李伟才, 魏永赞. 利用geNorm、NormFinder和BestKeeper软件进行内参基因稳定性分析的方法[J]. 现代农业科技, 2017(5): 278-281. WU J Y, HE B, DU Y J, LI W C, WEI Y Z. Analysis method of systematically evaluating stability of reference genes using geNorm, NormFinder and BestKeeper[J]. Modern Agricultural Technology, 2017(5): 278-281. (in Chinese)
[12] 张驰宇, 张高红, 杨敏, 贲昆龙. 四步法消除SYBR GreenⅠ实时定量RT-PCR中引物二聚体的影响[J]. 中国生物化学与分子生物学报, 2004(3): 387-392. ZHANG C Y, ZHANG G H, YANG M, BEN K L. Elimination of primer-dimer effect in SYBR green Ⅰ real-time RT-PCR using4-step program[J]. Chinese Journal of Biochemistry and Molecular Biology, 2004(3): 387-392. (in Chinese)
[13] HUGGETT J, DHEDA K, BUSTIN S, ZUMLA Z. Real-time RT-PCR normalisation; strategies and considerations[J]. Genes and Immunity, 2005, 6(4): 279-284.
[14] 蒋婷婷, 高燕会, 童再康. 石蒜属植物实时荧光定量PCR内参基因的选择[J]. 园艺学报, 2015, 42(6): 1129-1138. JIANG T T, GAO Y H, TONG Z K. Selection of reference genes for quantitative real-time PCR in[J]. Acta Horticulturae Sinica, 2015, 42(6): 1129-1138. (in Chinese)
[15] 齐香玉, 陈双双, 冯景, 王华娣, 邓衍明. 茉莉花实时荧光定量PCR内参基因的筛选与验证[J]. 华北农学报, 2020, 35(6): 22-30. QI X Y, CHEN S S, FENG J, WANG H D, DENG Y M. Selection and validation of candidate reference genes for quantitative real-time PCR inAiton[J]. Atca Agriculturae Boreali-Sinica, 2020, 35(6): 22-30. (in Chinese)
[16] 梁芳, 许申平, 张燕, 王默霏, 崔波. 蝴蝶兰基因作为低温胁迫内参基因的研究[J]. 热带作物学报, 2022, 43(7): 1338-1346. LIANG F, XU S P, ZHANG Y, WANG M F, CUI B.as reference gene inunder low-temperature stress[J]. Chinese Journal of Tropical Crops, 2022, 43(7): 1338-1346. (in Chinese)
[17] 曹映辉, 郑燕, 张燕萍, 胡美娟, 童妍, 朱尾银, 徐建球, 赵凯, 彭东辉, 周育真. 建兰花香物质合成相关基因qRT-PCR内参基因筛选[J]. 分子植物育种, 2023, 21(10): 3282-3289. CAO Y H, ZHENG Y, ZHANG Y P, HU M J, TONG Y, ZHU W Y, XU J Q, ZHAO K, PENG D H, ZHOU Y Z. Selection of suitable qRT-PCR reference genes for floral scent biosynthesis in[J]. Molecular Plant Breeding, 2023, 21(10): 3282-3289. (in Chinese)
[18] STAIGER C J. Signaling to the actin cytoskeleton in plants[J]. Plant Molecular Biology, 2000, 51: 257-288.
[19] 胡瑞波, 范成明, 傅永福. 植物实时荧光定量PCR内参基因的选择[J]. 中国农业科技导报, 2009, 11(6): 30-36. HU R B, FAN C M, FU Y F. Reference gene selection in plant real-time quantitative reverse transcription PCR[J]. Journal of Agricultural Science and Technology, 2009, 11(6): 30-36. (in Chinese)
[20] 李晗, 李治龙, 李晓屿, 李玉花, 蓝兴国. 羽衣甘蓝不同组织及柱头发育实时荧光定量PCR内参基因的筛选[J]. 植物研究, 2016, 36(4): 565-572. LI H, LI Z L, LI X Y, LI Y H, LAN X G. Selection of reference genes for real-time fluorescence quantitative PCR in different tissues and stigma development from ornamental kale[J]. Bulletin of Botanical Research, 2016, 36(4): 565-572. (in Chinese)
[21] 侯志强, 王庆国. 鲜切马铃薯实时荧光定量PCR分析中内参基因的选择[J]. 安徽农业科学, 2013, 41(12): 5207-5209. HOU Z Q, WANG Q G. Reference genes selection for real-time fluorescence quantitative PCR in fresh-cut potato[J]. Journal of Anhui Agricultural Science, 2013, 41(12): 5207-5209. (in Chinese)
[22] MAROUFI A. Selection of reference genes for real-time quantitative PCR analysis of gene expression inunder drought stress[J]. Biologia Plantarum, 2016, 60(4): 645-654.
[23] QU R, MIAO Y, CUI Y, CAO Y, ZHOU Y, TANG X, YANG J, WANG F. Selection of reference genes for the quantitative real-time PCR normalization of gene expression infortunel[J]. BMC Molecular Biology, 2019, 20(1): 9.
[24] MA L, WU J, QI W, COULTER J A, FANG Y, LI X, LIU L, JIN J, NIU Z, YUE J, SUN W. Screening and verification ofreference genes for analysis of gene expression in winter rapeseed (L.) under abiotic stress[J]. PLoS One, 2020, 15(9): e0236577.
[25] 仇志浪, 何美乾, 文壮, 杨鵾, 洪怡, 文晓鹏. 甜樱桃花芽不同发育时期内参基因的筛选与验证[J]. 种子, 2020, 39(2): 37-43. QIU Z L, HE M Q, WEN Z, YANG K, HONG Y, WEN X P. Selection and validation of reference genes in sweet cherry flower bud at different development stages[J]. Seed, 2020, 39(2): 37-43. (in Chinese)
[26] NICOT N, HAUSMAN J F, HOFFMANN L, EVERS D. Housekeeping gene selection for real-time RT-PCR normalization in potato during biotic and abiotic stress[J]. Journal of Experimental Botany, 2005, 56(421): 2907-2914.
[27] YUAN X Y, JIANG S H, WANG M F, MA J, ZHANG X Y. Evaluation of internal control for gene expression inby quantitative real-time PCR[J]. Applied Biochemistry and Biotechnology, 2014, 173(6): 1431-1445.
[28] CHUANG Y C, HUNG Y C, TSAI W C, CHEN W H, CHEN H H. PbbHLH4 regulates floral monoterpene biosynthesis inorchids[J]. Journal of Experimental Botany, 2018, 69(18): 4363-4377.
Selection of Suitable RT-qPCR Reference Genes for Floral Scent Biosynthesis inI-Hsin Venus
ZHANG Yangting1, ZHANG Yanping1, HUANG Jingyan1, WANG Wenjun1, TONG Yan1, ZHAO Kai2, ZHOU Yuzhen1*
1. College of Landscape Architecture and Art, Fujian Agricultural and Forestry University / Key Laboratory of National Forestry and Grassland Administration for Orchid Protection and Utilization, Fuzhou, Fujian 350002, China; 2. College of Life Sciences, Fujian Normal University, Fuzhou, Fujian 350117, China
I-Hsin Venus is an excellent variety of fragrant flowers, with elegant flower types, long flowering period and rich floral fragrance, which has high garden ornamental value.,,,,,,and, were selected as the candidate internal reference genes based on the transcriptome data of different flower development stages in. I-Hsin Venus to select the appropriate reference genes for RT-qPCR analysis of the correlated genes in the biosynthesis pathway of the floral scent inI-Hsin Venus. The expression of the candidate internal reference genes in inflorescences at different stages was detected by RT-qPCR. The candidate internal parameter genes were analyzed by combining three internal parameter gene stability analysis software: geNorm, NormFinder and BestKeeper. The results of comprehensive analysis showed thatwas the most stable and could be used as the best internal reference gene for the expression analysis of. I-Hsin Venus.
I-Hsin Venus; reference genes; RT-qPCR; expression stability
S682.31
A
10.3969/j.issn.1000-2561.2023.11.006
2022-06-05;
2022-09-15
福建省自然科学基金项目(No. 2023J01283);福建农林大学优秀硕士论文基金项目(No. 1122YS01009)。
章杨婷(1998—),女,硕士研究生,研究方向:园林植物与应用。*通信作者(Corresponding author):周育真(ZHOU Yuzhen),E-mail:zhouyuzhen@163.com。
