中药辛夷及其近缘种的DNA条形码分子鉴定
2023-12-14张心怡邱雨轩胡杨田荣赵勉严辉王吓长胡立宏
张心怡,邱雨轩,胡杨,田荣,赵勉,严辉,王吓长,胡立宏
(1.南京中医药大学江苏省中药功效物质重点实验室,江苏 南京 210023;2.南京中医药大学药学院,江苏 南京 210023)
中药辛夷为临床常用药材,《中国药典》2020年版规定辛夷为木兰科植物玉兰Magnoliadenudata、望春玉兰Magnoliabiondii或武当玉兰Magnoliasprengeri的干燥花蕾[1]。其味辛性温,归属于肺、胃经,具有发散风寒,通鼻窍的功效,主产于河南、四川、陕西、湖北等地[2],是兼药用及观赏价值的植物。化学研究表明,辛夷中有效成分主要包括金合欢醇、松油醇、樟脑等挥发油物质[3],具有抗炎[4]、抗氧化、抗菌等活性[5],还能够调节中枢系统,有助于提高记忆、提神醒脑[6]。但近年来辛夷药材存在混伪品较多、用药混乱、质量把控不过关等诸多问题,如采集样品中常混有紫玉兰、黄山木兰、二乔玉兰等近缘种药材,因它们的花被形状、大小以及数量较为相似,加大了鉴别难度[7]。此外,在生产实践中玉兰优良无性系常以嫁接方式进行扩大繁殖[8],不同来源的嫁接苗质量和成活率明显不同。因此,需要建立一种快速、简便、有效的方法鉴别辛夷药材,以确定其用药品种,保障临床用药安全。
DNA条形码技术是通过一段标准的、有足够变异的、易扩增且相对较短的DNA片段对物种进行识别和鉴定的技术,该方法快速准确、易于操作,是对传统中药鉴定方法的补充[9]。常见的鉴定植物的DNA条形码有ITS、ITS2、psbA-trnH、matK和rbcL等序列。目前辛夷的DNA条形码仅有ITS序列的报道[10],本课题组前期多次对ITS序列扩增和测序,但PCR扩增成功率极低,故本研究采用ITS、ITS2、psbA-trnH、matK和rbcL五种DNA条形码序列对中药辛夷及其近缘种进行分子鉴定,以期为辛夷药材的分子鉴定提供科学依据。
1 材料
1.1 植物样品
本研究于2022年8—10月分别于江苏南京、江苏宜兴、河南南阳、云南昆明等地采集玉兰植物样本,由南京中医药大学药学院严辉教授鉴定。提取采集到的21份玉兰植物样本DNA,并从Genbank网站下载玉兰和武当玉兰条形码序列,共计9个品种30份代表样本。样品信息详见表1。

表1 样品信息表
1.2 试剂与仪器
DNA提取试剂盒(X0422,北京天根生化科技有限公司)、β-巯基乙醇(M8210,北京索莱宝科技有限公司)、DL2 000 DNA marker试剂(SD0531,TaKaRa公司)、氯仿(20220311,国药集团化学试剂有限公司)、琼脂糖Agarose(IA0629,上海生工生物试剂公司)、Gelred核酸染料(A616694-0500,上海生工生物试剂公司)、50×TAE缓冲液(B548101-0500,上海生工生物试剂公司)。
数显恒温水浴锅(KQ-500DF,昆山市超声仪器有限公司)、核酸电泳仪(HE-120,上海天能科技有限公司)、凝胶成像仪(5200multi,上海天能科技有限公司)、高速离心机(5430R,Eppendorf公司)、NanoDrop One[ND-ONE-W(A30221),Thermo公司]、高压灭菌锅[GR60DA,致微(厦门)仪器有限公司]、PCR扩增仪(Biometra TOne 96G,Analytik jena公司)、微波炉(M1-211A,广东美的厨房电器制造有限公司)。
2 方法
2.1 DNA提取、PCR扩增及测序
取液氮冷冻保存的玉兰品种叶片30 mg、根皮50 mg,研碎后用植物基因组DNA提取试剂盒提取总DNA,NanoDrop One检测DNA浓度和纯度。提取的DNA的A260 nm/A280 nm值确保控制在1.8~1.9之间(A表示吸光度)。选用通用引物及标准流程扩增各样品ITS、ITS2、psbA-trnH、matK及rbcL条形码片段。PCR反应体系为50 μL,包括2×Taq PCR Mix 25 μL,正向、反向引物各2 μL,200~400 ng的模板DNA,加ddH2O补足至50 μL,扩增条件详见表2。扩增结束后以1%凝胶电泳检测,凝胶成像分析系统分析。PCR产物经凝胶电泳检测后,送往生工生物工程(上海)股份有限公司进行双向测序。
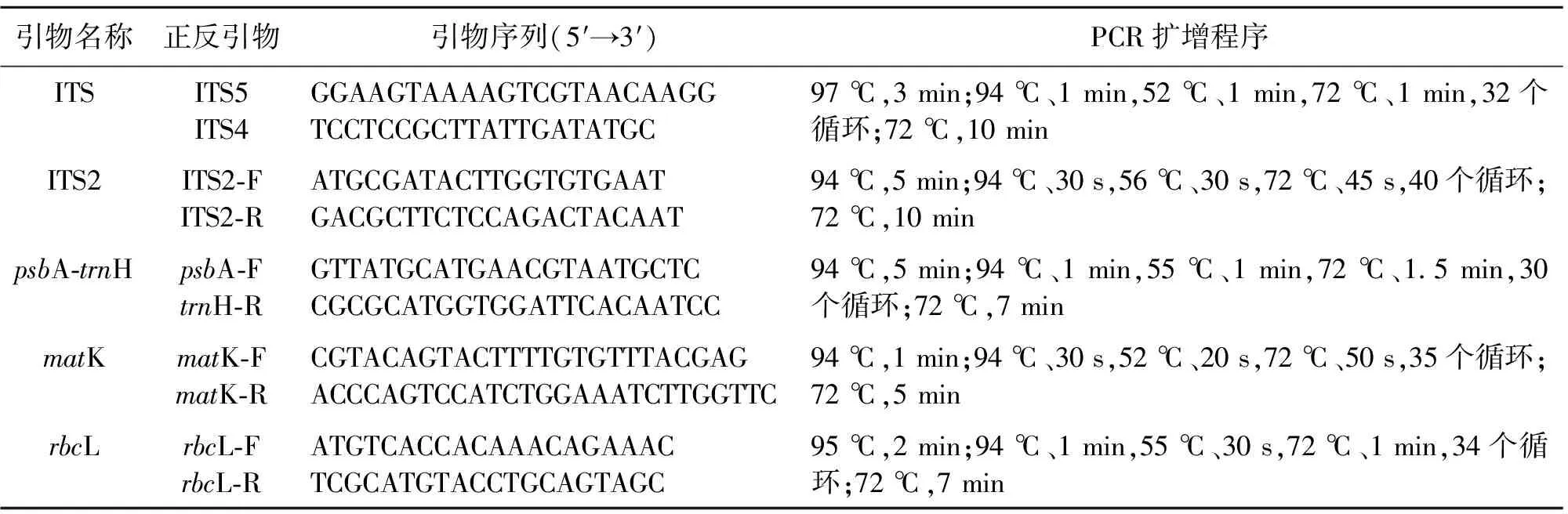
表2 DNA条形码PCR反应正反引物序列及扩增程序
2.2 数据分析
测序结果采用Codon Code Aligner V10.02(CodonCode公司,美国)进行正反拼接,参考序列峰图去掉低质量部分,得到完整的基因序列,并于NCBI数据库中进行Blast比对,保证样本鉴定成功率。从GenBank下载相似度高的相应种属序列,将所有候选序列导入MEGA7.0进行比对分析,计算序列比对长度、变异位点和简约信息位点等,并基于Kimura-2-Parameter(K2P)模型计算种内及种间遗传距离并建立邻接(NJ)聚类进化树,分析碱基之间的差异。聚类分析检测采用Bootstrap方法,重复1 000次(表3)。
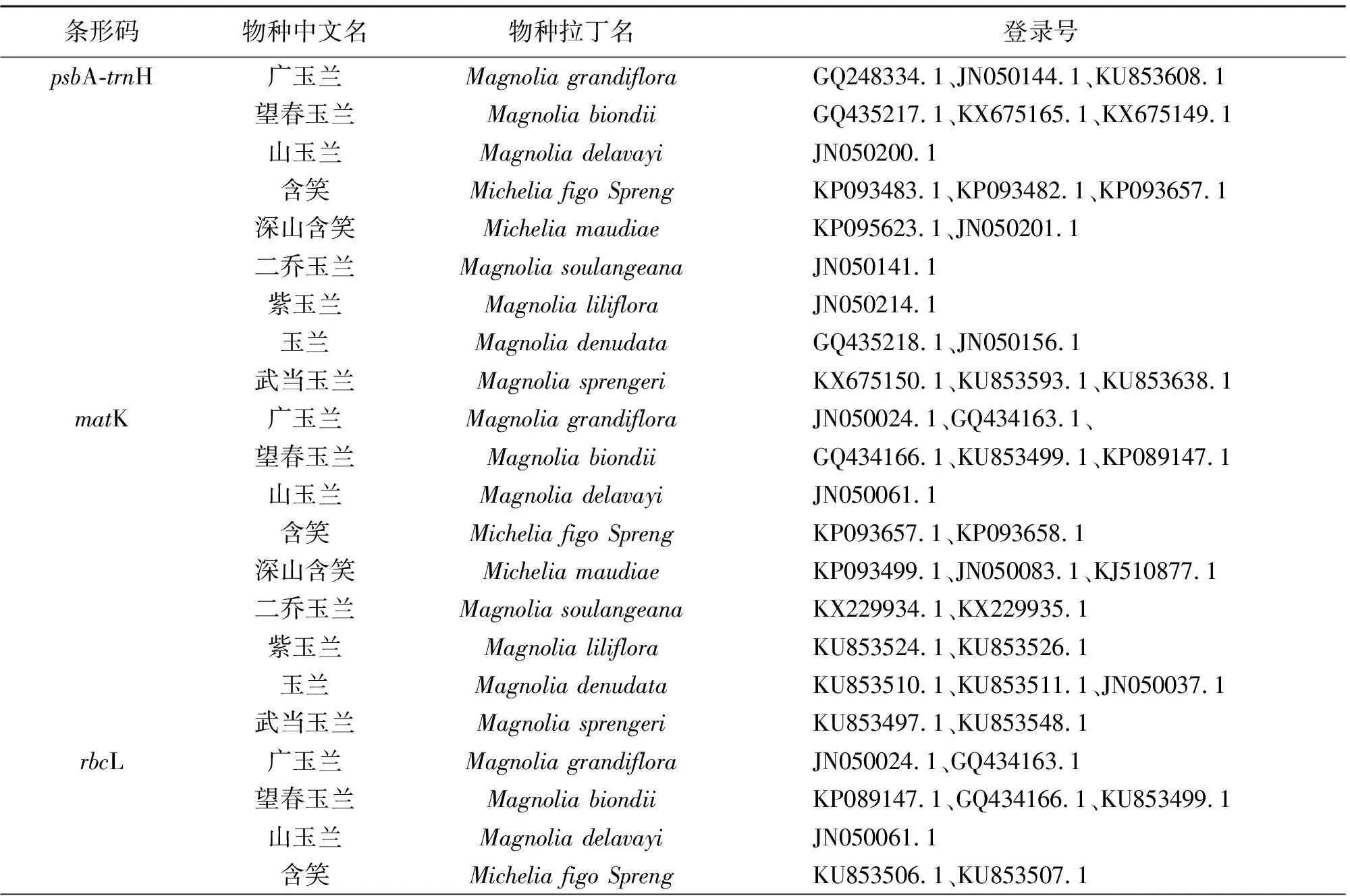
表3 从GenBank获取的辛夷及其近缘种psbA-trnH、matK和rbcL条形码序列
3 结果
3.1 PCR扩增、测序结果分析
为了进一步比较各DNA条形码序列的PCR扩增效果及变异程度,对5种条形码序列进行PCR扩增与测序,得到相应的扩增、测序成功率和序列信息。如表4所示,psbA-trnH,matK扩增较为成功,扩增成功率均大于98%,扩增结果详见图1~2。扩增产物经1.0%琼脂糖凝胶电泳检测,胶图呈现单一明亮条带且无拖尾现象,该结果提示该序列扩增效果良好。rbcL扩增成功率较低,为38.1%,且扩增条带不清晰;ITS、ITS2序列PCR扩增成功率极低,为0和28.5%,样品扩增后呈现条带极少。
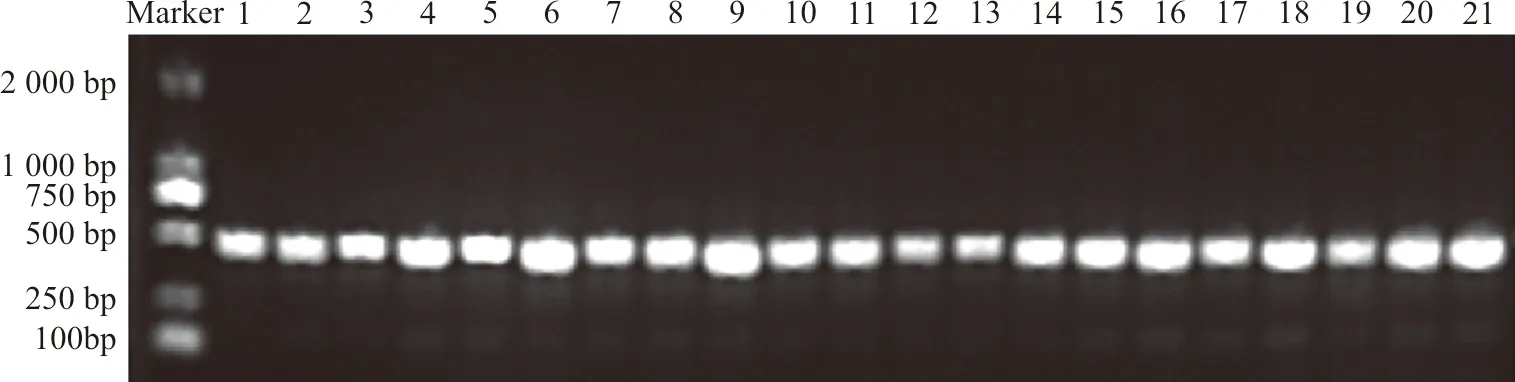
注:Marker是比对的目标条带,比对长度为500 bp;1~3.广玉兰;4~6.望春玉兰;7~9.山玉兰;10~12.含笑;13~15.深山含笑;16~18.紫玉兰;19~21.二乔玉兰

注:Marker是比对的目标条带,比对长度为750~1 000 bp;1~3.广玉兰;4~6.望春玉兰;7~9.山玉兰;10~12.含笑;13~15.深山含笑;16~18.紫玉兰;19~21.二乔玉兰

表4 中药辛夷及其近缘种的序列特征
3.2 序列特征分析
研究共获得psbA-trnH序列40条,matK序列40条,rbcL26条,其中包括Genbank下载的psbA-trnH序列19条,matK序列19条,rbcL序列18条。比对后的序列长度均小于1 000 bp,长度matK>rbcL>psbA-trnH,符合DNA条形码长度的要求。其中matK序列长度最长,为728 bp,变异位点和简约信息位点最多,说明matK变异程度最大,rbcL序列变异位点数少于matK。psbA-trnH序列变异位点数最少,可见psbA-trnH序列变异程度最低,此序列变异相对稳定,变异位点数占总位点数的12.3%。
3.3 DNA条形码Barcoding gap分析
利用MEGA7.0软件计算psbA-trnH、matK、rbcL序列的种内种间距离,并基于K2P遗传距离模型计算样品间的种间、种内遗传距离。理想的DNA条形码应符合种内遗传距离明显小于种间遗传距离,两者之间存在明显的界限,即“Barcoding gap”[11]。本文选择的psbA-trnH、matK、rbcL片段皆是进化速率较快的叶绿体间隔区[12],结果显示,psbA-trnH序列的种间平均距离为0.152,种内平均遗传距离为0.012;matK序列的种间平均距离为0.449,种内平均距离为0.021;rbcL的种间平均距离为0.538,rbcL种内平均距离为0.025(表4)。其中,psbA-trnH序列的种内种间距离重叠较少,Barcoding gap较matK更明显;rbcL序列种内种间遗传距离重叠多,Barcoding gap不明显,同时其序列扩增及测序成功率低,鉴定效果并不理想(图3)。因此,选择psbA-trnH和matK作为鉴别辛夷及其近缘种的候选序列,开展进一步实验。
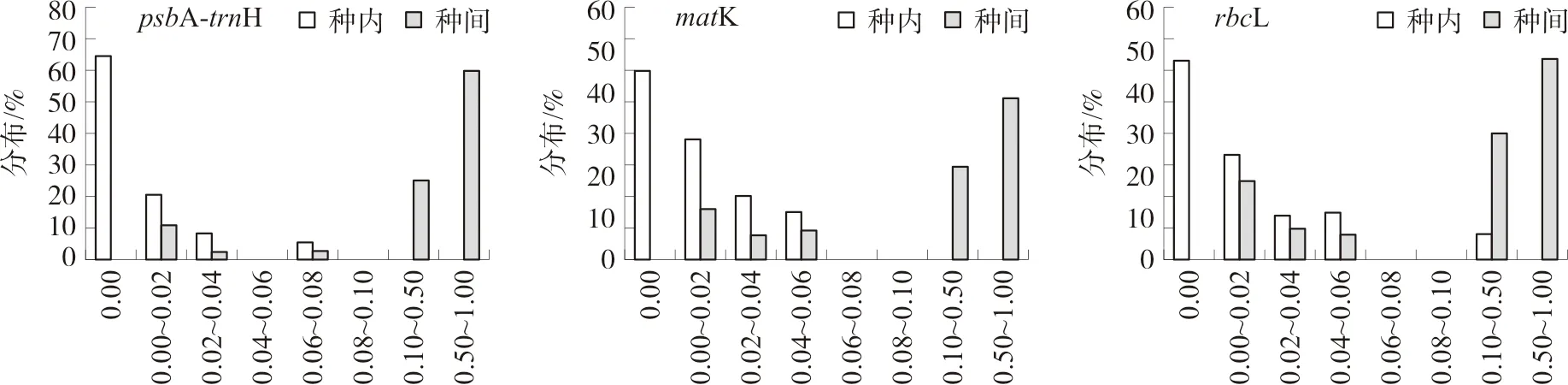
图3 psbA-trnH、matK、rbcL的遗传距离分布
3.4 辛夷及其近缘种序列psbA-trnH及matK二级结构
将psbA-trnH及matK碱基序列导入RNA fold预测辛夷及其近缘种的二级结构。如图4~5所示,各品种的psbA-trnH序列二级结构为1个主环、左右两侧各分布3个螺旋区,每个螺旋区上茎环的数量(即主环左、右侧螺旋区茎环数分别计数)、大小和开合角度均有差异。茎环数量如表5所示,茎环数紫玉兰最多,左侧为13,右侧为12;山玉兰最少,左侧为9,右侧为11。茎环从大到小排列为广玉兰>武当玉兰>含笑>紫玉兰>二乔玉兰>玉兰>望春玉兰>山玉兰>深山含笑。螺旋区开合角度深山含笑最大,含笑最小。matK序列二级结构均由2个主环、左侧主环4个螺旋区,右侧主环5个螺旋区组成。茎环数最多的是二乔玉兰,左侧为17,右侧为25;广玉兰最少,左侧为15,右侧为19。茎环从大到小排列为玉兰>含笑>深山含笑>二乔玉兰>望春玉兰>武当玉兰>紫玉兰>广玉兰>山玉兰。紫玉兰螺旋区开合角度略大,其余类似。结合psbA-trnH及matK两种序列的二级结构可以发现,不同物种螺旋区分支的茎环数量、大小、螺旋区的开合角度均有差别,故psbA-trnH和matK两种序列二级结构的建立可用于区分辛夷药材及其近缘种。
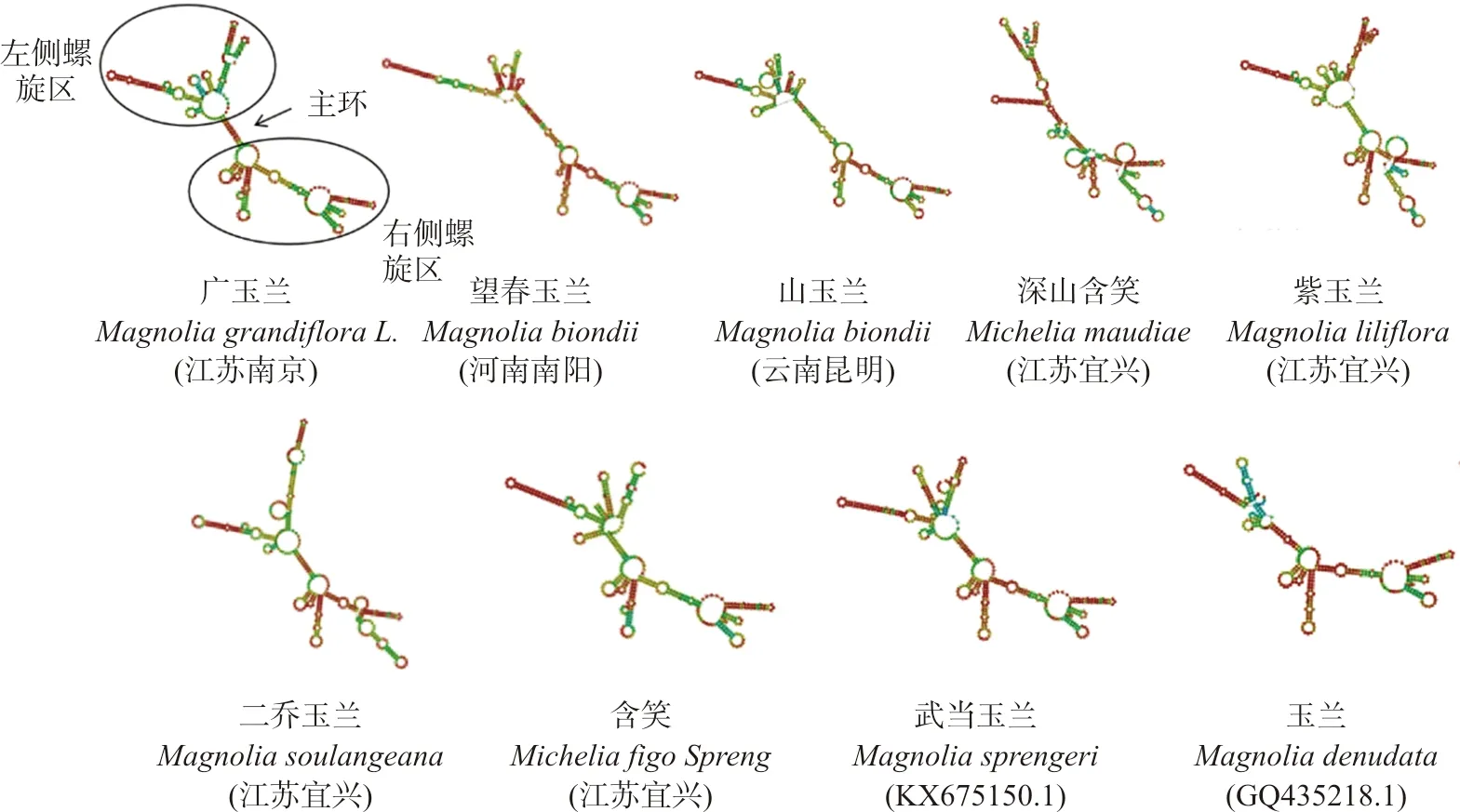
图4 辛夷及其近缘种psbA-trnH序列二级结构

图5 辛夷及其近缘种matK序列二级结构
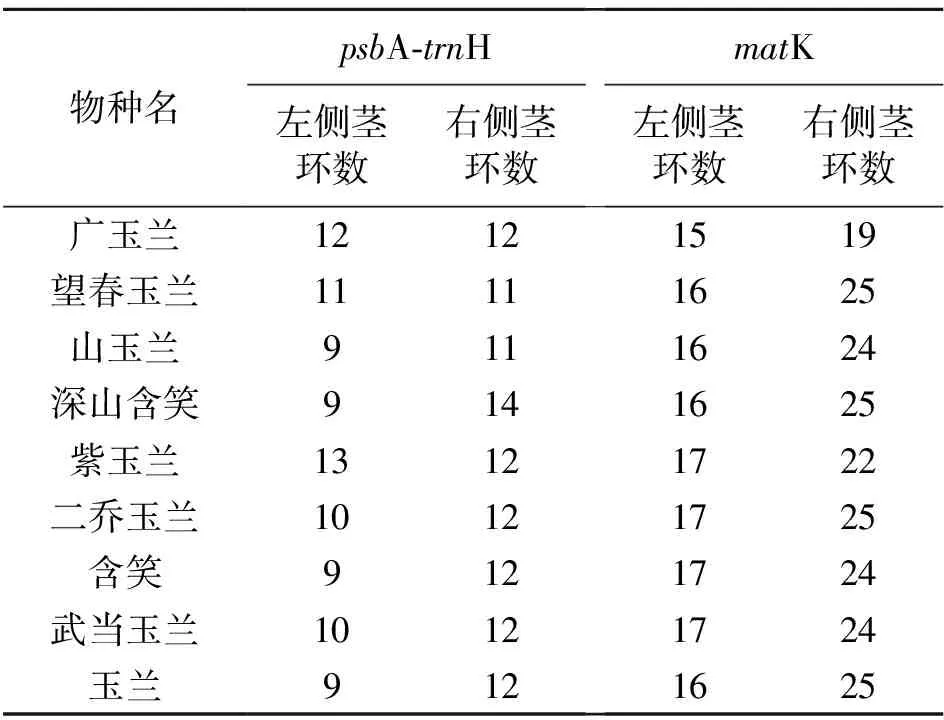
表5 psbA-trnH和matK序列螺旋区茎环数量
3.5 系统发育树分析
为了能够更直观清晰地反映出鉴定结果,我们利用MEGA7软件对psbA-trnH和matK两种条形码序列构建系统发育树。如图6所示,利用psbA-trnH和matK条形码独立建树,发现能将辛夷与其近缘种聚为不同分支。结果表明,psbA-trnH建立的系统发育树能够在9个物种中准确区分6个品种,辛夷药材的3个品种各自均能聚为一支,呈明显的单系性,且近缘种各自聚为不同分支,与辛夷药材明显区分开,但未能鉴别含笑、山玉兰、二乔玉兰这3个近缘种。matK建立的系统发育树能准确区分5个品种,均为辛夷近缘种,各自聚为一支,与其他品种区分开,但玉兰和武当玉兰混为一支,未能区分开。可见,psbA-trnH和matK两种单独的DNA片段并不能将9种植物全部鉴定出来,只能鉴别出部分植物。于是,我们将psbA-trnH和matK两种序列联合建树分析。如图6所示,基于psbA-trnH+matK基因组序列建立的系统发育树能够准确区分鉴定9种植物,辛夷药材与近缘种各自聚为不同分支,有明显区分。故利用psbA-trnH+matK在鉴定准确性、覆盖物种数更为优异,鉴别辛夷及其近缘种效果更好。
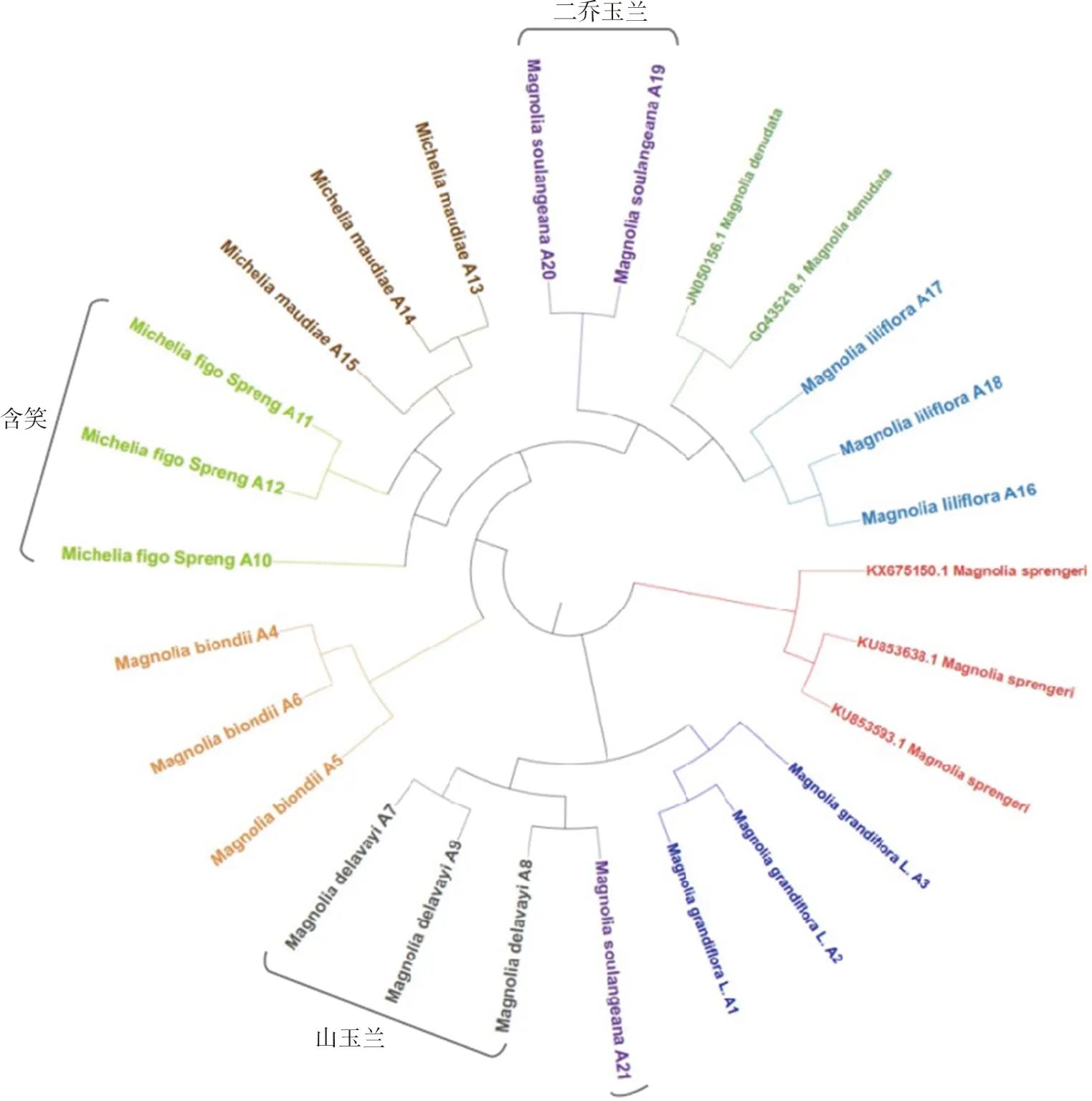
图6 基于psbA-trnH、matK、psbA-trnH+matK序列构建的系统发育树
3.6 基于psbA-trnH+matK组合条形码鉴定玉兰嫁接品种
根据上述实验结果,课题组选用psbA-trnH+matK组合条形码联合分析,对玉兰嫁接品种的砧木根皮部位进行分子鉴定,验证DNA条形码技术对于嫁接品种的鉴定可行性。如表6所示,通过psbA-trnH+matK组合条形码鉴定,显示采集批次中广玉兰、紫玉兰及二乔玉兰的砧木根段为望春玉兰嫁接。此方法提高了玉兰植物品种的鉴定效率,也为嫁接品种的鉴定提供了新思路。

表6 psbA-trnH+matK序列联合鉴定玉兰嫁接品种
4 讨论
中药辛夷以望春玉兰、玉兰、武当玉兰的花蕾入药,然而国内玉兰品种繁多,花蕾形状相似,难以从外观上准确鉴定,进而影响辛夷药材的鉴别。因此,本实验选用5种条形码(ITS、ITS2、psbA-trnH、matK和rbcL)进行鉴定辛夷及其近缘种最佳DNA条形码的筛选研究。结果显示,psbA-trnH和matK条形码序列PCR扩增、测序成功率均大于98%,而rbcL扩增及测序成功率较低,ITS、ITS2并不理想,故不宜用来作为辛夷的种间鉴定。考虑可能由于植物细胞内不同核糖体的进化水平存在差异,扩增产物可能被细菌、真菌等微生物的核糖体污染等[13],进而对实验结果产生影响。而叶绿体在结构、序列上相对保守[14],进化水平较为一致且扩增产物不受微生物影响。
随后根据K2P遗传距离、种内种间变异分析,辛夷及近缘种的种内遗传较为稳定,种间变异程度较大,且psbA-trnH、matK和rbcL三条序列种间种内距离均有一定的重叠,psbA-trnH种内种间距离重叠较少,Barcoding gap较matK明显。随着生物信息学的不断发展,遗传物质二级结构的相关研究不断增多,因其比一级结构在生物进化过程中序列更具有保守性,可作为系统发育分析的参考,逐渐成为研究热点[15-16]。尤其适用对原植物及其近似种之间二级结构形态上的差异进行种间鉴别,为科学分类提供参考[17]。结合二级结构的形态差异,选择psbA-trnH和matK作为鉴别辛夷及其近缘种的候选序列,实现了物种间的有效区分。
陈士林及其团队在药用植物及中药材研究中提出了以ITS为主,rbcL为辅的组合条形码鉴定物种的DNA条形码分子鉴定体系[18]。张明英等[19]提出通过多序列联合分析能够在一定程度上提高物种鉴定成功率,鉴别效果显著。本实验中,NJ聚类树显示,基于psbA-trnH序列建立的系统进化树能够准确区分6个品种,包括辛夷药材的3个品种,而matK变异位点最多,变异程度大,进化树能够准确区分5个品种,但不能区分玉兰和武当玉兰。在进一步探究组合条形码的鉴定潜能时,发现psbA-trnH+matK基因组序列联合分析构建的系统发育树能够准确鉴定9个品种,与单一条形码比较,无论物种鉴定成功率亦或是系统发育分析聚类中都有更为突出的指征。
最后,作为我国常见的绿化观赏树种,在生产实践中玉兰优良无性系常以嫁接方式进行扩大繁殖。研究表明,不同属的木兰科植物间具有广泛的嫁接亲和力[8,20],嫁接的砧木通常分为同属砧木和异属砧木2类。同属砧木如紫玉兰[21]、望春玉兰[22-23]等,植株生长适应性强、抗病虫害能力强于普通植株[24],且嫁接成活率较高。异属砧木通常指山玉兰[25]、黄兰含笑[26]等,不同的砧穗组合使得异属砧木的嫁接成活率存在较大差异,而近年来随着市场需求不断扩大,玉兰苗木开始出现供应短缺的问题。因此快速鉴别玉兰砧木品种,保证较高的嫁接成活率、育苗速度和质量,具有解决生产问题的实际意义。利用psbA-trnH+matK基因组序列首次对玉兰嫁接品种的根皮进行分子鉴定,显示采集批次中广玉兰、紫玉兰和二乔玉兰的砧木根段为望春玉兰嫁接。
综上所述,推荐使用psbA-trnH+matK组合条形码作为鉴定辛夷及其近缘种的首选序列。该组合条形码能提供更多遗传信息以准确鉴定辛夷药材,为辛夷药材的品种鉴定和分类提供分子生物学依据。
