基于桑格测序法建立高鼻羚羊鉴定方法的可行性研究
2023-11-25沙才华廖秀云周傲白雪蒋晓霞徐博文黄海超赵福振邵建宏
沙才华,廖秀云,陈 轩,周傲白雪,蒋晓霞,徐博文,黄海超,赵福振,邵建宏
(拱北海关技术中心,国家外来病检测重点实验室,国家口蹄疫猪瘟检测重点实验室,珠海,519001)
高鼻羚羊(Saiga tatarica)是与剑齿虎(Saber toothed tiger)同期的有蹄类食草动物[1],被视为具有重要生物学研究价值的活化石。高鼻羚羊的角俗称羚羊角,在中国传统医学中被视为珍稀药材。历史上,高鼻羚羊广泛分布于欧亚大草原,东北至蒙古,东南至中国新疆,横跨西北部的喀尔巴阡山脉和西南部的高加索山脉,在更为久远的更新世,高鼻羚羊也曾出没于白令区域和欧洲的不列颠群岛[2]。目前,高鼻羚羊在中国境内已灭绝,整合分类学资讯系统(ITIS)公布的资料显示[3],高鼻羚羊分布于南亚、北亚和欧洲,又被称为草原高鼻羚羊(steppe saiga),“steppe”一词特指西伯利亚一带没有树木的大草原。另有资料显示,高鼻羚羊现分布于俄罗斯、哈萨克斯坦、土库曼斯坦和乌兹别克斯坦[4]。由于分布范围小、数量少和人工驯养难度大,高鼻羚羊被世界自然保护联盟(IUCN)列为濒危物种红色名录极危(CR)物种[5],被保护野生动物迁徙物种公约(CMS)和濒危野生动植物种国际贸易公约(CITES)列入附录Ⅱ,是国家一级重点保护野生动物。
调查显示,高鼻羚羊的分布地区普遍存在盗猎行为,20世纪80年代从前苏联出口至中国、日本等地的高鼻羚羊角实际值为官方统计值的5~7倍,1994年从俄罗斯、哈萨克斯坦等地非法走私至东亚的高鼻羚羊角达44 t[6]。2005—2010年,分布有高鼻羚羊的土库曼斯坦、乌兹别克斯坦、哈萨克斯坦、俄罗斯联邦和蒙古5个国家相继签署CMS[7],全面取消高鼻羚羊及其制品的国际贸易。然而,非法走私高鼻羚羊角的行为仍屡禁不止,单次最高走私量超过1 000 根[8-9]。此外,由于供需关系长期严重不均衡,高鼻羚羊角的黑市价格已高达人民币8 万元/根[10-11]。因此,研究高鼻羚羊角的鉴定方法对保护濒危野生动物,打击违法犯罪和统一执法口径具有重要意义。
目前针对高鼻羚羊角鉴定方法的研究主要包括形态观察、切片观察等直接判断法,光谱、色谱等理化分析法,以及各类分子生物学法,由于技术原理和技术手段的区别,上述鉴定方法均有自身的适用性和局限性,基于同类技术建立的不同鉴定方法在鉴定结果上也可能存在偏差。分子生物学技术对微量、痕量样品的鉴定具有显著优势。自常规PCR 技术面世以来,基于分子生物学理念开发的各类检测、鉴定方法一直在物种鉴定领域中占据主导地位,因此,本研究基于桑格测序法建立一套鉴定高鼻羚羊及其制品的分子生物学检测方法并验证其可行性,以作为现有技术的进一步补充。
1 试验材料
1.1 高鼻羚羊样品
拱北海关技术中心动物检疫实验室存留的高鼻羚羊完整角2份,编号1和2;市场采购的商品化高鼻羚羊角粉3 份,厂商分别为北京同仁堂(亳州)饮片有限责任公司、成都岷江源药业有限公司和内蒙古普康药业有限公司,对应编号3、4和5。
1.2 对照样品
拱北海关技术中心动物检疫实验室存留的动物组织和动物制品,包括野牦牛(Bos mutus)肉、水牛(Bubalus bubalis)完整角、家牛(Bos taurus)完整角、梅花鹿(Cervus nippon)完整角、驯鹿(Rangifer tarandus)角切片、马鹿(Cervus elaphus)角切片、山羊(Capra hircus)完整角、绵羊(Ovis aries)完整角和白犀牛(Ceratotherium simum)角磨粉,各1 份,编号6~14。样品1、2和6~14均于较早前使用本实验室自行设计的分子生物学鉴定方法(经CNAS 认证)确认为所述物种。
2 试验方法
2.1 样品DNA提取
用蘸有95%乙醇的棉球均匀擦拭角制样品和肉制样品的表面数次,以清除附着在样品表面可能影响检测结果的异物,干燥后使用碎瓷片或手电钻(无菌钻头)刮/钻取适量角实质或使用无菌剪刀剪取适量肉组织制备待检样品。粉末状制品直接拆封称取适量粉末制备待检样品。上述待检样品均严格按照OMEGA 核酸提取试剂盒(OMEGA E.Z.N.A.®Tissue DNA Kit D3396-02)说明书提取DNA,并于-20 ℃保存备用。
2.2 PCR引物合成
由上海辉睿生物科技有限公司合成PCR 引物,大小约300 bp,引物序列:F-primer-300,5'-ACTTCTAGCATCTTCCATAGTTGAG-3';R-primer-300,5'-GGGAAGTGAAAGGAGTAGGAGG-3'[11]。
2.3 PCR扩增
以供试样品提取的DNA 为模板进行常规PCR扩增,反应体系:2×TaqPCR Mastermix 12.5 µL,上、下游引物各1.0 µL(10 µmol/L),DNA 模板2.0 µL,补水至总反应体积25.0 µL。反应程序:95 ℃预变性5 min;95 ℃变性30 s,64 ℃退火30 s,72 ℃延伸30 s,共33个循环;72 ℃完全延伸5 min。
2.4 电泳和测序
扩增产物经1.5%琼脂糖凝胶电泳检测,并采用桑格测序法双向测序(测序工作由本实验室完成)。
2.5 序列分析
通过美国国家生物技术信息中心(NCBI)的序列比对工具BLAST 进行物种相似性比对分析,验证物种来源。
3 结果
3.1 PCR扩增产物电泳结果
样品1~4 在约300 bp 处均呈现显著扩增条带,样品5在300 bp处呈现极微弱扩增条带,样品6~14、阴性对照和空白对照均未见扩增条带(图1)。
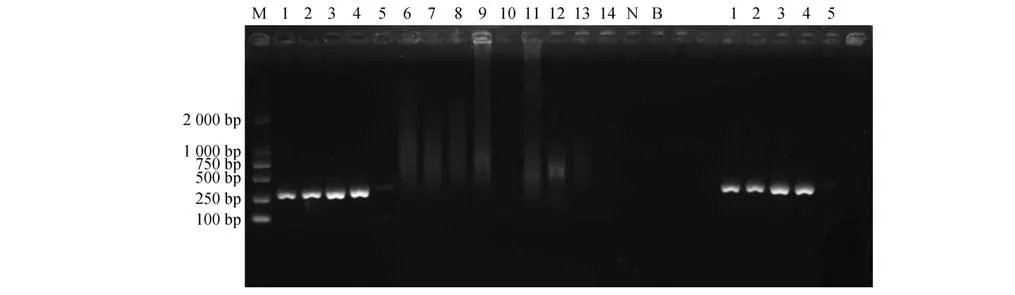
图1 PCR扩增产物电泳结果Fig.1 Electrophoresis results of PCR amplified products
3.2 NCBI比对结果
2份高鼻羚羊角样品(样品1、2)和3份商品化羚羊角粉样品(样品3、4 和5)的测序结果(表1)经BLAST 检索均显示为Saiga tatarica,且所有测序结果与Saiga tatarica参考序列(登录号:NC_020746.1)的相似性均大于99.42%(表2)。

表1 样品1~5的测序结果Tab.1 Sequencing results of sample 1 to sample 5

表2 样品1~5序列的BLAST结果Tab.2 Results of BLAST for the sequences of sample 1 to sample 5
4 讨论
在探索高鼻羚羊桑格测序鉴定方法的过程中,本研究曾根据NCBI 基因数据库公布的高鼻羚羊细胞色素c 氧化酶亚型Ⅰ(cytochrome c oxidase subunit Ⅰ,COI)基因序列,在高度保守区域设计两套特异性PCR 引物(表3),多次重复试验结果表明两套引物均能准确分辨供试样品是否含有高鼻羚羊成分,但PCR 扩增产物的凝胶电泳图呈现部分假阳性(图2),而使用上述对照样品的引物配合PCR 反应参数得到的扩增结果未出现假阳性。综合既往研究,发现较长的扩增产物片段有利于提高序列分析的准确性,但可能不利于凝胶电泳图的特异性呈现。
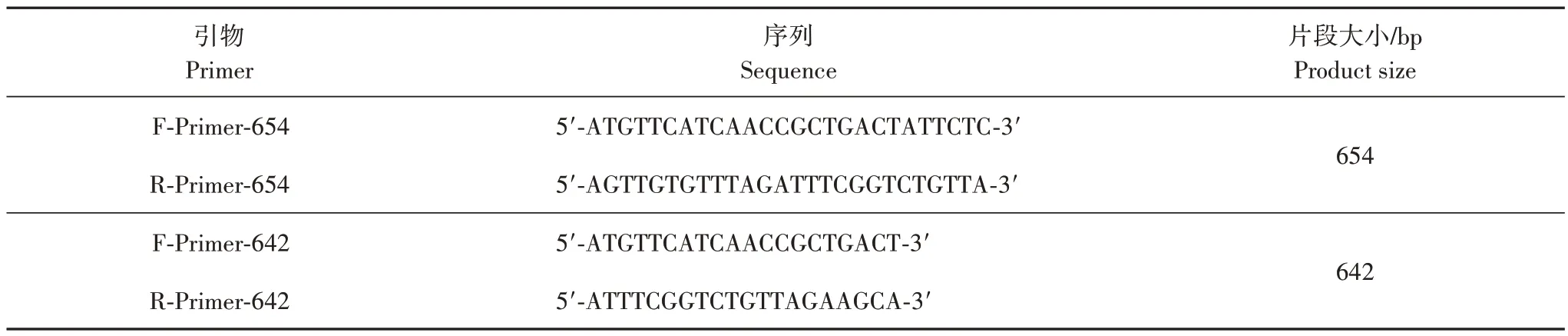
表3 高鼻羚羊细胞色素c氧化酶亚型Ⅰ基因扩增引物Tab.3 Cytochrome c oxidase subunit I gene amplification primers for Saiga antelope
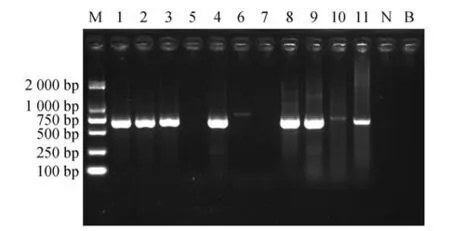
图2 部分样品的Primer-654检测结果Fig.2 Gel electropherograms of PCR amplification products of some of the samples using Primer-654
具有特征曲线的角是高鼻羚羊最具经济价值的组织,但多数动物的角组织由角质蛋白和胶原蛋白等成分组成,其线粒体DNA 含量远少于肉、皮和骨等其他组织,为确保鉴定结果的准确性,本研究先后尝试了全自动核酸提取仪和人工过柱提取2 种不同的方法,发现后者得到的DNA 质量略优于前者,DNA 质量浓度均大于20 ng/µL,该浓度能够满足PCR 扩增的基本要求,但DNA 的平均质量浓度仅为其他常见组织的6%~10%。如图1、表2 所示,本研究所有对照样品均未见扩增条带,而所有阳性样品与高鼻羚羊参考序列的相似性均大于99.42%,其中,样品5 的凝胶电泳条带极其微弱,但其扩增产物测得的序列仍能明确区分于林羚(Tragelaphus spekii)等其他近似物种。经过对蒋超等[11]公开的特异性引物进行反应条件优化并增加桑格测序,本研究建立的检测方法具有灵敏度高、特异性强的特点,能够准确扩增微量目标基因。
此外,高鼻羚羊属(Saiga)物种的分类在国际上仍未达成共识,查询ITIS、NCBI、IUCN、CMS、CITES 以及国家重点保护野生动物名录公布的最新物种信息,可归纳出两个主要分歧和一个高度统一。分歧一是将蒙古高鼻羚羊单列为一个物种(ITIS、CMS 和CITES),亦或归入高鼻羚羊的亚种(NCBI、IUCN);分歧二是若蒙古高鼻羚羊单列为一个物种,是否应将其进一步细分为Saiga borealis borealis和S.b.mongolica2 个亚种。统一之处为除国家重点保护野生动物名录外,其他机构/名录都从种或亚种的层面将高鼻羚羊与蒙古高鼻羚羊进行区分(表4)。然而,本研究测得的所有目标序列在NCBI 基因数据库的比对结果仅匹配至种,未显示具体亚种。为尝试区分,本研究通过NCBI 进一步查询S.tatarica、S.t.tatarica和S.t.mongolica的相关信息,发现NCBI 收录上述3 个物种/亚种的线粒体基因序列数分别为291、76、26 条,前者有长度大于16 000 bp的线粒体全基因序列,而后两者序列的最长长度均小于1 220 bp,且后两者均未登录线粒体的COI基因序列,也未查询到S.borealis的相关序列。因而,根据现有、可及的种和亚种序列,本研究无法进一步判断上述方法是否能够准确区分S.tatarica tatarica和S.t.mongolica,或S.tatarica和S.borealis。换言之,现阶段的材料无法证明本研究建立的方法能够在分子层面区分高鼻羚羊和蒙古高鼻羚羊。再者,本研究纳入的平行样品和对照样品数量和种类偏少,也未能基于明确、可信的证据分别获得高鼻羚羊和蒙古高鼻羚羊的样品,上述均有待后续加以拓展和验证。

表4 高鼻羚羊属物种分类Tab.4 Species classification of Saiga
