基于简化基因组测序的麋鹿遗传资源多样性及种群结构分析
2023-11-25张树苗田恒玖李夷平李俊芳郭青云王立波程志斌白加德
张树苗,田恒玖,李夷平,陈 颀,李俊芳,李 林,郭青云,王立波,程志斌,白加德
(1.北京麋鹿生态实验中心,北京,100076;2.北京松山国家级自然保护区,北京,102115;3.江苏省大丰麋鹿国家级自然保护区管理处,盐城,224136)
麋鹿(Elaphurus davidianus)是中国特有物种,国家一级重点保护野生动物,IUCN 红皮书野外灭绝物种[1-2],是世界物种重引进项目的成功范例。19世纪末,由于栖息地丧失、气候变化及过度捕猎,麋鹿在中国本土灭绝[3]。1900 年前后,中国仅存的麋鹿被欧洲各大动物园掠夺饲养。英国乌邦寺收集了分布于欧洲各地的18 只麋鹿进行繁殖,逐步建立起完整的小种群[4]。1985—1987 年,我国政府着手开展麋鹿重引入工作[5],同期,英国乌邦寺分两批将38只麋鹿重引入到中国,饲养在北京南海子麋鹿苑;1986年引入39 只到江苏大丰麋鹿国家级自然保护区。经过30 多年的人工繁殖、复壮及野外放归,中国麋鹿保护工作取得显著进展。目前人工繁殖的麋鹿种群已全面覆盖麋鹿原有栖息地,迁地保护地从当初的2 个增至现在的89 个,种群数量12 000 余只,其中有6个野生种群,数量达5 000余只[4,6]。中国麋鹿种群从本土灭绝经重引入,种群逐渐复壮,再到放归自然,成为保护野外灭绝物种的成功典范。
小种群易受遗传漂变的影响[7],特别是重引入的麋鹿种群具有显著的奠基者效应和瓶颈效应(bottleneck effect)[8-9]。奠基者效应对麋鹿重引入的扩张具有显著影响[10-11],而瓶颈效应发生在种群经历短暂的数量剧减后,种群的遗传信息完全来自少数存活下来的个体[12]。因此,瓶颈效应与奠基者效应非常类似,这种效应的严重后果就是破坏种群遗传结构,使原有种群动态特征失去平衡,加剧遗传多样性的丧失。正因如此,我国麋鹿种群存在遗传多样性缺失,近交衰退严重,抵抗疾病能力差,寿命降低及雌鹿流产率高等现象[13-14]。人类活动、气候变化、环境污染和生境破碎化等会导致野生麋鹿种群数量进一步下降,形成离散种群[15-18]。目前,我国麋鹿保护仍面临严峻挑战[19-20]。
近年来,随着高通量测序技术的快速发展,通过基因组重测序手段,在对物种鉴定、分类、遗传多样性和遗传结构的研究中发挥越来越重要的作用[21-26]。本研究基于简化基因组技术,对北京南海子麋鹿苑半散养种群的10 只麋鹿及江苏大丰麋鹿国家级自然保护区圈养种群和野生种群的8 只麋鹿,共18 只个体进行遗传多样性和遗传结构分析,在不同生境下,分析中国麋鹿的遗传多样性背景,并对中国重引入麋鹿工作作出科学而客观的评价,以期为麋鹿放归、种群基因交流等保护措施的开展提供科学支撑。
1 研究区概况
北京南海子麋鹿苑(以下简称“北京麋鹿苑”)位于华北平原北部,燕山山脉南缘。江苏大丰麋鹿国家级自然保护区(以下简称“江苏大丰”)地处黄海之滨,江苏省东部、盐城市南部,东临黄海,具体信息见表1。

表1 研究区基本信息Tab.1 The basic information of study areas
2 材料与方法
2.1 材料与基因组样本收集
共采集18 只麋鹿个体的血液样品,其中,10 只采集于北京麋鹿苑半散养种群,8只采集于江苏大丰圈养(2 区:4 只)和野生种群(3 区:4 只)。将血液样品各取5 mL 至抗凝管中,通过天根试剂盒(QIAamp Fast DNA Stool Mini Kit(50))提取血液样本DNA,并通过琼脂糖凝胶和NanoDrop 2000 紫外分光光度计(Thermo Fisher Scientific)对抽提的DNA 质量和浓度进行检测。DNA产物置于-20 ℃保存备用。
使用BGISEQ-500测序仪测序,随机打断检测合格的DNA 样品,筛选符合要求、大小适合的DNA 片段,并将纯化后的DNA 片段连接测序接头,滚环扩增制备DNB,然后在阵列化测序芯片上进行双端测序。
2.2 测序及参考基因组序列比对
原始下机数据(raw data)经过SOAPnuke过滤后,应用BWA 软件将得到的clean data 比对到参考基因组上(NCBI 数据库的Project ID:PRJNA391565[25]),然后使用SAMtools 排序,参数为“-q1-C50-g-tDP,SP,DP4-I-d250-L250-m2-p”,并利用Picard 软件对重复reads 标记,最后使用GATK(https://www.broadinstitute.org/gatk/)进行BQSR质量值校正,并移除低质量测序数据。
2.3 基因型鉴定
SNP检测:使用Genome Analysis Toolkit(GATK)[27]对比对得到的BAM 文件进行单核苷酸多态(single nucleotide polymorphisms,SNPs)检测和过滤。为获得高质量SNP,选用参数QD<2,MQ<30,MQRankSum<-12.5,FS>200,ReadPosRankSum<20,QUAL<30.0和AN<40 对低质量的SNP 进行过滤。过滤后,利用HaplotypeCaller 软件,以GVCF 文件生成并统计每只个体形成的SNP基因型。为进一步精准比较北京麋鹿苑和江苏大丰麋鹿SNP 位点的多态性,使用VCFtools 对种群间未覆盖位点、个体间多态性低位点及重叠缺失位点进行过滤,参数为MAF>0.05。
2.4 遗传多样性分析
利用R 软件包统计SNP 位点数目和观测杂合度(Ho)。
2.5 遗传结构和分化
使用完整的SNP 数据集,利用VCFtools 计算2 个种群间的群体遗传分化指数(FST),分析群体间遗传关系远近,并计算Tajima’sD值,检测选择效应。
使用fastStructure[28]对种群祖先遗传成分进行分析,首先利用structure Harvester 做K值评估,设置K=2~8,迭代数设为100,每个K值运行20 次,统计K值的最大似然值。Structure 计算结果用Structure Harvester网页工具分析并进行ΔK计算,公式为:ΔK=mean(|L″(K)|)/sd(L(K)),根据结果绘制ΔK折线图,找出最佳K值。
使用Plink软件对2个群体的SNP基因型进行主成分(PCA)分析,利用R 软件绘制第一主成分与第二主成分的二维图。
使用FastTree[29]中的邻接法(neighbor-joining,NJ)进行1 000 次Bootstrap 检验,构建系统发育树(phylogenetic tree),用以研究个体之间的遗传聚类分析。
3 结果
3.1 测序数据质量及与参考基因组比对
18 只麋鹿个体简化基因组测序reads 数量为448.98 Mb~711.08 Mb。过滤后reads有效数据比例为98.91%~99.78%。Raw bases为44.90 Gb~71.11Gb,Clean bases 为44.50 Gb~70.94 Gb。CleanQ30bases rate 为92.86%~95.14%,说明样本建库测序的质量达到重测序标准,测序数据质量信息见表2。
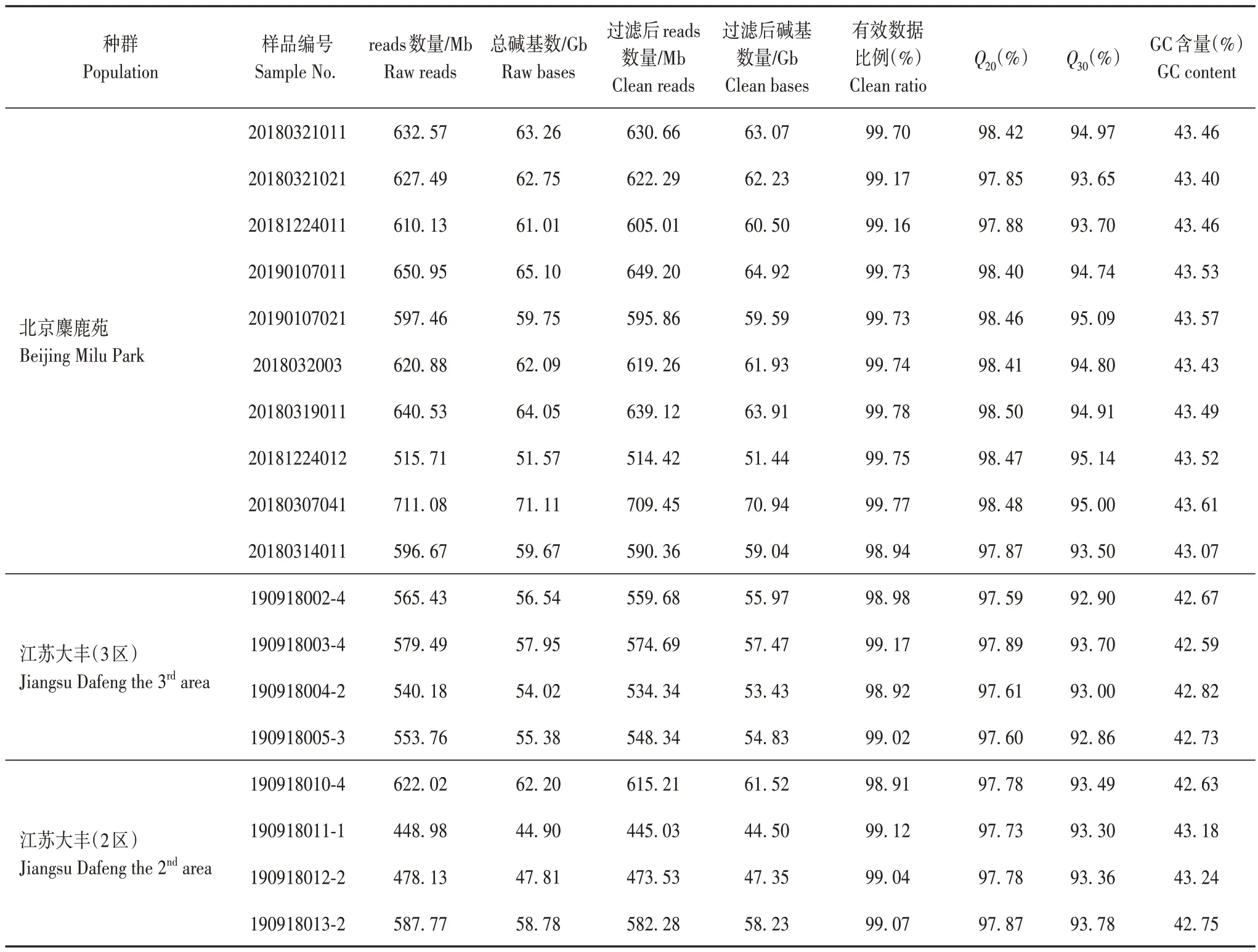
表2 测序数据质量信息汇总Tab.2 Summary of sequencing data quality information
本研究采用的参考基因组为麋鹿参考基因组,大小约为2.6 Gb(https://www.ncbi.nlm.nih.gov/assembly/GCA_002443075.1/),包 含194 812 条scaffold,GC 含量为40.5%[25]。18 份麋鹿样品简化基因组测序数据与参考基因组比对情况见表3。样本比对到参考基因组上的比例为98.18%~99.67%,比对到参考基因组唯一位置的比例为94.03%~94.68%,平均测序深度为15.70~24.37 倍,覆盖率为99.28%~99.54%,说明麋鹿简化基因组测序基本覆盖参考基因组,可用于后续分型和群体结构分析。
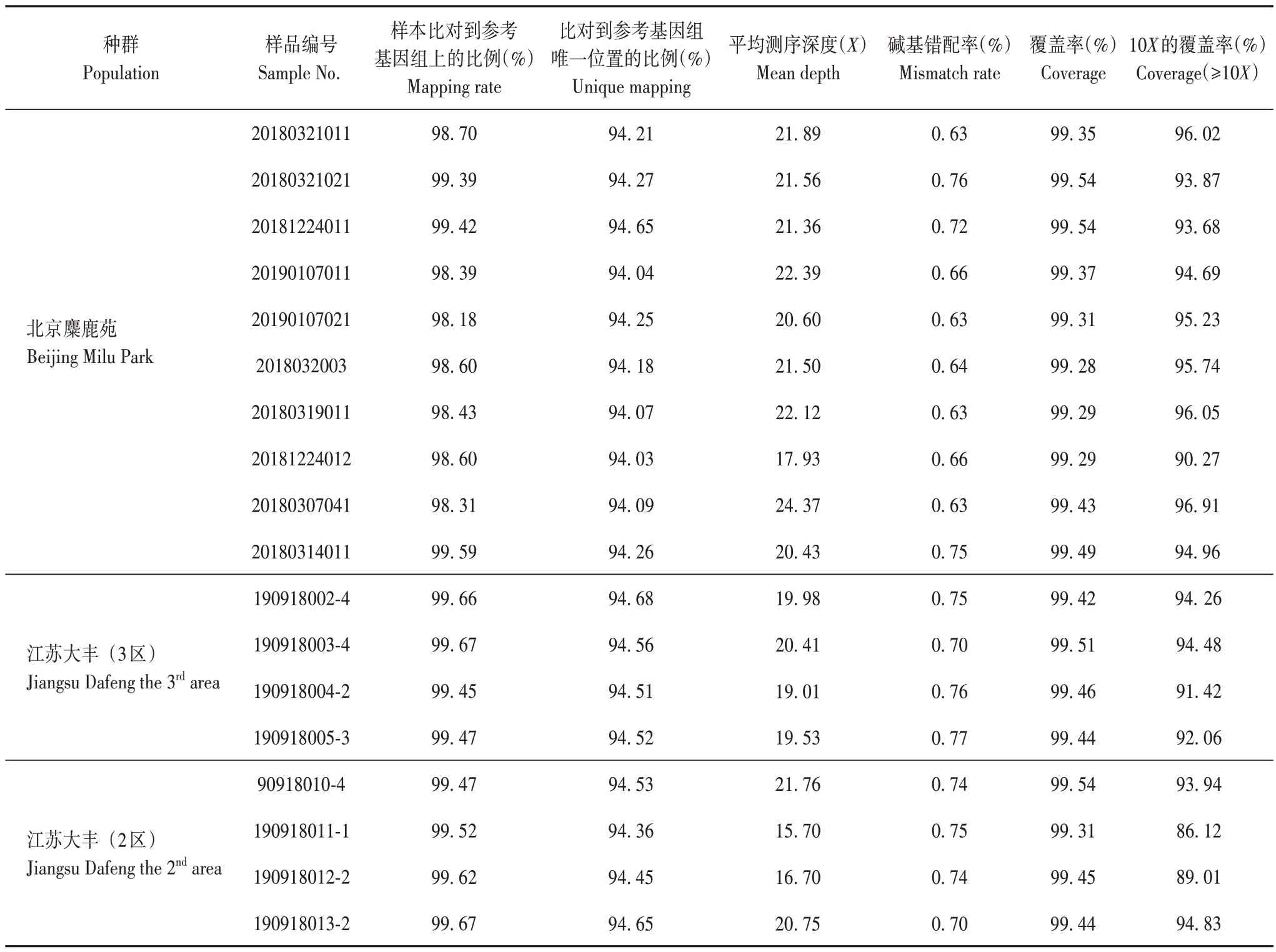
表3 麋鹿样品简化基因组测序数据与参考基因组比对Tab.3 Comparison of simplified genome sequencing data and reference genome of Père David’s deer
3.2 种群遗传多样性
经与参考基因组比较,样本共检测到SNP 位点37 581 752 个,遗传多样性统计见表4。北京麋鹿苑10 只麋鹿的SNP 平均值为2 166 903 个,江苏大丰8 只麋鹿的SNP 平均值为1 989 090 个。两地麋鹿SNP 位点的观测杂合度分别为0.59~0.68 和0.63~0.71。总体来说,北京麋鹿苑的麋鹿遗传多样性略低于江苏大丰。整合北京地区和江苏地区的两个麋鹿种群的SNP 标记,最终筛选到均匀分布于每条染色体、多态性好的共有SNP 位点1 456 457 个,用于后续遗传结构分析。

表4 麋鹿样本的SNP遗传多样性统计Tab.4 Statistics of SNP genetic diversity of Père David’s deer
3.3 种群遗传结构和分化
使用VCFtools 分析种群间FST,结果显示江苏大丰和北京麋鹿苑2 个种群间平均FST为0.085,表明2 个种群间存在中等程度的遗传分化。18 只麋鹿平均Tajima’sD为1.29,北京麋鹿苑麋鹿种群的平均Tajima’sD为0.90,江苏大丰麋鹿种群的平均Tajima’sD为0.89,各种群Tajima’sD分布如图1所示。
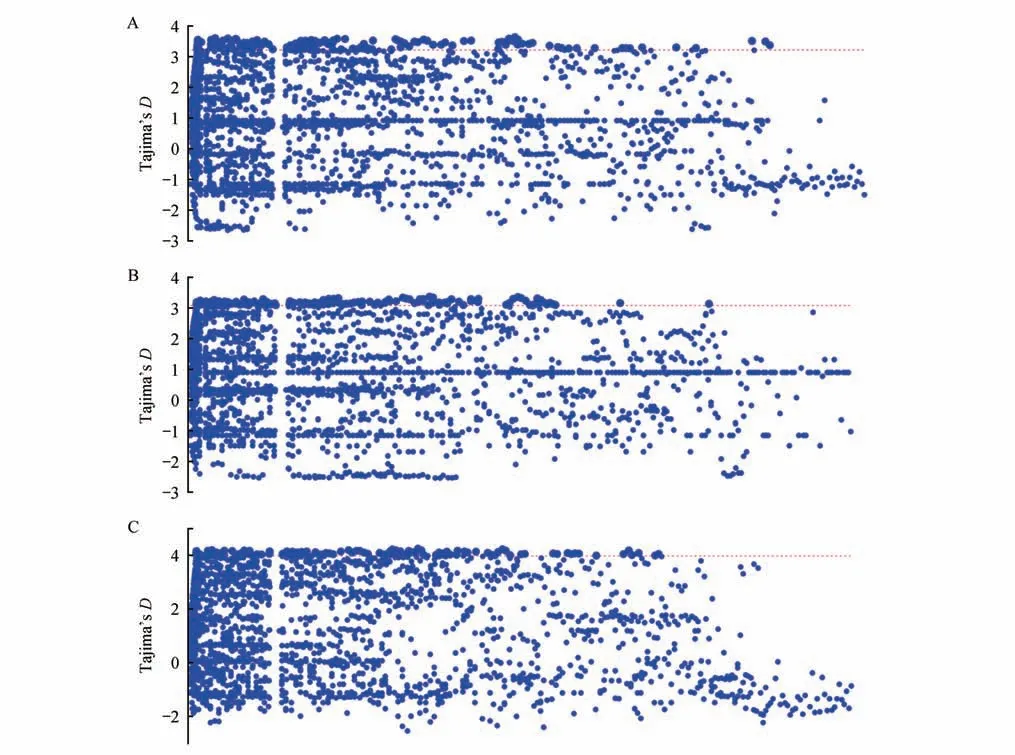
图1 不同种群Tajima’s D分布Fig.1 Distribution of Tajima’s D values in different populations
种群结构分析显示,当K=2 时,为最优K值,推测北京麋鹿苑和江苏大丰麋鹿种群有了初步分化(图2),并且北京麋鹿苑中,6 个个体显示出纯合性,表明相对于江苏大丰麋鹿种群来说,北京麋鹿苑个体的血统可能更为单一。
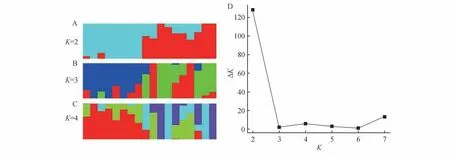
图2 麋鹿种群遗传结构分析Fig.2 Population structure analysis of Père David’s deer
基于SNP 标记的主成分分析结果显示,北京麋鹿苑和江苏大丰2 个种群之间具有一定的遗传分化,推测2 个种群地理引入个体可能存在一定的遗传差异(图3)。系统发育树结构也支持主成分分析结果,2 个种群分别形成单独的遗传聚类(图4)。总体来说,主成分分析与进化树聚类结果均表明北京麋鹿苑和江苏大丰2 个种群的遗传多样性已出现一定遗传的差异。

图3 麋鹿种群的主成分分析Fig.3 Principal component analysis diagram of Père David’s deer
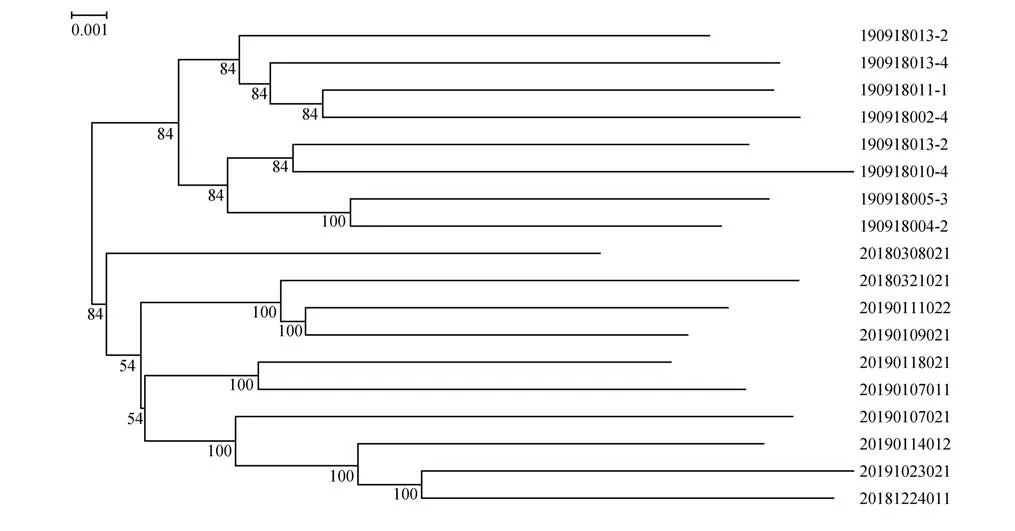
图4 麋鹿种群系统发育树Fig.4 Phylogenetic tree NJ-tree of Père David’s deer
4 分析与讨论
种群结构、群体分层及基因渗入是全面理解种群结构变异的重要指标。本研究从群体遗传结构、群体主成分分析和系统发育树,全面了解北京麋鹿苑和江苏大丰麋鹿种群间的分类关系及亲缘关系远近,显示出北京麋鹿苑半散养麋鹿与江苏大丰麋鹿种群存在中等程度的遗传分化。在群集最佳值为2(K=2)时,麋鹿种群分化较为显著。基于成对的SNP 差异NJ系统发育树也揭示2个重引入种群之间的独立遗传簇,这种遗传分化暗示了北京麋鹿苑与江苏大丰重引入群体之间存在一定的遗传差异。
使用遗传数据推断种群历史动态在群体遗传学研究中具有重要作用。种群中存在许多低频率的等位基因(稀有等位基因),表明群体存在定向选择或种群扩张,而种群中高等/中等频率的等位基因较多,则表明群体存在一定的平衡选择和瓶颈效应[30]。FST和Tajima’sD是衡量种群结构差异和动态变化的关键参数,FST适用于亚种群间的多样性比较,用于衡量种群分化程度,分化指数越大,差异越大。本研究结果显示江苏大丰和北京麋鹿苑2 个种群间平均FST为0.085,表明2个种群间存在中等程度的遗传分化。同时,本研究分析得出北京麋鹿苑与江苏大丰麋鹿种群的Tajima’sD>0,表明2个种群在种群重引进和迁地保护中可能都存在一定的瓶颈效应。
麋鹿群体尽管经历过严重的种群瓶颈,但仍存留较高的遗传多样性。本研究发现北京麋鹿苑与江苏大丰麋鹿种群的观测杂合度均大于0.50,表明2个种群的遗传多样性较丰富。目前,关于野生动物群体遗传多样性的研究已经进入基因组水平。Zhang等[25]通过第二代高通量基因组测序技术,构建了麋鹿第一个基因组草图。针对麋鹿[26]、朱鹮(Nipponia nippon)[31]、大熊猫(Ailuropoda melanoleuca)[32]和 北极熊(Ursus maritimus)[33],研究人员利用种群基因组测序和比较基因组进行遗传多样性的比较分析,结果显示朱鹮的近交程度最高,麋鹿次之,并认为麋鹿并没有存在很高的近交程度,这与多年来麋鹿群体的幼体存活率较高的结果[34]一致。
相比于单独繁育一个大群,多个小群分开繁育所面临的风险要大得多。中国在复壮麋鹿繁育种群时,不断对麋鹿实施多地迁地保护,根据长期的栖息地调查和麋鹿种群数量普查工作,目前已确认建立了89 个麋鹿繁育场所,为保护麋鹿储备了丰富的种源,但各种群间由于地理隔离而缺乏基因交流。本研究结果与这种现象一致,通过基因组测序技术,笔者发现北京麋鹿苑和江苏大丰种群之间产生了遗传分化,缺乏基因交流。因此,为更好地保护中国麋鹿种群的健康繁衍,加强各繁育场所的基因交流非常重要,应该增进不同种群间的基因交流。
本研究通过简化基因组测序技术,获得海量的第三代分子标记,利用高质量SNP 标记对18 只麋鹿的遗传资源进行遗传多样性和种群结构分析,发现北京麋鹿苑麋鹿的遗传多样性略低于江苏大丰。遗传结构与进化树结果均提示北京麋鹿苑与江苏大丰的麋鹿种群有初步分化,该分化可能由地理隔离因素、遗传漂变和瓶颈效应共同作用导致。由于本研究选用的麋鹿个体数量较少,SNP 标记组对麋鹿不同分布区的鉴定有效性还需进一步验证。
