环境RNA技术在水生生态系统生物多样性监测中的应用
2023-11-21李文琼代书勤线薇薇
李文琼, 代书勤, 张 辉, 2, 3, 线薇薇, 2
环境RNA技术在水生生态系统生物多样性监测中的应用
李文琼1, 3, 代书勤1, 张 辉1, 2, 3, 线薇薇1, 2
(1. 中国科学院海洋研究所 中国科学院海洋生态与环境科学重点实验室, 山东 青岛 266071; 2. 青岛海洋科学与技术试点国家实验室 海洋生态与环境科学功能实验室, 山东 青岛 266237; 3. 中国科学院大学, 北京 100049)
随着人类活动在全球范围内的不断增强, 水生生态系统的生物多样性丧失, 群落结构发生巨大变化。因此, 进行准确可靠的生物多样性监测来确定目标区域的物种丰度和群落结构对生态保护和资源可持续利用至关重要。环境DNA技术(eDNA)作为一种非入侵性的新手段, 正在迅速发展, 然而, eDNA技术易出现假阳性, 从而导致实时生物多样性监测不准确的现象仍然普遍存在。与eDNA技术相比, 环境RNA技术(eRNA)的快速降解使得它具有减少eDNA监测结果假阳性的潜力。本文概述了eRNA技术在水生生态系统的生物多样性监测中的可行性, 主要从eRNA技术在水生生态系统中的可检测性和提高检测分辨率的潜力两个方面来介绍; 综述了eRNA技术目前在水生生态系统生物多样性监测方面的研究进展; 指出了eRNA技术目前存在的问题并分析了未来的研究方向。本文对未来将eRNA技术实际应用于水生生态系统的生物多样性监测、生态保护和资源可持续利用具有一定的参考价值。
环境RNA; 生物多样性; 生态监测; 生态保护
随着网箱养殖、过度捕捞、矿物及油气资源开发等人类活动在全球范围内的不断增强, 水生生态系统的生物多样性丧失, 群落结构发生巨大变化[1]。为了在受影响区域实施及时、有效的措施以保护隐蔽的、脆弱的以及濒危的物种, 提前干预和监管外来物种的入侵行为并保证海洋资源的可持续利用, 探究准确可靠的生物多样性监测方法来确定目标区域的物种丰度和群落结构是至关重要的[2-3]。
上述挑战, 以及传统野外监测方法的局限性, 均促进了基于分子技术的生物多样性监测的快速发展。新型分子技术引入了对生物多样性进行非靶向监测的可能性, 它可以探索研究区域的所有群落, 并进一步提供了将某些稀有或未知物种和生态上重要的物种纳入生物多样性监测的可能性[4]。目前应用于生物多样性监测较为成熟的分子技术是环境DNA(environmental DNA, eDNA)技术, 该技术成本低、破坏性小、准确性高的特点使其越来越频繁的应用于水生生态监测, 其中包括生物多样性监测及生物量定量评估。例如, Zhang等[5]基于eDNA 技术研究了长江口及其邻近水域鱼类群落, 结果表明, 与传统方法相比, eDNA技术能够更为灵敏地监测到鱼类群落结构随季节的变化。Diao等[6]基于eDNA技术对中国南海的生物多样性进行监测, 结果表明, 通过eDNA技术成功实现了对三大类群的多样性监测。以上研究均证明了eDNA技术能够应用于水生生态系统生物多样性监测。在Salter等[7]对大西洋鳕鱼进行eDNA技术的定量研究中, 将eDNA技术与传统拖网调查的结果进行比较, 发现大西洋鳕鱼的捕获量与eDNA技术所得结果呈显著正相关关系, 因此, eDNA技术具有水生生态系统的鱼类种群的区域性定量评估的潜力。尽管eDNA技术日渐成熟且广泛应用于水生生态监测, 然而eDNA在生物体离开后仍在环境中持续存在数周以及在水柱中迁移的能力, 可能导致检测结果的假阳性, 从而导致实时生物多样性评估不准确[8-14]。而环境RNA技术(environmental RNA, eRNA)有望作为一种高敏感性的补充技术与eDNA技术结合使用来解决这一问题。
eRNA, 指在环境中存在的RNA, 由生物体释放到周围环境中, 主要以游离、囊泡或细胞的形式存在, 并在环境介质中发生一段距离的迁移, 之后大部分eRNA迅速降解, 还有极少的eRNA吸附在沉积物颗粒上向下沉降并在深海沉积物中积累[15-17](图1)。

图1 环境RNA在海洋中的活动轨迹
eRNA技术指从环境样本中直接提取RNA片段后进行相关生物多样性研究。eRNA技术的操作流程主要包括环境样品采集, 提取样品中的RNA并将其逆转录, 将获得的互补DNA(complementary DNA, cDNA)作为模板进行PCR扩增, 以及基于高通量测序技术来监测研究区域中的生物多样性(图2)。

图2 eRNA技术的操作流程——以水样为例
1 环境RNA技术在生物多样性监测中的可行性
与DNA相比, RNA是一种不太稳定的分子。RNA分子的单链结构、致使碱基催化水解的额外羟基的存在以及外源性和内源性RNA酶的普遍存在可能是eRNA的降解速率较eDNA更快的主要原因[18-19]。因此, 普遍认为环境介质中的RNA会因其过快的降解速率而无法检测到具有生物学意义的数量[20]。然而, 最近的实验研究表明, eRNA在水生生态系统中具有一定的可检测性、高释放率和快速周转率, 这些eRNA所特有的优势以及eRNA技术在提高检查分辨率方面的潜力, 使得eRNA技术在水生生态系统的生物多样性监测中具有可行性。
1.1 环境RNA在水生生态系统中的可检测性
Cristescu[15]在阐述eRNA持久性的综述性文章中表明, 代谢活跃的生物体可以将大量RNA释放到环境中, 而细胞外囊泡、衣壳等结构的存在能够防止RNA被RNA酶迅速降解, 从而大大延长了微生物或大型生物释放到周围环境中的RNA在环境中的存在时间; von Ammon等[21]在关于eDNA与eRNA分子信号的检测相关性的研究中表明, eRNA的可检测性随着eDNA拷贝数的增加而增加。这些研究为eRNA在水生生态系统中的可检测性提供了初步的理论依据。之后人们利用水族箱进行受控实验, 以更具体地探究eRNA在水体环境中的存在时间, 进一步证实了eRNA在水生生态系统中的可检测性。例如, Wood等[22]以两种海洋无脊椎动物(缨鳃虫和柄海鞘)为研究对象, 比较了eDNA和eRNA的可检测时间, 研究发现eRNA在移除生物体的水族箱中的可检测时间长达13 h, 比eDNA时间要短; Mérou等[23]在检测受感染牡蛎中的牡蛎寄生虫的eDNA和eRNA在海水中存在时间的实验中发现, 牡蛎寄生虫的eRNA能够持续存在30 d, 比eDNA的存在时间还长; Marshall等[24]基于eDNA技术和eRNA技术, 采集了移除斑马贻贝和条纹贻贝后的水族箱样品进行研究, 结果表明在移除生物体的3 d后仅检测到核糖体RNA(ribosomal RNA, rRNA)基因靶标, 而H2B信使RNA(messenger RNA, mRNA)基因靶标在24 h后就无法检测到。在最近的2项探究环境条件对eRNA降解速率影响的实验中, Qian等[25]调查了温度对中国对虾eRNA可检测时间的影响, 结果显示eRNA在低温下可检测时间较长(22 h左右), 而随着温度的升高, eRNA的可检测时间缩短至11 h左右; Kagzi等[26]在基于pH梯度探究蚤状溞eRNA的存在时间的研究中发现, pH值的变化对其eRNA的存在时间影响不大, 范围约为21~27 h。在最近的一项野外试验中, Littlefair等[27]发现, 野外采集水样后低温储存2~5 h仍然可以检测到来自eRNA的可靠信号。因此, eRNA可以足够持久的存在于水体环境中, 这使得eRNA具有一定的可检测性。
1.2 环境RNA技术的检测分辨率
eDNA技术的分辨率仅限于物种水平的检测或种群水平的遗传推断, 而eRNA技术在这方面具有超越eDNA技术的潜力[28]。多年来, 侵入性生物监测方法一直使用从组织或整个生物体产生的 mRNA图谱来阐明个体的生物学状态[29-30]。从微观上来说, mRNA反映了来源生物的转录图谱。个体随时间推移或者来自不同环境的个体之间所产生的mRNA图谱的变化可以作为生物体对特定环境应激源反应的特征。也就是说, 拥有相似基因图谱的生物体, 根据它们的发育阶段、生理状态、遗传背景或表型的不同, 可能会在转录图谱中表现出显著的差异[31-34]。
由于RNA序列只由在生物体组织内转录活跃的基因产生, 因此不同状态的同种生物之间相应的转录特征可以由生物体产生的eRNA来反映, 而这种生物特征可以在一段时间内检测到[22, 24]。因此, Yates[20]认为eRNA技术能够比eDNA技术以更高的检测分辨率来更精确地反映物种的存在, 并提供物种存在与否之外的信息, 这个过程可以通过使用高特异性靶向分析检测eRNA序列, 反映从环境样品中检测到产生的特异性或强烈上调的eRNA转录物相应的生理状态来实现。Tsuri等[35]的研究首次证明了eRNA技术具有提高检测分辨率的能力, 研究通过斑马鱼eRNA的mRNA分型来确认遗传分子的组织来源, 发现一部分特定的靶向组织的mRNAs是在水中检测到的eRNA的来源, 这进一步证明了eRNA技术具有监测水生脊椎动物生理状态的潜力。因此, eRNA技术有望通过检测不同生理状态特异性表达的mRNA分型来监测生物个体的生理状态, 从而提高生物多样性监测的检测分辨率, 以更好的进行生态保护, 进一步实现资源可持续利用。
2 环境RNA技术的研究进展
自2010年以来, 随着生物技术特别是高通量测序技术的飞速发展, 越来越多的研究利用eRNA技术监测复杂的生态系统的不同生物的多样性, 主要包括陆地以及水生生态系统等, 其中与水生生态系统有关的研究约占eRNA技术相关研究总数的89%[36-43](图3)。
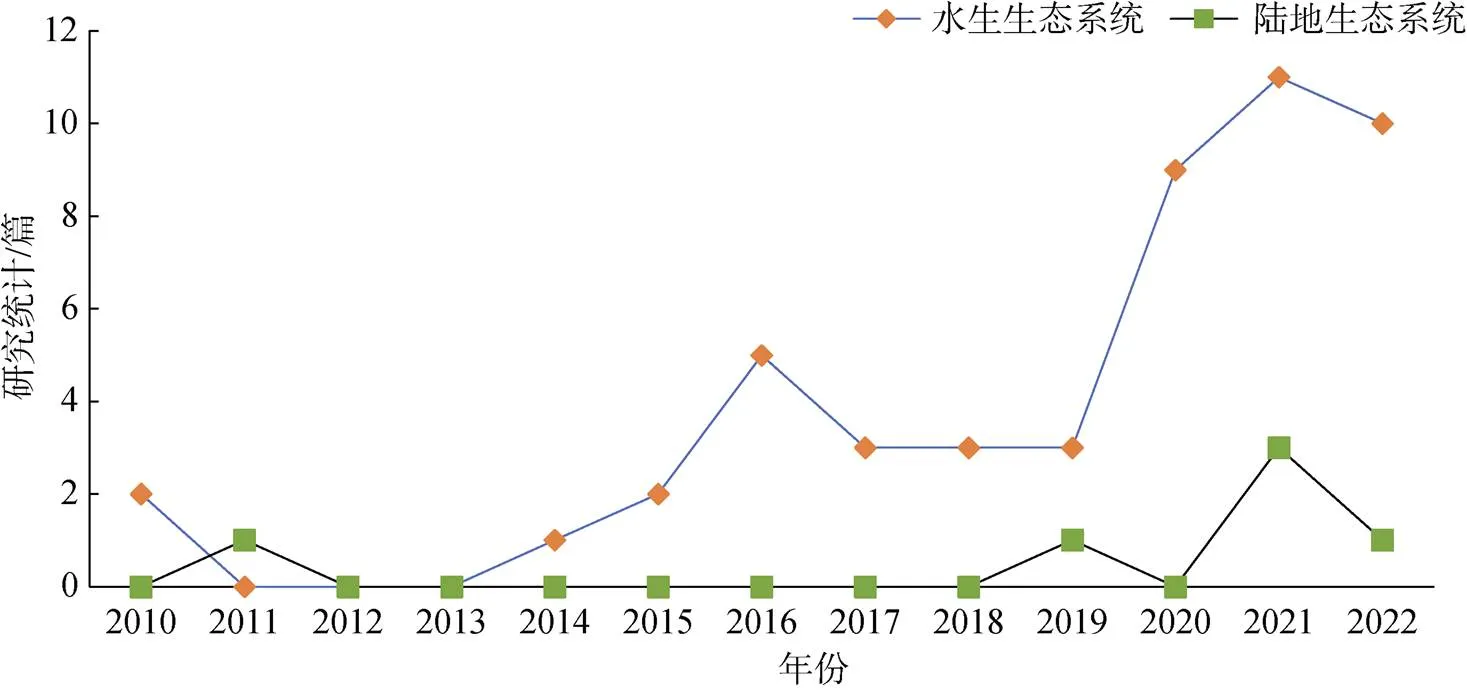
图3 环境RNA研究统计
截至2022年7月, 相比于较为成熟并已投入实际应用的eDNA技术, 大多数评估eRNA技术对生物多样性监测的适用性的研究都是概念验证研究, 还有一部分是与eDNA技术进行对比或作为传统监测方法和eDNA技术的补充方法进行的实验研究。虽然eRNA技术主要用于监测水生生态系统生物多样性, 但其中多数研究主要集中在海洋沉积环境中的原核生物、微型真核生物等小型生物的多样性监测上, 而利用eRNA技术监测海洋水体环境中的鱼类等大型生物的生物多样性的研究相对较少(图4)。
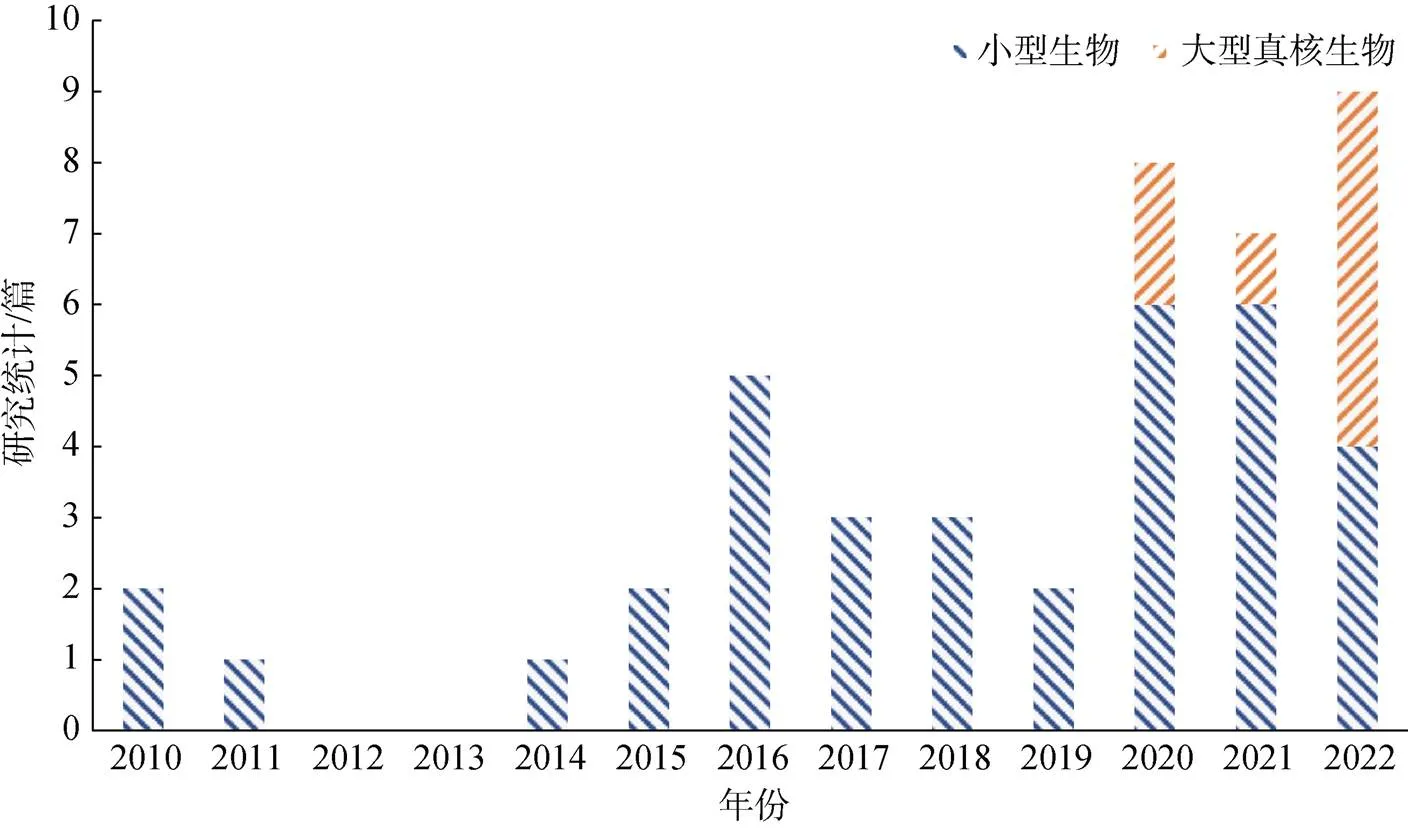
图4 以小型生物与大型真核生物为研究对象的环境RNA研究统计
2.1 环境RNA技术在小型生物多样性监测中的应用
自2014年以来, 许多研究将eRNA技术用于水生生态系统的沉积环境和水体环境中原核生物、微型真核生物等小型生物的多样性监测上, 并与基于eDNA技术和传统的监测方法所得数据进行了比较, 以反映沉积物和水体中小型生物的多样性受人类活动的影响, 评估eRNA技术在监测水生生态系统的生物多样性方面的可行性。尽管一部分研究认为, eDNA技术在进行生物多样性监测方面的可行性与eRNA技术一致甚至更高[41, 44-47]。但更多研究发现, eRNA技术在生物多样性监测方面有相较eDNA技术所特有的优势, 因此能够作为一种监测水生生态系统的小型生物多样性的有力工具(表1)。
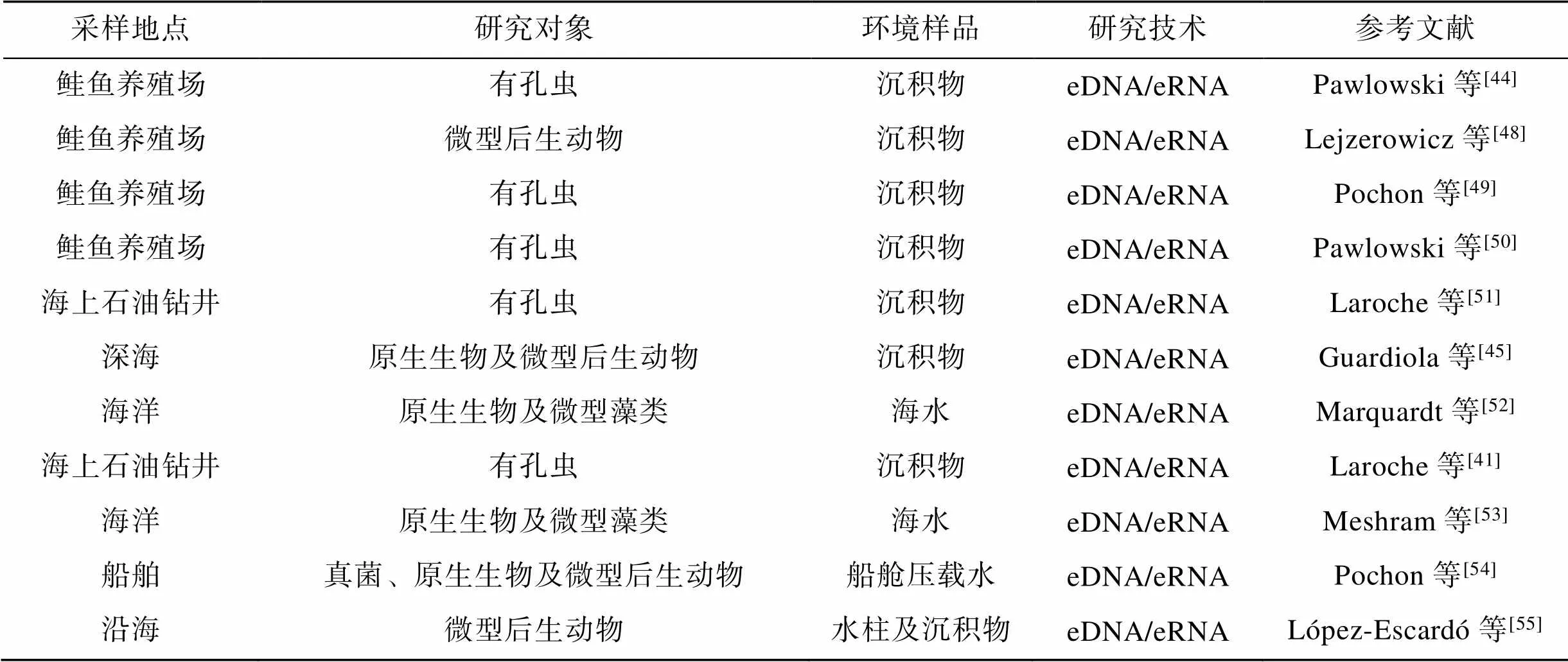
表1 eRNA 技术应用在小型生物群落监测中的文献统计
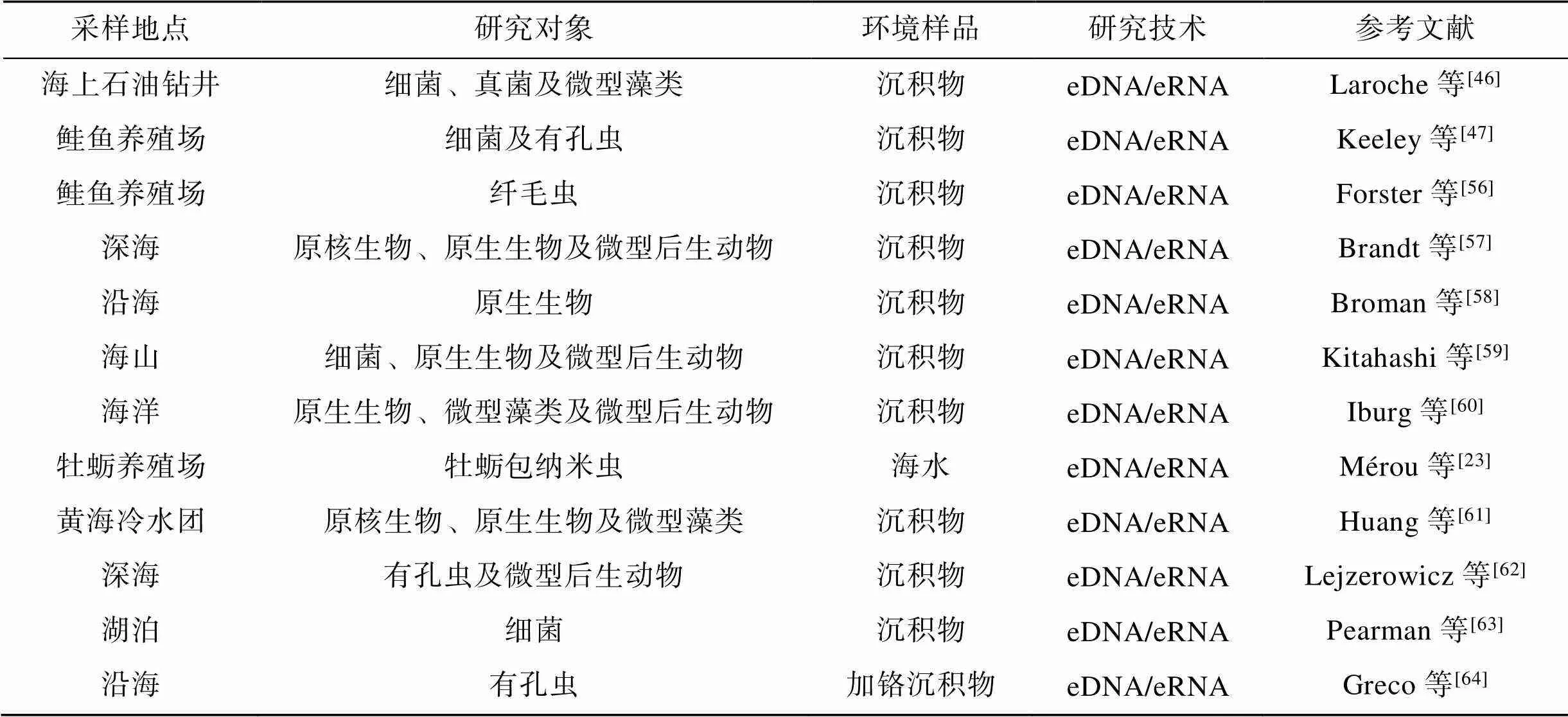
续表
在水生生态系统的沉积环境中, Pawlowski等[44, 50]和Pochon等[49]对海洋渔业养殖场沉积物中的有孔虫进行监测, 发现eRNA技术能够更加强烈地检测到有孔虫物种丰富度随着有机富集程度的增加而急剧下降; Pochon等[49]基于eRNA技术所推断的不同流量养殖场之间的有孔虫群落结构与基于eDNA技术及传统的监测方法的所得结果一致, 且eRNA技术所得数据与各项生物指数及环境变量之间存在比eDNA技术更强的相关性; Laroche等[41, 46, 51]在监测近海石油钻井对有孔虫群落影响的研究中发现eRNA技术所得数据与环境变量及空间梯度之间的相关性更强, 且能够更好地反映人类活动对α多样性的影响。Lejzerowicz等[48]监测了苏格兰鲑鱼养殖场中微型后生动物的生物多样性, 证明了基于eRNA技术推断的生物指数能更好地反映与基于传统监测方法所得生物指数之间的相关性; Forster等[56]以纤毛虫为研究对象, 发现基于eRNA技术的纤毛虫群落监测的分辨率比基于eDNA技术的更高; Huang等[61]在对黄海冷水团内外底栖微型真核生物群落结构进行调查的研究显示, eRNA技术分析在监测海洋底栖微型真核生物的空间分布方面具有很大优势。在水生生态系统的水体环境中, Marquardt等[52]和Meshram等[53]在监测挪威西斯皮茨卑尔根岛海水中的原生生物及微型藻类的生物多样性研究中得出, eRNA技术能够更好地反映微型真核生物群落中纤毛虫类群的存在; Pochon等[54]对新西兰附近码头的船舱压载水样品进行调查, 发现基于eRNA技术与eDNA技术所得数据之间存在差异, 且更为突出地反映了纤毛虫等微型后生动物的生物多样性。eRNA技术可以作为水生生态系统小型生物多样性监测的有效工具。
2.2 环境RNA技术在大型生物多样性监测中的应用
eRNA技术的相关研究多聚焦于海洋、淡水沉积物中的小型生物多样性的监测, 而以水生生态系统中的鱼类等大型生物为研究对象的相关研究较少。但自2020年开始, 一些利用eRNA技术来监测水生生态系统中大型生物多样性的研究证实, 其可以作为eDNA技术的一种补充工具来监测鱼类等大型生物的群落结构和物种多样性(表2)。
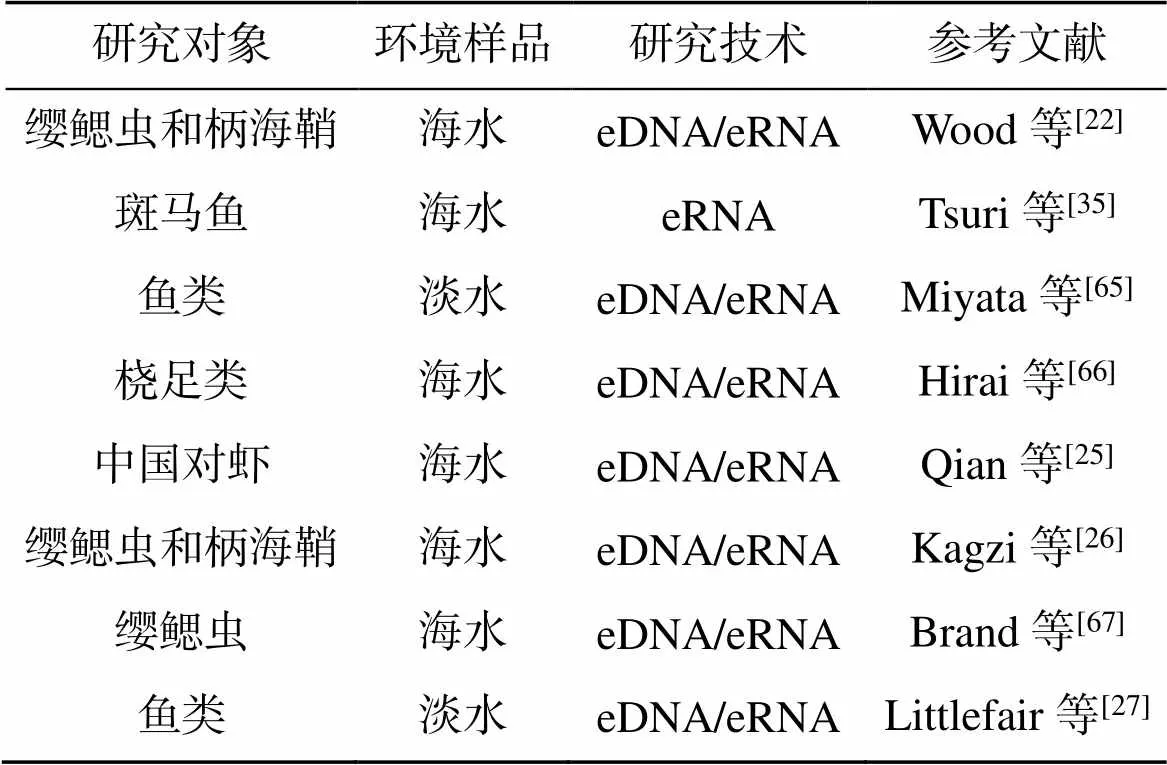
表2 eRNA技术应用在大型真核生物群落监测中的研究统计
在水族箱实验的基础上, Tsuri等[35]成功检测了斑马鱼的6个特异性mRNA靶标; Miyata等[65]通过eRNA技术靶向在日本那珂河中采集的水样中的线粒体12S rRNA基因, 成功地对鱼类进行了eRNA技术的分析, 结果显示eRNA技术对环境中存在物种的检测敏感性略高于eDNA技术, 而阳性预测性明显高于eDNA技术, 同时, eRNA技术所得数据与传统的实地调查所得物种丰度之间的相关性高于eDNA技术, 因此, 可以得出eRNA技术具有更强的潜力来识别实际存在于采样点的代谢活跃的鱼类物种组合; Littlefair等[27]利用eRNA技术来调查加拿大IISD实验淡水湖区中采集的水样, 研究成功确认eRNA来源于鱼类生物, 且实现了与eDNA技术相似的鱼类物种检测率, 甚至其检测到的真阳性率明显更高, 说明eRNA技术能够准确检测到更多的已知存在于湖泊中的物种。因此, eRNA技术在监测水生生态系统中包括鱼类在内的大型真核生物的多样性方面是切实可行的。
2.3 环境RNA技术与环境DNA技术相结合
自2016年以来, 大量研究对水生生态系统中的小型生物多样性进行监测, 证明了将eRNA技术与eDNA技术结合使用, 能够监测最近存在于研究区域的代谢活跃的生物类群, 有效改善eDNA技术所产生的假阳性, 以提高检测结果的准确性。Meshram等[53]的研究显示, 将二者结合使用可以解释已识别分类群的代谢活性水平; Pochon等[54]在调查船舶压载水样品的研究中, 进一步证实了eRNA技术能够确定环境样品中是否存在代谢活跃的生物体; Lejzerowicz等[62]监测太平洋上克拉里昂-克利珀顿区深海底栖微型真核生物的生物多样性, 结果表明eRNA技术可以更准确地描绘深海群落在空间和时间上的斑块性, 从而反映代谢活跃的、更短的时间尺度内的生物多样性。Laroche等[41, 46]研究表明, 结合eRNA技术和eDNA技术所得数据、建立共享数据集, 有望成为处理遗留DNA、减少假阳性更为有效的方法; Greco等[64]监测围隔生态系统中铬含量不同的沉积物中底栖有孔虫群落所受的影响, 结果表明结合使用eRNA技术和eDNA技术有利于更准确地捕捉生态影响; López- Escardó等[55]在基于eRNA技术调查欧洲六6个沿海地点采集的水柱和沉积物样品的研究中发现, 使用eRNA技术和eDNA技术所得的共享数据集能够更好地监测研究区域的生物多样性。因此, eRNA技术有望成为eDNA技术的有力补充。
3 环境RNA技术存在的问题及改进措施
eRNA技术可以与eDNA技术优势互补、结合使用来提高监测水生生态系统生物多样性的准确度, 但迄今为止, eRNA技术与eDNA技术仍然存在很多问题亟待解决。因此, 将eRNA技术作为新型补充工具投入实际应用还需要很多努力(表3)。
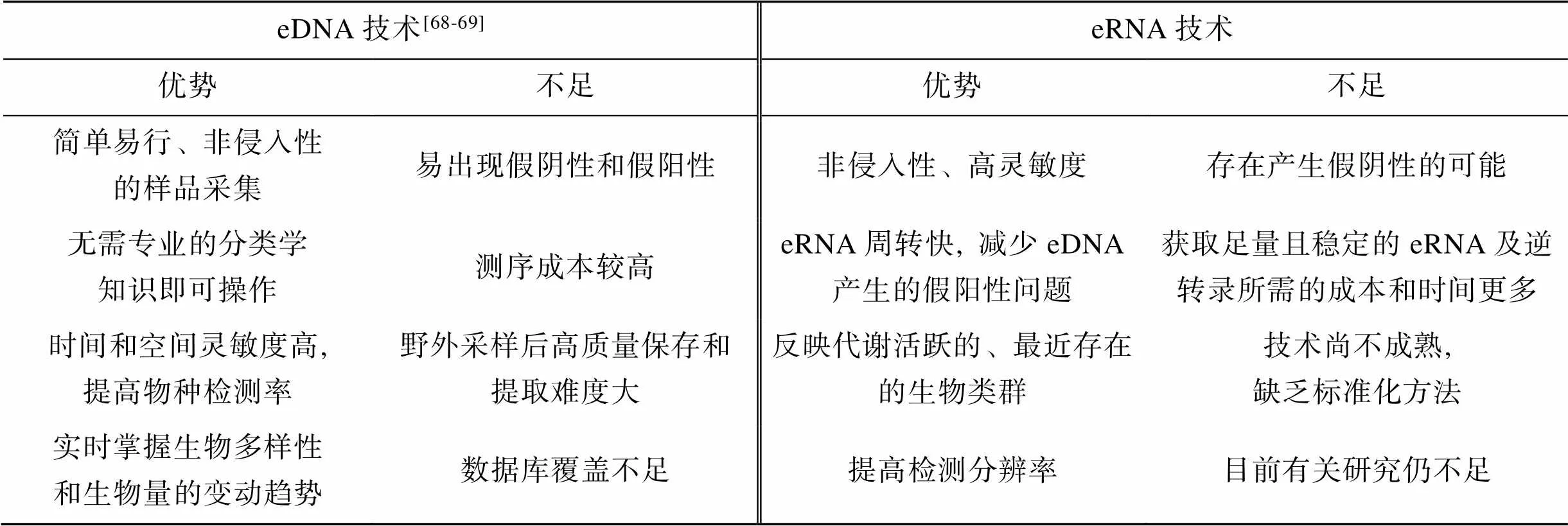
表3 eDNA和eRNA技术监测生物多样性的优缺点总结
一方面, 相比于eDNA技术, eRNA的快速降解使eRNA技术投入应用的条件更为严格[54]。具体表现在eRNA技术在样品采集、保存以及提取过程中, 为了确保能够获得足量的eRNA以用于下游分析, 需要付出比eDNA技术更多的成本和时间。在目前的研究中, 将样品低温保存或储存在RNA保存液中通常是最大限度地减少现场采样和实验室处理之间eRNA损失的方法。然而, 低温保存以及RNA保存液昂贵的成本, 使得eRNA技术的实际应用存在限制[70]。另一方面, 与eRNA的组成、来源、浓度等相关的问题还没有明确的结论, 且水生生态系统中eRNA释放和降解的相关研究均是在封闭的水族箱中针对有限的目标物种进行的, 并不能完全还原自然环境对eRNA的影响, 也无法推断各项研究结果之间存在差异甚至完全相反的原因[20]。
因此, 需要针对eRNA的敏感性来进一步研究专门、廉价且能够广泛应用的采样、保存以及提取方法, 从而得到浓度高、稳定性好的eRNA以用于下游分析。另外, 需要对eRNA自身存在的问题及自然环境中不同物种的eRNA的释放和降解进行进一步研究。
4 结论与展望
eRNA技术主要通过收集环境中的RNA片段来实现相关生物多样性的监测调查。作为一种新颖的分子技术, 目前关于eRNA技术的研究大多是概念验证和实验性研究。在研究对象上, 目前基于eRNA技术的研究多集中于水生生态系统沉积环境中的原生生物等小型生物, 而有关水生生态系统中水体环境中的鱼类等大型真核生物的研究相对较少; 在全球范围内, 目前基于eRNA技术监测水生生态系统生物多样性的研究主要集中在新西兰、日本、加拿大等发达国家, 而国内基于eRNA技术的相关研究相对空缺。但eRNA技术具有不容忽视的应用价值, 在未来仍需国内外的科研工作者们进一步开展相关研究, 特别是对海洋水体环境中的鱼类等大型真核生物多样性的研究, 以充分发挥eRNA技术的优势和潜力, 为海洋生物多样性监测提供理论支持和技术手段。
目前, 将eRNA技术投入实际应用还需要解决很多问题。第一, 需要进一步对生物所释放的eRNA在自然环境中的可检测性进行确定; 第二, 需要标准化eRNA技术的采集方法; 第三, 需要进一步解决eRNA本身所存在的问题。eRNA技术的投入使用将有望准确监测最近存在于研究区域的代谢活跃的生物类群的生物多样性, 减少eDNA技术带来的假阳性, 传递生物个体生理状态、环境压力响应和繁殖状态信息, 提高实际应用中的检测分辨率, 从而实现高灵敏度分析, 这有助于更准确地监测人类活动造成的环境扰动并为生态保护和资源可持续提供新的信息。但应当注意的是, eRNA技术并不能完全替代eDNA技术, 应将二者结合应用, 优势互补, 以有效减少误差的出现, 提高研究的准确性, 在水生生态系统生物多样性监测方面共同发挥作用。
[1] Zizka V M A, Koschorreck J, Khan C C, et al. Long-term archival of environmental samples empowers biodiversity monitoring and ecological research[J]. Environmental Sciences Europe, 2022, 34(1): 40.
[2] Ficetola G F, Miaud C, Pompanon F, et al. Species detection using environmental DNA from water samples[J]. Biology Letters, 2008, 4(4): 423-425.
[3] Rees H C, Maddison B C, Middleditch D J, et al. The detection of aquatic animal species using environmental DNA-a review of eDNA as a survey tool in ecology[J]. Journal of Applied Ecology, 2014, 51(5): 1450-1459.
[4] Darling J A, Galil B S, Carvalho G R, et al. Recommendations for developing and applying genetic tools to assess and manage biological invasions in marine ecosystems[J]. Marine Policy, 2017, 85: 54-64.
[5] Zhang H, Yoshizawa S, Iwasaki W, et al. Seasonal fish assemblage structure using environmental DNA in the Yangtze Estuary and its adjacent waters[J]. Frontiers in Marine Science, 2019, 6: 515.
[6] Diao C Y, Jia H, Guo S J, et al. Biodiversity exploration in autumn using environmental DNA in the South China sea[J]. Environmental Research, 2022, 204: 112357.
[7] Salter I, Joensen M, Kristiansen R, et al. Environmental DNA concentrations are correlated with regional biomass of Atlantic cod in oceanic waters[J]. Communications Biology, 2019, 2: 461.
[8] Barnes M A, Turner C R, Jerde C L, et al. Environmental conditions influence eDNA persistence in aquatic systems[J]. Environmental Science & Technology, 2014, 48(3): 1819-1827.
[9] Dejean T, Valentini A, Duparc A, et al. Persistence of environmental DNA in freshwater ecosystems[J]. PloS One, 2011, 6(8): e23398.
[10] Goldberg C S, Sepulveda A, Ray A, et al. Environmental DNA as a new method for early detection of New Zealand mudsnails ()[J]. Freshwater Science, 2013, 32(3): 792-800.
[11] Thomsen P F, Kielgast J, Iversen L L, et al. Monitoring endangered freshwater biodiversity using environmental DNA[J]. Molecular Ecology, 2012, 21(11): 2565-2573.
[12] Deiner K, Altermatt F. Transport distance of invertebrate environmental DNA in a natural river[J]. PloS One, 2014, 9(2): e88786.
[13] Shogren A J, Tank J L, Andruszkiewicz E, et al. Controls on eDNA movement in streams: Transport, retention, and resuspension[J]. Scientific Reports, 2017, 7: 5065.
[14] Cristescu M E, Hebert P D N. Uses and misuses of environmental DNA in biodiversity science and conservation[J]. Annual Review of Ecology Evolution and Systematics, 2018, 49(1): 209-230.
[15] Cristescu M E. Can environmental RNA revolutionize biodiversity science?[J]. Trends in Ecology & Evolution, 2019, 34(8): 694-697.
[16] Torti A, Lever M A, Jorgensen B B. Origin, dynamics, and implications of extracellular DNA pools in marine sediments[J]. Marine Genomics, 2015, 24: 185-196.
[17] Orsi W, iddle J F, Edgcomb V. Deep sequencing of subseafloor eukaryotic rRNA reveals active Fungi across marine subsurface provinces[J]. PloS One, 2013, 8(2): e56335.
[18] Li Y F, Breaker R R. Kinetics of RNA degradation by specific base catalysis of transesterification involving the 2’-hydroxyl group[J]. Journal of the American Chemical Society, 1999, 121(23): 5364-5372.
[19] Chee T S, Chin Y B. DNA, RNA, and protein extraction: The past and the present[J]. Journal of Biomedicine & Biotechnology, 2009, 2009: 574398.
[20] Yates M C, Derry A M, Cristescu M E. Opinion environmental RNA: A revolution in ecological resolution?[J]. Trends in Ecology & Evolution, 2021, 36(7): 601-609.
[21] Von Ammon U, Wood S A, Laroche O, et al. Linking environmental DNA and RNA for improved detection of the marine invasive fanworm[J]. Frontiers in Marine Science, 2019, 6: 00621.
[22] Wood S A, Biessy L, Latchford J L, et al. Release and degradation of environmental DNA and RNA in a marine system[J]. Science of the Total Environment, 2020, 704: 135314.
[23] MÉrou N, Lecadet C, Pouvreau S, et al. An eDNA/eRNA-based approach to investigate the life cycle of non-cultivable shellfish micro-parasites: the case of, a parasite of the European flat oyster[J]. Microbial Biotechnology, 2020, 13(6): 1807-1818.
[24] Marshall N T, Vanderploeg H A, Chaganti S R. Environmental (e)RNA advances the reliability of eDNA by predicting its age[J]. Scientific Reports, 2021, 11(1): 2769.
[25] Qian T Y, Shan X J, Wang W J, et al. Effects of temperature on the timeliness of eDNA/eRNA: A case study of[J]. Water, 2022, 14(7): 1155.
[26] Kagzi K, Hechler R M, Fussmann G F, et al. Environmental RNA degrades more rapidly than environmental DNA across a broad range of pH conditions[J]. Molecular Ecology Resources, 2022, 22(7): 2640-2650.
[27] Littlefair J E, Rennie M D, Cristescu M E. Environmental nucleic acids: A field-based comparison for monitoring freshwater habitats using eDNA and eRNA[J]. Molecular Ecology Resources, 2022, 22(8): 2928-2940.
[28] Sigsgaard E E, Jensen M R, Winkelmann I E, et al. Population-level inferences from environmental DNA-Current status and future perspectives[J]. Evolutionary Applications, 2020, 13(2): 245-262.
[29] Tom M, Auslander M. Transcript and protein environmental biomarkers in fish—a review[J]. Chemosphere, 2005, 59(2): 155-162.
[30] Raftos D A, Melwani A R, Haynes P A, et al. The biology of environmental stress: molecular biomarkers in Sydney rock oysters ()[J]. Environmental Science-Processes & Impacts, 2016, 18(10): 1359-1359.
[31] Akbarzadeh A, Gnther O, Houde A L, et al. Developing specific molecular biomarkers for thermal stress in salmonids[J]. BMC Genomics, 2018, 19(1): 749.
[32] Debes P V, Normandeau E, Fraser D J, et al. Differences in transcription levels among wild, domesticated, and hybrid Atlantic salmon () from two environments[J]. Molecular Ecology, 2012, 21(11): 2574-2587.
[33] Spanier K I, Mieke J, Ellen D, et al. Conserved transcription factors steer growth-related genomic programs in daphnia[J]. Genome Biology & Evolution, 2017, 9(6): 1821-1842.
[34] Watanabe Y, Tanaka R, Kobayashi H, et al. Metamorphosis-dependent transcriptional regulation of, a noveltype I keratin gene[J]. Developmental Dynamics, 2002, 225(4): 561-570.
[35] Tsuri K, Ikeda S, Hirohara T, et al. Messenger RNA typing of environmental RNA (eRNA): A case study on zebrafish tank water with perspectives for the future development of eRNA analysis on aquatic vertebrates[J]. Environmental DNA, 2020, 3(1): 14-21.
[36] Pareek C S, Smoczynski R, Tretyn A. Sequencing technologies and genome sequencing[J]. Journal of Applied Genetics, 2011, 52(4): 413-435.
[37] Soon W W, Hariharan M, Snyder M P. High-throughput sequencing for biology and medicine[J]. Molecular Systems Biology, 2013, 9: 640.
[38] Adrian-Kalchhauser I, Burkhardt-Holm P. An eDNA assay to monitor a globally invasive fish species from flowing freshwater[J]. PloS One, 2016, 11(1): e0147558.
[39] Clusa L, Ardura A, Gower F, et al. An easy phylogenetically informative method to trace the globally invasivemud snail from River’s eDNA[J]. PloS One, 2016, 11(10): e0162899.
[40] Eichmiller J J, Bajer P G, Sorensen P W. The Relationship between the distribution of common carp and their environmental DNA in a small lake[J]. PloS One, 2014, 9(11): e112611.
[41] Laroche O, Wood S A, Tremblay L A, et al. Metabarcoding monitoring analysis: the pros and cons of using co-extracted environmental DNA and RNA data to assess offshore oil production impacts on benthic communities[J]. Peerj, 2017, 5: e3347.
[42] Stoeckle M Y, Soboleva L, Charlop- Powers Z. Aquatic environmental DNA detects seasonal fish abundance and habitat preference in an urban estuary[J]. PloS One, 2017, 12(4): e0175186.
[43] Wood S A, Pochon X, Ming W, et al. Considerations for incorporating real-time PCR assays into routine marine biosecurity surveillance programmes: a case study targeting the Mediterranean fanworm () and club tunicate ()[J]. Genome, 2019, 62(3): 137-146.
[44] Pawlowski J, Esling P, Lejzerowicz F, et al. Environmental monitoring through protist next-generation sequencing metabarcoding: assessing the impact of fish farming on benthic foraminifera communities[J]. Molecular Ecology Resources, 2014, 14(6): 1129-1140.
[45] Guardiola M, Wangensteen O S, Taberlet P, et al. Spatio-temporal monitoring of deep-sea communities using metabarcoding of sediment DNA and RNA[J]. Peerj, 2016, 4: e2807.
[46] Laroche O, Wood S A, Tremblay L A, et al. A cross-taxa study using environmental DNA/RNA metabarcoding to measure biological impacts of offshore oil and gas drilling and production operations[J]. Marine Pollution Bulletin, 2018, 127: 97-107.
[47] Keeley N, Wood S A, Pochon X. Development and preliminary validation of a multi-trophic metabarcoding biotic index for monitoring benthic organic enrichment[J]. Ecological Indicators, 2018, 85: 1044-1057.
[48] Lejzerowicz F, Esling P, Pillet L, et al. High-throughput sequencing and morphology perform equally well for benthic monitoring of marine ecosystems[J]. Scientific Reports, 2015, 5: 13932.
[49] Pochon X, Wood S A, Keeley N B, et al. Accurate assessment of the impact of salmon farming on benthic sediment enrichment using foraminiferal metabarcoding[J]. Marine Pollution Bulletin, 2015, 100(1): 370-382.
[50] Pawlowski J, Esling P, Lejzerowicz F, et al. Benthic monitoring of salmon farms in Norway using foraminiferal metabarcoding[J]. Aquaculture Environment Interactions, 2016, 8: 371-386.
[51] Laroche O, Wood S A, Tremblay L A, et al. First evaluation of foraminiferal metabarcoding for monitoring environmental impact from an offshore oil drilling site[J]. Marine Environmental Research, 2016, 120: 225-235.
[52] Marquardt M, Vader A, Stubner E I, et al. Strong seasonality of marine microbial eukaryotes in a high-Arctic fjord (Isfjorden, in West Spitsbergen, Norway)[J]. Applied and Environmental Microbiology, 2016, 82(6): 1868-1880.
[53] Meshram A R, Vader A, Kristiansen S, et al. Microbial eukaryotes in an Arctic under-ice spring bloom north of Svalbard[J]. Frontiers in Microbiology, 2017, 8: 1099.
[54] Pochon X, Anastasija Z, Fletcher L M, et al. Wanted dead or alive? Using metabarcoding of environmental DNA and RNA to distinguish living assemblages for biosecurity applications[J]. PloS One, 2017, 12(11): e0187636.
[55] LÓpez-EscardÓD, Paps J, Vargas C D, et al. Metabarcoding analysis on European coastal samples reveals new molecular metazoan diversity[J]. Scientific Reports, 2018, 8: 9106.
[56] Forster D, Filker S, Kochems R, et al. A comparison of different ciliate metabarcode genes as bioindicators for environmental impact assessments of salmon aquaculture[J]. Journal of Eukaryotic Microbiology, 2019, 66(2): 294-308.
[57] Brandt M I, Trouche B, Henry N, et al. An assessment of environmental metabarcoding protocols aiming at favoring contemporary biodiversity in inventories of deep-sea communities[J]. Frontiers in Marine Science, 2020, 7: 234.
[58] Broman E, Bonaglia S, Norkko A, et al. High throughput shotgun sequencing of eRNA reveals taxonomic and derived functional shifts across a benthic productivity gradient[J]. Molecular Ecology, 2021, 30(13): 3023-3039.
[59] Kitahashi T, Sugime S, Inomata K, et al. Meiofaunal diversity at a seamount in the pacific ocean: A comprehensive study using environmental DNA and RNA[J]. Deep Sea Research Part I: Oceanographic Research Papers, 2020, 160: 103253.
[60] Iburg S, Nybom I, Bonaglia S, et al. Organic contaminant mixture significantly changes microbenthic community structure and increases the expression of PAH degradation genes[J]. Frontiers in Environmental Science, 2020, 8: 128.
[61] Huang P P, Zhao F, Xu K D, et al. Are marine benthic microeukaryotes different from macrobenthos in terms of regional geographical distribution? New insights revealed by RNA metabarcoding[J]. Continental Shelf Research, 2020, 209: 104255.
[62] Lejzerowicz F, Gooday A J, Angeles I B, et al. Eukaryotic biodiversity and spatial patterns in the Clarion-Clipperton Zone and other abyssal regions: Insights from sediment DNA and RNA metabarcoding[J]. Frontiers in Marine Science, 2021, 8: 671033.
[63] Pearman J K, Biessy L, Howarth J D, et al. Deciphering the molecular signal from past and alive bacterial communities in aquatic sedimentary archives[J]. Molecular Ecology Resources, 2022, 22(3): 877-890.
[64] Greco M, Lejzerowicz F, Reo E, et al. Environmental RNA outperforms eDNA metabarcoding in assessing impact of marine pollution: A chromium-spiked mesocosm test[J]. Chemosphere, 2022, 298: 134239.
[65] Miyata K, Inoue Y, Amano Y, et al. Fish environmental RNA enables precise ecological surveys with high positive predictivity[J]. Ecological Indicators, 2021, 128: 107796.
[66] Hirai J, Hidaka K, Nagai S, et al. DNA/RNA metabarcoding and morphological analysis of epipelagic copepod communities in the Izu Ridge off the southern coast of Japan[J]. Ices Journal of Marine Science, 2021, 78(9): 3444-3456.
[67] Brand S C, Jeffs A G, Von Ammon U, et al. Assessing the presence, settlement and growth of the invasive Mediterranean fanworm,, on mussel farms[J]. Journal of Experimental Marine Biology and Ecology, 2022, 554: 151767.
[68] 刁曹鋆, 王闻, 线薇薇, 等. 环境DNA技术在渔业资源生物量评估中的研究进展: 现状与展望[J]. 海洋科学, 2022, 46(2): 135-144. DIAO Caoyun, WANG Wen, XIAN Weiwei, et al. Role of the environmental DNA technology application in the biomass assessment of the fishery resource: current status and future perpectives[J]. Marine Sciences, 2022, 46(2): 135-144.
[69] 张辉, 线薇薇. 环境DNA在生态保护和监测中的应用[J]. 海洋科学, 2020, 44(7): 96-102. ZHANG Hui, XIAN Weiwei. Application of environmental DNA technology in ecological conservation and monitoring[J]. Marine Sciences, 2020, 44(7): 96-102.
[70] Bowers H A, Pochon X, Von Ammon U, et al. Towards the optimization of eDNA/eRNA sampling technologies for marine biosecurity surveillance[J]. Water, 2021, 13(8): 1113.
Application of environmental RNA technology in biodiversity monitoring of the aquatic ecosystem
LI Wen-qiong1, 3, DAI Shu-qin1, ZHANG Hui1, 2, 3, XIAN Wei-wei1, 2
(1. CAS Key Laboratory of Marine Ecology and Environmental Sciences, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China; 2. Laboratory for Marine Ecology and Environmental Science, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China; 3. University of Chinese Academy of Sciences, Beijing 100049, China)
The continuous increase in human activities worldwide has caused a loss of aquatic biodiversity and a dramatic change in the aquatic community structure. Therefore, accurate and reliable biodiversity monitoring is essential for ecological conservation and sustainable resource use to determine species abundance and community structure in target areas. Environmental DNA (eDNA) technology is a new and noninvasive monitoring method that is rapidly developing. However, eDNA technology is prone to false positives, which lead to inaccurate real-time biodiversity monitoring that is still widespread. Owing to its rapid degradation, environmental RNA (eRNA) is potentially less prone to false positives than eDNA. This paper summarized the feasibility of eRNA in biodiversity monitoring of the aquatic ecosystem, primarily focusing on its detectability and potential to improve detection resolution in the aquatic ecosystem. Furthermore, the current research progress in the field of eRNA was also summarized. Finally, the current challenges associated with using eRNA were discussed, and a possible future research direction was proposed. This paper can provide a valuable reference for the practical application of eRNA in biodiversity monitoring, ecological conservation, and sustainable use of resources in aquatic ecosystems in the future.
environmental RNA; biodiversity, ecological monitoring; ecological conservation
Nov. 1, 2022
[National Key Research and Development Project of China, No. 2022YFE0112800; National Natural Science Foundation of China, No. 42111540159; Youth Innovation Promotion Association CAS, No. 2020211]
P735
A
1000-3096(2023)8-0090-11
10.11759/hykx20221101002
2022-11-01;
2022-12-02
国家重点研发计划项目(2022YFE0112800); 国家自然科学基金(42111540159); 中国科学院青年创新促进会(2020211)
李文琼(1999—), 女, 山东泰安人, 硕士研究生, 主要研究方向为渔业生态, E-mail: liwenqiong@qdio.ac.cn; 张辉(1986—), 通信作者, 男, 山东安丘人, 研究员, 主要研究方向为渔业生态, E-mail: zhanghui@qdio.ac.cn
(本文编辑: 赵卫红)
