计算机虚拟筛选技术在害虫行为调节剂研究中的应用
2023-09-25付冠军周亚楠刘万学钱万强万方浩
付冠军, 黄 聪, 周亚楠, 刘 博, 刘万学, 钱万强, 万方浩*
1中国农业科学院农业基因组研究所,广东 深圳 518120; 2青岛农业大学植物医学学院,山东 青岛 266109; 3中国农业科学院植物保护研究所植物病虫害生物学国家重点实验室,北京 100193
自然环境中存在十分复杂的化学信息,昆虫能够利用其敏锐的嗅觉系统感受环境中的各种气味信息,并对其做出相应的生理响应和行为反应,例如觅食、求偶、寄主定位、躲避天敌和定位产卵场所等(娄永根和程家安,2001; 王桂荣等,2004)。利用气味信息干扰昆虫正常的行为活动是害虫绿色防控的手段之一,与化学农药相比,以气味化合物为活性物质的昆虫行为调节剂具有环境友好及抗性发展缓慢等特点(杨斌等,2020)。目前,昆虫行为调节剂活性物质的来源主要包括昆虫自身通过腺体释放的信息素以及寄主植物所释放的化合物(闫凤鸣等,2013)。明确对昆虫具有行为调控活性的化合物是昆虫行为调节剂开发的关键,过去实现这一目的主要借助于一系列生物测定的方法,例如:利用气相色谱质谱联用技术(gas chromatograph-mass spectrometer, GC-MS)进行昆虫生境中化学信息的成分鉴定;气相色谱触角电位联用(gas chromatography-electro antenna graphic detection, GC-EAD)、触角电位反应(electro antenna graphy, EAG)等技术验证昆虫对特定化合物刺激是否产生电生理响应;Y型嗅觉仪等行为活性测试装置验证目标化合物对昆虫行为的影响(尹海辰等, 2020);爪蟾卵母细胞、人类胚胎肾细胞以及果蝇空神经元等异源表达系统(Bengtssonetal.,2014; Grosse-Wildeetal.,2007; Liuetal.,2014; Sakuraietal.,2004)或RNAi、CRISPR/Cas9等基因编辑技术(Chenetal.,2020; Guoetal.,2020)来确定活性成分及其作用的分子靶标。然而,由于昆虫生境中的化学信息种类繁多,加上生物测定方法工作流程繁琐、耗时耗力,亟需发展一种新的方法并辅助传统方法进行活性化合物的筛选,进而提高昆虫行为调节剂先导化合物的筛选效率。
随着计算机技术的迅速发展,基于计算机的药物虚拟筛选为实现高通量筛选提供了可靠的解决方案,显著提高了先导化合物的发现效率(刘轲等,2018; 刘润哲等,2021)。近年来,昆虫嗅觉分子机制研究的不断深入、昆虫嗅觉信息数据化程度的不断提高,使得通过计算机进行昆虫行为调节剂先导化合物筛选成为可能,极大地推动了昆虫行为调节剂开发的进程。目前,基于计算机的昆虫行为调节剂虚拟筛选主要有以下2种策略(刘润哲等,2021):一是以理论计算为依据,基于配体的虚拟筛选,其设计思路是通过计算分析已知活性化合物的分子特征,采用数理统计方法揭示化合物分子特征与其活性之间的定量变化规律,即定量结构-活性关系(quantitative structure-activity relationship, QSAR);二是以分子对接为理论,基于结构的虚拟筛选,其设计思想是通过计算机模拟蛋白质与小分子的结合模式,根据蛋白质与配体结合特征和结合自由能判断结合强度。
在昆虫嗅觉识别过程中,气味结合蛋白(odorant binding proteins, OBP)负责将进入嗅觉感器的气味化合物分子运输至嗅觉感受神经元表面的气味受体(olfactory receptors, OR)和气味共受体(odorant receptor coreceptors, Orco)组成的复合体中,从而实现对气味分子的特异性识别(李慧等,2021; Zhaoetal.,2018)。因此,OBP与OR家族在昆虫与化学信息相互作用的研究中被重点关注。OBP为水溶性蛋白,相比于跨膜蛋白OR更容易获得晶体结构(Venthur &Zhou,2018),在先前的昆虫行为调节剂虚拟筛选研究中,OBP的潜在配体的预测常采用基于结构的筛选方法(李敏等,2019),OR气味反应谱的预测则多采用基于配体的虚拟筛选策略。
1 基于配体的昆虫行为调节剂虚拟筛选
基于配体的虚拟筛选策略以一系列通过实验确定的嗅觉数据为基础,通过建立能够正确拟合配体分子与受体蛋白结合活性关系的QSAR模型从化合物库中高通量筛选潜在的活性化合物(刘轲等,2018)。其一般流程如图1所示:第一步,数据获取,研究人员需针对不同的任务从文献或专业数据库中收集相应的嗅觉数据信息(Sharmaetal.,2022),其数量和质量往往直接影响模型的预测性能(Affonsoetal., 2013),因此,收集的数据尽可能全面、准确;第二步,数据转换,由于获取的嗅觉信息一般是受体和其对应的配体,计算机无法直接处理这类数据,因此,需要将受体和配体的理化特性转换为计算机可读的数据形式,且尽可能完整、有效地代表固有信息;第三步,模型训练与评估,对转化后的数据划分训练集和测试集,利用机器学习算法对训练集进行训练构建受体和配体分子的定量构效关系,并利用测试集评估模型质量;第四步,化合物筛选,训练获得高质量的预测模型之后,即可利用模型预测未知受体与配体的结合能力(刘润哲等,2021)。
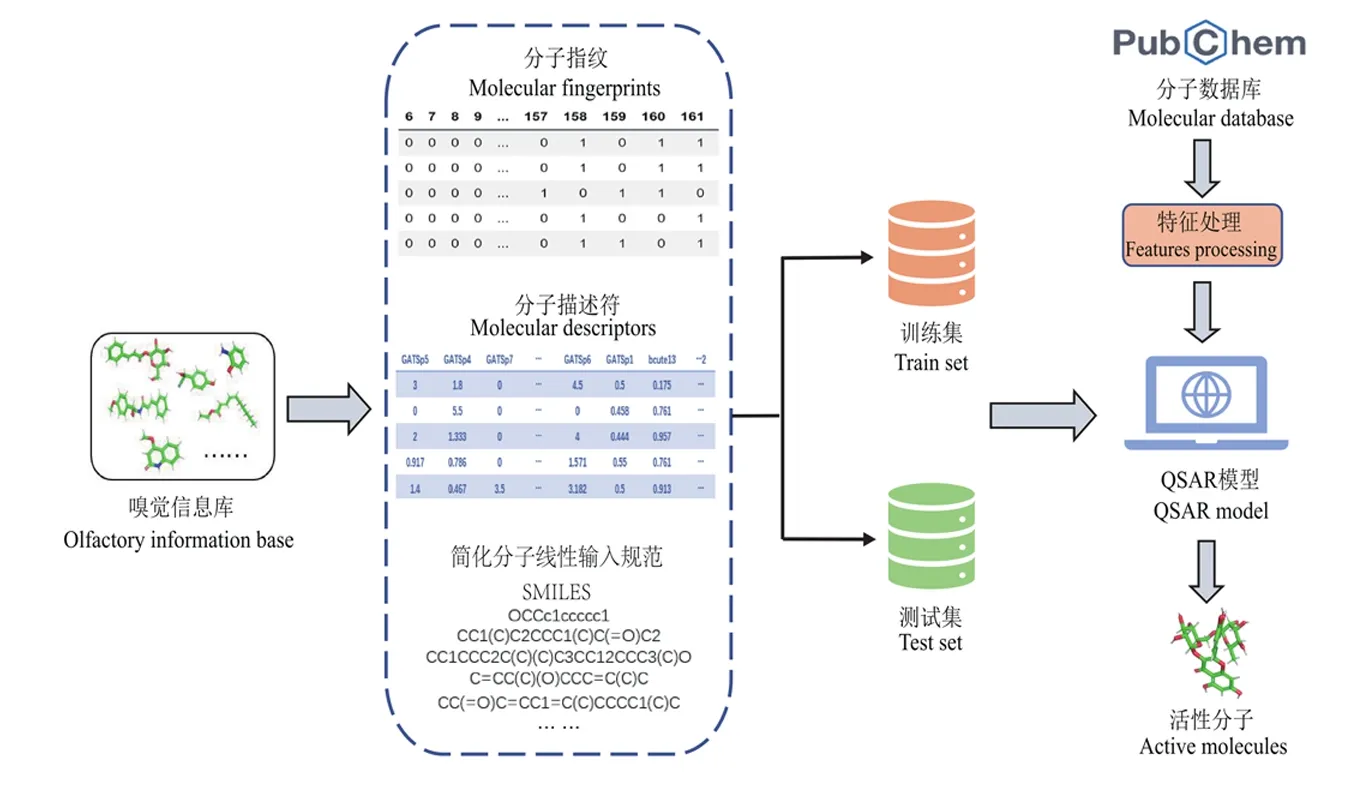
图1 基于配体的药物筛选基本流程
1.1 数据获取与处理
获取嗅觉受体的反应谱数据是开发QSAR模型的关键,数据来源有以下2个方面:1)嗅觉信息数据库,如DoOR (http:∥neuro.uni-konstanz.de/DoOR)数据库系统收录了黑腹果蝇Drosophilamelanogaster几乎所有的气味受体反应谱数据,涉及693种气味化合物(Munch &Galizia,2016);2)大量未收录在专业数据库但已报道的嗅觉反应数据(Chenetal.,2020; Guoetal.,2021; Miuraetal.,2010)。
分子特征表示作为分子化学信息的数学表示使得计算机对化学信息的处理更为方便,常用分子特征表示方法主要有分子描述符、分子指纹、简化分子线性输入规范(simplified molecular input line entry system, SMILES),在QSAR研究中以分子描述符和分子指纹较为常见。Mordred (Moriwakietal.,2018)、PaDEL-Descriptor (Yap,2011)、CDK (Steinbecketal.,2003)、RDKit、Dragon和alvaDesc (Mauri,2020)等工具都可实现由分子化学信息向其数学表示的转换,尽管在数量和表示类型上不尽相同,但其最终目标都是尽可能有效、全面地提供分子特征的数学表示,几种常见计算工具及其提供的分子特征数量见表1。然而,上述工具中部分存在软件配置困难或停止维护的情况,开源在线服务平台ChemDes (http:∥www.scbdd.com/chemdes)集成了多个分子特征计算工具,例如Chemopy、CDK、RDKit等,用户只需选择对应的计算工具并提交SMILES信息或上传分子结构文件即可得到分子特征,大大减少软件配置等问题(Dongetal.,2015)。

表1 分子描述符计算工具
1.2 机器学习与虚拟筛选
机器学习是人工智能的重要分支,能够通过特定算法拟合真实数据形成对人类经验的模仿,算法作为机器学习的核心对于“经验模仿”具有重要意义。Scikit-learn、PyTorch、TensorFlow (刘润哲等,2021)等开源科学计算库的发展,大大降低了编程难度,极大方便了科研人员对计算生物学的应用。根据算法处理数据机制的不同,机器学习算法分为传统机器学习算法和深度学习算法,相比于传统机器学习算法,深度学习算法通过模拟人脑的神经机制强调模型对数据更深入的理解。
人们通过异源表达系统研究明确了大量极具价值的嗅觉编码信息,为利用机器学习方法研究嗅觉受体的化学感受谱提供基础的数据支撑。例如,Kepchiaetal.(2019)将冈比亚按蚊AnophelesgambiaeGiles气味共受体ORco的83个配体结合数据(58个拮抗剂和25个非拮抗剂)的拓展连接指纹(extended connectivity fingerprints, ECFP)作为分子特征输入,并选择朴素贝叶斯分类算法(naive bayesian)对输入的数据集进行拟合,在对1280种气味分子进行虚拟筛选中发现了一种具有全新结构的气味分子2-tert-butyl-6-methylphenol (BMP)与ORco具有较好的结合活性,进一步利用电生理实验验证了BMP的生物学效应,为ORco拮抗剂的开发提供了新的方法。然而,受限于高度差异的昆虫气味受体以及嗅觉数据解析程度,基于配体的昆虫行为调节剂筛选策略主要应用于双翅目(Boyleetal.,2013,2016; Guptaetal.,2021; Oliferenkoetal.,2013; Tauxeetal.,2013),2020年,Caballero-Vidaletal.(2020)首次将这一策略扩展到鳞翅目昆虫海灰翅夜蛾Spodopteralittoralis(Boisduval)的气味受体功能研究中,应用支持向量机(support vector machine, SVM)算法对气味受体SlitOR25的气味反应数据(13个活性数据和39个非活性数据)的分子特征和标签进行拟合,对3306282个分子进行虚拟筛选以发现其全新的配体,选择32种具有潜在活性的化合物进行功能验证,揭示了11种具有生物学活性的配体分子。
2 基于结构的昆虫行为调节剂虚拟筛选
在结构生物学的推动下,研究人员通过特定的算法解析蛋白受体与配体分子在原子水平上的相互作用方式来推动先导化合物的发现及筛选效率(刘轲等,2018)。基于结构的虚拟筛选依赖于靶标蛋白和配体分子的三维结构,通过空间匹配和能量优化分析活性位点、预测受体和配体结合的优势构象,从而确定与受体蛋白具有结合能力的潜在配体。因此,基于结构的筛选主要是通过分子对接技术的数据库搜寻,即将数据库中的分子依次对接到靶标蛋白的活性口袋,通过构象搜索算法不断优化分子与受体氨基酸侧链、骨架结合的构象,并结合打分函数评估结合亲和性(图2)。

图2 基于结构的药物筛选流程
2.1 受体和配体的三维结构
随着后基因组时代的到来,结构生物学已成为当下的研究热点,蛋白质作为生命过程发挥生物学功能的主要大分子物质被广泛关注。核磁共振技术(nuclear magnetic resonance)、X 射线晶体衍射法(X ray crystalline diffraction)以及冷冻电镜技术(cryo-electron microscopy)作为解析蛋白质三维结构的主要手段(徐颢溪和刘磊,2020),奠定了结构生物学研究的基础,目前已有191565个蛋白质结构数据收纳于PDB数据库(https:∥www.rcsb.org)。尽管如此,随着高通量测序技术的发展,蛋白质序列呈井喷式增加,通过实验解析蛋白晶体结构已无法满足当前科研发展的需求,科学家将目光逐渐移向基于序列信息的蛋白质结构预测(Dengetal. 2016),经过半个多世纪的发展,以Swiss-model、Modeller和I-Tasser为代表的同源建模和以AlphaFold2、Rosetta为代表的从头建模策略均在蛋白质结构预测的准确性和精度方面取得了巨大成功。值得一提的是,AlphaFold2在2020年第14届国际蛋白质结构预测评估大赛(Critical Assessment of Techniques for Protein Structure Prediction, CASP)中斩获头筹(刘子楠等,2020)。
受体蛋白与配体的结合能力决定了昆虫对气味配体的识别,是引起昆虫行为变化的主要原因之一,因此,在分子对接研究中,获得精确的受体蛋白和配体分子的三维结构同等重要。相较于受体蛋白的三维结构,配体小分子的三维结构更易获得,如由美国国立卫生研究院(National Institutes of Health, NIH)开发的化学分子数据库ChemBank (https:∥pubchemdocs.ncbi.nlm.nih.gov/)存储了1607755个分子的结构信息以供用户免费下载,由美国加州大学旧金山分校(University of California, San Francisco, UCSF)药物化学系的Irwin和Shoichet实验室提供和维护支持的ZINC (https:∥zinc15.docking.org/)包含2.3亿个市场上可购买的化合物3D结构。
2.2 分子对接与虚拟筛选
自1894年Fischer提出蛋白和配体结合的“锁-钥模型”以来,分子对接技术发展迅速,目前,已有超过60种分子对接工具,例如:Dock、AutoDock、Gold、Glide、Moe Dock、AutoDock Vina等(Pagadalaetal.,2017)。基于结构虚拟筛选由于不受目的嗅觉蛋白化学感受范围解析程度的影响,应用更为广泛。例如:Thireouetal. (2018)在筛选冈比亚按蚊新型趋避剂的研究中,依据性状相似和化学相似性对42755个合成分子进行筛选,确定了33个与已知驱蚊剂相似的分子,使用AutoDock软件的拉马克遗传算法(Lamarckian genetic algorithm, LGA)将其与AgamOBP1晶体结构进行对接构象搜索,选择对接结果较好的16个分子对其进行功能验证,其中4个化合物能与AgamOBP1结合且具有趋避活性。Yietal. (2015)对斜纹夜蛾SpodopteralituraFabricius气味结合蛋白OBP1进行同源建模,并在CHARMM力场下对其初始结构分别执行梯度下降(steepest descend)和共轭梯度(conjugate gradient)能量最小化程序检查模型质量,在对接筛选中,先使用Ligandfit工具的蒙特卡罗法和能量网格对Available Chemicals Directory 数据库中的1536550种化学物质进行首轮对接,随后用Cdocker工具进行二次对接,并根据PLP1、Ligscore1 2种评分函数对复合物结合稳定性进行排序,选择对接合理的2040个化合物中的21个具有不同结构和结合模式的化合物进行荧光竞争结合分析,其中15个化合物均与OBP1具有良好的结合活性。分子对接研究在昆虫行为调节剂虚拟筛选研究中涉及双翅目、膜翅目、半翅目、鳞翅目、鞘翅目等类群,如表2所示。
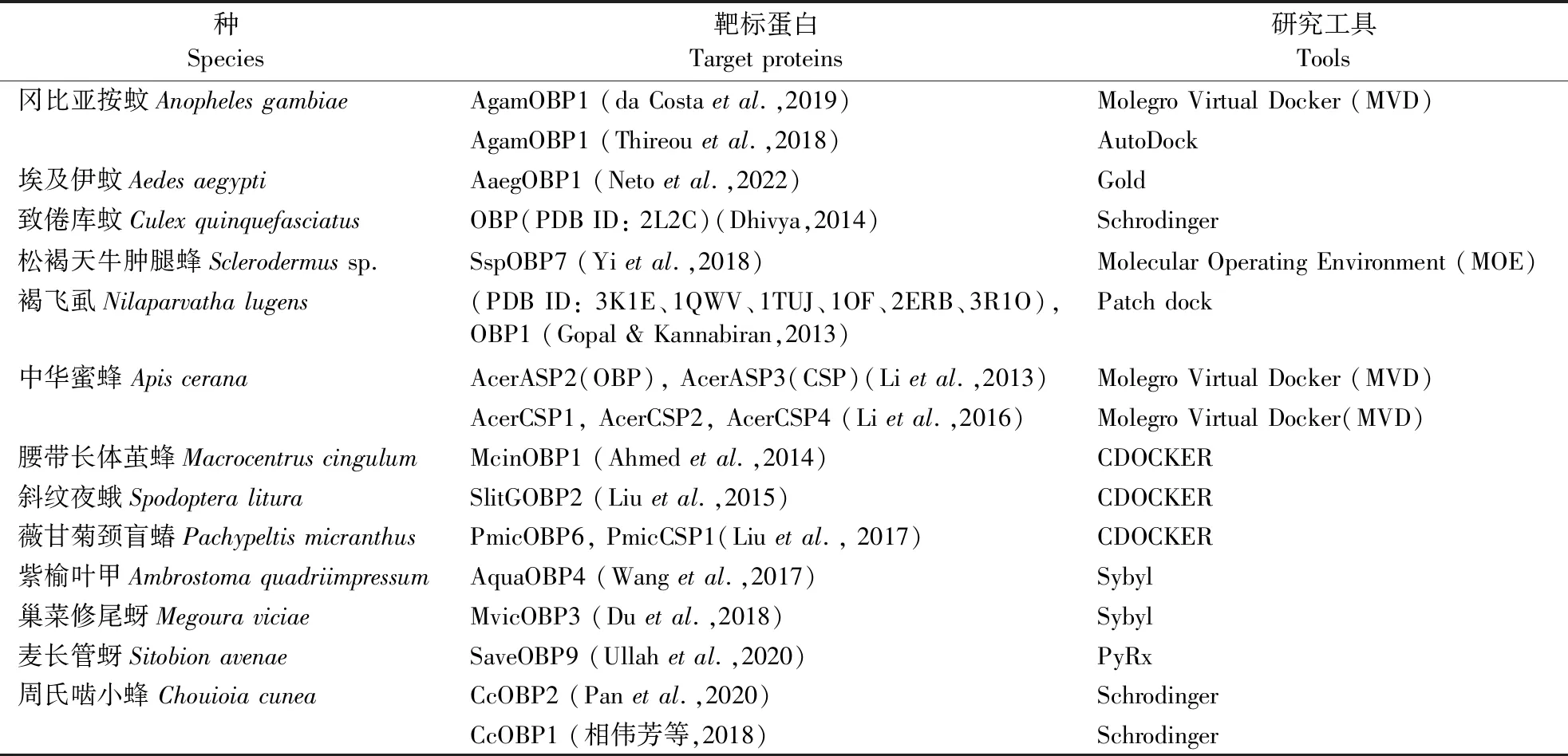
表2 分子对接在昆虫行为调节剂研究中的应用
3 讨论与展望
基于配体和基于结构的2种虚拟筛选都能促进先导化合物的发现进程,然而2种方法各有优劣,基于配体的虚拟筛选要求具备一定的编程基础,研究门槛相对较高,且模型开发过程繁琐,从模型设计到模型应用的时间较长,但运行速度较快,拟合好模型之后可短时间内完成对海量化合物的预测任务;基于结构的虚拟筛选往往借助于可视化的研究工具,研究门槛相对较低,能够在短时间内快速上手应用,但运行速度远低于基于配体的筛选方法。
在昆虫的行为调节剂的先导化合物发现研究中,基于配体的虚拟筛选:1)侧重于广谱性嗅觉受体蛋白,且严重依赖于嗅觉编码解析程度,无法对调谐范围较窄以及功能不明确的嗅觉受体蛋白进行研究;2)现有的嗅觉反应数据仅涉及与昆虫生境关系密切的气味化合物分子,仅代表了自然界中庞大且复杂化学信息库中的一小部分,且未经生物学功能测试的化合物无法对其是否具有活性做出定论,因此,在一定程度上限制了模型训练数据的多样性和模型泛化能力;3)仅有少数模式物种的嗅觉反应信息以高度集中的数据库形式存在,因此,对于非模式物种的气味反应数据,需要研究人员花费大量的时间和精力从海量文献中收集获得。ODORactor 数据库提供了大量哺乳动物的OR反应谱信息,并以此开发了小分子配体预测功能(Liuetal.,2011),极大程度上促进了大数据的利用。随着昆虫化学生态领域数据化研究的不断深入,构建嗅觉反应数据库和利用计算生物学的方法预测嗅觉反应谱是未来研究受体与配体结合关系的趋势,对掌握昆虫的化学感受谱以及应用具有深远意义。
基于结构的虚拟筛选:1)对接打分函数能够直接影响研究人员对结果的分析判断,然而现有的评分函数对蛋白受体与配体的结合能力评价准确性仍有待提高,在未来很长一段时间,需要依赖大量实验数据对评分函数进行优化,进一步提高对接结果在正常生理状态下的表现能力及其评估精度;2)分子对接的准确与否与蛋白三维结构的精度密切相关,蛋白建模虽然在一定程度上弥补了通过实验方法解析蛋白晶体结构的短板,但仍有部分蛋白质的结构预测达不到应用的标准(刘子楠等,2020);3)计算机模拟无法完全体现生物大分子与小分子在正常生理状态下的结合模式,因此,分子对接的准确性一直备受关注,完成分子对接后往往需要人们进一步基于先验知识进行判断,或利用微量热涌动、荧光竞争结合等手段验证其准确性(李敏等,2019; 刘航玮等,2017),尽管如此,该技术仍然大大降低了筛选的盲目性。随着实验数据丰度的不断上升和算法不断优化,分子对接的准确性将会进一步提高,基于结构的昆虫行为调节剂研究将会进一步发展。
