胰岛素分泌细胞诱导过程中miRNA-mRNA 调控网络的生物信息学分析
2023-09-20王涛,潘鑫
王 涛,潘 鑫
(锦州医科大学医疗学院医学系,辽宁 锦州 121013)
miRNA 作为一类小的非编码RNA,其长度大约在22 个核苷酸左右,能够与靶mRNA 3'端非翻译区互补,进而降解或阻止该mRNA 翻译出蛋白质,实现转录后水平的基因表达调控[1]。糖尿病作为一种慢性疾病,部分是由胰岛β 细胞受损而引起,胰腺或胰岛移植对于该类疾病治疗效果较好,但是由于供体缺乏以及免疫排斥导致临床应用受限[2,3],而干细胞分化为胰岛素分泌细胞(insulin-producing cells,IPCs)再行移植,有望彻底根治[4,5]。生物信息学通过整合多个学科领域信息和知识,能够从整体层面揭示疾病或生物过程的复杂分子机制[6-8]。本研究运用生物信息学方法分析人胚胎干细胞(human embryonic stem cells,hESCs)向IPCs 诱导分化过程中mRNA 的表达谱,预测靶向作用于该mRNA 的miRNA,构建miRNA-mRNA 调控网络。通过对调控网络的分析,拟找到IPCs 诱导分化的关键节点,为进一步开展分子靶向干预提供新思路。
1 资料与方法
1.1 资料来源 选择GEO(https://www.ncbi.nlm.nih.gov/geo/)数据库内的GSE42094 数据集作为研究对象,该数据集包含23 个样本数据(GSM1032319-41),涉及未分化hESCs、IPCs 分化的5 个时期、胰腺内胰岛和胎胰。选择未分化hESCs(hESCs 组)和IPCs 分化的5 个时期(Diff1、Diff2、Diff3、Diff4 和IPCs 组)研究mRNA 的表达谱。应用数据库自带软件GEO2R对6 组数据进行分析,获取差异表达的mRNA。
1.2 GO 功能和KEGG 路径分析 选用DAVID(https://david.ncifcrf.gov/tools.jsp)数据库进行功能富集分析,主要分析GO 功能以及KEGG 路径,以P<0.05为差异有统计学意义。
1.3 蛋白互作网络分析 选用STRING(https://stringdb.org/)数据库进行蛋白互作网络(protein-protein interaction,PPI)分析,选择Homo sapiens 作为“Organism”的条件,其余参数均为系统默认设置。
1.4 miRNA 预测 针对差异表达基因预测其靶miRNA,选用miRTarBase(https://mirtarbase.cuhk.edu.cn/)数据库,选择reporter assay、western blot、qPCR 和microarray4 种实验验证的miRNA-mRNA靶向匹配作为预测miRNA 的入选标准,并应用Cytoscape 3.9.1 软件对miRNA-mRNA 的调控网络进行可视化。
1.5 预测miRNA 的验证 将预测的miRNA 与PMID:20735361[9]的Table S4 数据取交集,分析miRNA 在人胚胎T3 干细胞向胰腺胰岛样细胞团分化过程中的表达,其Table S4 数据显示出胚胎T3 干细胞(hES-T3 cells grown on mouse embryonic fibroblast feeder,T3ES)、胚胎内胚层(embryoid bodies differentiated from T3 cells,T3EB)和胰腺胰岛样细胞团(pancreatic islet-like cell clusters derived from T3 cells,T3pi)3 组标准化的miRNA 表达量。
1.6 转录因子-靶基因调控网络的构建 针对验证的miRNA 以及显著下调基因查找调控其表达的转录因子,选用FunRich 3.1.3 软件分析转录因子,以P<0.05 为差异有统计学意义,并作为转录因子的入选标准,同时应用Cytoscape 3.9.1 插件iRegulon 预测转录因子,将二者分别与Mahmoud H 等[10]报道的β细胞分化相关的重要转录因子取交集,对转录因子-靶基因调控网络可视化。
2 结果
2.1 差异及因及其GO 功能和KEGG 路径分析 共得到差异表达的基因188 个,选取诱导后显著下调的基因共21 个(表1),对其进行GO 功能和KEGG路径分析,发现下调基因中POU5F1、DNMT3B、NANOG 和UCA1 集中在基因表达调控,POU5F1 和NANOG 与内胚层的决定以及成体干细胞种群维持有关,PPP1R16B 和PPP2R2C 与蛋白磷酸化调节活性有关。DAVID 数据库进行KEGG 路径显著性分析并没有找到有统计学意义的信号通路。
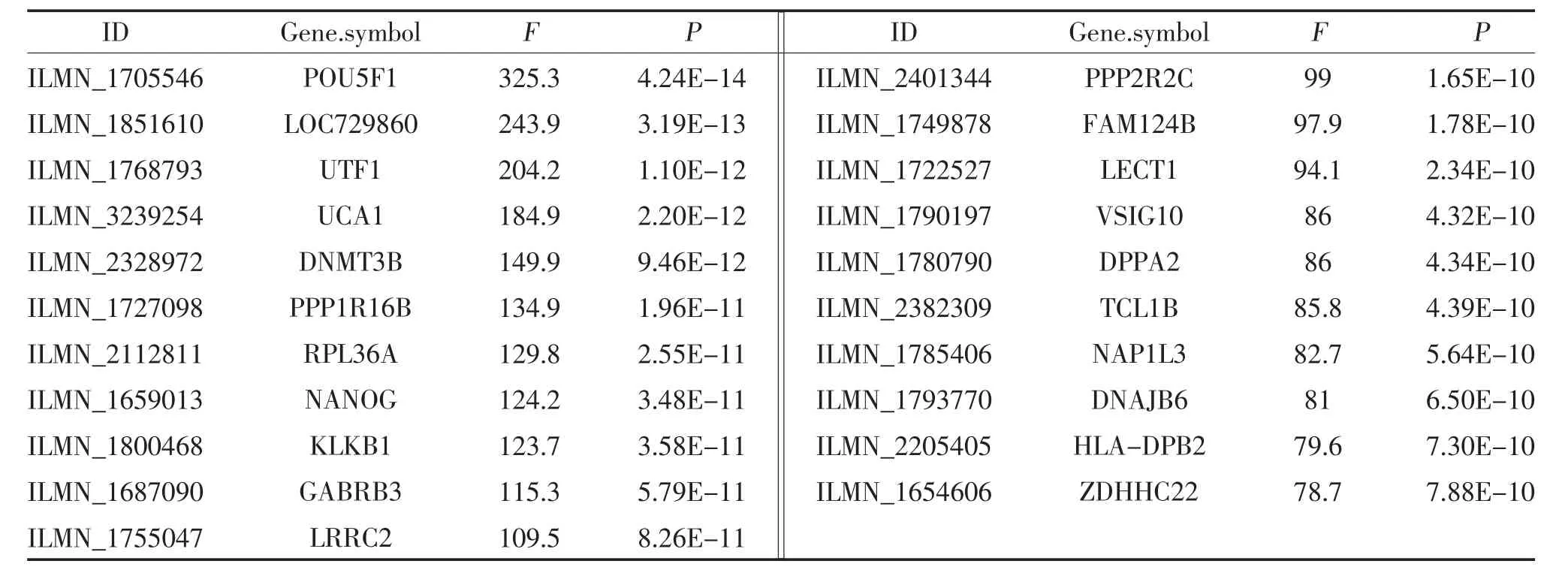
表1 表达下调的差异基因
2.2 蛋白互作网络分析 通过PPI 分析21 个诱导后显著下调的基因,发现LOC729860、UCA1 和HLADPB2 未被STRING 数据库识别,因此该网络有18个节点,14 条边,其PPI 富集P值为1.0e-09,差异有统计学意义。进一步分析发现POU5F1、UTF1、DNMT3B、NANOG、GABRB3、DPPA2 和TCL1B 形成互作网络,剩余11 个蛋白相互之间没有关联,见图1。
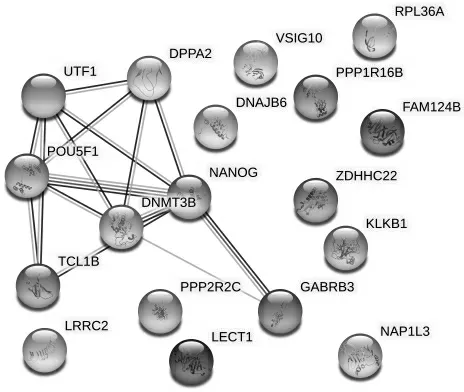
图1 蛋白互作网络分析
2.3 miRNA-mRNA 调控网络 通过miRTarBase 数据库查找靶向作用于显著下调基因的miRNA,选择reporter assay、western blot、qPCR 和microarray 4 种实验验证的miRNA-mRNA 靶向匹配作为预测miRNA 的入选标准,共得到25 个预测的miRNA,应用Cytoscape 3.9.1 软件对miRNA-mRNA 调控网络进行可视化(图2),发现PPI 网络的7 个关联蛋白也处于该调控网络的核心区域,预测的大部分miRNA 围绕其展开,其中,miR-335-5p 能够调控POU5F1、DNMT3B、NANOG、FAM124B 和LECT1 的表达,进一步在GenomeNet 数据库(https://www.genome.jp/pathway/hsa04550+5460)中对这5 个基因进行KEGG 路径分析,发现POU5F1 和NANOG 处于干细胞多能性调控信号通路(hsa04550)之中,调控干细胞的自我更新。
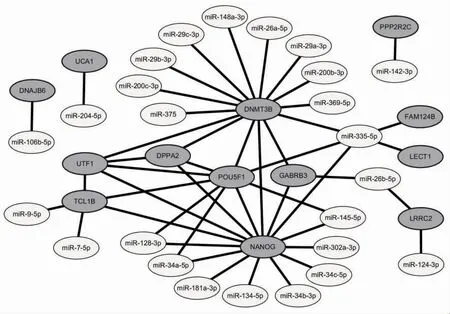
图2 miRNA-mRNA 调控网络
2.4 预测miRNA 的验证 将预测的miRNA 与PMID:20735361 的Table S4 数据取交集,得到24 个验证的miRNA,只有1 个预测的miRNA(miR-200b-3p)未被验证,大部分预测的miRNA 得到验证,即存在于干细胞及其诱导分化的过程中,表明这种基于4 种实验预测miRNA 的方法具备一定的可信度(表2)。表2 显示miR-26b-5p、miR-106b-5p、miR-200c-3p、miR-148a-3p、miR-375、miR-204-5p、miR-7-5p、miR-302a-3p 和miR-124-3p 表达量均在胚胎内胚层阶段(T3EB)显著升高而又在胰腺胰岛样细胞团阶段(T3pi)显著降低,miR-26a-5p、miR-335-5p 和miR-134-5p 表达量随着诱导进程逐渐升高。这两类miRNA 的表达特点与显著下调基因表达量在T3EB阶段后显著降低形成对照,提示二者之间存在负性调控。结合图2 分析发现miR-26b-5p 和miR-335-5p 将miRNA-mRNA 调控网络核心区域的7 个基因往外延伸,其中,LRRC2 通过miR-26b-5p、FAM124B 和LECT1 通过miR-335-5p 与核心区域关联。进一步将仅通过microarray 实验预测的miRNA 剔除后作调控网络图(图3),发现大部分miRNA 主要调控POU5F1、DNMT3B 和NANOG 的表达,该部分miRNA 是通过reporter assay、western blot 和qPCR 实验验证了其与靶基因的匹配关系,其中,miR-200c-3p、miR-148a-3p、miR-204-5p、miR-302a-3p、miR-26a-5p、miR-134-5p 和miR-335-5p与其靶基因表达模式存在负性调控,可得到进一步验证,miR-369-5p 也被上述3 种实验所验证,但是其在整个诱导过程中表达量未发生变化。结合表2发现,仅通过microarray 实验预测的miRNA,如miR-26b-5p、miR-106b-5p、miR-375、miR-7-5p 和miR-124-3p 与其靶基因的表达模式存在负性关系,或可佐证该部分miRNA 与其靶基因的靶向调控。
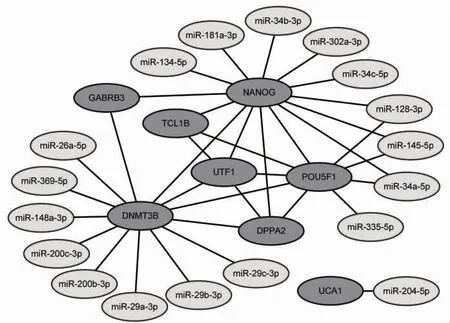
图3 3 种方法预测的调控网络
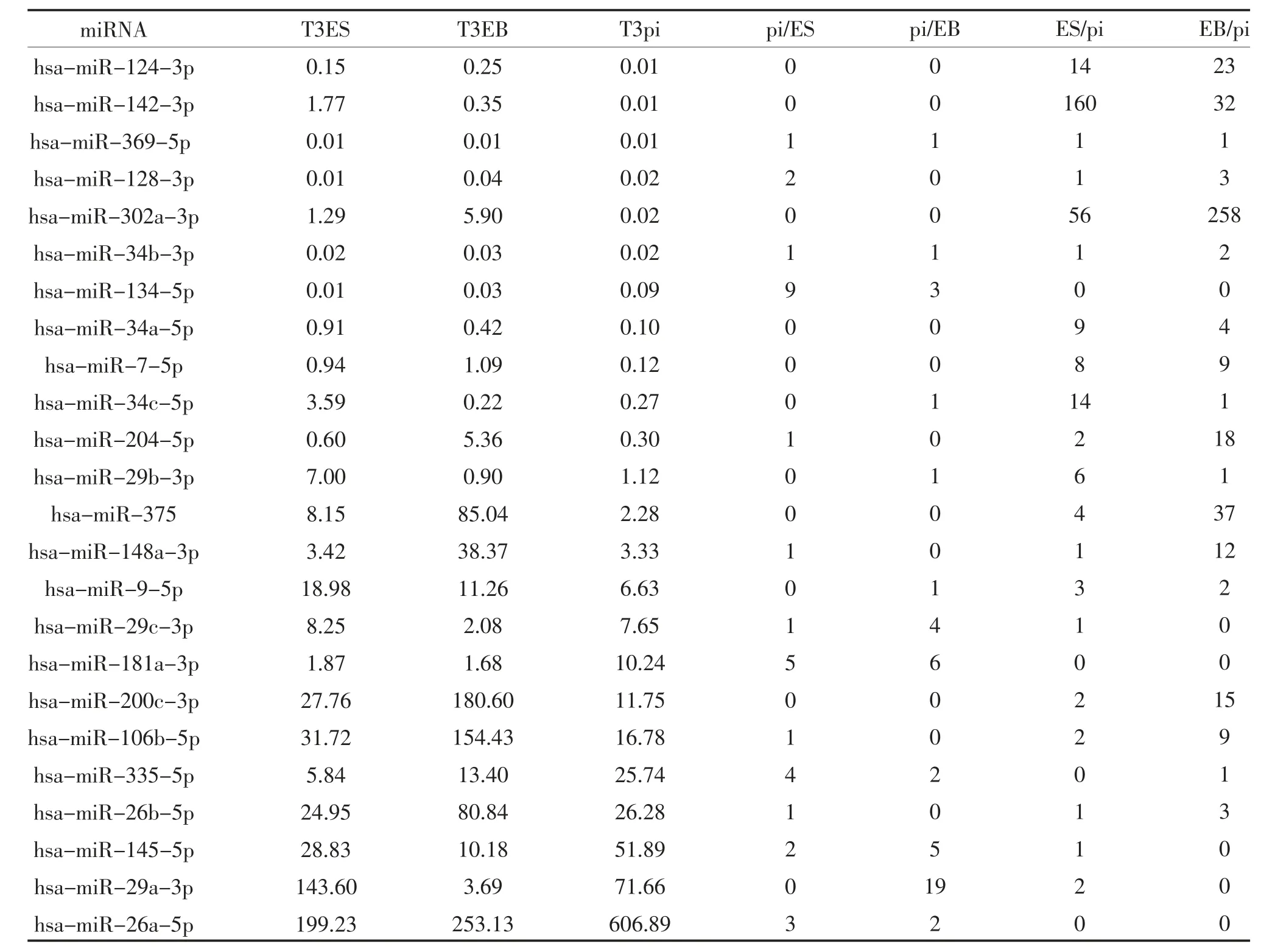
表2 PMID:20735361 结果验证预测的miRNA
2.5 显著下调基因的转录因子 针对21 个显著下调基因查找调控其表达的转录因子,FunRich 软件没有得到有统计学意义的转录因子,通过iRegulon 分析得到49 个转录因子,将其与188 个差异表达基因取交集,得到POU5F1、NANOG 和TFAP2A 3 个转录因子,并应用Cytoscape 可视化(图4A),发现POU5F1 和NANOG 本身即为显著下调基因,其表达量见图4B 和4C,其中NANOG 调控9 个基因表达,包括其自身亦即存在顺式作用调控,POU5F1 调控6个基因表达,也存在顺式作用调控,且二者之间互为对方转录因子,存在相互转录调控现象,而TFAP2A调控6 个基因表达,其中包含POU5F1 和NANOG基因。ZDHHC22、PPP1R16B 和GABRB3 3 个基因同时受上述3 个转录因子的调控,结合POU5F1 和NANOG 基因表达量显著下降的变化,可以理解该部分显著下调基因主要存在转录水平的调控。进一步将iRegulon 分析得到的49 个转录因子与Mahmoud H 等[10]报道的β 细胞分化相关的重要转录因子取交集,得到ONECUT1(HNF6)、NEUROD1和NKX2-2 3 个转录因子(图4D),发现ONECUT1能够调控8 个基因表达,其中包括NANOG 基因,NEUROD1 与 ONECUT1 共同调控 PPP2R2C、ZDHHC22 和LECT1 3 个基因,而LECT1 基因也受NKX2-2 转录因子调控,可见该部分基因除受转录水平调控外还存在较强的转录后水平调控,以至于其表达量在分化后期显著下降。
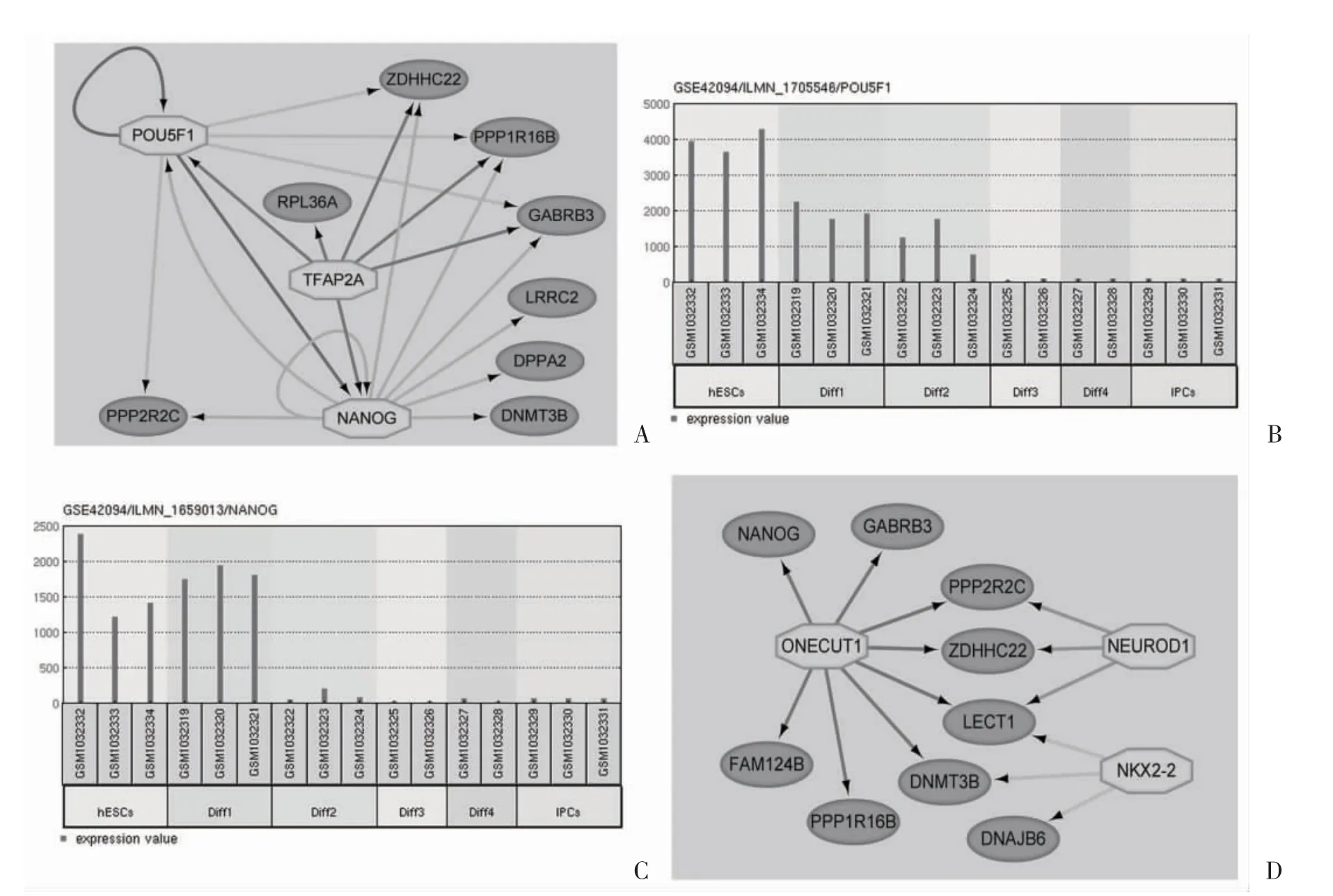
图4 转录因子-靶基因调控网络
2.6 验证miRNA 的转录因子 针对24 个验证的miRNA 查找调控其表达的转录因子,FunRich 软件得到158 个有统计学意义的转录因子,将其与Mahmoud H 等[10]报道的β 细胞分化相关的重要转录因子取交集,得到NKX6-1、ONECUT1、PAX6、MAFB、HNF4A、PDX1、PAX4 和MNX1(HB9)8 个转录因子(图5A)。iRegulon 插件分析得到52 个转录因子,将其与188 个差异表达基因取交集,得到TFAP2A、NR2F2 和FOXA2 3 个转录因子,进一步与Mahmoud H 等报道的β 细胞分化相关的重要转录因子取交集,得到ONECUT1、FOXA2 和PAX4 3 个转录因子,将上述两部分转录因子融合分析,发现FOXA2 既是β 细胞分化相关的重要转录因子也是188 个差异表达基因之一,其表达量见图5B,可见其在整个诱导分化过程中一直处于高表达状态,对整个诱导分化进程发挥转录水平的调控。应用Cytoscape 对转录因子-miRNA 可视化(图5C),发现转录因子TFAP2A 能够调控7 个miRNA 表达,其中miR-375、miR-148a-3p、miR-124-3p 和miR-200c-3p 表达量均在T3EB 阶段显著升高而又在T3pi 阶段显著降低,这与TFAP2A 的表达模式一致(图5D),表明存在较强转录水平的调控。而miR-142-3p、miR-9-5p 和miR-34a-5p 表达量在整个诱导分化过程中一直下降,提示这3 个miRNA 还受其他转录因子的调控或者越到分化后期其转录后水平的调控越强以至于检测到的miRNA 表达量越低。
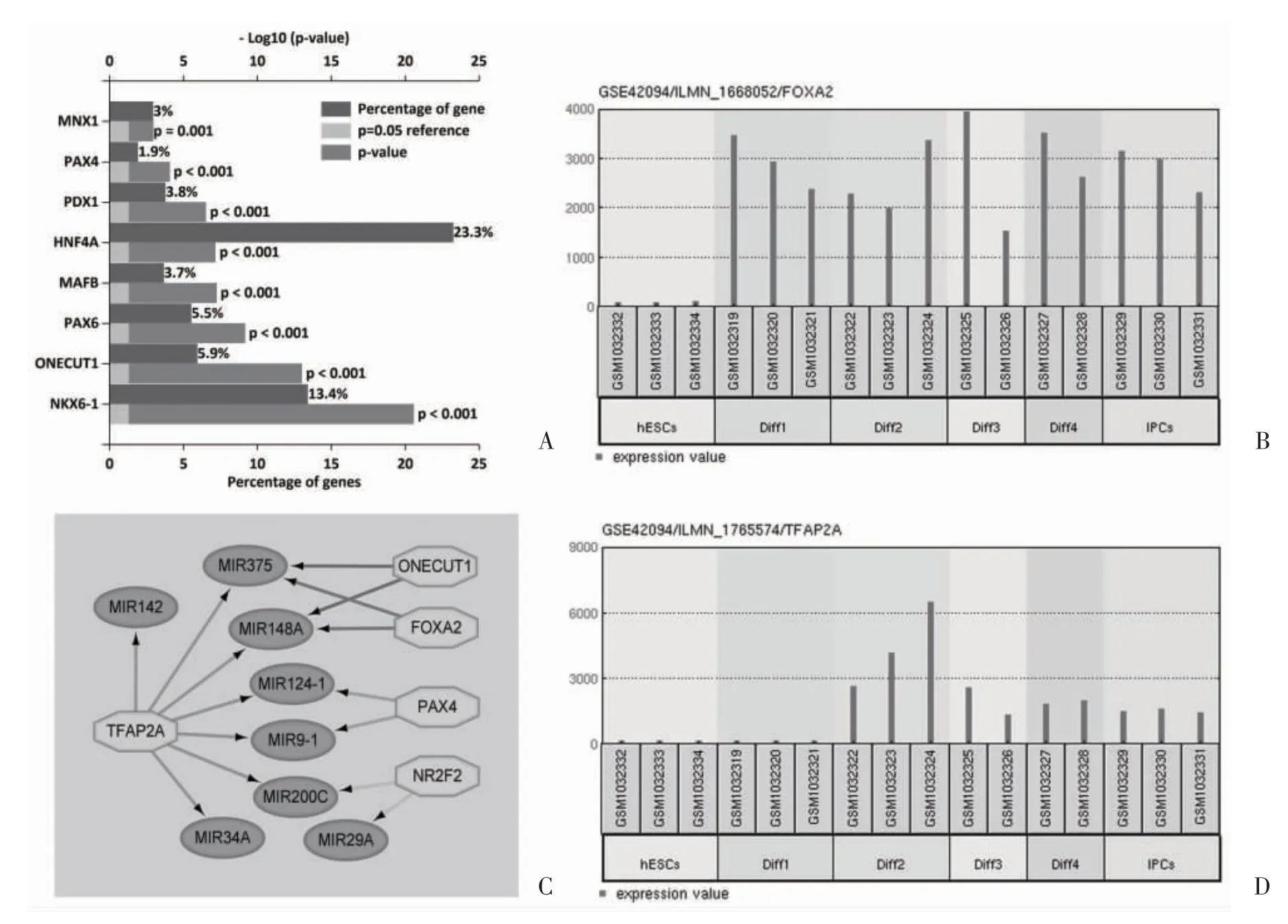
图5 miRNA 的转录因子
3 讨论
本研究通过对GSE42094 的6 组样本数据分析,得到188 个差异表达的基因,选取诱导后显著下调的21 个基因进行后续研究。选用DAVID 数据库对其进行GO 功能和KEGG 路径显著性分析,发现其中POU5F1 和NANOG 基因与基因表达调控、内胚层的决定和成体干细胞种群维持3 个功能均有相关性,但没有找到有统计学意义的KEGG 路径。进一步在GenomeNet 数据库中检索POU5F1 和NANOG 基因的KEGG 路径,发现二者同处于干细胞多能性调控信号通路,能够调控干细胞的自我更新,这与其GO 功能相呼应。另外,该通路与TGF-β、MAPK、PI3K-Akt 以及Wnt 4 条信号通路相关联,这与Mahmoud H 等[10]报道的体外β 细胞分化的关键信号通路相一致。
通过构建miRNA-mRNA 调控网络发现,LOC729860、UTF1、KLKB1、DPPA2、NAP1L3、HLA -DPB2 等6 个基因没有对应miRNA 调控,而PPP1R16B、RPL36A、VSIG10、ZDHHC22 等4 个 基因虽有相应miRNA 调控,但不满足设定的入选标准,因此,这10 个基因在应用Cytoscape 3.9.1 软件进行miRNA-mRNA 调控网络可视化过程中未有miRNA 呈现。网络显示miR-335-5p 能够调控POU5F1、NANOG、FAM124B、DNMT3B 和LECT1 5个基因的表达。其中,POU5F1 和NANOG 基因较为熟知[11-13],而FAM124B 在人HeLa 细胞内能够与染色质解旋酶DNA 结合蛋白7(chromodomain helicase DNA binding protein 7,CHD7)相互作用,而CHD7基因在小鼠ESCs 内与OCT4、NANOG 以及SOX2同处于一个增强子元件,提示FAM124B 作为一个关联因子参与增强子介导的转录[14,15]。DNA 甲基转移酶3B(DNA methyltransferases 3B,DNMT3B)是DNA 甲基转移酶蛋白家族成员之一,DNMT1 主要负责维持DNA 甲基化,DNMT3A 和DNMT3B 负责从头合成甲基转移酶[16,17]。在hESCs 内,DNMT3B 的表达量是已分化细胞的近30 倍,其缺失能够影响线粒体的生成以及提高细胞内α-酮戊二酸的水平,进而引起hESCs 的多向分化[18]。白细胞源性趋化蛋白1(leukocyte cell-derived chemotaxin 1,LECT1)又称软骨源性抑制因子-1(chondromodulin-1),是一个25 kDa 的糖蛋白,由跨膜蛋白经转录后修饰生成,其有助于蛋白聚糖的合成以及体外软骨细胞的生长[19]。在hESCs 内,LECT1 表达量是已分化细胞的近40 倍,其和POU5F1、NANOG 一样被作为干性维持基因[20]。因此,miR-335-5p 所靶向调控的这5 个基因均与干性维持有关,提示miR-335-5p 通过负性调控该类基因的表达,抑制hESCs 的干细胞多能性调控信号通路,促使其转向分化。另外,miR-335-5p 表达量在诱导分化过程中逐渐升高已由文献数据所验证,这与上述干性维持基因的显著下调形成对照。
2021 年,Sabouri E 等[21]报道miR-375、miR-7、miR-186、miR-26a、miR-690 以及miR-302a 6 个IPCs 诱导分化过程中的重要miRNA。本研究同样发现miR-375 和miR-26a 能够靶向调控DNMT3B 基因表达、miR-7 能够调控TCL1B 以及miR-302a 能够调控NANOG 基因表达。可见该miRNA-mRNA调控网络具备一定可信度,通过进一步查找双方的转录因子也可部分地佐证,因此,基于显著下调基因构建的该miRNA-mRNA 调控网络在干细胞诱导分化过程中发挥重要作用。
综上所述,本研究基于显著下调基因构建的miRNA-mRNA 调控网络参与干细胞诱导分化进程并发挥重要作用。
