贵州蜜环菌资源的分子标记与遗传多样性分析
2023-09-18王彩云武新玲成忠均张翔宇
王彩云, 武新玲, 侯 俊, 王 永, 成忠均, 张翔宇*
(1.毕节市中药研究所, 贵州 毕节 551700; 2.山东中医药大学 药学院, 山东 济南 250355; 3.中国医学科学院 药用植物研究所, 北京 100193; 4.大方县乡村振兴局, 贵州 大方 551600)
0 引言
【研究意义】蜜环菌属〔Armillaria(Fr.) Staude〕是担子菌纲伞菌目口蘑科中具有重要药用、经济价值的属,也是全球森林生态系统真菌区系中重要组成部分。蜜环菌(Armillariamellea)不仅是我国传统名贵中药材猪苓(Grifolaumbellata)和天麻(Gastrodiaelatal)栽培必不可少的共生菌,而且具有较高的食药用价值。另外,蜜环菌在全世界寒带和温带侵染600多种树木,可导致林木根朽病,阔叶树及针叶树尤为严重[1]。目前,全球蜜环菌生物种为北美10个、欧洲7个、非洲5个、澳洲5个,亚洲至少存在19个生物种,我国分布有15种[2]。自1978年Korhonen首次发现蜜环菌生物种以来,生物种已成为蜜环菌系统分类的基础,根据生物种建立的分类种,已得到森林病理学和菌物分类学界的广泛认可[3]。而蜜环菌属的系统发育学和遗传多样性研究是现代蜜环菌生物种研究的热点之一,具有重要意义。【前人研究进展】国内外学者在蜜环菌属的分子鉴定及遗传多样性方面开展了大量研究,COETZEE等[4-5]基于ITS和IGS-1序列进行了来自印尼、马来西亚以及南美的部分蜜环菌属种的系统进化地位分析和分子鉴定;KIM等[6-7]采用RFLP、ITS、AFLP等多种标记手段对北美的部分蜜环菌属种进行了鉴定以及系统进化分析,部分菌株得到有效区分;国内关于蜜环菌报道中,王守现等[8]用RAPD技术进行了13个蜜环菌菌株的遗传多样性分析,提出RAPD技术可以有效区分蜜环菌菌株并分析其遗传多样性;孙立夫等[9]应用ISSR技术分析了采自东北的53个法国蜜环菌菌株的遗传多样性,提出ISSR标记在蜜环菌中存在较高的多态性。【研究切入点】目前,有关蜜环菌的研究报道主要集中于生物学特征、化学成分分析等方面[10-16],有关贵州蜜环菌菌株分子标记及遗传多样性分析的报道相对较少。【拟解决的关键问题】利用分子标记技术对贵州天麻主产区毕节地区19个蜜环菌居群的菌株遗传多样性进行分析,构建聚类分析树状图,弄清亲缘关系,并讨论其形成的可能原因,以期为蜜环菌的优良资源筛选及后续研究奠定基础。
1 材料与方法
1.1 材料
1.1.1 蜜环菌 供试蜜环菌为毕节市中药研究所从贵州天麻主产区收集的蜜环菌资源(表1),经分离、提纯后的试管种。
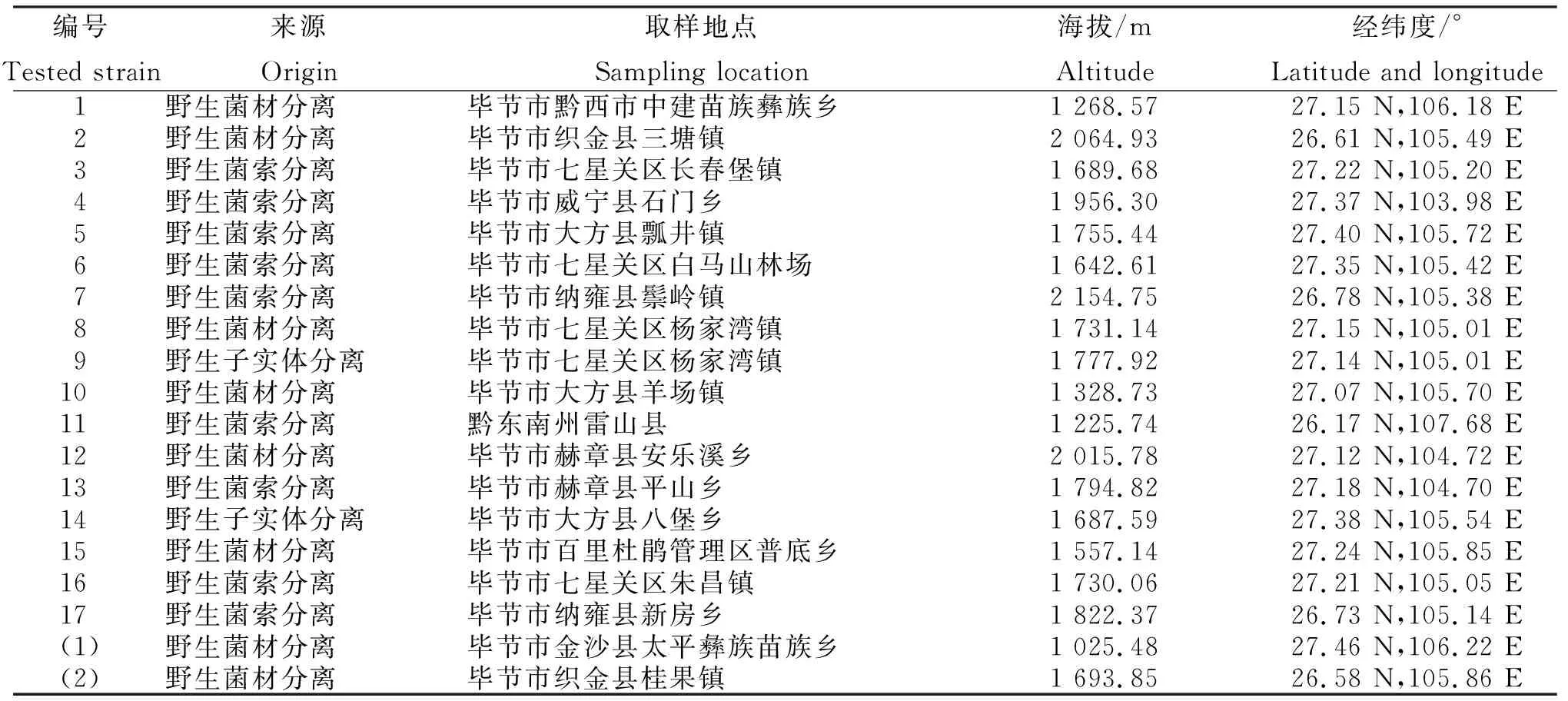
表1 供试蜜环菌资源信息与来源
1.1.2 主要试剂和仪器 用于PCR反应的2×M5HiPer plus Taq HiFi PCR mix 购于北京聚合美生物科技有限公司,引物由生工生物工程(上海)股份有限公司合成。标准DNA分子量(Marker)DL5000购于宝日医生物技术(北京)有限公司(TaKaRa)。DNA提取试剂盒(货号DP305)购于天根生化科技(北京)有限公司。PCR扩增仪(Appliedbiosystems,Veriti)、凝胶成像系统(Bio-Rad,XRS+)、NanoDrop分光光度仪(Thermo Scientific,NanoDrop2000)、电泳仪(Bio-Rad,通用电泳仪164-5070)。
1.2 方法
1.2.1 DNA提取及浓度测定 取适量蜜环菌的新鲜菌索,置于液氮中研磨后利用天根生化科技(北京)有限公司的植物基因组DNA提取试剂盒(货号DP305)提取蜜环菌DNA,将DNA原液保存于-20 ℃备用。再利用微量核酸蛋白分析仪Nanodrop 2 000测定DNA浓度及纯度,并利用琼脂糖凝胶电泳检测DNA的完整性。
1.2.2 分子标记的扩增引物及筛选 依据参考文献[17-19],选择3对引物进行蜜环菌分子标记扩增并进行分析,其中,1#引物[17]序列为AR1,5′-CTGACCTGTTAAAGGGTATGTGC-3′;AR2,5′-AAGCTGAATCCTTCTACAAAGTCAA-3′;2#引物[18]序列为EF595F,5′-CGTGACTTCATCAAGAACATG-3′;EF1160R,5′-CCGATCTTGTAGACGTCCTG-3′;3#引物[19]序列为LR12R,5’-CTGAACGCCTCTAAGTCAGAA-3′;O-l:5′-AGTCCTATGGCCGTGGAT-3′。以此3对引物分别对蜜环菌DNA进行分子标记扩增,选择合适引物。
1.2.3 PCR扩增 以蜜环菌DNA为模板,分别利用1.2.2确定的引物进行PCR扩增。PCR扩增反应在Bio-Rad PCR扩增仪上进行,反应程序为95 ℃预变性5 min;95 ℃变性30 s,57 ℃退火30 s,72 ℃延伸30 s,40个循环;72 ℃延长7 min,4 ℃保存。PCR扩增体系为DNA 3 μL,F/R引物各0.5 μL,10 μL 2× M5HIPer plus Taq HIFI PCR mix,ddH2O 6 μL,总体系为20 μL。PCR产物用1.0%琼脂糖凝胶,电泳仪180 V电压15 min,测定引物是否能扩增出目的片段。
1.2.4 序列比对及聚类分析 将PCR扩增所得原液送至中国农业科学院进行Sanger测序。用ContigExpress拼接测序结果,并去除两端不准的部分。将拼接好的序列在NCBI数据库中进行比对,并下载同源度较高的序列用于构建系统发育树。运用DNASTAR中的EditSeq对碱基长度、GC含量等进行测量;利用MEGA7.0进行聚类分析并构建进化树。
2 结果与分析
2.1 蜜环菌菌株的DNA质量
用植物基因组DNA试剂盒(DP305)提取蜜环菌基因组DNA,OD260/OD280值在1.63~1.96,DNA浓度为24.9~117.8 ng/μL(表2),其纯度和产量均较高,说明所提取的DNA可用于后续试验。

表2 供试蜜环菌样品的DNA浓度与OD260/OD280值
2.2 蜜环菌菌株的适宜引物
3对引物以蜜环菌DNA为模板进行PCR扩增、电泳检测后发现,2#、3#引物均能有效对19份蜜环菌DNA模板进行扩增(图1)。2#、3#引物适用于蜜环菌种群,对蜜环菌DNA模板产生各自特有且清晰的目的基因带。因此,选用2#、3#引物进行分子标记扩增分析。

图1 蜜环菌样品用2#(上)、3#(下)引物扩增的目的片段图谱
2.3 蜜环菌菌株的碱基组成
由表3可知,19株蜜环菌以2#、3#引物扩增获得序列的碱基长度、GC含量等均不同。其中,以2#引物扩增的序列,碱基片段长度为574~667 bp,平均值为593 bp;GC含量为51.79%~54.27%,平均含量为53.56%。C含量为23.43%~30.13%,平均含量为26.38%;G含量为23.43%~30.34%,平均含量为27.18%;A含量为25.21%~22.15%,平均含量为23.31%;T含量为22.01%~24.62%,平均含量为22.96%。以2#为引物时,各供试菌株的GC含量差异不明显,表明其亲缘关系较近,其中,10号、13号菌株的碱基长度以及GC含量、各碱基含量均相同,表明2个菌株的亲缘关系极为接近。以3#引物扩增的序列,碱基片段长度为828~925 bp,平均值为891 bp;GC含量为43.01%~45.00%,平均含量为44.02%。C含量为18.15%~25.42%,平均含量为22.74%;G含量为17.51%~25.16%,平均含量为21.26%;A含量为23.82%~33.57%,平均含量为28.68%;T含量为23.62%~33.06%,平均含量为27.20%。以3#为引物时,各供试菌株的GC含量差异不明显,表明其亲缘关系较近,其中,1号菌株与3号菌株的GC含量相同,碱基长度相同,表明其亲缘关系极为接近。

表3 2#、3#引物扩增19个蜜环菌菌株的碱基组成
2.4 蜜环菌菌株的相似率
由表4可见,各供试蜜环菌菌株以2#引物扩增得到的序列比对覆盖率不低于82%,序列相似率不低于96.08%。其中,8、14、17的DNA序列与A.mellea的同源性最高,序列相似率均达99%以上,基因覆盖度均达96%以上,其余16个菌株的DNA序列与A.gallica的同源性最高,序列相似率均高于96%,基因覆盖度均达82%以上。

表4 各菌株用2#、3#引物扩增序列的相似率
各供试蜜环菌菌株以3#引物扩增得到的序列比对覆盖率不低于93%,序列相似率不低于98.45%。其中,10号菌株的序列比对覆盖率为93.00%,序列相似率为99.88%,与A.sinapina的同源性最高;11号菌株的序列比对覆盖率为99.00%,序列相似率为99.54%,与A.cepistipes的同源性最高;8、14、17的DNA序列与A.mellea的同源性最高,序列相似率均达98%以上,基因覆盖度均达94%以上;其余14个菌株的DNA序列与A.gallica的同源性最高,序列相似率均高于99%,基因覆盖度均达94%以上。综上,贵州天麻主产区毕节市野生蜜环菌资源,大部分为A.gallica,少部分为A.mellea。
2.5 蜜环菌菌株的聚类
如图2所示,2#引物将19份天麻样品分为7大类,样品(2)、4、2、(1)、16、17聚为第1类,其中4、(2)、2、(1)的遗传距离最近;第2类为样品7、12;第3类为样品9、15;第4类为样品8、3、11;样品6单独聚为一类;第6类为样品1、5、14;第7类为样品10、13。与上述分类不同,3#引物将19份样品分为5大类,样品4、13、9、11聚为第1类;样品6、12、17聚为第2类;样品14单独为1类,与A.mellea的遗传距离最近;样品(1)、10单独为第4类;样品2、(2)、8、16、7、5、15、1、3聚为第5类,其中,样品2、(2)遗传距离最近,5、15、1、3遗传距离最近。

图2 2种引物扩增蜜环菌样品DNA目的片段的进化树
从2#、3#引物的扩增序列构建的系统发育树看,19个菌株在系统发育树上的聚类结果与表3的GC含量分析基本一致;与表4结果接近。来自不同地区的蜜环菌菌株未聚为一类,说明蜜环菌资源遗传多样性丰富,与地理环境关系不密切。8号和9号菌株均来自毕节市七星关区杨家湾镇,8号分离于菌材,9号分离于子实体,但是遗传距离较远,亲缘关系不密切,可能是相同地区的菌株自身发生遗传变异,导致菌株的遗传多样性。
3 讨论
近年来,世界各地关于蜜环菌的报道越来越多,欧洲、北美等地对蜜环菌的研究较早也比较深入,我国有关蜜环菌的研究进展较快,蜜环菌的鉴定方法随之从传统的形态学鉴定发展到如今的分子生物学鉴定。作为传统经典的鉴定方法,形态学有着其独特的优越性,但由于蜜环菌分类特征不明显,表观形态多样,而且需要结合孢子、菌索、子实体等样本才能鉴定,受到较大限制;在分子生物学鉴定中,采用单个的IGS、ITS、EF-1α序列无法准确鉴定每个蜜环菌种。基于此,研究选择特异性引物2#(翻译延伸因子1α基因,EF-1α)以及3#引物(基因间隔区,IGS),其中,翻译延伸因子1α基因参与蛋白质的合成。研究结果表明,2个引物扩增序列得到的聚类结果不一样,可能是因为2种引物扩增的区域进化速度不相同,EF-1α的进化相对保守。对采自贵州毕节市的19份野生蜜环菌资源进行基于分子标记的遗传多样性分析后,2对引物均能有效扩增蜜环菌的基因组DNA,并能对贵州毕节产区蜜环菌资源进行有效分类,对蜜环菌遗传多样性研究提供了宝贵资源,该分子标记体系的建立为今后利用分子标记对蜜环菌遗传多样性研究、亲缘关系分析及种质资源鉴定等研究工作奠定了基础。
研究种质资源遗传多样性的有效方法是聚类分析和GC含量,在生物体的DNA序列中,GC含量能够反映出种属间亲缘关系的遗传特性,每个物种的DNA中都有其特定的GC含量,不同物种的GC含量不同,GC含量差异越小,亲缘关系越接近[20]。基于聚类和GC含量等对来源于贵州主产区的19份蜜环菌种质进行分析,以2#引物扩增的序列分为7大类群,以3#引物扩增的序列分为5大类群,不同地区来源的种质相互交错,亲缘关系与地理来源无明显相关性,但贵州毕节市野生蜜环菌资源大部分为A.gallica,少部分为A.mellea。黄万兵等[21]研究提出,A.gallica、A.cepistipes能促进天麻生长;CHA等[22-23]研究提出,A.gallica的共生效果相对较好。研究发现,贵州毕节市天麻主产区存在丰富的A.gallica资源,因此完全有条件筛选最适宜当地天麻种植的蜜环菌菌种来发展天麻产业,无需从外地引种、购种,从而有效避免长期跨地区引种带来的产量不稳、菌种退化等问题。
4 结论
2#、3#引物能有效扩增蜜环菌的基因组DNA,并能将采自贵州毕节市的19份野生蜜环菌资源进行有效分类。通过序列比对、GC含量分析以及聚类分析发现,不同地区的蜜环菌资源相互交错,亲缘关系与地理来源无明显的相关性,贵州毕节市野生蜜环菌资源大部分为A.gallica,少部分为A.mellea。
