干旱胁迫下油用牡丹‘凤丹’基因组DNA 甲基化分析
2023-09-14樊明月李昱莹侯小改郭丽丽
樊明月,李昱莹,王 璨,侯小改,郭丽丽
(河南科技大学农学院,洛阳 471023)
全球变暖所导致的干旱等气候问题对植物造成了严重影响[1],降水量不足以及降水模式改变使干旱成为非生物胁迫中最具破坏性及最能影响作物产量的重要因素[2-5],极大限制了植物的正常生长发育[6-8]。 干旱对植物的影响贯穿植物生长发育的各个阶段,不仅影响植物的生长形态,还可引起植物体内生理生化发生变化[9-11]。 已有研究证明,干旱胁迫可诱导植物基因组DNA 甲基化状态发生改变[12-13]。DNA 甲基化是植物自身应对干旱胁迫重要的途径之一,可通过改变植物基因表达水平,引起不同生理反应,从而适应干旱[14-15]。
DNA 甲基化作为表观遗传学的重要研究内容,其在调控基因表达、维持基因组稳定性等方面具有重要作用[16-17]。 随着对DNA 甲基化的研究日益精进和分子研究技术手段的不断发展,DNA 甲基化检测的方法和手段日新月异。 其中,甲基化敏感扩增多态性(Methylation sensitive amplification polymorphism,MSAP)技术是利用限制性内切酶HpaII 和MspI 对基因组DNA 进行特异性酶切,酶切后获得不同的DNA切割片段,以此来揭示植物基因组DNA 的甲基化状态[18]。 该方法中的限制性内切酶HpaII 和MspI 可识别相同的5’-CCGG 序列[19],基于其对胞嘧啶的敏感程度不同,HpaII 选择性酶切一条链被甲基化的位点,而MspI 选择性酶切双链内侧被甲基化的位点,HpaII 和MspI 均可切割无甲基化位点[20-21]。
牡丹(Paeonia suffruticosaAndrews)作为中国传统名花,在我国有着悠久的人工栽培应用历史[22],其观赏价值和药用价值被广泛肯定[23]。 油用牡丹‘凤丹’(P.ostii‘Fengdan’)作为新兴的油用作物,籽油品质高,且其生态适应性强[24-25],于2011 年被国家卫生部批准为新资源食品[26-28]。 随着国家对新型油料作物的大力扶持和油用牡丹产业链的不断完善,人们对油用牡丹的关注度越来越高,不断深入研究挖掘其潜在价值。 本试验研究不同干旱处理下‘凤丹’基因组DNA 的甲基化差异及其变异模式,分析干旱胁迫对‘凤丹’基因组DNA 甲基化带来的影响,以期为其在培植繁育等方面的研究提供理论依据。
1 材料与方法
1.1 试验材料
试验材料为河南省洛阳市国际牡丹园内4 年生油用牡丹‘凤丹’。 挑选长势良好且生长情况一致的4年生‘凤丹’,移栽于规格为20 cm×16 cm×13 cm 的花盆中,所有植株的光照条件一致。
1.2 试验方法
1.2.1 试验设计
试验设对照组(按80%的日蒸腾量57.4 mL 浇水)、轻度干旱处理组(按40%的日蒸腾量28.7 mL 浇水)及轻度干旱复水处理组、重度干旱处理组(干旱过程中不浇水)及重度干旱复水处理组。 不同处理组植株均在干旱处理14 d 后出现表型;对出现表型的轻度干旱复水处理组和重度干旱复水处理组植株进行复水处理,对其正常浇水即为复水,复水处理3 d 后,轻度干旱处理组植株恢复至正常状态的表型,复水处理5 d 后,重度干旱处理组植株恢复至正常状态的表型。 随机选取同一处理组不同植株上的完整叶片,混合放在锡箔纸上,包裹、标记,放入液氮中低温冷冻保存、待用。
1.2.2 甲基化敏感多态性分析
(1)采用改良的CTAB 法提取处理后的‘凤丹’叶片基因组DNA[29]。
(2)采用付胜杰等[30]建立的MSAP 反应体系及程序对‘凤丹’基因组DNA 进行酶切、连接、预扩增和选择性扩增。 选择EcoRI∕MspI 和EcoRI∕HpaII 作为双酶切组合,对‘凤丹’基因组DNA 进行双酶切,MSAP分析所用接头和预扩增引物序列见表1。

表1 MSAP 分析的接头序列和引物序列Table 1 Adapter sequences and primer sequences for MSAP analysis
(3)为方便酶切体系的配制,需将DNA 样品质量浓度保持一致(400 ng∕μL),酶切体系共20 μL,包含1 μL 基因组DNA、1 μLEcoRI、1 μLHpaII∕MspI、2 μL 10 ×EcoRI Buffer、2 μLHpaII∕MspI Buffer,用无菌双蒸水补齐。
(4)酶切后应立即进行连接反应,连接反应体系共20 μL,包含2 μL 10 ×T4 连接酶Buffer、1 μL T4连接酶、0.4 μLHpaII-MspI 接头、0.4 μLEcoRI 接头、10 μL 酶切产物,用无菌双蒸水补齐,连接产物需16 ℃过夜;过夜连接后的产物于65 ℃温育10 min 后终止反应。
(5)以连接产物作为模板进行预扩增,预扩增反应体系共20 μL,包含10 μL 连接产物、预扩增引物各0.5 μL、0.2 μLTaqDNA 聚合酶、0.4 μL dNTPs、2 μL PCR Buffer,用无菌双蒸水补齐。
(6)预扩增完成后,用稀释40 倍的预扩增产物作为模板进行选择性扩增,MSAP 选择性扩增反应体系共20 μL,包含2 μL 预扩增产物、特异性扩增引物各1 μL、10 μLTaqDNAMix,用无菌双蒸水补齐。
(7)配制浓度为6%(体积比)的变性聚丙烯酰胺凝胶,将选择性扩增获得的PCR 产物上样于聚丙烯酰胺凝胶中进行垂直电泳,电压220 V,电泳时间140 min。 电泳结束后对凝胶进行银染,观察条带。
1.2.3 数据分析
染色显影后,观察统计每个PAGE 胶图版的条带,在同一位点处有清晰条带显示记为“1”,无条带显示处记为“0”。 通过Quantity One 软件读取MSAP 条带信息并转化成表型数据0∕1 矩阵,统计结果制成Excel 表格。
2 结果与分析
2.1 干旱胁迫对‘凤丹’牡丹表型变化的影响
在干旱胁迫下,‘凤丹’植株的叶片形态变化显著,叶片长势明显变差,具体表现为:轻度干旱时,植株叶片开始下垂,并出现萎蔫和发黄;重度干旱时,叶片出现严重的干枯卷曲,叶片数量减少,枝条下垂。 对不同程度干旱处理后的植株进行复水处理,轻度干旱复水的植株仍能恢复其生命活力,外观上基本恢复正常,叶片皱缩和萎蔫消失,叶片舒展,但与对照植株相比,仍表现出叶片发黄、叶片数量减少的现象;重度干旱的植株复水后也仍具有生命力,但与对照植株相比,叶片发黄且较为皱缩,枝条下垂(图1)。

图1 干旱胁迫对‘凤丹’表型变化的影响Fig.1 Effects of drought stress on phenotypic changes of P.ostii ‘Fengdan’
2.2 不同干旱胁迫下‘凤丹’牡丹总甲基化状态
使用EcoRI∕MspI 和EcoRI∕HpaII 组合对样品DNA 酶切后,检测出4 种甲基化类型(图2),分别为I 型(超甲基化):H、M 均无带,DNA 双链外部甲基化或双链内外部都甲基化,记为(0,0);II 型(全甲基化):H无带、M 有带,DNA 双链内部甲基化,记为(0,1);III 型(半甲基化):H 有带、M 无带,DNA 单链外部甲基化,记为(1,0);IV 型(非甲基化):H、M 均有带,即DNA 双链的CCGG 位点无甲基化发生,记为(1,1)。

图2 选择性扩增凝胶电泳图谱Fig.2 Selectively amplification gel electrophoresis
以30 对选择性引物进行扩增,通过观察比对筛选出多态性丰富、重复性较好的26 对引物组合,使用MSAP 检测分析不同处理下‘凤丹’牡丹基因组DNA 甲基化状态(表2)。 结果显示,26 对选择性引物共扩增出932 个CCGG 位点。 对照植株半甲基化位点(III 型)有115 个,半甲基化率为12.34%;全甲基化位点(II 型)86 个,全甲基化率为80.90%。 在不同程度干旱处理组中,轻度干旱和重度干旱的‘凤丹’牡丹半甲基化位点分别为103 个和101 个,半甲基化率分别为11.05%和10.84%;全甲基化位点分别为140 个和174 个,全甲基化率均为85.09%。 轻度干旱诱导‘凤丹’牡丹基因组DNA 的半甲基化率下降了1.2%;重度干旱诱导其半甲基化率下降了1.5%;轻度干旱和重度干旱胁迫下,‘凤丹’牡丹的全甲基化率均表现为上升趋势,升幅为4.2%。
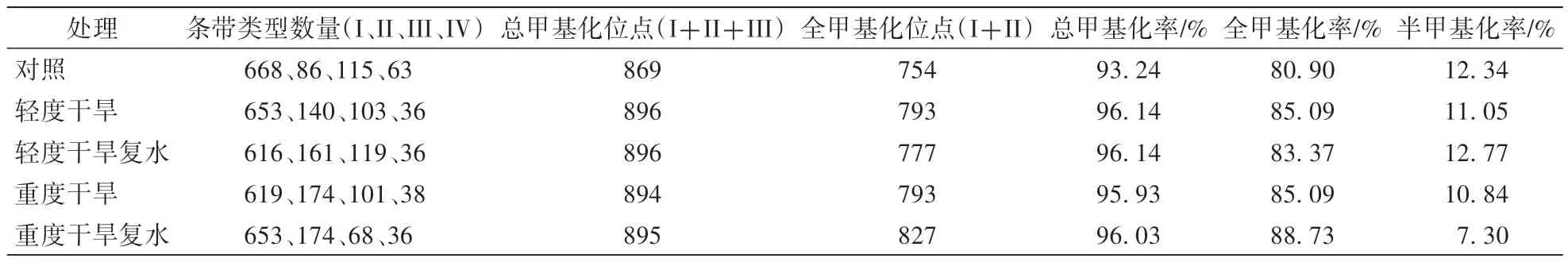
表2 不同干旱条件下‘凤丹’基因组DNA 甲基化水平Table 2 Genomic DNA methylation levels of P.ostii ‘Fengdan’under different drought stress
干旱复水处理组的‘凤丹’牡丹基因组DNA 半甲基化位点数和全甲基化位点数均发生改变。 统计结果显示:轻度干旱复水后,‘凤丹’牡丹的半甲基化位点有119 个,半甲基化率为12.8%,全甲基化位点有161 个,全甲基化率为83.4%,与对照相比,半甲基化率和全甲基化率均呈现上升趋势;与轻度干旱处理组相比,半甲基化率升高了1.7%,全甲基化率降低了1.7%。 重度干旱复水后,‘凤丹’牡丹的半甲基化位点有68 个,半甲基化率为9.7%,全甲基化位点有174 个,全甲基化率为85.0%,与对照相比,半甲基化率降低了2.6%,全甲基化率升高了4.1%;与重度干旱处理组相比,半甲基化率降低了1.1%,全甲基化率降低了0.1%。
2.3 干旱诱导的‘凤丹’牡丹甲基化变化和去甲基化变化
分析不同程度干旱处理后下‘凤丹’牡丹基因组DNA 甲基化的变异模式,发现26 对选择性引物在不同处理的样本中扩增产生了4 种变异模式,共15 种条带类型(表3 和表4)。
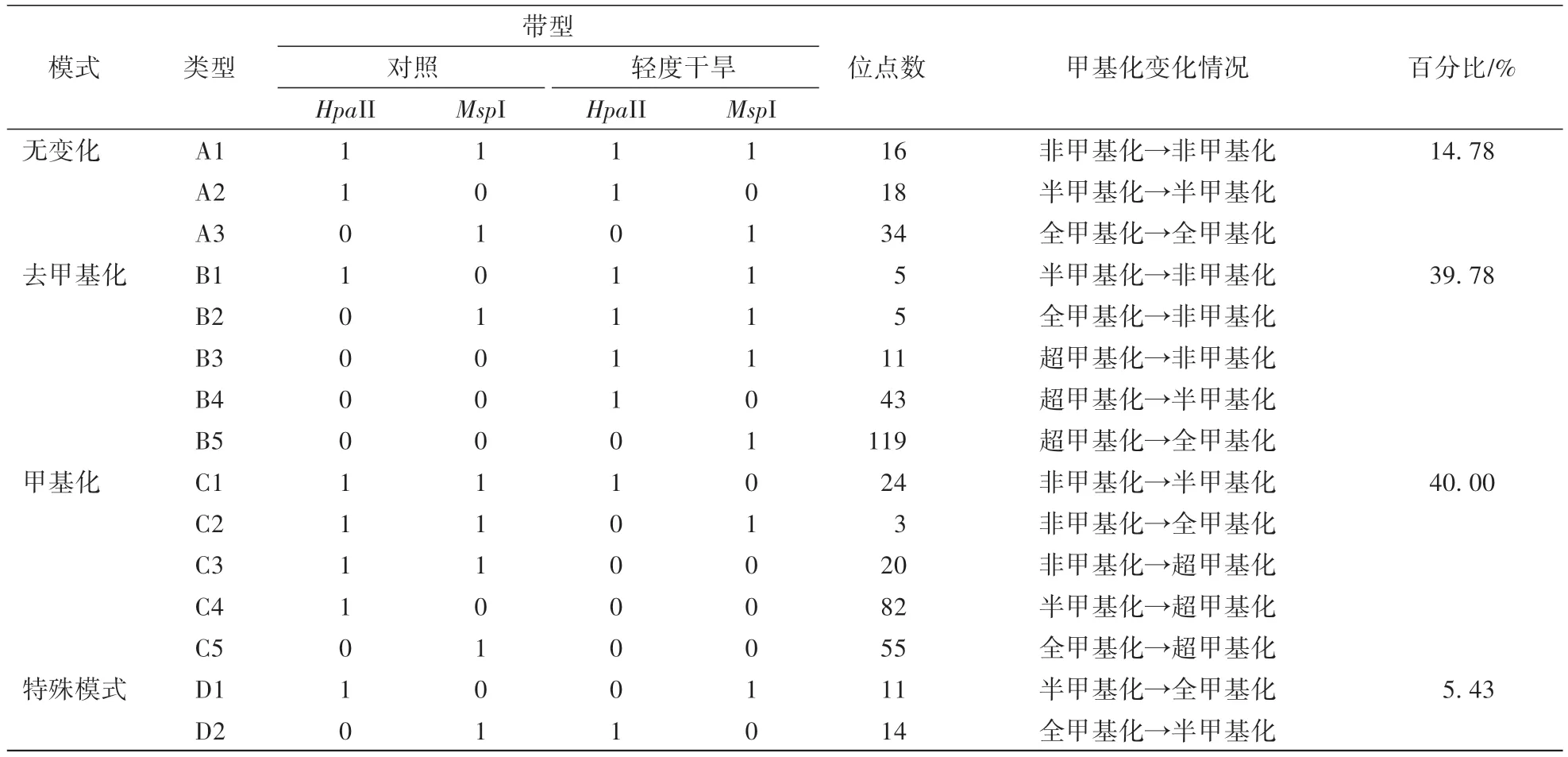
表3 轻度干旱处理下‘凤丹’基因组DNA 甲基化模式的变化Table 3 Changes in genomic DNA methylation patterns of P.ostii ‘Fengdan’ under slight drought treatment

表4 重度干旱处理下‘凤丹’基因组DNA 甲基化模式的变化Table 4 Changes in genomic DNA methylation patterns of Paeonia ostii ‘Fengdan’ under severe drought treatment
模式A:对照和干旱处理组有相同的甲基化CCGG 位点,甲基化位点无变化,该模式包含3 种条带类型A1、A2、A3,在轻度干旱处理组中占14.78%,在重度干旱处理组中占11.69%。 模式B:与对照相比,处理组DNA 甲基化水平下降,为去甲基化,该模式包含5 种条带类型,B1—B3 为对照中的甲基化位点变成非甲基化位点;B4 表示与对照相比,超甲基化位点变为半甲基化位点,在重度干旱处理组中该变化高于轻度干旱处理组;B5 表示超甲基化位点变为全甲基化位点。 该模式在轻度干旱处理组中占39.78%,在重度干旱处理组中占46.00%。 模式C 为甲基化模式,包含5 种条带类型C1、C2、C3、C4、C5,其中C1—C3为对照中的非甲基化位点变成了甲基化位点;C4 为半甲基化位点变为超甲基化位点;C5 为全甲基化位点变成了超甲基化位点。 该模式在轻度干旱处理组中占40.00%,在重度干旱处理组中占38.60%。 模式D为特殊模式,在此类别下,对照与处理组之间的甲基化变异水平不是单纯的升高或降低。
3 结论与讨论
‘凤丹’牡丹在遭受干旱胁迫后,其生长发育受到显著影响。 在不同干旱处理下,‘凤丹’牡丹的表型发生改变,叶片会出现失水皱缩,对其进行复水处理后,可恢复活力,表明面对干旱胁迫时‘凤丹’牡丹总体趋向于休眠以应对外界的危害。 大部分高等植物基因组DNA 甲基化水平在30%—50%[31-35],本研究表明,‘凤丹’牡丹基因组DNA 甲基化水平明显高于其他植物。 干旱胁迫下的‘凤丹’牡丹基因组DNA 半甲基化率随着干旱程度的增加呈现逐渐下降的趋势,全甲基化率呈现增加的趋势;在进行干旱复水后,轻度干旱复水处理组的半甲基化程度和对照基本相同,出现该现象的原因可能是原来某些非甲基化位点发生了甲基化使半甲基化水平略微增加;而重度干旱复水处理组与对照相比,半甲基化水平显著降低,表明重度干旱对其影响较大,可能导致牡丹基因组DNA 产生了一些不可逆转的变化。
在长期进化中,植物形成了自身的抗旱机制[36]。 DNA 甲基化在植物抵抗非生物胁迫的过程中发挥着至关重要的作用,使植物得以在逆境中生存[37]。 DNA 甲基化作为一种潜在的可遗传的表观遗传现象,在防御外源DNA 入侵和阻遏转座子在基因组移动以及调控高等植物基因表达方面具有双重作用[38-39]。许多研究表明,DNA 甲基化参与了植物对抗非生物胁迫的过程,且已证明DNA 甲基化是植物参与适应干旱胁迫的调节系统中的重要一环。 植物通过调节基因组的甲基化水平,适应外界的非生物胁迫,以保证植物自身能够适应外界环境而正常生长[40]。
本试验对干旱胁迫下‘凤丹’牡丹基因组DNA 甲基化水平变化进行了探究,旨在揭示油用牡丹抗逆能力与其DNA 甲基化程度之间的关系。 ‘凤丹’牡丹作为多年生木本植物,其基因组庞大而复杂,甲基化水平与植物自身基因组复杂程度紧密相关[34,41],‘凤丹’牡丹基因组的高甲基化率可能与其自身基因组包含大量转座子及其他重复序列有关。 面对干旱胁迫,‘凤丹’牡丹改变自身甲基化水平,其基因组甲基化率上调;在解除胁迫后,‘凤丹’基因组总甲基化率仍高于对照,表明DNA 甲基化参与了其对抗干旱胁迫的过程。 植物基因组DNA 的甲基化和去甲基化往往还影响着其基因转录的激活和抑制[42-45],即影响基因的表达,探究参与干旱胁迫调控DNA 甲基化的基因,挖掘其功能,将成为改良油用牡丹抗性及品质后续工作的重要思路。
