Maize 6H-60K芯片在玉米实质性派生品种鉴定中的应用分析
2023-09-14田红丽张如养范亚明张云龙易红梅邢锦丰王凤格赵久然
田红丽 张如养 范亚明 杨 扬 张云龙 易红梅 邢锦丰 王凤格 赵久然
Maize 6H-60K芯片在玉米实质性派生品种鉴定中的应用分析
田红丽**张如养**范亚明**杨 扬 张云龙 易红梅 邢锦丰 王凤格*赵久然*
北京市农林科学院玉米研究所/ 农业农村部农作物DNA指纹创新利用重点实验室(部省共建) / 玉米DNA指纹及分子育种北京市重点实验室, 北京 100097
玉米实质性派生品种鉴定已成为当前种业知识产权保护的热点之一。为加快其精准高效分子鉴定技术的建立, 本文利用多种类型派生品种为研究材料: 京2416与京2416C (两者为遗传背景高度相近的两个自交系), 京724与京72464 (两者为遗传背景相近的两个自交系), 以及由京724与京72464两者构建的893个DH系遗传群体等。研究分析了Maize 6H-60K芯片(包含61,214个SNP位点集合)应用于玉米派生品种鉴定的潜力。结果显示: (1) 京2416与京2416C间存在829个SNP位点差异, GS值(遗传相似度)为98.7%, 56.7%的差异位点集中分布在5号染色体长度约39 Mb区域内。(2) 京724与京72464之间差异位点数目为4912个, GS值为90.1%, 44.8%的差异位点集中分布在3号染色体上。(3) 893个DH系与2个亲本京724及京72464之间的GS值分布均呈现连续性, 其中与京724之间的GS值范围88.0%~97.0%, 平均值为92.6%; 与京72464之间的GS值范围88.3%~98.6%, 平均值为94.5%。(4) 893个DH系进行两两成对比较, 共比较398,278对, 所有DH系之间均有明确的SNP位点差异; GS值最小为87.5%, 最大为99.9%, 平均值为94.3%。结果表明Maize 6H-60K包含的SNP位点集能够精准评估派生、近似或极近似自交系及DH系的遗传背景, 将所有材料一一鉴别明确区分开来, 并具有进一步锁定与派生性状连锁标记的潜力。建议亟需基于Maize 6H-60K SNP位点集合, 利用高效芯片、靶向测序等平台建立玉米实质性派生品种分子鉴定技术规程, 为玉米品种知识产权保护、品种创新等提供技术支撑。
玉米; 实质性派生品种; 分子鉴定; Maize 6H-60K芯片; 高密度SNP位点集合
玉米是我国种植面积最大、品种数量最多、种业市值最高的农作物, 是确保国家粮食安全的战略支撑。随着玉米种业的快速发展和生物育种技术的普及应用品种数量剧增, 同时派生或近似品种正在逐步增多, 品种同质化问题严重。截至目前我国已有审定玉米品种2万个以上, 每年仍有近1万个品种组合在参试; 申请植物新品种权超过15,000件, 其中授权品种6000多件, 在农作物中排名第一(http://202.127.42.145/bigdataNew/)。创新是引领发展的第一动力, 保护知识产权就是保护创新。面对种业现状迫切需要推行严格的品种知识产权保护制度, 使其成为种业创新的核心保障。2022年3月1日公布实施的新种子法, 进一步加强了品种权保护力度, 扩大了品种权保护的环节、范围。该种子法借鉴国际经验, 对接UPOV国际植物新品种保护联盟(International Union for the Protection of New Varieties of Plants)发布的植物新品种保护公约1991年本文, 建立了EDV (Essentially Derived Variety)实质性派生品种保护制度, 解决当前品种同质化问题, 激励种业原始创新。
针对实质性派生品种保护制度的实施, 亟需建立精准高效的鉴定技术, 高质量支撑种质资源、育种材料的合理合法利用, 激励品种创新。EDV指由原始品种实质性派生或者由该原始品种的实质性派生品种派生出来的品种, 除派生过程产生的差异外, 与原始品种基因型或基因型组合控制的基本性状相同, 即遗传背景相同或近似、个别性状有差异。获得派生品种的途径主要有自然变异或人工诱变, 从原始品种中选变异单株, 以原始品种为受体进行多次回交, 高代姊妹系, 遗传工程转化等。EDV品种判定的核心为品种间遗传组成的相似程度, 可采用表型性状测试、育种系谱分析以及基于分子标记计算遗传相似度等方法。表型测试存在受环境影响、耗时长、成本高、数据难以共享与标准化等问题; 系谱分析法则由于难以获取品种详细准确的系谱信息而很少应用[1]。分子标记法可直观反映品种间遗传组成的相似程度, 在鉴定EDV品种方面具有精准、快速、成本低等优势。近20年来基于分子标记法玉米EDV品种鉴定的报道主要为利用AFLP (Amplified Fragment Length Polymorphisms)、SSR (Simple Sequence Repeats)、SNP (Single Nucleotide Polymorphism)以及MNP (Multiple Nucleotide Polymorphism)标记建立的快速鉴定方法[2-10]。SNP标记由于具有在基因组中分布密度高, 易实现数据整合共享以及与功能基因关联强等优势被首选, 并被国际组织UPOV和ISF国际种子联盟(International Seed Federation)推荐应用[8,11]。
分子标记位点的密度和基因组覆盖度是决定EDV鉴定精准性的关键, 因而须利用基因组均匀分布的高密度位点集合反映品种间的遗传组成相似度[12]。重测序数据通量和质量的提高促进了全基因组水平SNP位点的挖掘评估与利用, 在玉米中开发了几款高密度芯片如Maize SNP50、Maize 600K、Maize 55K[13-15]。Maize SNP50和Maize 600K为商业化芯片, 主要以美国和欧洲材料进行评估, 因此位点具有偏好性; 定制芯片主要以应用于基因定位、分子育种而开发。北京市农林科学院玉米研究所基于388份国内外代表性玉米自交系的全基因组重测序数据, 挖掘优异变异位点, 评估确定了高质量、均匀分布的位点集(包含核基因组61,214个位点、叶绿体基因组68个位点), 最终研制定型集品种鉴定、确权等多用途为一体的高密度芯片Maize 6H-60K[16]。2021年颁布的行业标准“玉米品种真实性鉴定SNP标记法”推荐采用该芯片包含的61,214个SNP位点进行玉米品种真实性身份鉴定[17]。
本研究选用了一组派生自交系、893个由相近自交系构建的遗传群体为材料, 利用Maize 6H-60K芯片包含的61,214个SNP位点(以下简称Maize 6H- 60K SNP位点集合)对供试材料从全基因组相似、差异度, 后代群体与亲本材料之间关系等角度, 分析Maize 6H-60K芯片对玉米实质性派生品种鉴定的有效性和精准性, 以期为我国玉米派生品种鉴定、品种知识产权保护以及品种创新等提供关键技术支撑。
1 材料与方法
1.1 供试材料
选取由北京市农林科学院玉米研究所选育或创制的2组材料, 研究分析Maize 6H-60K SNP位点集对于鉴定玉米EDV品种/材料的有效性和精准性。一组是自有知识产权的优良自交系京2416、京2416C。京2416具有抗旱、抗倒伏、配合力高、熟期早、灌浆速率高、脱水快等优点, 已成为玉米育种广泛应用的骨干自交系, 利用京2416已组配选育出京农科728、NK718、MC121等优良玉米新品种20多个[18-19]。以京2416为受体、高抗咪唑类除草剂玉米种质为供体, 通过多代回交结合分子育种和严格的田间鉴选, 选育获得抗咪唑类除草剂的自交系京2416C。另一组是由自主知识产权的优良自交系京724、京72464构建的DH系(doubled haploid lines)遗传群体, 共包含893个DH系。京724和京72464同为X杂优群的相近优良自交系, 40个SSR位点中差异位点数为6个, 形态性状差异主要表现在穗位高、雄穗分枝数、生育期(开花期)以及果穗性状的不同[20-21]。京724具有抗病抗逆性强、籽粒品质优、配合力高等优点, 已组配出京科968、MC738等优良杂交种。京72464为京724与京464同群优系聚合选育的优良自交系, 已组配育成MC121、MC817等玉米新品种。
1.2 总DNA提取
采用改良CTAB法提取总DNA, 并除去RNA[20]。用1%的琼脂糖凝胶电泳和紫外分光光度计测定DNA质量和浓度(Nanodrop 2000, Thermo Scientific),工作液浓度统一调整为50 ng µL–1。所制备的DNA无蛋白质、RNA以及其他杂质污染, 琼脂糖凝胶电泳显示DNA条带单一, 无降解。
1.3 利用Maize6H-60K芯片对供试材料进行基因分型
Maize 6H-60K芯片为从全基因组重测序数据中挖掘评估优异位点, 集成已有芯片研究基础, 研制形成的具有高密度、高质量、高鉴别力、高灵活性、高兼容性、高性价比等特征的芯片产品[16]。本研究利用Maize 6H-60K芯片, 基于Affymetrix Axiom芯片描述试验流程, 在Gene Titan平台上对供试材料进行基因分型(Axiom 2.0 Assay for 384HT Array Format Automated Workflow User Guide rev5)。获得的原始CEL文件导入Axiom Analysis Suite软件(Affymetrix, Thermo Fisher Scientific)中, 采用“best practices”流程分析获得基因型数据, 参数设置为DQC > 0.82且QC call rate > 97%。
1.4 数据分析
利用SNP比对统计工具(V1.0) (软著登记号: 2018SR026743, 北京市农林科学院玉米研究所)统计京2416和京2416C之间、京724和京72464之间差异位点、遗传相似度参数, 分析893个DH系分别与京724、京72464之间遗传相似度参数, 893个DH系两两之间遗传相似度参数。遗传相似度值采用了相同等位基因比值(GS)和位点相似度(LS) 2种方案。GS (%) = (1–/)×100, 其中为比较总等位基因数目,为差异等位基因数目; LS (%) = (1–/)×100, 其中为比较总位点数目,为差异位点数目[17]。利用Python3语言, 采用Biopython图形库绘制京2416和京2416C之间、京724和京72464之间差异位点在染色体上的物理位置示意图(https://biopython.org/)。针对京724和京72464及其形成DH系群体, 基于Python3语言、利用Pandas数据计算库, 采用matplotlib库中的violinplot模块绘制每条染色体上的交换次数图; 采用Biopython库中的BasicChromosome分析多态性标记的分离情况。利用ChromPlot程序绘制从京724和京72464构建群体中抽取的3个DH系的基因型图谱[22]。
2 结果与分析
2.1 京2416与京2416C的全基因组遗传背景比较
京2416、京2416C两个自交系的纯合位点百分比达99.8%, 两者GS值为98.7%, 差异位点数目为829个。差异位点的物理位置呈现集中分布, 56.7%的差异位点分布在5号染色体长度约39 Mb区域(127,844,907~166,897,241 bp之间); 其次是在1号、7号和9号染色上差异位点数目分别为100、70和111个(图1)。两者SNP标记差异位点分布规律、形态性状差异以及品种选育方式均体现了派生品种的特征。
2.2 京724与京72464全基因组遗传背景比较
京724和京72464同为X群内的相近优良自交系, 基于60K芯片分析显示京724和京72464之间GS值为90.1%, 有效差异位点数目为4912个。差异位点的物理位置呈现相对集中分布, 44.8%的差异位点(2203个)分布在3号染色体上; 其次是在1号、2号、4号和9号染色上差异位点数目分别为586、363、1084和349个(图2-A, B)。
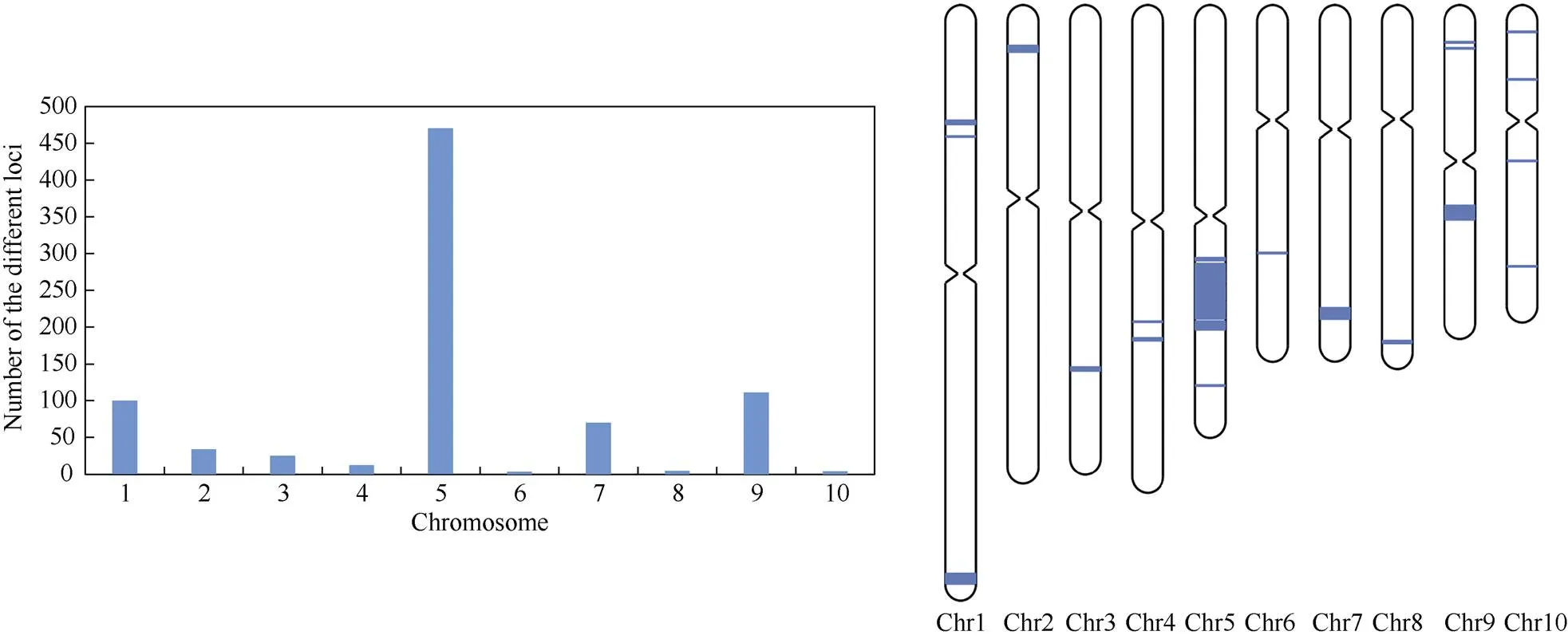
图1 京2416和京2416C之间差异位点在全基因组中的分布
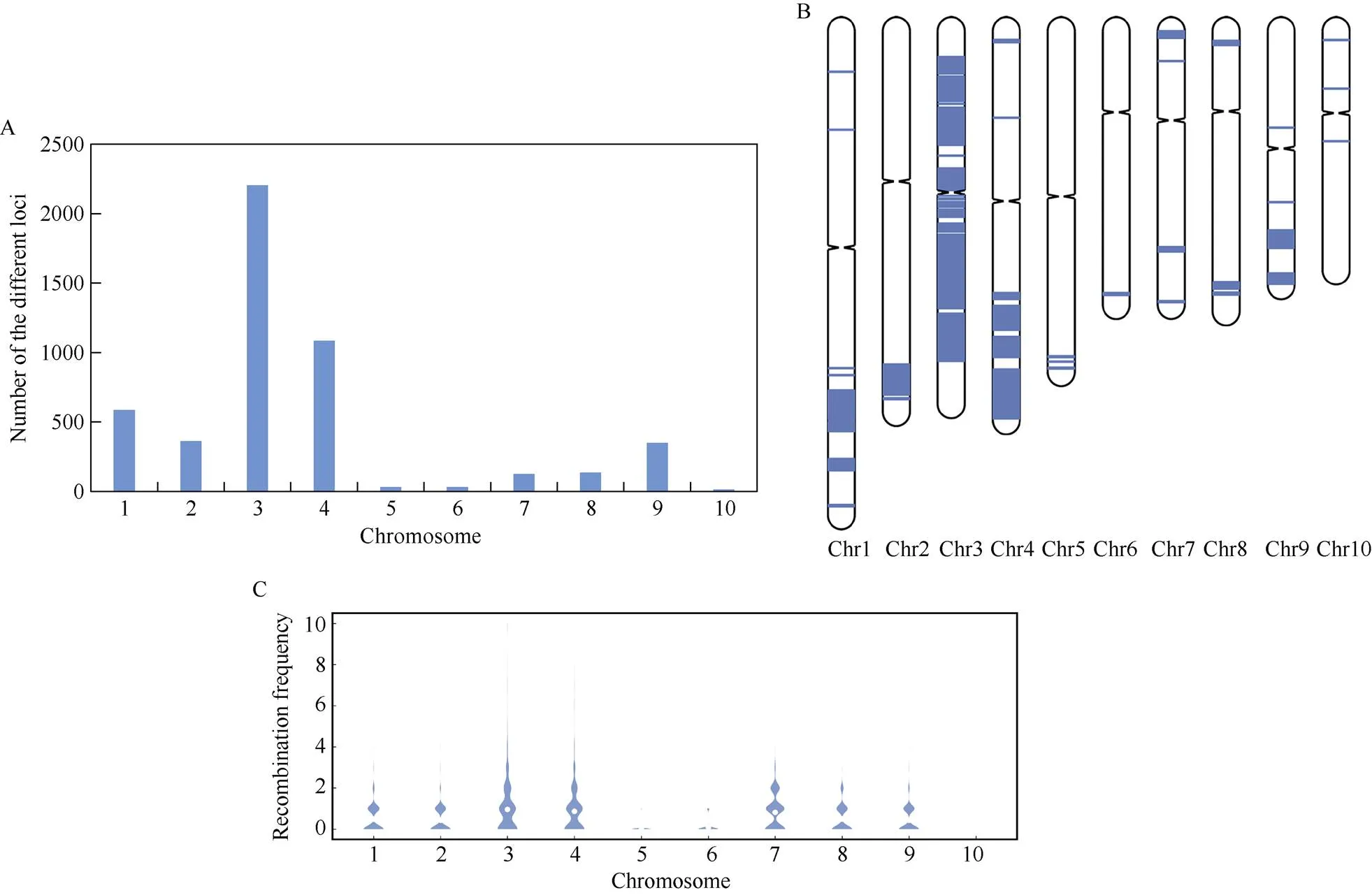
图2 京724和京72464差异位点分布(A和B)及基于DH系群体分析的染色体重组交换次数(C)
2.3 京724与京72464构建的DH群体的全基因组遗传背景分析
基于京724和京72464及其构建的DH群体在2个亲本上差异位点的基因型数据, 分析每条染色体的重组交换频次, 进而验证60K SNP标记的精准区分能力。与图2-A和B显示的京724和京72464差异位点分布一致; 3号、4号和7号染色体重组交换次数相对高, 所有DH系平均发生0.97、0.87和0.82次, 3号和4号染色体有的样本交换次数高达3次; 1号、2号、8号和9号染色体平均交换0.49、0.44、0.50和0.44次; 5号、6号和10号染色体基本没有发生交换(图2-C)。
893个后代DH系与奠基者材料京724、京72464之间遗传相似度分布情况可直观反映60K芯片对近似或派生材料的区分能力。图3显示893个DH系与2个亲本与之间的遗传相似度值分布具有连续性, 群体材料呈现了遗传背景具有偏向于父本或母本的情况、也具有介于两者之间的情况, 与实际调查的形态数据反映的规律一致。893个DH系与京724间GS值范围为88.0%~97.0%, 平均值为92.6%; LS值范围为83.7%~95.1%, 平均值为90.3%。893个DH系与京72464间GS值范围为88.3%~98.6%, 平均值为94.5%; LS值范围为82.9%~97.4%, 平均值为92.9%。
对893个DH系进行两两成对比较, 共比较398,278对, 所有两两比对样品均能被明确区分开。GS值范围为87.5%~99.9%, 平均值为94.3%; 其中GS值≥92.0%的比对对数占总比对数的89.5%, GS值≥95.0%的占比36.3%, GS值≥97.0%的占比8.6%, GS值≥99.0%的占比1.3%, GS值在92.0%~97.0%之间的占比79.7% (图4)。LS值范围为82.6%~99.9%, 平均值为93.4%; 其中LS值≥92.0%的比对对数占总比对数的74.7%, LS值≥95.0%的占比22.4%, LS值≥97.0%的占比5.2%, LS值≥99.0%的占比0.9%, LS值在92.0%~97.0%之间的占比67.9% (图4)。
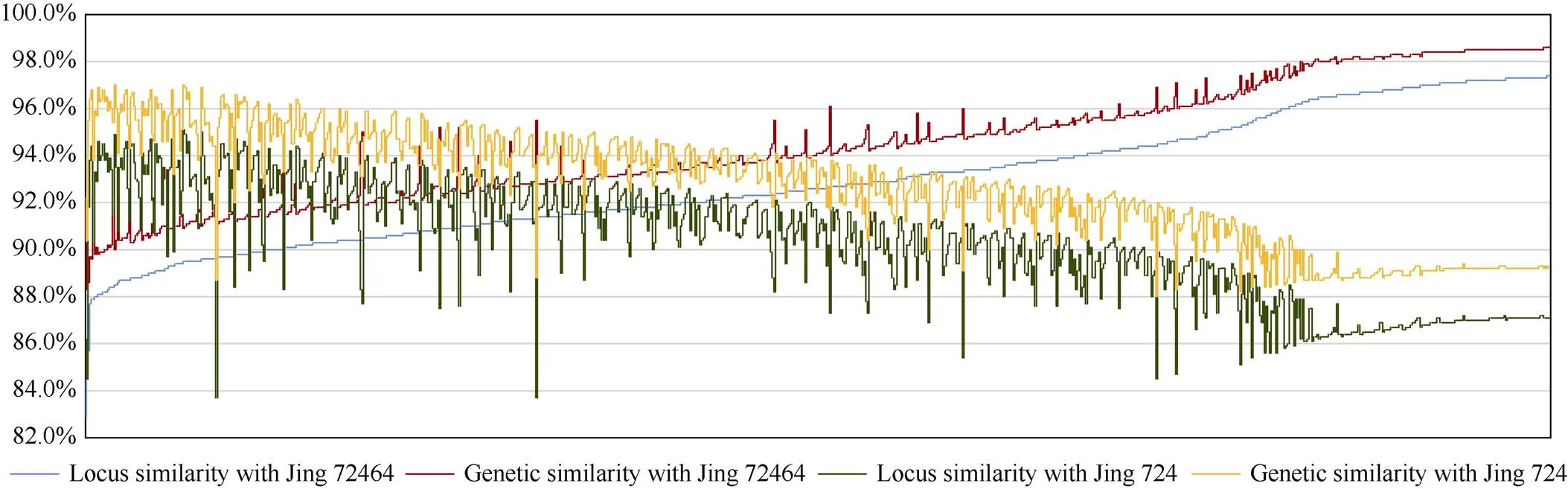
图3 893份DH系分别与京724、京72464之间遗传相似度分析
横坐标为893个DH系, 按照与京72464之间的LS值从小到大排序; 纵坐标为遗传相似度值。
The abscissa is the 893 DH lines, sorted from small to large according to the LS value with Jing 72464. The ordinate is the genetic similarity value.
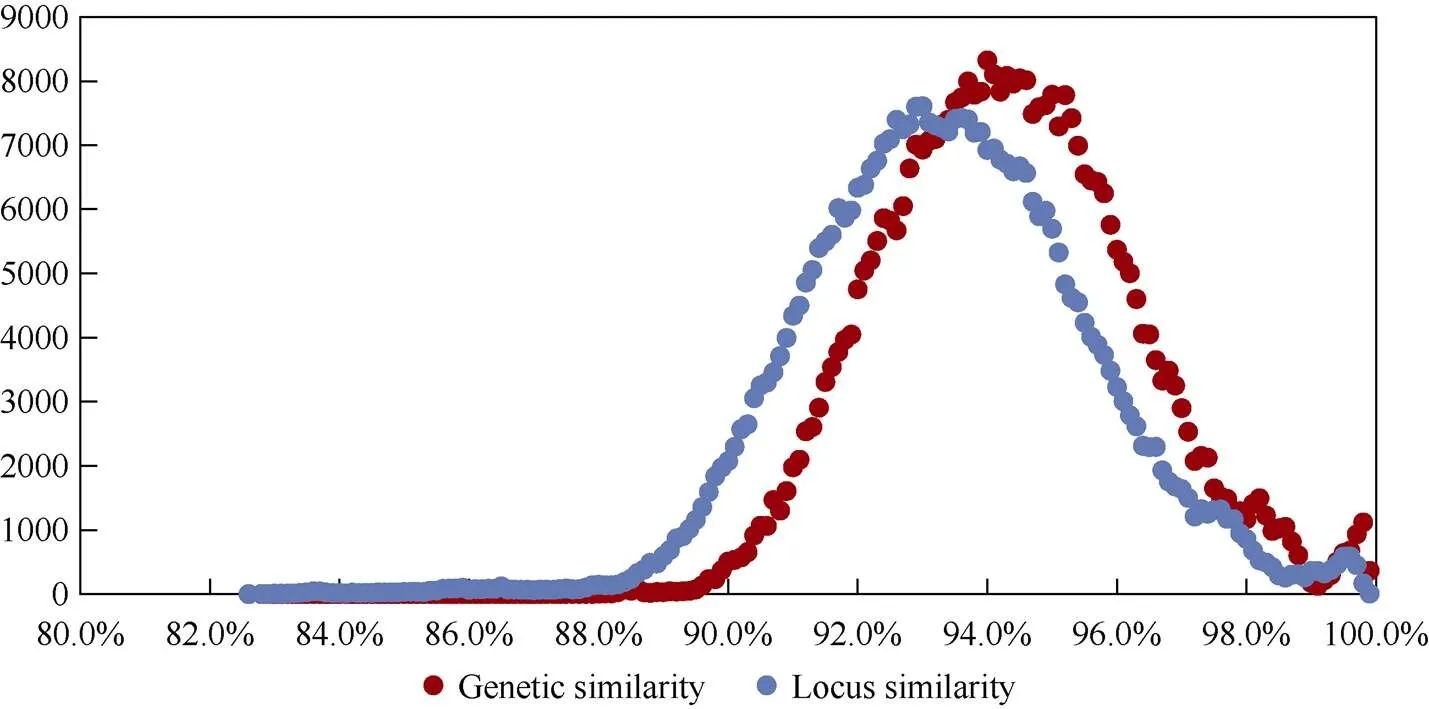
图4 893份DH系两两之间遗传相似度分布
横坐标为遗传相似度值, 纵坐标为成对分析的DH对数。
The abscissa is the value of genetic similarity and locus similarity, and the ordinate is the number of paired analysis DH lines.
2.4 京724与京72464构建DH群体的遗传分离分析
基于京724和京72464及其构建的DH群体在2个亲本上差异位点的基因型数据, 统计标记位点的遗传分离情况, 展示2个亲本的遗传贡献率, 进而验证60K SNP标记对派生或近似品种进行精准鉴定的能力。图5-A显示在双亲有差别的位点中, 88.7%的位点为1∶1分离, 11.3%的位点为偏母型分离, 少量位点的偏分离应该与DH群体的形成有关。
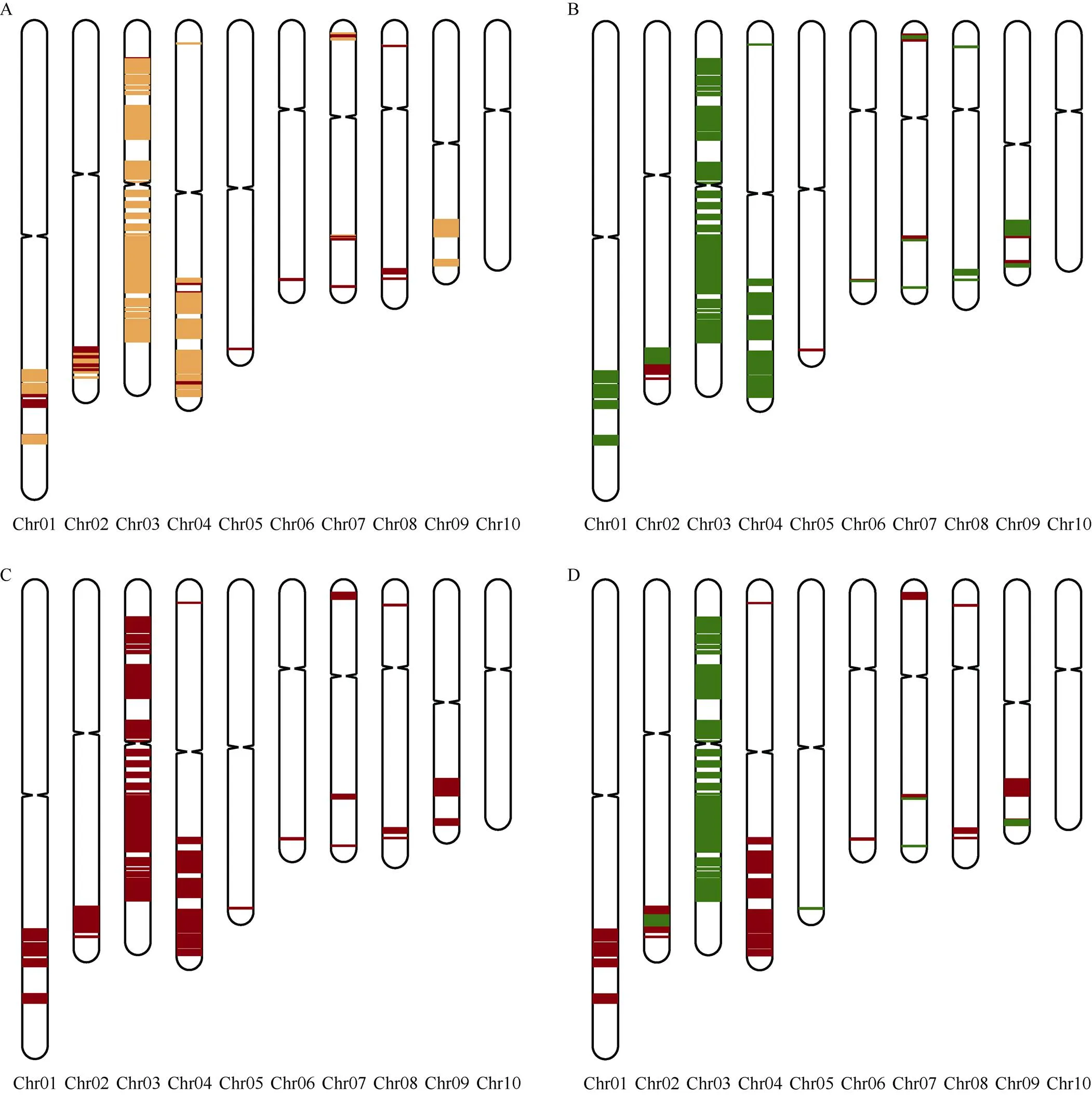
图5 京724和京72464构建的DH系群体的遗传分离图
图A为多态标记的遗传分离图, 红色为偏母型分离位点, 橙色为无偏分离位点。图B、C、D分别为DH388、DH677、DH543遗传背景图示, 其中白色区域为与京724和京72464均相同的纯合基因型, 深绿色区域为与京724相同的纯合基因型, 红色区域为与京72464相同的纯合基因型。
Fig. A is the genetic segregation diagram of polymorphic markers, red is the partial maternal segregation loci, and the orange is the unbiased segregation loci. Figs. B, C, and D are genetic backgroundof three DH lines (DH388, DH677, and DH543). Genotypes are indicated as follows: white area, the same homozygous genotype as Jing 724 and Jing 72464; green, the same homozygous genotype as Jing 724; red, the same homozygous genotype as Jing 72464.
在893个群体材料中选取了3个DH系, 图示化反映后代DH系与奠基者材料在基因组上的遗传背景情况(图5-B~D)。选取的3个DH系的纯合位点百分比均在99.5%以上, 与京724和京72464遗传相似度值代表了不同遗传分离情况。DH388遗传背景偏向于京724, GS值为97.0%; 与京72464之间GS值为90.0% (图5-B)。DH677遗传背景偏向于京72464, GS值为98.6%; 与京724之间GS值为89.2% (图5- C)。DH543与京724、京72464之间GS值分别为93.3%、93.7% (图5-D)。3个DH系在生育期、株高、穗行数与2个亲本之间形态性状偏向性与基于芯片分析的结果一致。
3 讨论
3.1 Maize6H-60KSNP位点集合在玉米实质性派生品种鉴定中的应用优势
基于Maize 6H-60K包含的SNP位点集合, 利用高效基因分型平台建立玉米实质性派生品种的鉴定技术具有以下优势。第一, 标记位点均匀覆盖全基因组, 不仅可以评估原始和派生品种之间的遗传相似度, 而且还可以挖掘与派生性状连锁的标记位点, 从而进一步验证派生品种特征。品种派生关系鉴定是分析待测样品是独立选育品种还是与已申请品种权保护品种存在派生关系, 以准确评价品种间遗传组成相似程度为目标; 所选用的标记位点具有全基因组均匀分布、兼容多平台特点, 数目一般数千至数万个[23]。欧美玉米派生品种鉴定选用了3072个SNP位点, 与ISF颁布的基于SNP标记进行玉米派生品种鉴定指南中选用的标记相同; 基于MNP标记法的玉米派生品种鉴定推荐了935个MNP标记[8-10]。最适标记位点集合需选用具有广泛代表性的材料进行评估确定, 理论上代表性标记数目越大, 遗传相似度评估结果越准确, 即需要代表全基因组的标记集合[12]。Maize 6H-60K SNP位点集是利用国内外代表性材料进行评估筛选的, 位点具有广适性尤其是能够精准评估国内玉米品种、材料[16]。同时6万个位点是基于物理和遗传位置相对均匀选取的, 具有覆盖全基因组特征; 根据转换的HTP (haplotype-tag polymorphism)标记统计覆盖了B73参考基因组的98.85%[24]。在本研究中Maize 6H-60K位点集合能够精准评估极近似自交系京2416和京2416C的遗传相似度, 两者差异位点显示了集中分布特征, 可进一步锁定与派生性状连锁标记。第二, 标记位点具有高质量和高品种鉴别力。基因分型完美位点占比90%以上, 位点重复一致性达99%以上, 90%以上位点的PIC值(多态信息含量)大于0.2, 可区分玉米已知品种[16]。本研究结果显示该芯片可以精准分析京724、京72464及群体DH系群体的遗传背景, 重组交换和遗传分离情况; 精准区分、评估遗传相似度值在82%~99%之间的每个DH系, 并且结果与形态性状差异和育种过程方法高度一致。第三, 可基于高效基因分型平台如芯片、靶向测序(genotyping by target sequencing, GBTS), 建立高通量、标准化、低成本的检测技术流程[25]。在样本通量上, 一次实验流程可分析数百、数千个样品, 6万多个位点的分型试验, 产生指纹数据近亿; 可集成工作站与数据管理系统, 实现试验流程、基因型数据分析的自动化和标准化; 相同位点数量级别下, 性价比高。
3.2 玉米实质性派生品种分子鉴定迫切需要研究和解决的关键问题
自1997年发布实施植物新品种保护条例以来, 我国品种权保护相关制度逐步完善, 工作体系不断健全, 品种保护意识持续提高[26-28]。尤其是近年来种业知识产权日益受到重视, 国家在各个层面作了系列重要部署。2021年中央一号文件中强调“打好种业翻身仗, 加强育种领域知识产权保护”; 《种业振兴行动方案》提出“加强知识产权保护, 优化营商环境”等。2022年3月颁布实施新修改的《种子法》, 将实质性派生品种概念纳入法律范畴, 育种者权利的范围由受保护品种延伸至受保护品种的实质性派生品种。EDV制度的实施将对种业健康发展起到正确的引导作用, 同时也给EDV分子鉴定技术带来了空前的机遇和挑战。
目前, 国内关于实质性派生品种的研究比较多, 但是多数为基于法律、政策角度的讲述, 与分子鉴定技术相关报道较少。玉米实质性派生品种分子鉴定技术的成熟应用仍需要解决以下核心关键问题[29-30]。
第一, 标记类型、优异标记集合、分型平台的确定。综合AFLP、SSR、SNP等标记特点, SNP标记具有全基因组均匀分布、密度高、数据易整合共享以及与功能基因关联性强等优势被推荐为理想标记, 可利用的分型平台有芯片、靶向测序(GBTS)、GBS (genotyping by sequencing)、重测序等[25]。重测序方法存在不同批次挖掘获取的位点不同, 数据分析周期长、且对硬件和人员要求高等因素而不是最适合的分型方法。即使在开始研究时利用的是重测序方法, 但是最后也需要评估确定一套优异位点集合[31]。GBS分型方法同样存在不同试验批次间获取标记位点集合的差异性的问题。基于芯片或靶向测序(GBTS)分型平台是利用特异探针获得固定位点集合,可以分析数千、数万乃至几十万标记, 具有高通量、自动化、标准化特点。
第二, 遗传相似度评估参数的选择。评估品种间遗传相似度参数有多种, 如LS (相同位点即相同基因型百分比)、GS (相同等位基因百分比)、IS (特性分值Identity Scores)、IBD (Identity by Descent), 以及基于Rogers、Jaccard遗传距离获得的相似度值等[7-8,12,31-34]。选择参数不同分析获得的遗传相似度值则不同, 但是均表现为与品种间相似程度呈正相关性。IS是一种计算样品间DNA序列一致性程度的分析方法, 是基于群体遗传学开发的相同起源基因比较的特性分值方法[31-32]。IBD参数为血缘同一性分析, 偏向于系谱追踪、重建的分析。基于Jaccard遗传距离评估相似度值对于显性标记如AFLP是适合的, 但是对于共显性标记SSR、SNP则不适合[34]。ISF发布的玉米EDV品种鉴定指南选用的是基于Rogers遗传距离评估样本间的相似度[7-8]。LS、GS两个参数均是以所有位点的权重都是相同的情况下进行计算, 当位点纯合率达到100%时LS和GS值相同, 否则GS值大于LS值。“玉米真实性鉴定SNP标记法”和“植物品种鉴定MNP标记法”标准均推荐的LS参数, 即利用位点或者基因型进行统计[10,17]。综合各参数特点, 建议选择LS、GS以及Rogers遗传距离3个评价指标开展后续研究, 确定最适于我国玉米派生品种鉴定的遗传相似度评估参数。
第三, EDV品种判定阈值线的确定。派生品种与原始品种相似度达到多少可判定为EDV, 相似度低于多少可判定为独立品种。ISF发布的基于SSR和SNP标记的玉米EDV鉴定指南中给出的阈值线为82%、90%和91%、95%, 国内还没有统一的判定标准[7-8,30,33]。判定阈值标准需综合考虑已知品种间的遗传背景、品种内变异、育种创新程度、技术误差等因素, 需平衡原始品种权与后续育种开发者之间利益, 通过充分征求意见之后设定一个科学合理、达成共识的判定阈值。为了从多个角度展示品种之间的关系, 出具EDV分子鉴定结果时除了遗传相似度参数值之外, 建议出具差异位点在基因组上分布图, 直观展示2个品种之间差异分布情况, 如本文中展示的京2416和京2614C, 京724和京72464差异位点分布。
第四, 申请或获得植物新品种权的玉米自交系指纹库构建。基于40个SSR标记和96个SNP标记, 已经系统构建了玉米杂交种指纹数据并面向全国公开共享, 为品种真实性鉴定、打击假冒伪劣套牌侵权提供技术支撑。建议基于Maize 6H-60K SNP位点集合建立申请或获得植物新品种权的玉米自交系的标准指纹, 有利于待测样品与标准原始品种指纹的直接比较, 为开展玉米EDV品种鉴定提供核心数据。同时对自交系指纹数据进行系统深入分析有利于阈值线的确定, 有利于摸清玉米遗传资源情况, 为品种持续创新提供数据支撑[31]。
第五, 在上述问题都解决的情况下, 如有详细的系谱、育种方法的记录数据, 则更有利于结果的综合判定, 然而目前准确详细的系谱等数据缺乏也是一个关键问题[29]。
概而言之, 在种业市场活跃、系列政策支持的情况下, 实质性派生品种分子鉴定技术为其发挥优势作用的关键时期。迫切需要基于高密度高质量标记如Maize6H-60K SNP位点集合等, 对核心问题进行充分广泛的研究, 系统全面建立鉴定技术规范, 为玉米种业创新提供技术支持。
4 结论
利用派生品种、相近自交系构建的遗传群体材料, 从多角度分析证明Maize 6H-60K SNP位点集合能够精准评估玉米派生、近似或极近似材料的遗传相似度, 并具有进一步锁定与派生性状连锁标记的潜力, 相关分析结果与形态、系谱或育种记录一致。建议基于高密度高质量SNP标记集合, 利用高效芯片、靶向测序(GBTS)等平台开展玉米实质性派生品种鉴定的核心问题研究, 建立鉴定技术规程, 为玉米品种知识产权保护、品种创新等提供关键技术支撑。
[1] 褚云霞, 陈海荣, 邓姗, 黄志城, 李寿国. 实质性派生品种鉴定方法研究进展. 上海农业学报, 2017, 33(5): 132–138. Chu Y X, Chen H R, Deng S, Huang Z C, Li S G. Development of the identification of essentially derived varieties., 2017, 33(5): 132–138 (in Chinese with English abstract).
[2] Heckenberger M, Bohn M, Ziegle J S, Joe L K, Hauser J D, Hutton M, Melchinger A E. Variation of DNA fingerprints among accessions within maize inbred lines and implications for identification of essentially derived varieties: I. Genetic and technical sources of variation in SSR data., 2002, 10: 181–191.
[3] Heckenberger M, Voort J R, Melchinger A E, Peleman J, Bohn M. Variation of DNA fingerprints among accessions within maize inbred lines and implications for identification of essentially derived varieties: II. Genetic and technical sources of variation in AFLP data and comparison with SSR data., 2003, 12: 97–106.
[4] Heckenberger M, Bohn M, Klein D, Melchinger A E. Identification of essentially derived varieties obtained from biparental crosses of homozygous lines: II. Morphological distances and heterosis in comparison with simple sequence repeat and amplified fragment length polymorphism data in maize., 2005, 45: 1132–1140.
[5] Heckenberger M, Muminović J, Voort J R, Peleman J, Bohn M, Melchinger A E. Identification of essentially derived varieties obtained from biparental crosses of homozygous lines: III. AFLP data from maize inbreds and comparison with SSR data., 2006, 17: 111–125.
[6] Kahler A L, Kahler J L, Thompson S A, Ferriss R S, Jones E S, Nelson B K, Mikel M A, Smith S. North American study on essential derivation in maize: II. Selection and evaluation of a panel of simple sequence repeat loci., 2010, 50: 486–503.
[7] ISF. Guidelines for Handling Disputes on Essential Derivation of Maize Lines, 2008. www.worldseed.org.
[8] ISF. Guidelines for Handling Disputes on Essential Derivation of Maize Lines, 2014. www. worldseed.org.
[9] Rousselle Y, Jones E, Charcosset A, Moreau P, Robbins K, Stich B, Knaak C, Flament P, Karaman Z, Martinant J R, Fourneau M, Taillardat A, Romestant M, Tabel C, Bertran J, Ranc N, Lespinasse D, Blanchard P, Kahler A, Chen J, Kahler J, Dobrin S, Warner T, Ferris R, Smith S. Study on essential derivation in maize: III. Selection and evaluation of a panel of single nucleotide polymorphism loci for use in European and North American Germplasm., 2015, 55:1170–1180.
[10] 彭海, 方治伟, 李论, 马爱进, 周俊飞, 温常龙, 李甜甜, 唐浩,陈红, 崔野韩, 张嘉楠, 贾英民, 许娜, 宋书锋, 胡美霞, 符习勤, 赵治海, 梁勇, 徐振江, 高利芬, 陈利红, 韩瑞玺, 张蝶, 张静, 余进文. 植物品种鉴定MNP标记法. 中华人民共和国国家标准, GB/T 38551–2020, 2020. Peng H, Fang Z W, Li L, Ma A J, Zhou J F, Wen C L, Li T T, Tang H, Chen H, Cui Y H, Zhang J N, Jia Y M, Xu N, Song S F, Hu M X, Fu X Q, Zhao Z H, Liang Y, Xu Z J, Gao L F, Chen L H, Han R X, Zhang D, Zhang J, Yu J W. Identification of plant varieties: MNP marker method. National Standards of the People’s Republic of China, GB/T 38551–2020, 2020 (in Chinese).
[11] UPOV (International Union for the Protection of New Varieties of Plants). Possible Used of Molecular Markers in the Examination of Distinctness, Uniformity and Stability (DUS). Geneva, Switzerland: UPOV, 2011.
[12] 徐云碧, 王冰冰, 张健, 张嘉楠, 李建生. 应用分子标记技术改进作物品种保护和监管. 作物学报, 2022, 48: 1853–1870. Xu Y B, Wang B B, Zhang J, Zhang J N, Li J S. Enhancement of plant variety protection and regulation using molecular marker technology., 2022, 48: 1853–1870 (in Chinese with English abstract).
[13] Ganal M W, Durstewitz G, Polley A, Bérard A, Buckler E S, Charcosset A, Clarke J D, Graner E M, Hansen M, Joets J, Paslier M C L, McMullen M D, Montalent P, Rose M, Schön C C, Sun Q, Walter H, Martin O C, Falque M. A large maize (L.) SNP genotyping array: development and germplasm genotyping, and genetic mapping to compare with the B73 reference genome., 2011, 6: e28334.
[14] Unterseer S, Bauer E, Haberer G, Seidel M, Knaak C, Ouzunova M, Meitinger T, Strom T M, Fries R, Pausch H, Bertani C, Davassi A, Mayer K F, Schön C C. A powerful tool for genome analysis in maize: development and evaluation of the high density 600k SNP genotyping array.s, 2014, 15: 823.
[15] Xu C, Ren Y H, Jian Y Q, Guo Z F, Zhang Y, Xie C X, Fu J J, Wang H W, Wang G Y, Xu Y B, Li P, Zou C. Development of a maize 55K SNP array with improved genome coverage for molecular breeding., 2017, 37: 20.
[16] Tian H L, Yang Y, Yi H M, Xu L W, He H, Fan Y M, Wang L, Ge J R, Liu Y W, Wang F G, Zhao J R. New resources for genetic studies in maize (L.): a genome-wide Maize6H-60K single nucleotide polymorphism array and its application., 2021, 105: 1113–1122.
[17] 王凤格, 晋芳, 田红丽, 易红梅, 赵久然, 金石桥, 杨扬, 王蕊,葛建镕, 支巨振, 赵建宗. 玉米品种真实性鉴定SNP标记法. 中华人民共和国农业行业标准, NY/T 4022–2021, 2021. Wang F G, Jin F, Tian H L, Yi H M, Zhao J R, Jin S Q, Yang Y, Wang R, Ge J R, Zhi J Z, Zhao J Z. Maize (L.) variety genuineness identification: SNP based method. Agricultural Industry Standards of the People’s Republic of China, NY/T 4022-2021, 2021 (in Chinese).
[18] 赵久然, 王元东, 宋伟, 张如养, 李春辉, 刘新香. 玉米骨干自交系京2416的选育与应用. 植物遗传资源学报, 2020, 21: 1051–1057. Zhao J R, Wang Y D, Song W, Zhang R Y, Li C H, Liu X X. Breeding and application of maize founder inbred line Jing 2416., 2020, 21: 1051–1057 (in Chinese with English abstract).
[19] 赵久然, 李春辉, 宋伟, 刘新香, 王元东, 张如养, 王继东, 孙轩, 王夏青. 玉米骨干自交系京2416杂种优势及遗传重组解析. 中国农业科学, 2020, 53: 4527–4536. Zhao J R, Li C H, Song W, Liu X X, Wang Y D, Zhang R Y, Wang J D, Sun X, Wang X Q. Heterosis and genetic recombination dissection of maize key inbred line Jing 2416., 2020, 53: 4527–4536 (in Chinese with English abstract).
[20] 王凤格, 易红梅, 赵久然, 刘平, 张新明, 田红丽, 堵苑苑. 玉米品种鉴定技术规程SSR标记法. 中华人民共和国农业行业标准, NY/T 1432–2014, 2014. Wang F G, Yi H M, Zhao J R, Liu P, Zhang X M, Tian H L, Du Y Y. Protocol for the Identification of Maize Varieties: SSR Marker Method. Agricultural Industry Standards of the People’s Republic of China, NY/T 1432-2014, 2014 (in Chinese).
[21] 赵久然, 李春辉, 宋伟, 王元东, 张如养, 王继东, 王凤格, 田红丽, 王蕊. 基于SNP芯片揭示中国玉米育种种质的遗传多样性与群体遗传结构. 中国农业科学, 2018, 51: 626–634. Zhao J R, Li C H, Song W, Wang Y D, Zhang R Y, Wang J D, Wang F G, Tian H L, Wang R. Genetic diversity and population structure of important Chinese maize breeding germplasm revealed by SNP-Chips., 2018, 51: 626–634 (in Chinese with English abstract).
[22] Oróstica K Y, Verdugo R A. ChromPlot: visualization of genomic data in chromosomal context., 2016, 32: 2366–2369.
[23] 赵久然, 王凤格, 田红丽, 易红梅, 王蕊, 葛建镕. 适于农作物品种分子身份鉴别和确权鉴定的检测方法. 中国发明专利, 2017, ZL 201710527354.5. Zhao J R, Wang F G, Tian H L, Yi H M, Wang R, Ge J R. Detection Methods for Identity Distinguish and Intellectual Property Confirmation of Crop Varieties Using Molecular Markers. Chinese Invention Patent, 2017, ZL 201710527354.5 (in Chinese).
[24] Zhao Y K, Tian H L, Li C H, Yi H M, Zhang Y L, Li X H, Zhao H, Huo Y X, Wang R, Kang D M, Lu Y C, Liu Z H, Liang Z Y, Xu L W, Yang Y, Zhou L, Wang T Y, Zhao J R, Wang F G. HTPdb and HTPtools: exploiting maize haplotype-tag polymorphisms for germplasm resource analyses and genomics-informed breeding., 2022, 3: 100331.
[25] 徐云碧, 杨泉女, 郑洪建, 许彦芬, 桑志勤, 郭子锋, 彭海, 张丛, 蓝昊发, 王蕴波, 吴坤生, 陶家军, 张嘉楠. 靶向测序基因型检测(GBTS)技术及其应用. 中国农业科学, 2020, 53: 2983–3004. Xu Y, Yang Q N, Zheng H J, Xu Y F, Sang Z Q, Guo Z F, Peng H, Zhang C, Lan H F, Wang Y B, Wu K S, Tao J J, Zhang J N. Genotyping by target sequencing (GBTS) and its applications., 2020, 53: 2983–3004.
[26] 崔野韩, 温雯, 陈红, 杨扬, 堵苑苑, 卢新. 我国农业植物新品种保护工作回顾与展望. 中国种业, 2019, (2): 9–11. Cui Y H, Wen W, Chen H, Yang Y, Du Y Y, Lu X. Review and respect of the plant new variety protection in China., 2019, (2): 9–11 (in Chinese).
[27] 邓伟, 崔野韩. 中国农业植物新品种保护制度及发展的研究. 中国种业, 2020, (11): 1–7. Deng W, Cui Y H. Study on the protection system and development of the plant new variety protection in China., 2020, (11): 1–7 (in Chinese).
[28] 温雯, 闫东哲, 刘衎, 崔野韩. 健全我国农业植物新品种保护制度体系的思考. 农业科技管理, 2022, 41(1): 71–75. Wen W, Yan D Z, Liu K, Cui Y H. Considerations on perfecting agricultural protection system of new plant varieties in China., 2022, 41(1): 71–75 (in Chinese with English abstract).
[29] Smith J S C. The future of essentially derived variety (EDV) status: predominantly more explanations or essential change., 2021, 11: 1261.
[30] 万志前, 张媛. 实质性派生品种制度的缘起、困境与困应. 浙江农业学报, 2020, 32: 2067–2076. Wan Z Q, Zhang Y. Origin, implementation difficulties and countermeasures of essential derived variety system., 2020, 32: 2067–2076 (in Chinese with English abstract).
[31] 张上都, 袁定阳, 路洪凤, 简燕, 李秀欣, 黄安平, 罗正良, 吕启明, 谭炎宁, 张勇飞, 袁隆平, 柏连阳. 基因组学方法用于水稻种质资源实质派生的检测结果和应用讨论. 中国科学: 生命科学, 2020, 50: 633–649. Zhang S D, Yuan D Y, Lu H F, Jian Y, Li X X, Huang A P, Luo Z L, Lyu Q M, Tan Y N, Zhang Y F, Yuan L P, Bai L Y. The results of rice germplasm EDV test by genomic analysis and related discussions., 2020, 50: 633–649 (in Chinese with English abstract).
[32] 简燕, 李小波, 王博, 赵静, 索海翠, 黄安平, 胡柏耿, 曹春梅,张勇飞. 马铃薯实质派生品种鉴定的基因组学技术. 中国马铃薯, 2020, 34: 321–328. Jian Y, Li X B, Wang B, Zhao J, Suo H C, Huang A P, Hu B G, Cao C M, Zhang Y F. Genomics technique for detection of potato essentially derived variety., 2020, 34: 321–328 (in Chinese with English abstract).
[33] 田红丽, 杨扬, 王璐, 王蕊, 易红梅, 许理文, 张云龙, 葛建镕,王凤格, 赵久然. 兼容型maizeSNP384标记筛选与玉米杂交种DNA指纹图谱构建. 作物学报, 2020, 46: 1006–1015.Tian H L, Yang Y, Wang L, Wang R, Yi H M, Xu L W, Zhang Y L, Ge J R, Wang F G, Zhao J R. Screening of compatible maizeSNP384 markers and the construction of DNA fingerprints of maize varieties., 2020, 46: 1006–1015 (in Chinese with English abstract).
[34] Noli E, Teriaca M S, Conti S. Criteria for the definition of similarity thresholds for identifying essentially derived varieties., 2013, 132: 525–531.
Application of maize 6H-60K chip in identification of maize essentially derived varieties
TIAN Hong-Li**, ZHANG Ru-Yang**, FAN Ya-Ming**, YANG Yang, ZHANG Yun-Long, YI Hong-Mei,XING Jin-Feng, WANG Feng-Ge*, and ZHAO Jiu-Ran*
Maize Research Institute, Beijing Academy of Agriculture and Forestry Sciences / Key Laboratory of Crop DNA Fingerprinting Innovation and Utilization (Co-construction by Ministry and Province) / Beijing Key Laboratory of Maize DNA Fingerprinting and Molecular Breeding, Beijing 100097, China
Theidentification of maize essentially derived variety has become the hot spot in the intellectual property protection of seed industry. In order to speed up the establishment of its accurate and efficient molecular identification technology, this article used multiple types of derived varieties as research materials: Jing 2416 and Jing 2416C (two inbred lines with highly similar genetic backgrounds), Jing 724 and Jing 72464 (two inbred lines with similar genetic backgrounds), as well as 893 DH lines of genetic population constructed by Jing 724 and Jing 72464. The study researched and analyzed the potential of maize 6H-60K chip including 61,214 SNPs in identification of maize essentially derived varieties. The results were as follows: (1) There were 829 SNPs differences between Jing 2416 and Jing 2416C, the GS value (genetic similarity) was 98.7%, and 56.7% of the difference loci were concentrated in the 39 Mb region of chromosome 5. (2) There were 4912 SNPs differences between Jing 724 and Jing 72464, the GS value was 90.1%, and 44.8% of the difference loci were concentrated on chromosome 3. (3) The distribution of genetic similarity values between 893 DH lines and two parents (Jing 724 and Jing 72464) was continuous. The GS value range between 893 DH lines and Jing 724 was 88.0%–97.0%, with an average of 92.6%. The GS value range between 893 DH lines and Jing 72464 was 88.3%–98.6%, with an average of 94.5%. (4) 893 DH lines were paired comparison, a total of 398,278 pairs were compared. There were specific SNP differences between all DH lines. The GS values of 893 DH lines in pairs ranged from 87.5% to 99.9%, with an average of 94.3%. Among them, the proportion of GS value ≥ 97.0% was 8.6%, and the proportion of GS value ≥ 99.0% was 1.3%. This study showed that maize 6H-60K SNP sets could accurately evaluate the genetic background of maize derived, similar or extremely similar inbred and DH lines, identify and distinguish all materials one by one, and had the potential to further lock the linkage markers of derived traits. It is suggested that the technical system for maize essentially derived variety molecular identification based on Maize6H-60K SNP sets using chip, genotyping by target sequencing (GBTS) and other platforms should be urgently established, so as to provide technical support for intellectual property protection and variety innovation of maize varieties.
maize; the essentially derived variety; molecular identification; maize 6H-60K chip; the high-density SNP loci set
10.3724/SP.J.1006.2023.23066
本研究由国家科技创新重大项目(2022ZD04019)和北京市农林科学院创新能力建设专项(KJCX202303)资助。
This study was supported by the National Scientific and Technological Innovation—Major Projects (2022ZD04019) and the Science and Technology Innovation Capacity Building Project of BAAFS (KJCX20230301).
赵久然, E-mail: maizezhao@126.com; 王凤格, E-mail: gege0106@163.com
**同等贡献(Contributed equally to this work)
田红丽, E-mail: tianhongli9963@163.com; 张如养, E-mail: ruyangzhang2009@126.com; 范亚明, E-mail: 13718078547@163.com
2022-09-30;
2023-05-24;
2023-06-06.
URL: https://kns.cnki.net/kcms2/detail/11.1809.S.20230605.1342.002.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
