基于U1-net网络的放疗脑肿瘤靶区分割
2023-09-05张本健王桂林
张本健, 林 辉, 郭 栋, 王桂林, 胡 敏
(1.合肥工业大学 电子科学与应用物理学院,安徽 合肥 230601; 2.合肥工业大学 计算机与信息学院,安徽 合肥 230601)
0 引 言
脑神经多形性胶质母细胞瘤(glioblastoma multiforme,GBM)是最常见的一类脑肿瘤,分为高级胶质瘤(high grade glioma,HGG)和低级胶质瘤(low graded glioma,LGG),其中LGG又分为低级星型细胞瘤和少突神经胶质瘤。HGG寿命通常不超过2 a,且一经发现需要立即治疗;LGG具有几年的预期生存期,积极治疗通常可以延长患者的寿命。手术切除是脑肿瘤治疗最常见的手段,此外,放疗、化疗可用于减缓不能手术移除的肿瘤组织。放疗作为肿瘤治疗的一种手段,主要通过电离辐射直接或间接作用肿瘤细胞,在照射的区域内沉积一定的能量从而达到破坏“靶区”肿瘤细胞的作用。
磁共振成像(magnetic resonance imaging, MRI)是一种断层功能影像成像方法,具有高软组织成像分辨率、无电离辐射损伤等优点,在肿瘤的诊断、治疗过程中发挥着重要作用。MRI通过在图像采集期间改变激发脉冲强度和重复时间,以生成4种包含T1加权成像(T1-weighted,T1)、增强T1加权成像(T1 with post-contrast,T1c)、T2加权成像(T2-weighted,T2)和液体衰减翻转恢复(fluid attenuated inversion recovery,FLAIR)的4个相关联模态图像。将这些不同模态MRI图像相结合,可提供肿瘤形状、大小、位置等内部结构信息。为改进放疗的实施精度,近年来出现了MRI图像引导的自适应放射治疗(MRI-guided adaptive radio therapy,MRIgART),即利用MRI多模态图像进行放疗过程中的图像引导。
文献[1-2]对精准放射治疗关键物理与技术进行了研究,开发了具有完全自主知识产权的精准放疗计划系统 KylinRay“麒麟刀”,已通过国家药监局认证检验。该系统发展了高精度剂量计算、快速计划优化、患者智能摆位、多维精准建模、射束精准调控、实时剂量反演与验证等一系列放疗关键技术,提出了“精准计划、精准定位、精准照射、精准测评”耦合的四精放疗技术体系,可为肿瘤放射治疗的全过程提供高效解决方案。MRIgART是KylinRay“麒麟刀”目前正在研发的产品之一,属于其前端基于先进人工智能深度学习的靶区自动勾画方法的研究。
肿瘤放疗靶区勾画在本质上属于图像分割,肿瘤放疗优质计划建立的前提是对放疗靶区的准确勾画。目前临床上常用的方法是医师的手工勾画,工作量巨大且具有很强的人为主观性,且脑肿瘤的形状多样性、位置不固定、结构复杂以及在不同患者身上具有非常大的差异,给人工分割脑肿瘤带来了许多实际困难,因此自动分割脑部肿瘤的研究具有重要的应用价值。
深度学习(deep learning,DL)是近年来兴起的一项人工智能技术,无论在科研还是其他领域都受到广泛的关注,并在医学肿瘤图像领域得到了应用。基于深度学习的医学图像分割方法不需要人工设计特征,而是由模型自己去学习数据中的深层信息,以加快诊断的速度与提升诊断的准确度,避免人为因素造成的误诊。肿瘤放疗优质计划建立的前提是对放射靶区的准确勾画,手工勾画工作量巨大且具有很强的人为主观性。利用深度学习方法辅助肿瘤靶区的自动勾画是人工智能技术在医学领域的渗透与应用。
深度学习中最典型的算法是卷积神经网络(convolutional neural network,CNN)[3],CNN是一种基于深度学习理论的多阶段全局可训练模型,可以从图像中学习深层的特征结构,获取高维的图像特征,对自然图像的识别与定位非常有效。目前,脑肿瘤分割方面的深度学习研究,主要集中在网络模型结构设计上[4]。文献[5]建立了多通道的网络结构,每次通过获取肿瘤图片的局部特征对每个像素分类,经过反复训练后恢复1张完整的分割图;文献[3]基于CNN完成脑肿瘤的分割任务,在其设计的网络中使用较小的3×3的卷积核,该模型取得了较好的结果;文献[6]提出将深度CNN框架用于脑肿瘤的分割。通过训练2个CNN模型分别对恶性胶质瘤和良性胶质瘤进行分割,提高了神经网络的处理速度,在CNN模型中使用尺寸较小的卷积核,不仅提高了速度,更有利于图像特征的获取。因为脑肿瘤MRI图像是三维结构,所以一些文献也提出了3D-CNN应用于脑肿瘤分割任务,例如文献[7]提出了用三维图像作为CNN模型的输入来训练三维CNN模型。这种3D-CNN模型虽然保留了MRI图像的空间信息,但是很大程度上增加了计算量,且训练速度慢。
CNN提取的抽象特征对图像进行分类,对图像中包含的对象进行分类,以及对图像中对象的粗略定位是非常有效的,然而由于采用了感受野,对图像特征的提取更多地是以“1小块临域”为单位的,很难做到精细(像素级)的分割,不能准确地划定物体具体的轮廓。鉴于CNN在图像精细分割方面的局限性,文献[8]提出了将全卷积网络用于语义分割问题的研究。
原始的U-net网络正是基于全卷积神经网络(fully convolutional network,FCN)的架构,是文献[9]提出使用编码-解码的结构,U-net网络与FCN都可应用于很小的数据集,网络最初提出时是用于细胞壁的分割,并且在肺结节检测和眼底视网膜上的血管提取等方面都有着出色的表现。U-net的优点有:① 通过对每个像素点进行分类,获得了更高的分割准确率;② 支持少量数据训练模型;③ 可实现现端到端,输入1张图像,输出1张图像。
本文基于MATLAB平台MatConvNet[9]公共源深度学习工具箱,针对CNN模型在图像分割领域很难做到精细分割的缺点,并结合脑肿瘤丰富的MRI图像信息,在全卷积神经网络的U-net网络的基础上,通过调整U-net网络,构建了适用于脑肿瘤图像的U1-net网络模型(以下简称为U1-net网络)。该网络克服了CNN模型忽略全局信息和丢失浅层网络信息的缺点,能够保留脑部MRI的全面信息,获得MRI的丰富特征,并且在公共数据集BRATS 2015上得到实验验证,表明了该网络的有效性,及其在勾画脑部肿瘤靶区精度上具有的良好效果。
1 材料与方法
1.1 BRATS数据集
本实验的脑肿瘤图片数据来源于BRATS 2015数据集[10],该数据集包含训练集和测试集。训练数据集由220个HGG病例和54个LGG病例组成。测试数据集由110个等级未知的病例组成。不同数据集的图像对齐到相同模板,插值成1 mm×1 mm×1 mm 体素分辨率的图像。由T1、T1c、T2和FLAIR的4个相关联模态图像形成了患者的MRI图像。BRATS 2015数据集如图1所示。所有图像标注整合了FLAIR、T1、T1c、T2等信息。每个图像数据在像素级上被标记为5类,分别是正常组织、坏死、水肿、非增强肿瘤区域、增强肿瘤区域。此外,BRATS数据集还提供了由临床专家手工分割的脑肿瘤勾画图作为自动勾画的真值标签(判别标准)(ground truth,GT)。
1.2 MatConvNet工具箱
MatConvNet为英国VLfeat小组开发的基于MATLAB平台的深度学习算法工具箱,简单易用、扩展性强。MatConvNet主题框架清晰,模块之间耦合性较小,便于用户根据需要对模块进行方便调整。其部分核心函数(如卷积、子采样等)使用C++编写,从而保证了运行速度。
MatConvNet 可提供构建深度学习模型需要的模块化的卷积层(convolution layer)、池化层(pooling layer)(子采样层)、反卷积层(deconvolution layer)(转置卷积层)、激活函数(activation function)等。
卷积层由若干卷积核组成,卷积运算的目的是提取输入的不同特征,并输出提取的特征,浅层的卷积层可能只能提取一些低级的特征,深层的网络能从低级特征中迭代提取更复杂的特征;池化层一般位于卷积层之后,由于卷积层提取到维度很大的特征,需要池化操作来特征降维、减小特征规模、减少参数量、减小过拟合,同时提高模型的鲁棒性,且池化分为最大池化和平均池化; 反卷积层中的反卷积操作通常用于将低维特征映射成高维输入,反卷积其实就是卷积的逆过程,通过反卷积操作并不能还原出卷积之前的图片,只能还原出卷积之前图片的尺寸;激活函数主要对卷积层的输出进行一个非线性映射,如果不用激励函数,CNN中每一层输出都是上层输入的线性函数,激活函数是用来增加非线性因素,用来提高线性模型的表达能力。
1.3 U-net网络
原始的U-net网络的U形结构如图2所示[9],由卷积层、最大池化层(下采样)、反卷积层(上采样)和ReLU函数4个部分组成。整个U-net网络的运行过程具体如下:
1) 下采样阶段。原始输入图像大小为572×572,经过2次由64个3×3的卷积核组成的卷积层后,得到特征图(feature map,FM)的大小变为568×568×64,经过2×2的最大池运算后,FM的大小变为284×284×64;根据上述过程重复4次,即(3×3卷积+2×2池化)×4次,每个池化后的第1个3×3卷积运算,3×3卷积核数成倍增加;到达最底层时,即在第4次最大池化之后,FM大小变为32×32×512,然后执行3×3×1 024的卷积操作2次,最终FM大小变化为28×28×1 024。
2) 上采样阶段。此时,FM的大小为28×28×1 024。首先进行2×2反卷积操作,使FM的尺寸变为56×56×512;然后对最大池化层之前的图像进行复制和剪切,再将反卷积得到的图像拼接在一起,得到1张56×56×1 024的图像,然后再进行3×3×512的卷积操作;重复上述过程4次,即进行(2×2反卷积+3×3卷积)×4次,每次拼接后的第1个3×3卷积操作后,3×3个卷积核的数目会翻倍;当到达顶层时,即经过第4次反卷积后,FM的大小将变为392×392×64,进行复制剪裁后再拼接,得到尺寸为392×392×128。然后再进行2次3×3×64的卷积操作得到388×388×64大小的图像,最后再进行1次1×1×2的卷积操作。
1.4 面向GBM靶区勾画的U1-net网络
加载原始的预训练U-net网络,并对U-net网络进行调整,构建适用于脑肿瘤图像分割的U1-net网络。U1-net网络由下采样阶段和上采样阶段组成,U1-net网络的框架结构如图3所示。

图3 U1-net网络结构图
整个U1-net网络的运行过程如下:
1) 下采样阶段。该阶段由5个卷积块和4个池化层组成。这5个卷积块都包含2个卷积层,卷积层中卷积核大小为3×3,步长为1。每个卷积层后都使用ReLU激活函数增加非线性因素。除了最后1个卷积块,在每个卷积块后使用相同的池化尺寸为2×2的最大池化操作。为了获取脑肿图像较高的分辨率,将U1-net网络的输入图像尺寸调整为320×320,经过下采样阶段,特征图大小转变为20×20。
2) 上采样阶段。该阶段由4个反卷积和4个卷积块组成,每个卷积块开始于反卷积层,其中卷积核大小3×3,步长为2,使得特征图大小增加1倍,数量减少1倍,最后特征图的大小再由下采样阶段的20×20变为320×320。U1-net网络在上采样阶段的卷积块中采用2个卷积层,用于减少特征图的数量。上采样阶段中的特征图来源于上一级上采样的特征图和对应的下采样的特征图。相比于原始的U-net网络,U1-net网络还使用了零填充来保证下采样和上采样阶段中的卷积层的输出维度。
1.5 输入图片数据的预处理
对于所有病例,有FLAIR、T1、T1c、T2共4个MRI序列。由于MRI图像强度的偏场畸变效应,引起图像中在同一区域的灰度值也会有差异,即使是同一扫描仪,也会在不同时间或者从不同患者身上采集到的切片通常具有不同的强度范围。
偏移场校正是校正由于磁场不均匀导致的图像对比度,强度标准化是将所有图像的强度值映射到标准或参考标度的过程。
为了解决这种强度归一化的问题,采用N4ITK处理方法。N4ITK是一款医学图像处理软件包,采用了多模态数据分割配准算法,用于处理图像配准和分割的问题,N4ITK方法主要是通过减去最高频率的灰度值,再除以校正后的灰度值来对图像进行归一化处理[11]。U1-net网络的输入图像尺寸为320×320,因此将所有图像数据全部归一化到320×320。MRI预处理前后对比图如图4所示。
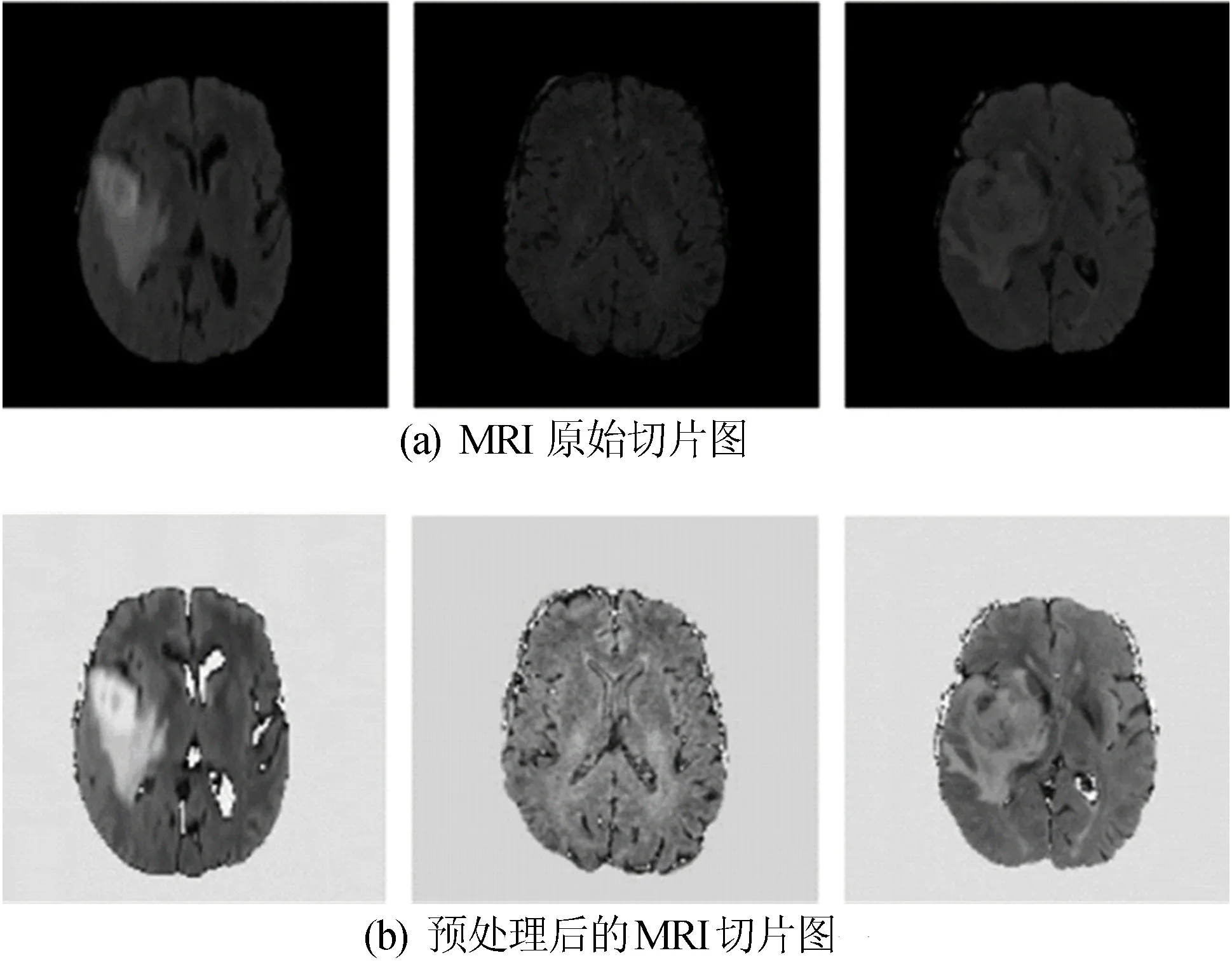
图4 MRI预处理前后对比图
1.6 U1-net网络训练及参数设置
本文针对脑肿瘤图像分割构建的U1-net网络的输入是一整张图像,以FLAIR、T1、T1c和T2 4个模态图像作为训练图像,取同一层切片组成的4通道图像。最后的输出类别设置为5,对应脑肿瘤的5个类别。训练参数设置为:初始学习率为5×10-4,权值衰减为0.005,迭代9 000次以上。
U1-net网络的准确度与迭代次数的关系如图5所示。

图5 U1-net网络的准确度随迭代次数的变化
由图5可知,网络的准确度随着迭代次数的增加越来越趋于稳定,逼近100%。
U1-net网络的损失函数值与迭代次数的关系如图6所示。
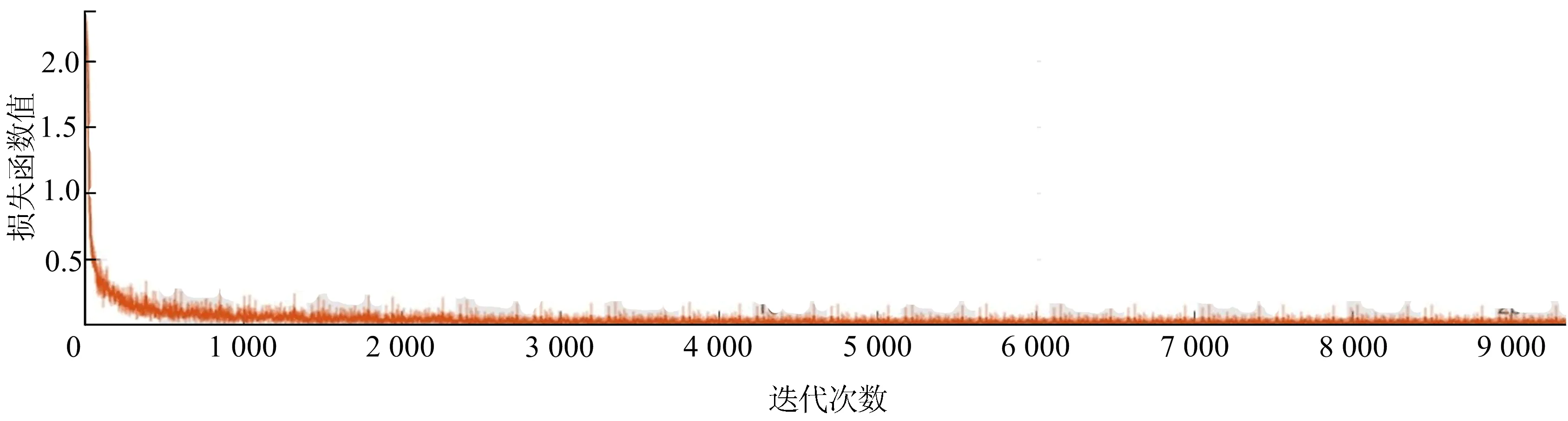
图6 U1-net网络的损失函数值随迭代次数的变化
从图6可以看出,损失函数值趋于0,表明预测结果与真实结果具有较高的一致性,U1-net网络训练效果较好,能完全适用于脑肿瘤图像分割任务。
1.7 分割的评估标准
利用Dice相似系数(Dice similarity coefficient,DSC)、阳性预测值(positive predictive value,PPV)、灵敏度S对分割方法进行评估[12],每种指标都是根据3种肿瘤区域来计算的。这3种区域包括完整肿瘤区域、核心区域和增强区域。完整的肿瘤区由坏死区、水肿区、非强化区和强化区组成; 核心区域由坏死区、非强化区和增强区组成; 增强区域只包括增强核心。这3个指标的计算公式为:
(1)
(2)
(3)
其中,tp、fp、fn分别是检测为真阳性、假阳性、假阴性的肿瘤点的数量。
2 实验结果讨论与分析
本文选取3个病人的经过U1-net网络后的分割预测图,分割结果如图7所示。
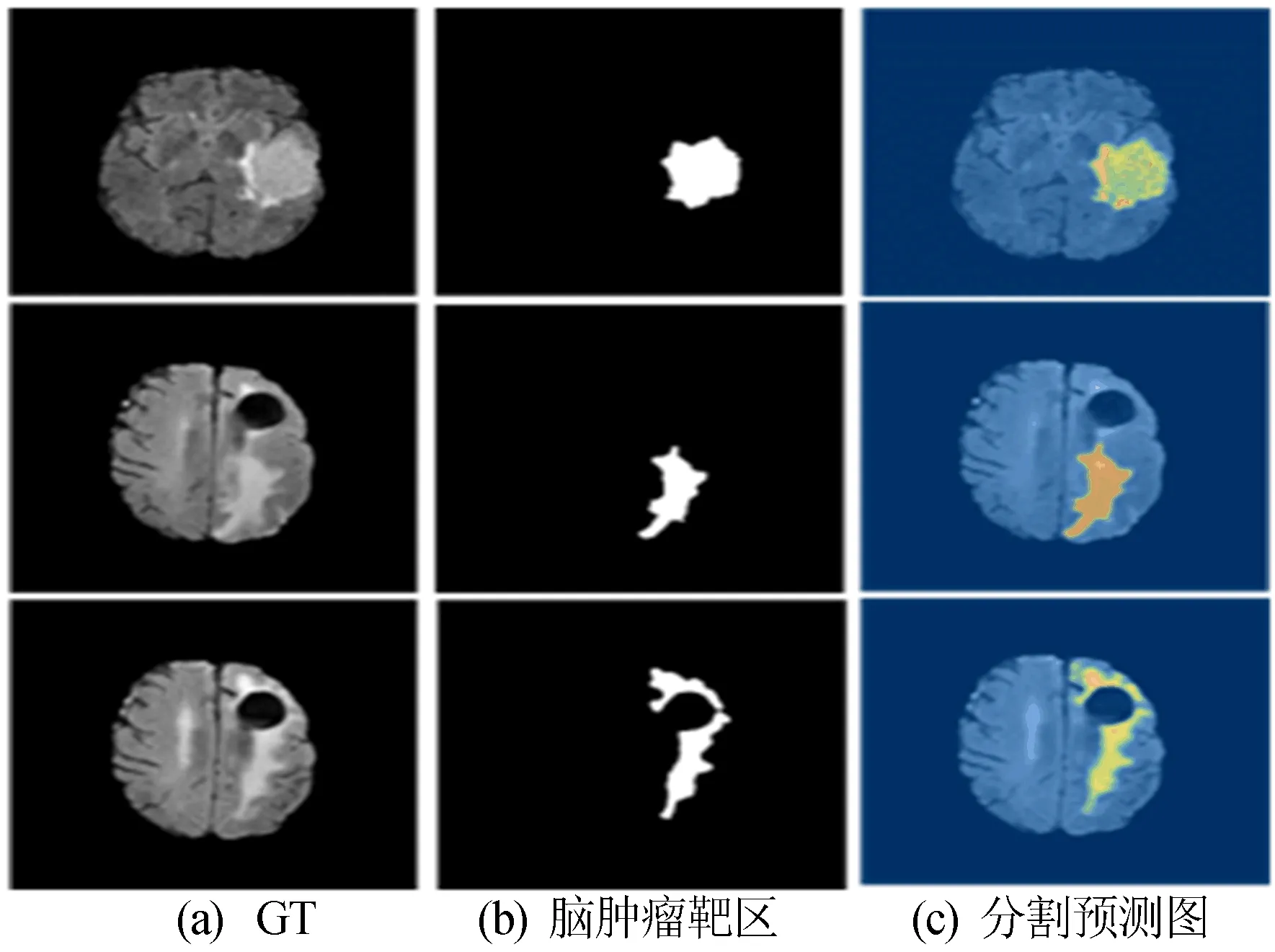
图7 U1-net网络的GT图与分割图的对比
图7中:黄色区域代表水肿;红色区域代表非增强肿瘤结构;绿色区域代表囊性坏死部分。由图7可知,U1-net网络可以分割出完整脑肿瘤区域,且与GT较为相似,获得了较好的匹配率,接近脑肿瘤的分割轮廓,分割效果较好。
针对脑肿瘤图像构建的U1-net网络与文献[6,13-15]方法,基于BRATS 2015/BRATS 2013数据集分割结果的DSC值对比见表1所列。表1中,前3种实验方法和本文的分割方法都是基于BRATS 2015进行的实验,第4种方法基于BRATS 2013进行的实验,BRATS 2015数据集和 BRATS 2013数据集在肿瘤图像特点上几乎没有区别。

表1 不同方法在BRATS 2015/BRATS 2013数据集的DSC值
从表1可以看出,U1-net网络分割效果较好,在完整肿瘤区、核心肿瘤区、增强肿瘤区的DSC值分别为0.92、0.85、0.83。与文献[14]方法构建的网络模型相比,完整肿瘤区和增强肿瘤区的DSC值高于其提出的网络模型,说明U1-net网络对完整肿瘤区和增强肿瘤区有良好的特性,但是在核心肿瘤区的DSC值略低于其建立的网络模型,可能是由于在1幅图像中,正常组织区域所占的比例较大,而肿瘤内部其他子区域还要进一步分为 4个类别,因此肿瘤每个子区占的比例就更小,导致类别学习不平衡。
从表1还可以看出,文献[15]方法构建的网络模型在完整肿瘤区、核心肿瘤区、增强肿瘤区的DSC数值相比于本文提出的U1-net网络明显低很多,这是由于其建立的网络是一种基于CNN的分割模型,而CNN模型存在易忽略全局信息和丢失浅层网络信息的缺点,从而导致其建立的网络获得了较低的DSC值。本文提出的U1-net网络是基于全卷积神经网络的网络模型,克服了CNN模型存在的缺点,在脑肿瘤图像上分割效果较好,相比于CNN模型,也做到了精细的像素级分割。
相关文献方法的分割结果如图8所示[6,13-14]。图8中,每幅图从左到右依次是原始脑肿瘤靶区、分割标准GT、分割结果。对比图7的效果图可知,虽然都区分出了脑肿瘤区域和正常组织,但是本文提出的分割方法在脑肿瘤边界处的分割结果相对更光滑,而且在脑肿瘤内各个子区域的分割效果也相对较好。

图8 相关文献方法的分割结果
U1-net网络和文献[16]提出的方法基于BRATS 2015数据集分割结果的DSC定量比较结果见表2所列。
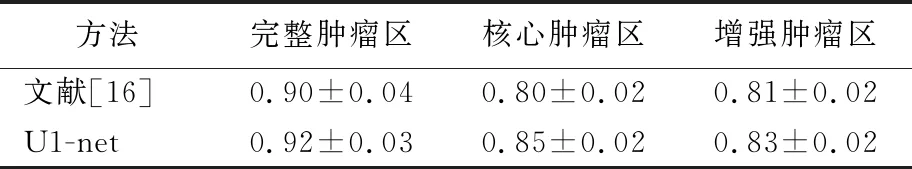
表2 不同方法在BRATS 2015数据集的DSC值
文献[16]针对脑肿瘤构建的基于图像块分类的分割模型(6-CNN网络),其思路是将肿瘤靶区的图像分割问题转化为肿瘤与非肿瘤图像块的分类问题来解决。图像块是从整张图像中依次提取的局部区域,并将其中心像素值作为整个图像块的像素值,同样中心像素的标签也作为整个图像块的标签。
在训练阶段,提取大量的图像块训练CNN;在测试阶段,从测试图像中提取的图像块被训练的CNN分类。每个图像块将通过CNN获得1个标签,图像块及其对应的标签将组成1个新的带有标签的大脑MRI,以此标签将被用来分割相应的区域,即所有图像块的分类结果整合构成测试图像的分割结果,然而6-CNN网络存在易丢失数据空间结构、训练过程繁琐、分割效率低下等弊端,且不能做到像素级精细分割。由表2可知,U1-net网络整体分割效果优于6-CNN网络,特别在核心肿瘤区的精度有明显提高,约提高了5%,U1-net网络对比6-CNN网络更能做到精细分割。
文献[17]除了在基于BRATS 2015数据集做过关于脑肿瘤分割方法的研究,还在基于BRATS 2013数据集提出了分层脑肿瘤的分割方法。本文构建的U1-net网络与文献[17]提出的分层脑肿瘤分割方法的3个指标对比结果见表3所列。
从表3可以看出,U1-net网络在完整肿瘤区、核心肿瘤区的DSC、PPV、灵敏度数值都有不同程度的提高,在增强肿瘤区的灵敏度数值相等。
3 结 论
U-net网络作为深度学习分割网络,也应用于其他生物医学图像处理领域,且已在具有差异性的其他医学图像分割任务中取得了优异的分割表现,而本文在结合MRI多模态丰富信息和基于全卷积神经网络的U-Net网络的基础上,通过调整并针对脑肿瘤图像分割构建了U1-net网络,其本质上属于U-Net网络模型的一种,因此在脑肿瘤医学图像分割任务中获得了良好的表现,能适应脑肿瘤因以任何形状存在于任意位置带来的差异性,从而能自动、准确地分割脑肿瘤靶区。
因此,该方法可以帮助医生在人工分割脑肿瘤时由于人为因素(如疲劳度、不同医师的个体化差异等)影响而导致的正确率下降。U1-net网络靶区分割效率也较高,一般处理一套人体脑部MRI切片(一般155张)平均只需要10~30 s,在医学成像应用中常常需要处理大量的数据而造成时间瓶颈,因此本文提出的U1-net网络具有重要的实用价值。
U1-net网络仅以MRI图像数据集进行模型训练与肿瘤靶区的分割,不需要参考病人的CT图像,这正是目前国际上普遍认可的MRI图像引导放疗概念的优势。本文工作所解决的问题是放疗肿瘤靶区的勾画,作为放疗计划制定的预工作,U1-net网络在完整肿瘤区的指标值较高,完整肿瘤区也正是肿瘤放射靶区,完整肿瘤区的分割精度越高,肿瘤放射靶区的放射效果越好。U1-net网络为放疗的自动化实施提供了基础,也为靶区自动勾画领域的研究提供了更多方向和借鉴思路。
