菠萝SLF/SFB基因家族全基因组鉴定与表达分析
2023-08-14井敏敏李小针李栋梁戴小红顾帅磊马朝明陈志辉陈晶晶
井敏敏 李小针 李栋梁 戴小红 顾帅磊 马朝明 陈志辉 陈晶晶
摘 要:自交不亲和性是高等植物通过抑制自花授粉来阻止近亲繁殖的一种繁殖机制。在以茄科、蔷薇科、车前草科为代表的配子体自交不亲和机制中,花粉和雌蕊之间的自我/非自我识别由多态性S 位点决定,其中SLF(S-locus F-box)/SFB(S-haplotype-specific F-box)基因是配子体自交不亲和花粉S 决定因子。本研究通过生物信息学方法从菠萝基因组中筛选鉴定出21 个SLF/SFB 家族基因(AcSLF1~AcSLF21),各AcSLF 基因在N 端具有F-box 保守结构域,其氨基酸长度、理论等电点、分子量存在较大差异,编码氨基酸序列大小范围为242~612 aa,分子量为27.778~69.070 kDa,其中18 个AcSLFs 蛋白偏碱性,总体蛋白稳定性较差,预测18 个AcSLFs 蛋白亚细胞定位于细胞核中,蛋白二级结构60%以上由α-螺旋和无规则卷曲组成。通过染色体定位分析发现其不均匀地分布在14 条染色体上,其中AcSLF21 未定位到具体染色体位置,并发现AcSLF4、AcSLF5、AcSLF6、AcSLF8、AcSLF14 和AcSLF15 与菠萝RNase T2 家族基因发生连锁。从进化关系中发现,菠萝SLF/SFB 家族与蔷薇科、车前科、茄科植物SLF/SFB 进化关系较远。利用qRT-PCR对AcSLFs 基因在菠萝雌蕊、雄蕊、叶片、果肉、茎和根的表达量进行分析,结果发现AcSLFs 基因在菠萝不同组织中具有明显的表达差异性,其中AcSLF1、AcSLF2、AcSLF4、AcSLF9、AcSLF10、AcSLF11、AcSLF15、AcSLF19、AcSLF20、AcSLF21 基因在菠萝雄蕊中呈高表达量,AcSLF3、AcSLF7 在果肉中表达量最高,AcSLF5、AcSLF12、AcSLF18 在根中表达量显著高于其他组织,总体来说,大部分菠萝AcSLFs 基因主要在菠萝雄蕊或叶片中高表达。同时分析了雌蕊自花、异花授粉前后的表达变化,发现AcSLF1、AcSLF2、AcSLF15 三个基因在授粉雌蕊中的表达量较未授粉显著上调,尤其在授粉6 h 表达量急剧上调,随着花粉管的伸长其表达量降低,但在授粉24 h 仍具有较高的表达量,推测这些基因可能在菠萝自交不亲和中起到重要作用。该研究为菠萝SLF/SFB 家族基因的克隆提供参考,并为菠萝自交不亲和机制研究奠定良好基础。
关键词:菠萝;SLF/SFB 基因家族;生物信息学;表达量分析
中图分类号:S668.3 文献标识码:A
植物自交不亲和性(self-incompatibility, SI)是指绿色开花植物能产生正常的配子,但自花不能正常授粉或授粉后不能正常结籽的一种生殖隔离现象[1]。菠萝属于典型的自交不亲和植物,除了部分野生种外,多数菠萝品种自交不亲和,无法产生种子,而菠萝类群Ananas comosus var.Comosus(主要是栽培菠萝)自交不亲和表现尤为突出。目前普遍认为菠萝属于配子体自交不亲和类型[2-3],配子体自交不亲和是植物界分布最广泛的自交不亲和调控机制,目前在蔷薇科、玄参科、茄科、罂粟科等配子体自交不亲和类型植物的研究中表明,花柱自交不亲和的决定因子被确定为S-RNase 基因,花粉决定因子为编码F-box蛋白的SLF(S-locus F-box)/SFB(S-haplotype-specificF-box)基因[4-5]。花柱分泌的S-RNase 可进入花粉管,通过自身的核糖核酸酶活性降解自花花核糖核酸酶活性降解自花花粉管中的rRNA,造成花粉管细胞骨架解体[6-7],从而抑制花粉管生长,造成自交不亲和,与S-RNase 互作的SLF/SFB 可通过形成SCF 复合体泛素化非自我S-RNase,泛素化的S-RNase 最终被26S 蛋白酶体降解[8],从而消除了S-RNase 的细胞毒性作用,花粉管可在花柱中继续生长。
迄今为止,已经从茄科、蔷薇科和玄参科等多种配子体型自交不亲和植物中分离出近百个花柱关键基因S-RNase[9],且其作用机制已研究较深入,但花粉关键控制基因研究稍滞后。LAI 等[10]通过对金鱼草S 基因座进行长片段测序,首次发现了1 个与S2-RNase 紧密连锁的编码F-box 蛋白的基因AhSLF-S2,该基因在花粉中特异性表达。
蔷薇科植物梅、扁桃中也鉴定出多个在花粉中特異性表达,同时具有较高序列多态性的编码F-box蛋白的基因[11-12]。在金鱼草及矮牵牛中利用转基因技术证实了S 基因座中的F-box 基因决定了花粉自交不亲和特异性反应,并命名为S-locus F-box(SLF)[13]。在蔷薇科植物中,花粉S 决定因子缺乏统一的命名,苹果亚科花粉S 候选基因被命名为S-locus F-box Brothers(SFBB)[14],李属植物被命名为S-haplotype-specific F-box(SFB)[15]。
目前,关于菠萝自交不亲和的研究主要集中在组织细胞学和田间杂交试验上,菠萝自交不亲和的主要控制基因尚不明确。本研究利用生物信息学技术在菠萝基因组中分离出21 个SLF/SFB家族成员,并对其理化特性、基因结构、染色体分布、保守结构域以及系统进化关系进行详细分析,并对各基因在菠萝不同组织中的表达模式进行分析,同时对雌蕊授粉前后AcSLF 基因的表达变化进行分析,以期为菠萝自交不亲和机制等方面研究提供理论基础。
1 材料与方法
1.1 材料
供试材料金菠萝(MD2)、巴厘菠萝(ComteDe Paris)种植于广东省湛江市南亚热带作物研究所国家热带果树种质资源圃,2 种材料均为自交不亲和材料,且巴厘菠萝授粉金菠萝为亲和可育。
收集金菠萝、巴厘菠萝成熟花粉至离心管,用小毛刷蘸取花粉授于金菠萝柱头,分别于授粉2、6、24 h 后取花柱,立即液氮冷冻,同时采集金菠萝叶片、雄蕊、未授粉雌蕊、果肉、茎、根组织样品进行冷冻,于–80 ℃保存备用。
1.2 方法
1.2.1 菠萝SLF/SFB 基因全基因组鉴定及理化特性分析 菠萝基因组数据从Pineapple GenomicsDatabase(http://pineapple.zhangjisenlab.cn/pineapple/html/index.html)[16]下载。从NCBI 中下载已报道的SLF/SFB 序列,采用MEGA 5 软件[17]进行ClustalW 核苷酸序列比对,然后利用HMMER 软件的Hmmbuild 生成SLF/SFB 的HMM 种子文件,基于此种子文件,筛选菠萝SLF/SFB 基因。
利用Expasy(https://web.expasy.org/protparam/)在线软件进行理论等电点、蛋白分子量、不稳定指数、脂溶系数、疏水指数等分析。利用Cell-PLoc2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)在线软件预测亚细胞定位。利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)在线软件分析蛋白质二级结构。
1.2.2 菠萝AcSLF 基因染色体定位 利用TBtools[18]软件对菠萝AcSLF 基因进行染色体定位作图。
1.2.3 菠萝AcSLF 系统进化树构建 利用MEGA5.0软件构建菠萝AcSLF 系统发育树,进化树构建方法为邻接法(Neighbor-joining method),bootstrap 值设置为1000。
1.2.4 菠萝AcSLF 基因结构及蛋白结构域分析从菠萝基因组数据库获得21 个AcSLFs 的基因组和CDS 序列, 使用GSDS ( http://gsds. cbi.pku.edu.cn/)[19]在线软件绘制各基因的内含子–外显子结构图。蛋白结构域采用SMART(http://www.smart.embl-heidelberg.de/)在线软件进行分析,并由TBtools 软件进行可视化作图。
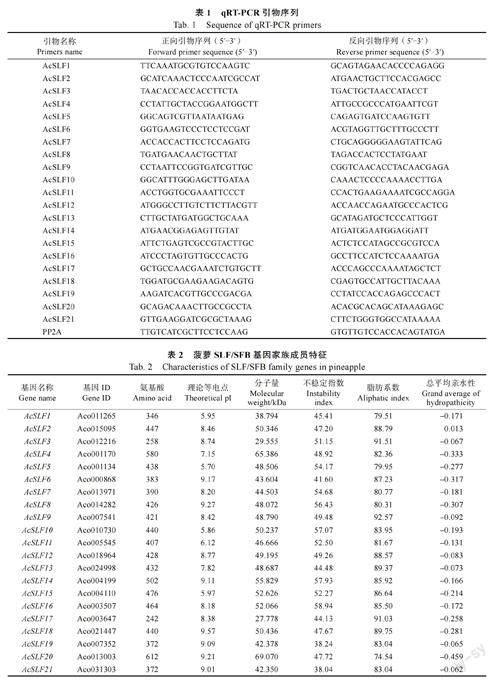
1.2.5 菠萝AcSLF 基因表达分析 利用OMGARNA 提取试剂盒从菠萝不同组织(叶片、雄蕊、雌蕊、果肉、茎、根以及授粉后雌蕊)中提取总RNA,使用逆转录试剂盒合成cDNA。利用SYBRGreen qPCR Master Mix 在Quant StudioTM 6 FlexSystem 实时荧光定量PCR 系统进行qRT-PCR 分析,内参基因为PP2A[20](引物序列信息见表1)。
使用2–ΔΔCT 方法[21]计算各基因的相对表达水平。
2 结果与分析
2.1 菠萝SLF/SFB 基因鉴定与理化特性分析
通过对菠萝基因组检索和鉴定,共获得21 个SLF/SFB 基因, 按照其在染色体位置命名为AcSLF1~AcSLF21。对21 个菠萝AcSLFs 进行理化特性分析,结果显示,AcSLFs 在氨基酸长度、理论等电点、分子量有较大差异,AcSLFs 的氨基酸序列大小范围为242~612 aa,分子量介于27.778~69.070 kDa 之间,预测的AcSLFs 的理论等电点(pI)为5.70~9.57,除AcSLF1、AcSLF5、AcSLF10、AcSLF15 外,其他蛋白偏堿性(pI>7)。总体AcSLFs 蛋白稳定性较差,仅AcSLF19、AcSLF21较稳定(表2)。
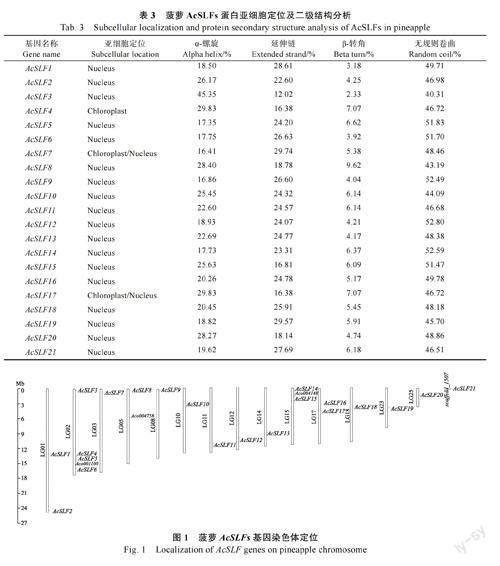
利用Cell-PLoc 2.0 软件预测蛋白质亚细胞定位,预计有18 种AcSLFs 蛋白只定位于细胞核,AcSLF4 定位在叶绿体,AcSLF7、AcSLF17 在细胞核与叶绿体中均有分布。通过分析AcSLFs 蛋白二级结构,发现由α-螺旋(alpha helix)、延伸链(extended strand)、β-转角(beta turn)和无规则卷曲(random coil)4 种构型组成,其中α-螺旋和无规则卷曲占60%以上,延伸链和β-转角占30%左右,无规则卷曲占比最大约50%(表3)。
2.2 菠萝AcSLFs 基因染色体定位
为明确菠萝AcSLFs 基因在染色体上的分布,利用菠萝基因组数据库中的基因位置信息,使用Tbtools 软件对基因在染色体上位置进行可视化(图1)。结果显示,AcSLF21 位于scaffold_1507,尚未定位到具体染色体位置,其余染色体不均匀地分布在14 条染色体上,其中AcSLF3、AcSLF4、AcSLF5、AcSLF6 位于LG02 号染色体,大部分基因位于染色体两端。同时发现部分AcSLFs 基因与菠萝RNase T2 家族基因发生连锁,其中AcSLF4、AcSLF5、AcSLF6 与Aco001100 紧密连锁,AcSLF8与Aco004758 连锁,AcSLF14、AcSLF15 与Aco00-4148 连锁。
2.3 菠萝AcSLFs 进化树构建
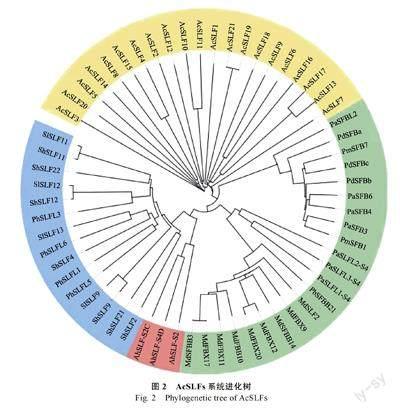
为了解菠萝AcSLFs 蛋白之间进化关系,将21 个AcSLFs 蛋白序列与苹果、梅、樱桃等蔷薇科植物、金鱼草车前科植物、番茄、矮牵牛等茄科植物的SLF/SFB 蛋白序列进行多重序列比对,通过NJ 的方法使用MEGA 6.0 软件构建系统进化树,结果如图2 所示(蓝色表示茄科植物,红色表示金鱼草车前科植物,绿色表示蔷薇科植物,黄色表示菠萝)。通过进化树分支聚类关系可以看出,番茄、多毛番茄和矮牵牛的SLF、SLFL 聚为一类,苹果、梨、甜樱桃的SLFL、FBX 和SFBB聚为一类,甜樱桃、扁桃、梅的SFB 聚为一类,而菠萝AcSLFs 与已报道的SLF/SFB 蛋白均未聚类到一起,说明菠萝AcSLFs 与蔷薇科、车前科、茄科植物SLF/SFB 进化关系较远,在进化树分支上聚类程度较低,这也说明了单子叶植物菠萝与双子叶植物亲缘关系较远。
2.4 菠萝AcSLFs 基因结构及蛋白结构域分析
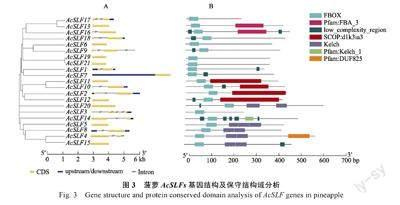
在21 个SLF/SFB 家族成员中,11 个AcSLFs基因均含有内含子,其中AcSLF3 含有4 个内含子,其他10 个基因只包含1~2 个内含子(图3A),不同的AcSLFs 基因含有内含子数量不同,而内含子与基因分类无显著相关性。对21 个菠萝AcSLFs基因进行蛋白结构域分析,结果表明各AcSLFs基因在N 端具有F-box 保守结构域, 其中AcSLF17、AcSLF19、AcSLF21、AcSLF3 只含有F-box 结构域, 不含其他结构域, AcSLF13、AcSLF16 在C 端具有F-box 关联的FBA 结构,另外AcSLF20、AcSLF4、AcSLF15、AcSLF5、AcSLF8基因含有Kelch 结构域(图3B)。
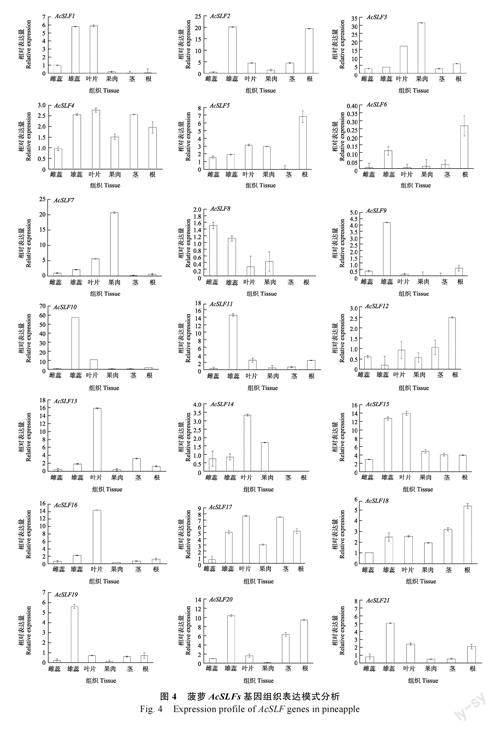
2.5 菠萝AcSLFs 基因组织表达分析
为了研究菠萝AcSLFs 基因组织表达模式,通过qRT-PCR 技术对21 个AcSLFs 基因在菠萝雄蕊、雌蕊、叶片、果肉、茎和根不同组织的表达量进行分析。结果显示(图4),不同的AcSLFs基因在菠萝不同组织中表现出明显的表达差异,其中,AcSLF1、AcSLF4、AcSLF13、AcSLF14、AcSLF15、AcSLF16 和AcSLF17 在叶片中呈最高表达量,AcSLF3、AcSLF7 在果肉中表达量最高,AcSLF5、AcSLF12、AcSLF18 在根中表达量高于其他组织,AcSLF6 在6 个组织部位表达量均较低,AcSLF1、AcSLF2、AcSLF4、AcSLF9、AcSLF10、AcSLF11、AcSLF15、AcSLF19、AcSLF20、AcSLF21在雄蕊中呈高表达量。总体来说,大部分菠萝AcSLFs 基因主要在菠萝雄蕊或叶片中表达量高。
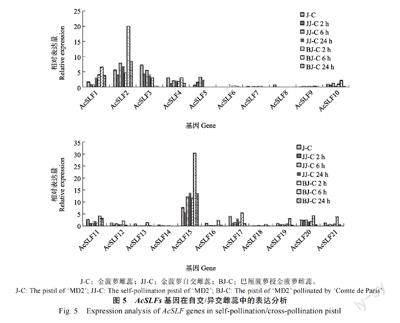
2.6 菠萝AcSLFs 基因授粉前后的表达差异分析
通过对21 个AcSLFs 基因在未授粉金菠萝雌蕊、自花授粉2、6、24 h 以及巴厘菠萝授金菠萝雌蕊2、6、24 h 的表达分析发现,AcSLF1、AcSLF2、AcSLF15 三个基因在授粉后表达显著上调,尤其是异花授粉后6 h,表达量显著高于未授粉雌蕊,在授粉后24 h 同样具有较高表达量,同时异花授粉表达量高于自花授粉(图5)。AcSLF5 在自花授粉后表达上调,而异花授粉后几乎无表达,AcSLF10、AcSLF11、AcSLF17、AcSLF19、AcSLF20和AcSLF21 在异花授粉6 h 稍上调表达。在配子体自交不亲和植物中,亲和与不亲和花粉均可在柱头上萌发生长,并伸入花柱,S-RNase 主要聚集在花柱上端, 当花粉管受到雌蕊分泌的S-RNase 胁迫时,会启动相应保护机制,花粉管编码多个识别非自我S-RNase 的SLF 蛋白,SLF蛋白可识别并泛素化非自我S-RNase,使S-RNase丧失毒性,从而花粉管可继续生长,而“自身”花粉管在S-RNase 的毒性下丧失活性停止生长[22-23]。
在菠萝授粉后,AcSLF1、AcSLF2、AcSLF15 基因异花授粉显著高于自花授粉,这说明“非自我”花粉SLF 基因的高表达,有助于其更好地发挥解毒作用,暗示这3 个基因在菠萝自交不亲和中发挥重要作用。
3 讨论
本研究通过全基因组检索鉴定了21 个菠萝AcSLFs,与矮牵牛16~20 个SLF 基因数目相当[24],与柑橘中报道113 个SLF/SLFL 基因相差较多[25]。
AcSLFs 在氨基酸长度、理论等电点、分子量有较大差异,且绝大多数AcSLFs 蛋白为碱性蛋白,总体AcSLFs 蛋白稳定性较差,仅AcSLF19、AcSLF21 较稳定。基于S-RNase 介导的自交不亲和反应,雌蕊S 基因S-RNase 周围存在多个SLF/SFB 共同行使花粉S 基因功能,通过形成SCF复合体泛素化非自我S-RNase,从而S-RNase 被26S 蛋白酶体降解,而SLF/SFB 无法识别降解自我S-RNase,导致自交不亲和性[26-27]。通过染色体定位分析,发现20 个AcSLFs 基因定位在14 条染色体上,AcSLF21 未定位到具体染色体位置,同时发现AcSLF4、AcSLF5、AcSLF6、AcSLF8、AcSLF14、AcSLF15 与菠萝RNase T2 家族基因连锁,ZHAO 等[28]在菠萝LG15 染色体上发现存在RNase T2 家族基因与F-box 基因紧密连锁。
WILLIAMS 等发现矮牵牛SLF 基因均位于S-RNase 基因序列的下游,且与S-RNase 具有相反的转录方向[29-31],目前尚不清楚是否所有配子体自交不亲和植物的S 基因均具有此特性,在菠萝中与S-RNase 连锁的SLF 基因AcSLF6 、AcSLF15 位于S-RNase 基因下游,其余几个基因位于上游,且在菠萝中发现与S-RNase 连锁的F-Box基因较少,远远少于矮牵牛、柑橘等其他配子体自交不亲和物种。
菠萝AcSLFs 基因组织表达分析发现AcSLF1、AcSLF2、AcSLF4、AcSLF8、AcSLF9、AcSLF10、AcSLF11、AcSLF15、AcSLF19、AcSLF20、AcSLF21在雄蕊中具有较高表达量,其中AcSLF1、AcSLF2、AcSLF15 基因在授粉后的花柱中表达显著上调,且异花授粉显著高于自花授粉,尤其在异花授粉6 h 表达量急剧升高,这说明异花授粉诱导了其表达,随着花粉管在雌蕊中的伸长其表达量降低,但在授粉24 h 后仍然具有较高的表达量。张伟等[32]研究发现神湾与亲和性不同的父本进行授粉7 h后,亲和性强的花粉管已伸长至花柱中部。本研究前期利用苯胺蓝染色发现亲和花粉管在授粉24 h已达到花柱底端,异花授粉后AcSLF1、AcSLF2、AcSLF15 的表达趋势与花粉管的生长相吻合,其表達模式表明AcSLF1、AcSLF2、AcSLF15 可能是调控菠萝自交不亲和反应的重要因子。本研究只分析了AcSLFs 在授粉后花柱包含花粉管一起的表达模式,还需进一步建立体外花粉管培养及诱导不亲和反应,分析其在花粉管中的表达模式及作用机制。本研究对菠萝SLF/SFB 家族成员进行鉴定和表达分析,有助于进一步寻找菠萝自交不亲和花粉S 决定因子,为研究菠萝自交不亲和机制研究提供良好的理论基础。
參考文献
[1] MCCLURE B A, CRUZ-GARCIA F, ROMERO C. Compatibility and incompatibility in S-RNase-based systems[J].Annals of Botany, 2011, 108(4): 647-658.
[2] BREWBAKER J L, GORREZ D D. Genetics of self- incompatibility in the monocot genera, Ananas (pineapple) and Gasteria[J]. American Journal of Botany,1967, 54: 611-616.
[3] MAJUMDAR S K, KERNS K R, BREWBAKER J L. Assessing self-incompatibility in pineapple by a pollen fluorescence technique[J]. Proceedings of the American Society for Horticultural Science, 1964, 84: 217-223.
[4] LEE H S, HUANG S, KAO T H. S proteins control rejection of incompatible pollen in Petunia inflata[J]. Nature, 1994,367: 560-563.
[5] SIJACIC P, WANG X, SKIPAN A L, WANG Y, DOWD P E, MCCUBBIN A G, HUANG S, KAO T. Identification of the pollen determinant of S-RNase-mediated self-incompa tibility[J]. Nature, 2004, 429: 2-5.
[6] JUAN A R, HERNAN J R, GOLDRAIJ A. Disorganization of F-actin cytoskeleton precedes vacuolar disruption in pollen tubes during the in vivo self-incompatibility response in Nicotiana alata[J]. Annals of Botany, 2012, 110(4): 787-795.
[7] YANG Q, MENG D, GU Z, LI W, CHEN Q J, LI Y, YUAN H, YU J, LIU C S, LI T Z. Apple S-RNase interacts with an actin-binding protein, MdMVG, to reduce pollen tube growth by inhibiting its actin-severing activity at the early stage of self-pollination induction[J]. The Plant Journal,2018, 95(1): 41-56.
[8] SUN P, KAO T H. Self-incompatibility in Petunia inflata: the relationship between a self-incompatibility locus F-box protein and its non-self S-RNase[J]. Plant Cell, 2013, 25(2):470-485.
[9] 何敏, 谷超, 吴巨友, 张绍铃. 果树自交不亲和机制研究进展[J]. 园艺学报, 2021, 48(4): 759-777.
[10] LAI Z, MA W, HAN B, LIANG L Z, ZHANG Y S, HONG G F, XUE Y B. An F-box gene linked to the self-incompatibility (S) locus of Antirrhinumis expressed specifically in pollen and tapetum[J]. Plant Molecular Biology,2002, 50(1): 29-41.
[11] ENTANI T, IWANO M, SHIBA H, Che F S, ISOGAI A,TAKAYAMA S. Comparative analysis of the self-incompatibility(S-) locus region of Prunus mume: identification of a pollen-expressed F-box gene with allelic diversity[J].Genes Cells, 2003, 8(3): 203-213.
[12] USHIJIMA K, YAMANE H, WATARI A, KAKEHI E,IKEDA K, HAUCK N R, IEZZONI A F, TAO R. The Shaplotype-sepecific F-box protein gene, SFB, is defective in self-compatible haplotypes of Prunus avium and P. mume[J].The Plant Journal, 2004, 39(4): 573-586.
[13] QIAO H, WANG F, ZHAO L, ZHOU J L, LAI Z, ZHANG Y S, ROBBINS T P, XUE Y B. The F-box protein AhSLF-S2 controls the pollen function of S-RNase-based self-incompatibility[J]. Plant Cell, 2004, 16(9): 2307-2322.
[14] KAKUI H, KATO M, USHIJIMA K, KITAGUCHI M,KATO S, SASSA H. Sequence divergence and loss-of-function phenotypes of S locus F-box brothers genes are consistent with non-self recognition by multiple pollen determinants in self-incompatibility of Japanese pear (Pyrus pyrifolia)[J]. Plant Journal, 2011, 68(6): 1028-1038.
[15] YAMANE H, IKEDA K, USHIJIMA K, SASSA H, TAO R.A pollen-expressed gene for a novel protein with an F-boxmotif that is very tightly linked to a gene for S-RNase in two species of cherry, Prunus cerasus and P. avium[J]. Plant Cell Physiology, 2003, 44(7): 764-769.
[16] XU H, YU Q, SHI Y, HUA X T, TANG H B, YANG L,MING R, ZHANG J S. PGD: pineapple genomics database[J]. Horticulture Research, 2018, 17(5): 66.
[17] TAMURA K, PETERSON D, PETERSON N, STECHER G,NEI M, KUMAR S. MEGA5: molecular evolution arygenetics analysis using maximum likelihood, evolutionary dis-tance, and maximum parsimony methods[J]. Molecular Biology Evolution, 2011, 28(10): 2731-2739.
[18] CHEN C J, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y H, XIA R. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202.
[19] HU B, JIN J P, GUO A Y, ZHANG H, LUO J C, GAO G.GSDS 2.0: an upgraded gene feature visualization server[J].Bioinformatics, 2015, 31(8): 1296-1297.
[20] CHEN H H, HU B Y, ZHAO L H, SHI D D, SHE Z Y,HUANG X Y, PRIYADARSHANI S V G N, NIU X P, QIN Y. Differential expression analysis of reference genes in pineapple (Ananas comosus L.) during reproductive development and response to abiotic stress, hormonal stimuli[J].Tropical Plant Biology, 2019, 12(2): 67-77.
[21] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2?ΔΔCT method[J]. Methods, 2001, 25(4): 402-408.
[22] KAO T H, TSUKAMOTO T. The molecular and genetic bases of S-RNase-based self-incompatibility[J]. Plant Cell, 2004, 16 (Suppl): S72-S83.
[23] AHMAD M H, RAO M J, HU J B, XU Q, LIU C C, CAO Z H, LARKIN R M, DENG X X, BOSCH M, CHAI L J. Systemsand breakdown of self-incompatibility[J]. Critical Reviews in Plant Sciences, 2022, 41(3): 209-239.
[24] WU L H, WILLIAMS J S, SUN L H, KAO T. Sequence analysis of the Petunia inflata S-locus region containing 17 S-Locus F-Box genes and the S-RNase gene involved in self-incompatibility[J]. The Plant Journal, 2020, 104:1348-1368.
[25] 梁梅. 柑橘自交不亲和相关基因鉴定及其演化[D]. 武汉:华中农业大学, 2019.
[26] HUA Z, KAO T H. Identification and characterization of components of a putative petunia S-locus F-box-containing E3 ligase complex involved in S-RNase-based self-incompatibility[J]. Plant Cell, 2006, 18(10): 2531-2553.
[27] LIANG M, CAO Z, ZHU A, LIU Y L, TAO M Q, YANG H Y, XU Q, WANG S H, LIU J J, LI Y P, CHEN C W, XIE ZZ , DENG C L, YE J L, GUO W W, XU Q, XIA R,LARKIN R M, DENG X X, BOSCH M, FRANKLINTONG V E, CHAI L J. Evolution of self-compatibility by a mutant Sm-RNase in citrus[J]. Nature Plants, 2020, 6:131-142.
[28] ZHAO H, ZHANG Y, ZHANG H, SONG Y Z, ZHAO F, ZHANG Y E, ZHU S H, ZHANG H K, ZHOU Z D, GUO H,LI M M, LI J H, HAN Q Q, COPSEY L, LI Q, CHEN H,COEN E, ZAHNG Y J, XUE Y B. Origin, loss and regain of self-incompatibility in angiosperms[J]. Plant Cell, 2022,43(1): 579-596.
[29] WILLIAMS J S, DER J P, DEPAMPHILIS C W, KAO T.Transcriptome analysis reveals the same 17 S-locus F-box genes in two haplotypes of the self-incompatibility locus of Petunia inflate[J]. Plant Cell, 2014, 26(7): 2873-2888.
[30] WILLIAMS J S, NATALE C A, WANG N, LI S,BRUBAKER T, SUN P L, KAO T. Four previously identified Petunia inflata S-locus F-box genes are involved in pollen specificity in self-incompatibility[J]. Molecular Plant,2014, 7(3): 567-569.
[31] IKEDA K, IGIC B, USHIJIMA K, YAMANE H, HAUCK N R, NAKANO R, SASSA H, IEZZONI A F, KOHN J R, TAO R. Primary structural features of the S haplotype-specific F-box protein, SFB, in Prunus[J]. Sexual Plant Reproduction, 2004, 16(5): 235-243.
[32] 張伟, 何业华, 栾爱萍, 李晶晶, 吴竞, 谢桃, 丁雅琦, 薛彪, 刘朝阳. ‘神湾’ 菠萝与亲缘关系不同的父本的交配亲和性研究[C]. 郑州: 中国园艺学会, 2019: 90.
