环境DNA宏条形码在浮游动植物多样性研究中的应用
2023-08-14吴泳江蔡明成孙翰昌胡慧蝶邓雅心龙应根
吴泳江,蔡明成,孙翰昌*,胡慧蝶,3,邓雅心,3,龙应根,3
(1.重庆文理学院园林与生命科学学院,重庆402160;2.重庆文理学院生态渔业产业化技术创新中心,重庆 402160;3.重庆三峡学院生物与食品工程学院,重庆 404100)
浮游植物是水生生态系统的初级生产者,其生产力占地球总生产力的一半以上[1]。浮游动物是浮游植物的主要消费者,是连通初级生产者与高营养级动物的关键营养纽带[2]。浮游动植物种类丰富、数量庞大、个体微小、分布广泛,导致基于传统形态学鉴定方法难以准确区分其种类,主要有以下原因:1)对鉴定人员形态学分类知识要求较高,基于主观形态判断难以标准化,且费时、费力;2)浮游动植物的生命周期短,生命周期内形态变化大;3)浮游动植物种间形态特征的趋同进化,导致形态学差异不明显;4)浮游动植物的形态表型存在可塑性,形态区分不精确。而最新的环境DNA(Environmental DNA,eDNA)宏条形码技术针对上述难题提供了一套崭新的解决方案,其无需分离生物个体,仅需提取浮游动植物遗留在水体环境中的DNA,基于特定的条形码基因设计通用引物完成PCR扩增,然后通过高通量测序和数据库序列比对,进行物种注释,最终实现物种多样性评估[3]。因此,本文重点围绕eDNA宏条形码技术在浮游动植物多样性研究中的应用进行综述,旨在为eDNA宏条形码技术的发展提供理论基础资料。
1 eDNA宏条形码技术概述
eDNA是指生物体遗留在环境中的DNA的总和,可直接从环境样品(水体、空气、土壤等)中提取获得,不具备任何明显的生物源[4]。水环境中的DNA,包括生物体经由粪便、皮肤、黏液等路径释放到水体中的DNA,主要为线粒体DNA和核DNA[5],其中利用线粒体DNA分析水生生物多样性的研究[6-7]较多。eDNA在所处环境中会经历衰变、浓度下降、直至无法再被检测的过程[8]。DNA条形码技术由Hebert P D N等[9]于2003年首次提出,是利用不同物种在特定基因序列(条形码)上存在的差异来开展物种鉴别的技术。随着高通量测序技术的快速发展,为能够一次性检测宏量的基因序列并且可高效检测生物多样性,eDNA宏条形码技术应运而生,相较于DNA条形码技术,其优势体现在“宏”字上、以eDNA为基本检测单位[10]。
eDNA宏条形码技术首先要采集和提取eDNA,根据检测目的选择合适的条形码基因来设计通用引物,再利用引物对eDNA进行PCR扩增和高通量测序;其次拼接和合并测序获得的双端测序序列,并过滤掉低质量、嵌合体以及小于150 bp的噪音序列,之后通常根据97%的相似度阈值将序列聚类成可操作分类单元(Operational taxonomic units,OTUs),再进行比对;最后通过生物信息学分析完成物种注释,达到物种鉴定的目的[11]。eDNA宏条形码技术研究浮游动植物具备以下优势:1)无需采集生物样本,对环境破坏少;2)不受物种性别和生理发育阶段的影响;3)能准确区分形态相似和表型可塑性高的物种;4)有统一的条形码数据库,鉴定标准统一,避免受鉴定人员主观经验的影响[2,12]。
2 eDNA宏条形码在浮游动物植物多样性研究中的采集方法
eDNA宏条形码技术研究浮游生物多样性,其采样的关键是尽可能多的采集浮游生物的eDNA。水体中eDNA的分布易受季节、自然环境、人类活动等因素的影响,因此需要依据水体环境特征,使用具有针对性的采样方案。1)溪流、湍急的河水和江水属于流动水体,eDNA混合较为均匀,其采样只需采集表层水[13],通常以主观确定的距离间隔从河岸和沿着水道取样,并在一个采样点采集多个样品。随着水体自上游向下游流动,河口处造就了丰富的物种,因此在河口处采样可以较为全面地整合河流网络的生物多样性信息[14]。2)湖泊、水库、池塘等静止水体,通常采集沿岸表层水即可[15]。但对于大型湖泊,还需进行精细尺度的eDNA采样或深水区采样。3)浮游动植物是海洋食物网的基础[16],但海水中的eDNA较为破碎,且降解比例较高,因而有必要综合采用基于滤网富集样品和水样的eDNA宏条形码技术[2]。仅仅在海岸线或近海海域采样,导致采样空间覆盖高度较低,生成的物种列表不全,因此需要采用覆盖所调查区域的网格抽样设计方法[17]。而且海洋的垂直空间很大,海水深度对浮游动植物的eDNA组成也有很大的影响,采样时最好覆盖全深度范围的eDNA样本。通常情况下每个采样点采集水样1~2 L,如果浮游生物分布范围广或丰度低,则需要加大取样范围和取水量[18-19]。为降低实验误差和提高目标物种检出率,通常要设置至少3个重复。
水样采集后还需要从中收集eDNA,主要采用过滤法、离心法、沉淀法。过滤法指使用抽滤装置和滤膜对水样进行抽滤,使eDNA富集在滤膜上;离心法指使用离心机对水样做离心处理,使eDNA沉降在离心管底部;沉淀法指使用无水乙醇和乙酸钠对水样中的eDNA进行聚集沉淀。使用过滤法可获得最高浓度的eDNA,其次是离心法,最低的是沉淀法[20-21]。此外,可将过滤法、离心法和沉淀法相结合,获得更为丰富的eDNA。为减少污染和降解,可将收集到的eDNA置于95%乙醇中,放入-20 ℃冰箱中保存,并及时完成扩增和宏条形码测序。在eDNA样品采集过程中,要尽量避免采样环境污染而造成的假阳性结果的出现,因而建议使用10%漂白剂清洗采样器械并消毒,采样过程中操作人员要戴好手套和口罩,并设置阴性对照(空白对照),实时监控污染状况;尽量使用无人机械技术进行采样操作,避免人为采样造成的交叉污染。
3 eDNA宏条形码在浮游植物多样性研究中的应用
浮游植物处于水体食物链的最底端,作为浮游动物和部分水体动物的食物[22-23],是水生生态系统能量传递以及物质流动的基础[24-26],具有细胞结构简单、生长周期短、个体微小等典型特点[27-28],因而其种群结构和丰度能敏锐地反映水域生态环境变化情况[29-31]。eDNA宏条形码技术在浮游植物多样性研究中的应用关键在于选择合适的条形码基因,再针对选定的条形码设计通用引物,完成扩增和测序。目前使用较为广泛的是单基因条形码,其来源于核糖体基因、线粒体基因或叶绿体基因。核糖体基因组主要为内转录间隔区(Internal transcribed space,ITS)、18S rRNA、16S rRNA;线粒体基因组主要为细胞色素C氧化酶Ⅰ(Cytochrome oxidase Ⅰ,COⅠ)、细胞色素C氧化酶Ⅲ(Cytochrome oxidase Ⅲ,CO Ⅲ);叶绿体基因组主要为matK、rbcL、atpB;其他条形码基因包括rpoB、trnL、tufA、rpoC1等。张丽娟等[32]、张莉等[33]、刘卫东等[34]、王晨等[35]在使用eDNA宏条形码技术研究我国海洋、江河、湖泊中的浮游植物多样性时均使用的是18S rRNA基因。这是因为该基因的物种覆盖率较高且条形码数据库较为完善,从而被优先用于浮游植物多样性研究。18S rRNA基因又分为V4可变区和V9可变区,它们均被广泛用作目标基因而完成引物设计[32-35]。基于18S rRNA条形码基因设计的通用引物在利用eDNA宏条形码研究浮游植物多样性中的应用示例见表1。为了提高单基因条形码的分辨率,也可将几种条形码基因联合使用,例如多基因条形码rpoC1+rpoB+matK,但与单基因条形码相比,其分析更为复杂。此外,还有超级条形码和特定条形码,这些在浮游植物多样性研究中均具有巨大的潜力[1]。利用eDNA宏条形码技术研究浮游植物多样性的报道较多,张丽娟等[32]评估了eDNA宏条形码监测真核浮游藻类结果的精确性,发现eDNA宏条形码技术能准确监测到滇池和抚仙湖的真核藻类多样性;Malviya S等[40]和王靖淇等[37]采用eDNA宏条形码技术获得的浮游藻类类群数量要远远高于显微镜观测,且发现了很多新物种;郭婷等[41]基于eDNA宏条形码技术快速监测出鄱阳湖中的浮游植物丰富度、均匀度及其空间分布,有效评估了鄱阳湖的生态系统健康状况。eDNA技术具有简便、高效、准确等优势,在浮游植物研究和多样性监测中被广泛应用。

表1 基于18S rRNA条形码基因设计的通用引物在利用eDNA宏条形码研究浮游植物多样性中的应用示例Tab.1 Application examples of universal primer based on 18S rRNA barcode gene in eDNA metabarcoding for monitoring phytoplankton diversity
4 eDNA宏条形码在浮游动物多样性研究中的应用
浮游动物以浮游植物为食,是连通水体中的初级生产者(浮游植物)和高营养级动物(鱼、虾、鸟等)的关键营养枢纽[2,42-43],具有个体微小、生活史短、代谢旺盛、种类繁多、数量庞大、分布范围广等特点,因而对水体环境变化的响应同样十分敏感[44-47],其种群结构和数量是衡量水体生态健康状况重要的依据[48-51]。利用eDNA宏条形码研究浮游动物与研究浮游植物相似,关键之处都在于选择合适的条形码基因,据此设计最佳的PCR扩增通用引物。与研究浮游植物相同,利用eDNA宏条形码研究浮游动物也使用核糖体基因中的ITS、18S rRNA、16S rRNA[2,52-53],以及线粒体基因中的COⅠ[54-55]。18S rRNA中的V9可变区比较适用于高通量测序平台,可覆盖更多的浮游动物类群,但对物种的分辨率较低,在种水平上较难区分物种[2]。COⅠ的进化率要高于18S rRNA,在物种水平上的分辨率较高,但对更高分类层级的分辨率较低[56]。高旭等[53]比较了18S rRNA-V9、COⅠ、16S rRNA条形码基因的通用引物在利用DNA研究浮游动物多样性中的差异,发现COⅠ条形码的引物更适用,这是因为其检测的物种特异性、物种覆盖度和物种识别敏感性均适中,且检出的浮游动物种群数量高于18S rRNA-V9引物和16S rRNA引物。基于18S rRNA和COⅠ条形码基因设计的通用引物在利用eDNA宏条形码研究浮游动物多样性中的应用示例见表2。孙晶莹等[57]研究表明,基于eDNA宏条形码技术可有效实现对浮游动物的半定量检测,在浮游生物多样性的研究和监测以及生物完整性评估中具有较大的实际应用价值。唐晟凯等[61]利用eDNA技术研究邵伯湖浮游动物物种,发现该技术适用于浅水湖泊浮游动物群落的监测,且其结合传统形态学观察的检出效果最佳。冯芸芝等[2]对eDNA宏条形码技术在海洋浮游动物多样性和生态学研究中的应用作了总结,发现该技术能够快速、准确、经济地分析大规模环境样本,并在海洋浮游动物生态学研究中得到了越来越广泛的应用。总之,利用eDNA宏条形码技术可快速而准确地反映浮游动物类群的多样性、分布特征、数量变化等。
5 浮游生物DNA条形码数据库
目前利用eDNA宏条形码技术研究浮游动植物多样性主要用到的数据库为生命条形码数据库(Barcode of life data systems,BOLD,http://www.boldsystems.org/index.php)和NCBI中的GeneBank数据库(https://www.ncbi.nlm.nih.gov/genbank/),二者均为开放数据库。截止2023年3月31日,BOLD数据库已收集到640 578种物种的条形码序列;录入至BOLD数据库的条形码需经过严格质量筛查和公示,也必须具备物种名称、凭证数据(存储机构和目录信息)、采集记录、标本标识符、序列大于500 bp、引物信息以及原始序列数据文件。GeneBank数据库已更新至2023年2月,其种的数据量比BOLD大,包括241 830 635条序列;录入至GeneBank中的序列也要进行基本的质量检查,但与BOLD相比,不存储序列色谱图、采集元数据或物种照片[62]。BOLD是一种具备存储功能的条形码管理工具,而GeneBank主要完成数据存储,这两个库中的很多数据是重合的,BOLD中的序列会自动传递到GeneBank,BOLD又从GeneBank中周期性挖掘条形码数据[63]。对测序获得的eDNA序列,需要使用比对工具检索比对到相应的数据库,使用BOLD-ID工具将测序获得的eDNA序列比对到BOLD数据库,可查询出与序列最匹配的物种名称、分类单元以及相近物种等;使用NCBI-BLAST工具将测序获得的eDNA序列比对到GeneBank数据库,可查询到与参考序列的相似度并打分,实现物种鉴定。除以上2个主要的数据库外,很多国家都建有本国的DNA条形码数据库,例如中国科学院建立的中国生命条形码信息管理系统和近海海洋DNA条形码资源库[64]、韩国生命条形码数据库[65]、澳大利亚淡水大型无脊椎动物的DNA条形码数据库[66]、中国淡水大型底栖无脊椎动物条形码数据库[67]等。研究者将eDNA测序后得到的序列比对到这些开放或者自有数据库,查询比对率,进而确定物种类型。构建高质量、高准确度的参考数据库是eDNA 宏条形码技术的关键,将测序获得的数据比对到目标序列,如果数据库出现错误或缺失,就会严重影响研究结果的准确性。因此,构建一个具备世界共识的高质量、高准确度数据库是一项规模庞大且艰巨的任务,需要在研究过程中不断更新完善DNA条形码数据库,以提高其精确度和可信程度。
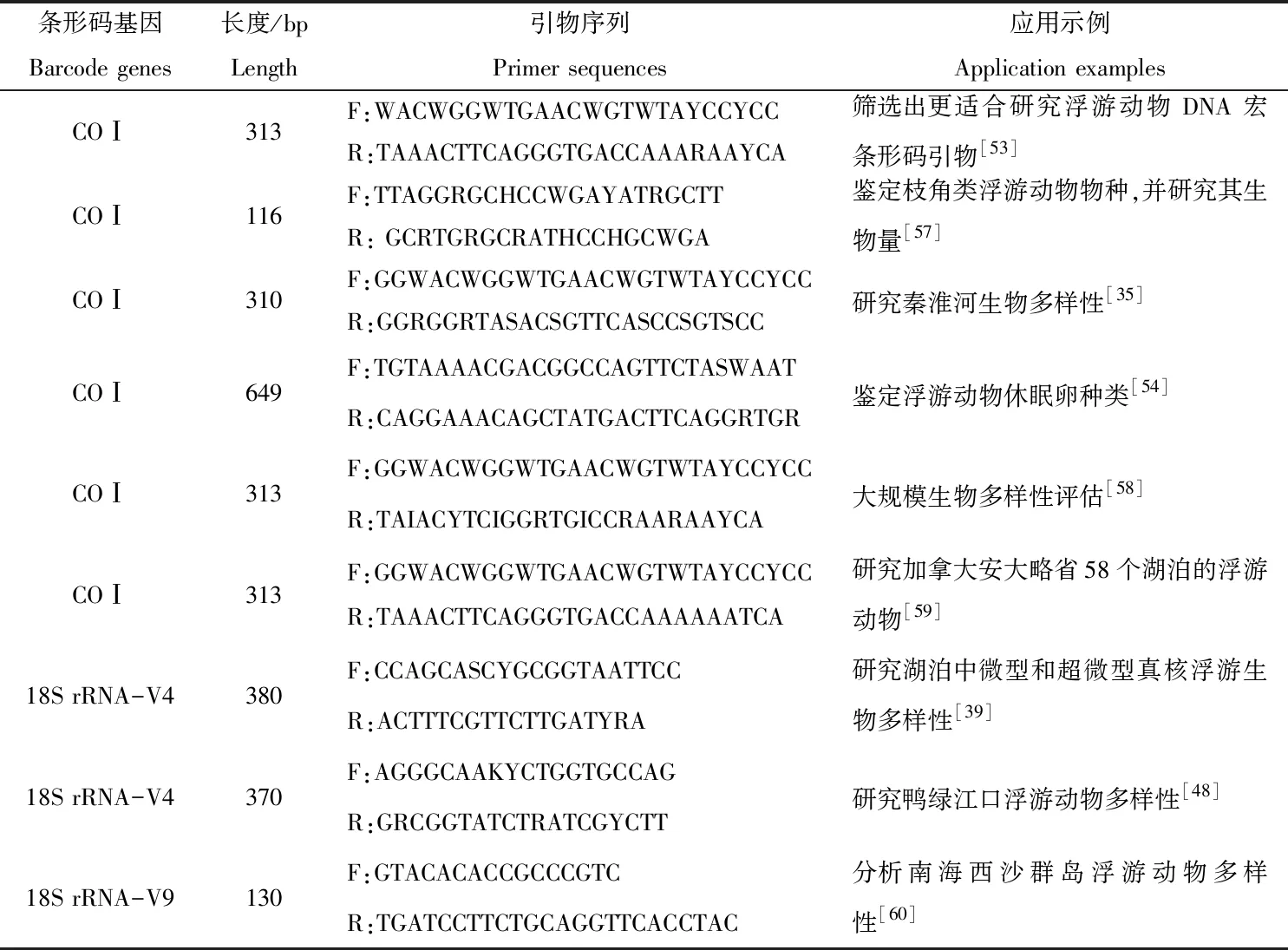
表2 基于18S rRNA和COⅠ条形码基因设计的通用引物在利用eDNA宏条形码研究浮游动物多样性中的应用示例Tab.2 Application examples of universal primer based on 18S rRNA and COⅠbarcode genes in eDNA metabarcoding for monitoring zooplankton diversity
6 小结与展望
传统浮游动植物的形态鉴定容易受鉴定人员的专业水平的影响,而eDNA宏条形码作为一种经济实惠的基因鉴别技术,具备便捷可靠、灵活实用、信息量大等优点,能快捷有序地应用于浮游动植物的鉴定与分类管理。目前eDNA宏条形码技术的应用主要通过研究浮游动植物多样性,以进一步评估水生生态系统物种多样性和健康状况。eDNA宏条形码技术主要包括eDNA样品采集、DNA提取、PCR扩增、高通量测序、序列对比与物种注释等,其准确性从样品采集开始便受影响,要根据不同的水体环境选择合适的采样方法,并在采样的过程中注意样品的代表性、避免样品被污染的情况发生。利用eDNA宏条形码技术研究浮游植物多样性通常使用18S rRNA条形码基因,而研究浮游动物多样性通常使用COⅠ条形码基因,但二者均根据目标基因设计通用引物完成PCR扩增。高质量、高准确度的参考数据库是eDNA 宏条形码技术研究应用的关键,需要在研究过程中不断完善数据库,使其具有更高的准确度且覆盖更为广泛的多基因片段,实现浮游动植物种群结构和数量的精准量化。
