三种游离核酸提取试剂盒对血浆cfDNA提取性能的比较
2023-07-31应超蔡燕宁郝淑文王婷刘沅贺赵立芳
应超 蔡燕宁, 郝淑文,5 王婷 刘沅贺 赵立芳
首都医科大学宣武医院1神经生物学研究室,4临床样本中心(北京 100053);2教育部神经变性病重点实验室(北京 100053);3国家老年疾病临床医学研究中心(北京 100053);5河北医科大学第一医院神经内科(石家庄 050031)
游离DNA(cell-free DNA,cfDNA)指体液中游离于细胞外的高度片段化DNA,其来源,包括核基因组(cell-free nuclear DNA,cf-nDNA)、线粒体基因组(cell-free mitochondrial DNA,cf-mtDNA)等[1]。cfDNA 片段大小分布可能提示其来源。例如,细胞凋亡产生160~180 bp DNA,而大于200 bp 的DNA 片段则通常来自坏死[2]。在一些病理状态下,如肿瘤、炎症等,血液中cfDNA 浓度会异常增加[3]。在癌症研究中,cfDNA 是一种具有很大潜力的生物标志物,尤其是其包含从肿瘤中脱落的循环肿瘤DNA(circulating tumor DNA,ctDNA),而ctDNA 携带与癌细胞相同的突变和甲基化模式[4-5],这使得微创地进行肿瘤早期诊断、动态监测以及治疗药物反应成为了可能[6-8]。
cfDNA 的低浓度和高度片段化限制了其在实际应用中的发展[9-10]。为了最大程度提高cfDNA产量并减少异常疾病信号的损失,有效地提取方法至关重要。商业化的cfDNA 提取试剂盒通常基于硅胶膜离心柱法以及磁珠法两种原理。然而不同品牌之间的差异导致提取效果良莠不齐,因此我们需要对不同试剂盒进行评价。在本研究中,我们采用了Agilent 2100 生物芯片分析仪、微滴式数字PCR(droplet digital PCR,ddPCR)、实时荧光定量PCR(quantitative Real-time PCR,qPCR)等常规实验技术,从提取效率、片段大小分布、回收率等方面评价了德国Qiagen 公司的Circulating Nucleic Acid Kit(硅胶膜离心柱法)以及两种基于磁珠法的国产试剂盒,旨在找到一种操作简单、省时、成本低、提取效率和回收率均较高的cfDNA提取试剂盒,以期为cfDNA 的临床应用提供实用选择。
1 资料与方法
1.1 研究对象本研究于2022 年4 - 5 月在首都医科大学宣武医院招募了10 名健康参与者,其中5 名为男性,5 名为女性。在参与研究之前,每个参与者都签署了书面知情同意书。将血液样本收集到10 mL EDTA-K2 抗凝管中,并在2~8 ℃条件下存储。在1 h 内对血液样本进行两步法离心(1 600 ×g,10 min,4 ℃;16 000 ×g,10 min,4 ℃),血浆充分混匀并分成3 个1 mL 的等分试样,立即储存在-80 ℃直至cfDNA 提取,所有血浆只冻融一次,以免影响结果的准确性。
1.2 cfDNA 提取采用3 种不同品牌的游离核酸提取试剂盒,分别为Circulating Nucleic Acid Kit(以下简称Kit A)、FineMag 大体积磁珠法血浆游离DNA 提取试剂盒(以下简称Kit B)以及核酸提取或纯化试剂(以下简称Kit C)。根据不同厂商说明书,以1 mL 血浆样本作为起始,利用上述3 种游离核酸提取试剂盒分别进行cfDNA 的提取。使用65 μL ddH2O 进行洗脱,分装cfDNA 以避免多次冻融。采用Qubit Fluorometer 4.0(美国Thermo Fisher Scientific 公司)对提取出的cfDNA 进行浓度检测。同时,使用Agilent 2100 生物芯片分析仪(美国Agilent 公司)分析样本的cfDNA 的片段大小和丰度信息[11]。
1.3 cfDNA 中ND1、ND4、RPP30 基因拷贝数的检测使用MicroDrop-100 液滴数字PCR 仪(广州永诺公司)对线粒体基因ND1、ND4 以及核基因RPP30 拷贝数进行分析,计算ND1 与ND4 拷贝数比值以评估cfDNA 中线粒体DNA 相对缺失情况;计算ND1/RPP30 拷贝数比值以分析cfDNA 中线粒体DNA 相对于细胞核DNA 的含量[12-13]。ddPCR 检测在20 μL 反应体系中进行,其中含有10 μL ddPCR Supermix 探针预混液(广州永诺公司)、4 μL 的引物探针混合液、0.3 μL 的Hind-Ⅲ(日本Takara 公司)、3.7 μL ddH2O 以及2 μL DNA 模板。根据制造商的说明,使用MicroDrop100A 液滴生成器(广州永诺公司)生成液滴,并将生成的液滴移入96 孔板中。PCR扩增采用以下条件:95 ℃10 min,1个循环;95 ℃30 s,64 ℃1 min,45 个循环。扩增后,将96 孔板放入MicroDrop-100B 检测器(广州永诺公司)中的液滴读取器并测量荧光信号,使用QuantDrop分析软件(广州永诺公司)进行分析,ddPCR 结果以copy number/mL 血浆表示。空白对照样本要求≤3 个阳性液滴。引物以及探针由上海生工生物工程有限公司合成,具体序列见表1。

表1 本研究中使用的引物和探针序列Tab.1 Primer and Probe Sequences used in this Study
1.4 cfDNA 浓度及完整性的检测使用qPCR 检测血浆cfDNA 中ALU 115 和ALU 247 两个片段的浓度,其中,ALU 115 的qPCR 结果代表样本的总cfDNA 浓度,ALU 247 的qPCR 结果用于测量坏死细胞所贡献的大片段cfDNA 含量,并通过ALU 247/ALU 115 的比值来评估cfDNA 片段的完整性[14-15]。每个20 μL 反应体系由10 μL LightCycler 480 SYBR Green I Master(瑞士Roche 公司)、4 μL DNA 模板、10 μmol/L 上下游引物各1 μL、4 μL ddH2O 组成。PCR 热循环条件如下:95 ℃10 min,1 个循环;95 ℃30 s,60 ℃1 min,共40 循环。在扩增后,进行熔融曲线分析以及琼脂糖凝胶电泳检测,以确认PCR 产物的特异性。通过标准曲线(5.2 × 100至5.2 × 10-5ng/μL)计算cfDNA 中ALU 115 和ALU 247 片段的浓度,qPCR 结果以ng/mL 血浆表示。每次运行时都包括cfDNA 样本以及空白对照,反应一式两份。ALU 115 以及ALU 247 引物的序列可见表1,引物由上海生工生物工程有限公司合成。
1.5 试剂盒对不同大小DNA 片段回收率及提取偏好性的检测在9 mL 健康人混合血浆中掺入终浓度为500 ng/mL 的Low Molecular Weight DNA Ladder(美国New England Biolabs 公司,该Ladder 包含11 条不同大小的DNA 片段,从25 bp 到766 bp),混匀并等量分成9 份,使用上述3 种试剂盒分别进行cfDNA 提取。如前所述[16],取1 μL 样本进行Agilent 2100 生物芯片分析仪检测,并用2100 Expert 软件计算Low Molecular Weight DNA Ladder 中50~766 bp 范围内10 条特定大小片段的DNA 浓度,以比较3 种试剂盒对不同大小DNA 片段的回收率和提取偏好性。
1.6 统计学方法采用SPSS 26.0 以及R(version 4.2.1)软件进行数据分析。定量资料使用Shapiro-Wilk 检验评估数据正态性,使用Levene's 方差齐性检验评估数据的方差齐性,符合正态分布的数据采用()形式表示并用单因素方差分析,进一步两两比较采用Tukey-Kramer 检验或Games-Howell 检验;非正态分布的数据采用M(P25,P75)表示并用Kruskal-Wallis H 检验,进一步两两比较采用Dwass-Steel-Critchlow-Fligner 检验。定性资料采用Fisher 精确概率法进行比较。此外,采用Spearman 秩相关系数法进行相关性分析,并使用R中的“corrplot”、“cocor”包来比较相关系数。检验水准α = 0.05。
2 结果
2.1 不同游离核酸提取试剂盒的基本特征对比见表2。

表2 3 种游离核酸提取试剂盒的基本特征Tab.2 Basic characteristics of the three cell-free DNA extraction kits
2.2 不同试剂盒提取的cfDNA 浓度及片段大小分布使用Agilent 2100 生物芯片分析仪评估了3 种试剂盒提取到的cfDNA 片段大小及浓度。如表3以及图1A 所示,3 种试剂盒提取到的cfDNA 在100~300 bp 范围内表现出显著的浓度差异(P= 0.006)。其中,Kit A 及Kit B 提取到的cfDNA 浓度显著高于Kit C(P=0.008、P=0.024)。另外,如图1B 所示,我们比较了3 种试剂盒提取的cfDNA 中单核小体主峰的检出率。其中,Kit A 及Kit B 检测到了60%的cfDNA 样本中的标志性单核小体主峰,Kit C 只检测到了30%的cfDNA 样本中的标志性单核小体主峰,但差异并无统计学意义(P=0.467)。
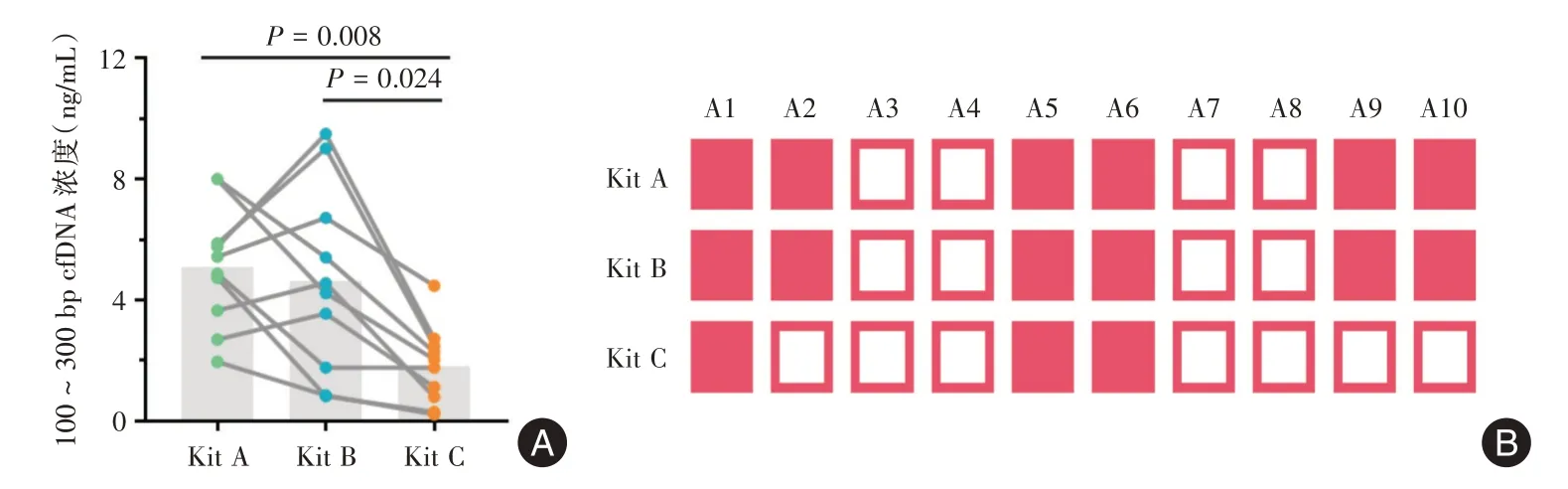
图1 基于Agilent 2100 生物芯片分析仪分析3 种游离核酸提取试剂盒的提取效率Fig.1 The extraction efficiency of the three cell-free nucleic acid extraction kits determined by Agilent 2100 bioanalyzer analysis
表3 本研究中的实验结果汇总Tab.3 Summary of experimental results in this study ±s
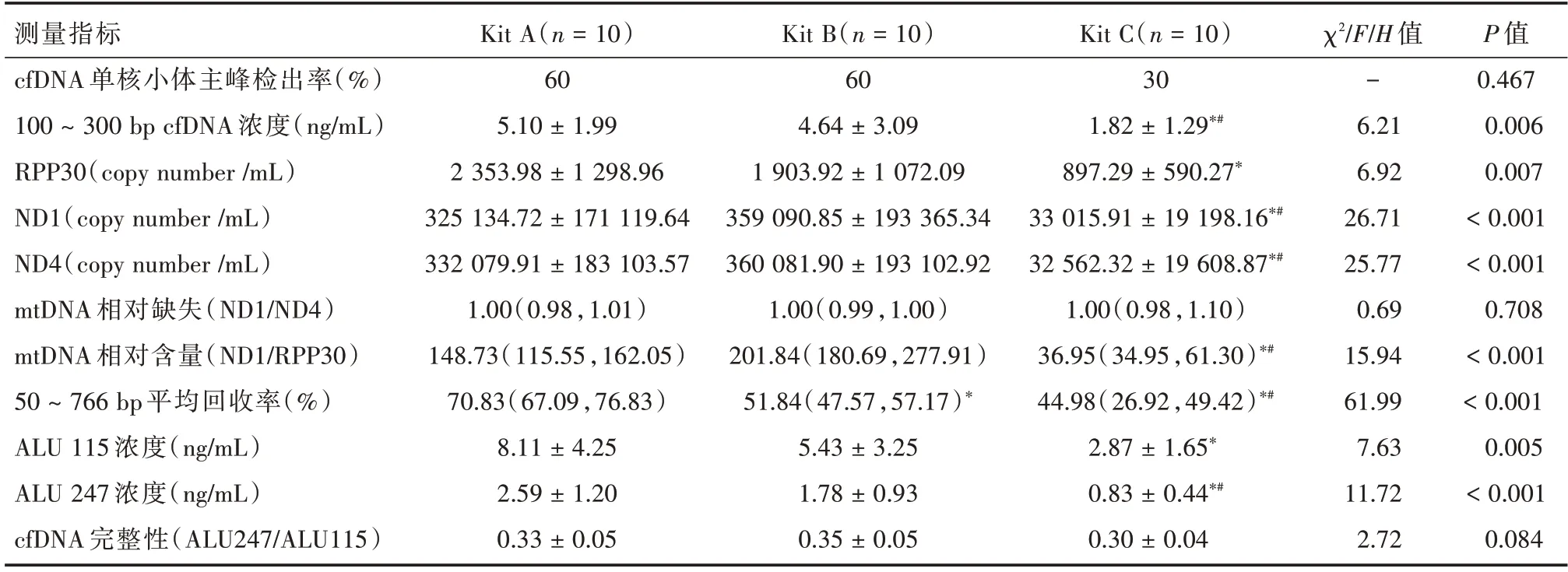
表3 本研究中的实验结果汇总Tab.3 Summary of experimental results in this study ±s
注:与Kit A 比较,*P<0.05;与Kit B 比较,#P<0.05
测量指标cfDNA 单核小体主峰检出率(%)100~300 bp cfDNA 浓度(ng/mL)RPP30(copy number /mL)ND1(copy number /mL)ND4(copy number /mL)mtDNA 相对缺失(ND1/ND4)mtDNA 相对含量(ND1/RPP30)50~766 bp 平均回收率(%)ALU 115 浓度(ng/mL)ALU 247 浓度(ng/mL)cfDNA 完整性(ALU247/ALU115)Kit A(n = 10)60 5.10 ± 1.99 2 353.98 ± 1 298.96 325 134.72 ± 171 119.64 332 079.91 ± 183 103.57 1.00(0.98,1.01)148.73(115.55,162.05)70.83(67.09,76.83)8.11 ± 4.25 2.59 ± 1.20 0.33 ± 0.05 Kit B(n = 10)60 4.64 ± 3.09 1 903.92 ± 1 072.09 359 090.85 ± 193 365.34 360 081.90 ± 193 102.92 1.00(0.99,1.00)201.84(180.69,277.91)51.84(47.57,57.17)*5.43 ± 3.25 1.78 ± 0.93 0.35 ± 0.05 Kit C(n = 10)30 1.82 ± 1.29*#897.29 ± 590.27*33 015.91 ± 19 198.16*#32 562.32 ± 19 608.87*#1.00(0.98,1.10)36.95(34.95,61.30)*#44.98(26.92,49.42)*#2.87 ± 1.65*0.83 ± 0.44*#0.30 ± 0.04 χ2/F/H 值-6.21 6.92 26.71 25.77 0.69 15.94 61.99 7.63 11.72 2.72 P 值0.467 0.006 0.007<0.001<0.001 0.708<0.001<0.001 0.005<0.001 0.084
2.3 ddPCR 测定cf-nDNA 以及cf-mtDNA 浓度为了检测不同试剂盒所提取的cfDNA 中cf-nDNA和cf-mtDNA 浓度是否存在差异,采用ddPCR 分析了cfDNA 中具有代表性的相关基因拷贝数,并比较了使用不同试剂盒对cf-mtDNA 相对含量以及相对缺失的影响。如表3 以及图2A-E 所示,3 种试剂盒提取的cf-nDNA(RPP30)和cf-mtDNA(ND1、ND4)浓度均存在显著差异(P= 0.007、P<0.001、P<0.001)。对于cf-nDNA,Kit A 的提取效率显著高于Kit C(P= 0.018);对于cf-mtDNA,Kit A 及Kit B 的提取效率显著高于Kit C(P= 0.001、P= 0.001、P= 0.001、P= 0.001)。此外,我们发现使用不同试剂盒导致了cf-mtDNA 相对含量(ND1/RPP30)出现假阳性差异(P<0.001),但cf-mtDNA 相对缺失(ND1/ND4)在3 种试剂盒之间差异无统计学意义(P= 0.708)。
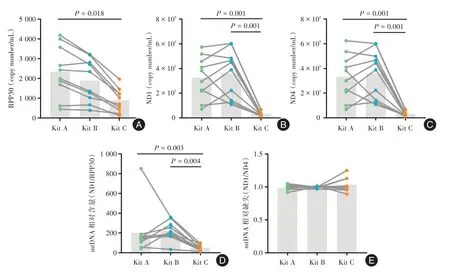
图2 基于ddPCR 分析的3 种游离核酸提取试剂盒比较Fig.2 Comparison of three cell-free nucleic acid extraction kits based on ddPCR analysis
2.4 qPCR检测血浆cfDNA浓度以及片段完整性采用qPCR 比较了3 种试剂盒提取的cfDNA 中多拷贝基因片段的浓度,并探究使用不同试剂盒进行提取是否会导致cfDNA 片段的完整性存在差异。如表3 及图3A-C 所述,3 种试剂盒所提取的cfDNA 中ALU 115 以及ALU 247 的浓度存在统计学差异(P= 0.005、P<0.001),但据此计算的cfDNA完整性无显著差异(P= 0.084)。具体而言,对于cfDNA 总浓度(ALU 115),Kit A 的提取效率显著高于Kit C(P= 0.009);对于坏死细胞来源的大片段cfDNA(ALU 247),Kit A 和Kit B 的提取效率均显著高于Kit C(P= 0.003、P= 0.030)。
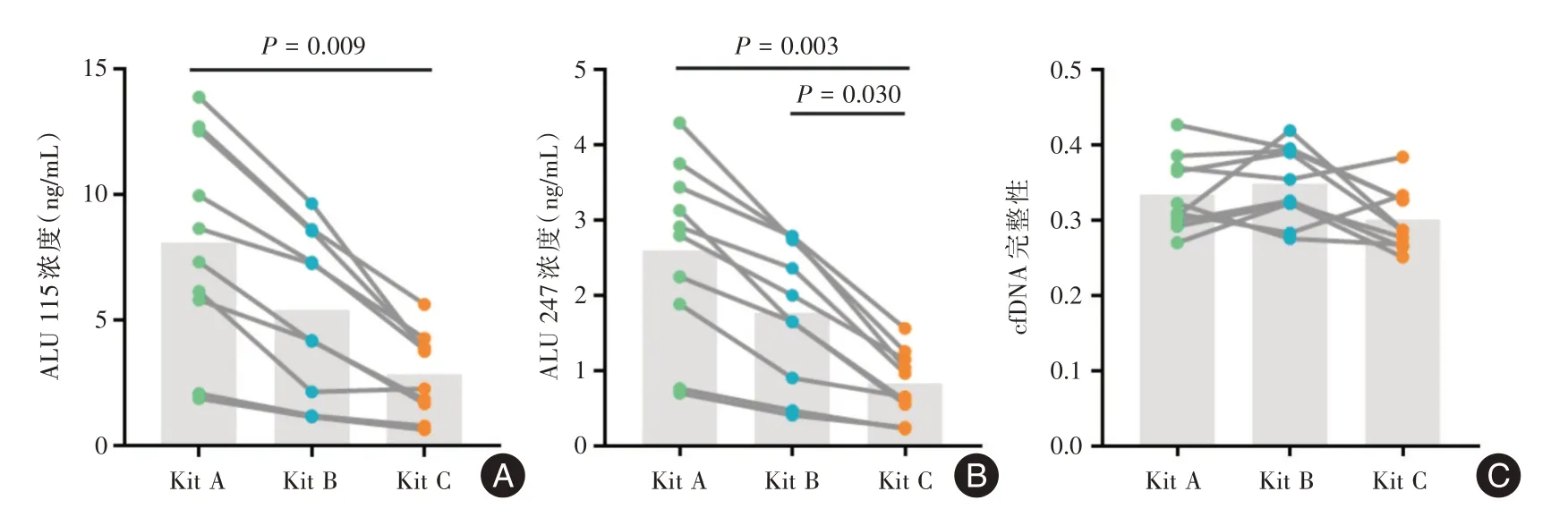
图3 基于qPCR 分析的3 种游离核酸提取试剂盒比较Fig.3 Comparison of three cell-free nucleic acid extraction kits based on qPCR analysis
2.5 不同游离核酸提取试剂盒对DNA Ladder 的回收率及提取偏好性的影响采用Agilent 2100生物芯片分析仪评估了3 种试剂盒对50~766 bp范围内10 个不同大小DNA 片段的回收率及提取偏好性的差异。如图4A 所示,所有试剂盒都不同程度地回收了75~766 bp 范围内的所有DNA 片段。我们比较3 种试剂盒对这些DNA 片段的回收率发现,除了766 bp 片段外,Kit A 对其他各片段回收率均显著高于Kit B(均P<0.05)以及Kit C(均P<0.05)。Kit A 对766 bp 片段的回收率显著高于Kit C(P= 0.007),但与Kit B 比较差异无统计学意义(P=0.058)。值得注意的是,Kit B对75 bp以及100 bp 片段的回收率显著高于Kit C(P= 0.007,P<0.001)。同时,Kit B对150、200 bp以及350 bp片段的回收率也存在高于Kit C 的趋势(P= 0.072,P= 0.062,P= 0.073)。对于50 bp 片段的回收,Kit A 的回收率约为59.51%,Kit B 的回收率仅为1.08%,而Kit C未能回收到该片段。就平均回收率而言,如表3 及图4B 所示,在50~766 bp 范围内,3 种试剂盒的平均回收率存在显著差异(P<0.001),Kit A 的平均回收率显著高于Kit B(P<0.001)以及Kit C(P<0.001)。同时,Kit B 的平均回收率显著高于Kit C(P= 0.002)。提取偏好性方面,在50~766 bp 范围内,Kit A 不存在明显的片段大小回收偏好性,而另外两种试剂盒在50~100 bp 范围内的回收率较低。但随着回收片段长度的增加,两种试剂盒回收率逐渐提升并趋于稳定。
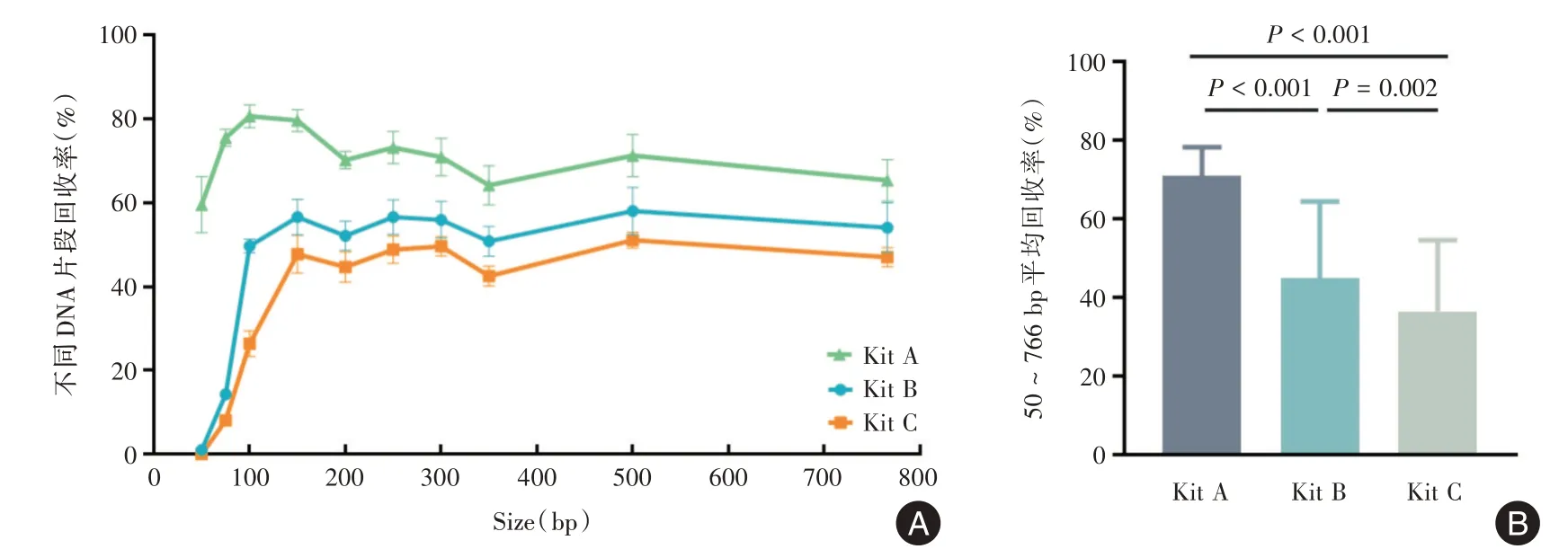
图4 3 种游离核酸提取试剂盒对外源性DNA Ladder 的回收效率比较Fig.4 Comparison of the recovery efficiency of the three cell-free nucleic acid extraction kits for the spiked-in DNA ladder
2.6 不同游离核酸提取试剂盒的cfDNA 提取相关性分析为了比较3 种试剂盒在ddPCR 以及qPCR 实验中所得到结果的相关性,进行了基于Kit A 的相关矩阵分析。如图5A-B 所示,在ddPCR方面,Kit A 与Kit B 在RPP30、ND1、ND4 拷贝数方面均显示出显著正相关(P<0.05),其Rho 值分别为0.96、0.73、0.74。同时,Kit C 也取得了类似的显著正相关(P<0.05),其Rho 值分别为0.91、0.80、0.76,且Kit C 与Kit A 的相关系数和Kit B 与Kit A的相关系数相比差异无统计学意义(P>0.05);在qPCR 方面,Kit A 分别与Kit B 在ALU 115 及ALU 247片段浓度方面也呈现出显著的正相关(P<0.05),相应的Rho 值分别为0.95、0.96。同时,Kit C 也表现出类似的显著正相关性(P<0.05),其Rho 值分别为0.84、0.89,且Kit C 与Kit A 的相关系数和Kit B与Kit A 的相关系数相比无统计学差异(P>0.05,P=0.055)。此外,由于RPP30、ALU115以及ALU247均来自于细胞核基因组,我们也探究了利用三种游离核酸提取试剂盒获得的cf-nDNA 拷贝数和浓度之间的相关性。结果显示,Kit A 的RPP30拷贝数与Kit B 的ALU115 及ALU247 浓度均呈现出显著正相关性,相应的Rho 值分别为:0.95、0.97(P<0.05),而Kit A 的RPP30 拷贝数与Kit C 的ALU115 及ALU247 的Rho 值分别为:0.94、0.90(P<0.05),两种试剂盒与Kit A 的相关系数比较无显著差异(P>0.05);同样地,Kit A 的ALU115及ALU247与Kit B 的RPP30 也均呈现出显著正相关性,相应的Rho 值分别为:0.96、0.89(P<0.05),Kit A 的ALU115 及ALU247 与Kit C 的RPP30 的Rho 值分别0.89、0.78(P<0.05),两种试剂盒与Kit A 的相关系数比较同样无显著差异(P>0.05)。
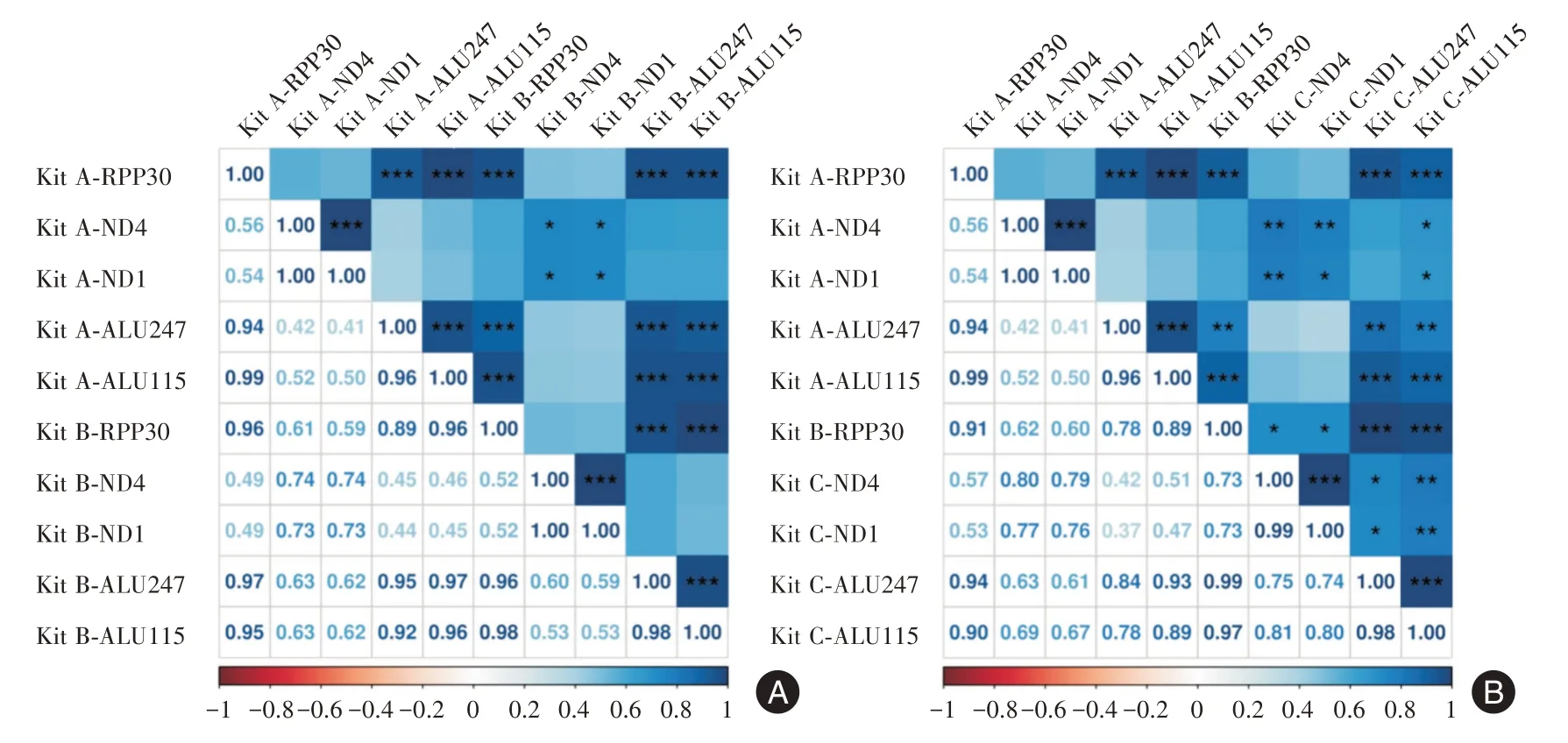
图5 3 种游离核酸提取试剂盒实验参数相关系数矩阵分析Fig.5 Analysis of the correlation coefficient matrix of the experimental parameters for the three cell-free nucleic acid DNA extraction kits
3 讨论
自1948年MANDEL和MÉTAIS首次描述cfDNA[17]以来,cfDNA 逐渐成为肿瘤领域的研究热点[18-22]。传统的穿刺活检不仅具有侵入性,而且难以完全获得肿瘤异质性和疾病进展等信息。基于cfDNA的液体活检提供了一种可重复评估患者癌症基因组特征的非侵入性方法,因为cfDNA 具有与同源组织相同的序列特征(例如染色体重排、基因突变、甲基化等)[23]。然而,cfDNA 含量低以及高度片段化的性质给下游实验分析带来了挑战。最大限度提高cfDNA 的回收效率有助于确保实验结果的可靠性,特别是针对ctDNA 的相关分析,因为ctDNA 通常只占总cfDNA 的一小部分[24]。此外,部分ctDNA通常较短(132~145 bp,甚至<100 bp),且携带更多的癌症相关突变[25-27]。若无法最大程度地回收这部分cfDNA,将严重影响cfDNA 作为癌症或其他疾病生物标志物的临床应用。因此,有必要在实验进行前,考虑提取方法的不同以及试剂盒选择所带来的局限性。
不同cfDNA 提取试剂盒在提取效率以及回收率等方面存在巨大差异。已有研究比较了各种商业试剂盒对cfDNA 的提取效率,认为Qiagen 的Circulating Nucleic Acid Kit 是性能较好的试剂盒之一[28-29]。然而,进口试剂盒往往价格昂贵、货期长、提取过程耗时。本研究将Qiagen 的Circulating Nucleic Acid Kit 试剂盒纳入了本次实验中,并与两种基于磁珠法的国产试剂盒进行对比。在成本方面,Qiagen试剂盒单人次价格远高于另外两种,且在操作时间上存在劣势。Agilent 2100 生物芯片分析仪结果表明,Kit A 及Kit B 所提取的100~300 bp 大小的cfDNA 浓度显著高于Kit C。在ddPCR 和qPCR的定量分析实验中,无论是cf-nDNA 的扩增还是cf-mtDNA的扩增,Kit B取得了与Kit A类似的结果。最后,我们比较了3 种试剂盒在50~766 bp 范围内的回收率。与之前研究报告的回收率相似[16],在50~766 bp 范围内,基于硅胶膜离心柱法的Kit A 取得了71.01%的平均回收率,显著高于基于磁珠法的两种国产试剂盒;考虑到ctDNA 通常小于150 bp,我们比较了3 种试剂盒对50、75、100 bp 和150 bp 这4 个片段的回收率。同样,Kit A 表现最佳,取得了60%~81%不等的回收率,显著高于Kit B 以及Kit C;而Kit B 对75 bp、100 bp 片段的回收表现也显著优于Kit C,但Kit B 和Kit C 对50 bp以及150 bp 片段的回收表现类似。因此,在提取偏好方面,两种基于磁珠法的试剂盒对小片段cfDNA,特别是<100 bp 的cfDNA 回收率不高,但随着所回收片段长度的增加,其回收率趋于稳定。
综上所述,本研究利用qPCR、ddPCR 和Agilent 2100 生物芯片分析仪对3 种试剂盒所提取的cfDNA 进行全面评估,并分析比较各试剂盒的提取效率、回收率以及偏好性。结果表明,Kit A 在以上几项结果的表现均优于Kit B 和Kit C;若考虑时间及经济成本,Kit B 可能是Kit A 的一种替代选择。
