反硝化细菌Y39-6的基因组及生理生化特征研究
2023-07-21韩国秀张多英张彦龙
韩国秀, 张多英, 张彦龙, 张 军
曾伟民1,2, 赵丹丹1,2, 雷 虹1,4
(1.黑龙江大学 农业微生物技术教育部工程研究中心, 哈尔滨 150500; 2.黑龙江大学 生命科学学院 黑龙江省普通高校微生物重点实验室, 哈尔滨 150080; 3.黑龙江大学 建筑工程学院, 哈尔滨 150080;4.黑龙江大学 生命科学学院 黑龙江省普通高校分子生物学重点实验室, 哈尔滨 150080)
0 引 言
假单胞菌(Pseudomonas)是革兰氏阴性菌中种数最多的一个属,目前该属有368 个种[1]。该属的大多数成员都有共同的特征,如革兰氏染色阴性、杆状、有鞭毛、不形成芽孢、过氧化氢酶阳性以及氧化酶阳性等。假单胞菌可存在于水、土壤、空气、植物以及人体肠道中,生活环境范围广泛,代谢途径多样。由于假单胞菌属分类非常复杂,因此在种水平上对其进行鉴定仍然是一项艰巨的任务。
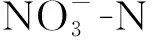
1 材料与方法
1.1 试验材料
菌株Y39-6从哈尔滨周边地下水中分离筛选获得[4]。试验用LB培养基:胰蛋白胨 10 g·L-1,酵母粉 0.5 g·L-1,NaCl 10 g·L-1,pH调为6.9~7.0。
1.2 试验方法
1.2.1 基因组的DNA提取、测序与拼接
配制100 mL LB液体培养基,在121 ℃下灭菌30 min,冷却后,以2%接种量将菌株Y39-6接入100 mL灭菌的LB培养基中,在20 ℃、160 r·min-1条件下过夜培养,获得新鲜菌液。采用Ezup柱式细菌基因组DNA抽提试剂盒(上海生工生物工程股份有限公司)从菌株Y39-6中提取基因组DNA[7]。
委托上海美吉生物医药科技有限公司(Shanghai Majorbio Bio-pharm Technology Co., Ltd) 对提取到的基因组DNA进行全基因组序列测序。通过SOAPdenovo 2.04[8]软件对菌株Y39-6的基因序列进行组装;通过Glimmer[9]软件(http://ccb.jhu.edu/software/glimmer/index.shtml)对基因组中的编码序列(CDS)进行预测;通过RNAmmer[10]软件对基因组中包含的 rRNA 进行预测,获得基因组中所有rRNA的种类、位置和序列信息;通过tRNAscan-SE v2.0[11]软件(http://trna.ucsc.edu/software/)对基因组中包含的tRNA 进行预测,获得基因组中tRNA的核苷酸序列信息、反密码子信息及二级结构信息,并通过CGView[12]软件绘制菌株Y39-6的基因组圈图。
1.2.2 基因组注释
通过Diamond[13]软件,将菌株Y39-6的蛋白质序列与 eggNOG[14](Evolutionary genealogy of genes: Non-supervised Orthologous Groups, http://eggnog.embl.de/)数据库中的物种蛋白序列进行比对,得到基因COG(Clusters of Orthologous Groups of proteins)注释。通过Blast2go[15]软件,使用Gene ontology (http://www.geneontology.org/)数据库对菌株Y39-6的基因组进行基因GO注释。
1.2.3 氮代谢基因
菌株Y39-6具有异养反硝化和自养脱氮能力,通过KEGG基因注释结果分析菌株Y39-6的氮代谢相关基因。
1.2.4 比较基因组学
利用前期获得的菌株Y39-6(CP069594)的16S rRNA序列建立系统发育树,获得与菌株Y39-6近缘的模式菌株。将近缘的菌株基因组信息与菌株Y39-6的基因组信息进行对比分析,并通过在线Venn图制作软件(https://orthovenn2.bioinfotoolkits.net/home)绘制菌株Y39-6和近缘模式菌株基因序列间的同源关系图。
1.2.5 生理生化特征
通过Biolog细菌自动鉴定系统[16]分析菌株Y39-6对不同碳源的利用与代谢情况,并比对其与近缘模式菌株在碳源利用和代谢方面的差异。利用美国MIDI公司研发的SHERLOCK全自动微生物鉴定系统鉴定菌株Y39-6,用美国Agilent公司的6890型气相色谱仪分析菌株Y39-6,将所得的气相色谱图谱与软件SHERLOCK MIS4.5(Microbial Identification System,美国MIDI公司)和LGS4.5进行分析比对,获得菌株Y39-6的脂肪酸种类及含量。
2 结果与讨论
2.1 菌株Y39-6的全基因组信息
经测序,得到菌株Y39-6的基因组完成图,菌株Y39-6全基因组序列长度为7 021 566 bp,DNA的G+C含量为60.75%。菌株Y39-6的全基因组序列已上传至NCBI(https://www.ncbi.nlm.nih.gov/),SUBID为SUB8969722,登录号为CP069594。CGView基因组圈图能够全面展示基因在正、反义链上的分布情况、基因的COG功能分类情况、G+C含量和基因组岛等信息,使人们更直观、更全面地认识菌株的基因组特征[17]。由CGView软件绘制的基因组圈图如图1所示。
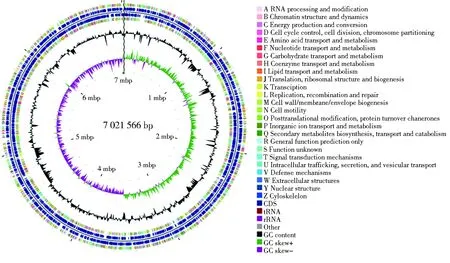
图1 菌株Y39-6的基因组圈图
2.2 菌株Y39-6的基因组注释
通过与EggNOG数据库中物种的蛋白序列进行比对,可以对菌株Y39-6的预测蛋白进行基因家族的归类,并给出该家族相应的功能注释信息。不同数据库中的基因功能描述可能存在差异,GO注释能够对基因的功能进行标准描述[19],COG是对同源蛋白簇进行注释,除获得基本功能、推导代谢途径外,还可以分析进化关系。COG和GO注释结果分别如图2和图3所示。

图2 菌株Y39-6的COG分类统计直方图

图3 菌株Y39-6的GO分类统计直方图
COG注释的基因一共有5 378个,将蛋白分为21个类型,COG注释到的功能已知基因占所有基因的84.44%。由图2可知,功能未知基因占比最高,为1 537个。在功能已知的基因中,在氨基酸的运输和代谢,转录,无机离子的运输和代谢,能量生产和转换,细胞壁、膜、包膜生物合成,信号转导,碳水化合物的运输和代谢方面的基因较多,基因数量分别为497、461、369、360、293、290和259个。菌株Y39-6在进行氨基酸运输和代谢方面的基因最多,氨基酸是蛋白质组成的基本物质,因此,菌株Y39-6可能在蛋白质合成方面具有独特优势。
GO注释将基因分为细胞组成、分子功能和生物过程三大类。菌株Y39-6基因组的GO注释结果一共有4 533个,与细胞组成、分子功能和生物过程相关的基因数量分别为2 025、3 748和2 059个,GO注释的基因占所有基因的71.17%。由图3可知,在细胞组成方面,膜组成相关基因数量最多,为1 177个,占GO注释基因总数的18.48%;在生物过程方面,转录调控、DNA模板相关基因数量最多,为146个,占GO注释基因总数的2.29%;在分子功能方面,DNA结合相关基因数量最多,为549个,占GO注释基因总数的8.62%。
根据菌株Y39-6的COG和GO注释结果可以发现,该菌株在膜的组成、形成方面的基因数量均较多,可能是因为该菌株从低温地下水中分离,菌株的生长容易受到环境胁迫而发生变化,生物膜的组成和形成与微生物适应不良环境相关[20],这可能也是菌株Y39-6适应低温环境的原因之一。
2.3 氮代谢基因分析
通过基因注释结果分析,得到菌株Y39-6具有Nar、Nir、Nor、Nos和Nap 5种与氮代谢相关的基因簇。Nar为硝酸盐还原酶,用于将硝酸盐还原成亚硝酸盐;Nir为亚硝酸盐还原酶,用于将亚硝酸盐还原成氧化氮;Nor为氧化氮还原酶,用于将氧化氮还原成氧化亚氮;Nos为氧化亚氮还原酶,用于将氧化亚氮还原成氮气;Nap为周质硝酸盐还原酶,用于将硝酸盐还原成亚硝酸盐。基因分布情况如表1所示,其中norV、norR、norD、norQ、norC、norB、napC、napB、napA、napD、napE、nosR、nosZ、nosD、nosL、narI、narJ、narH、narK、narL和nirS参与异养反硝化过程,napC、napB、napA、napD、napE、nirD和nirB参与自养脱氮过程。
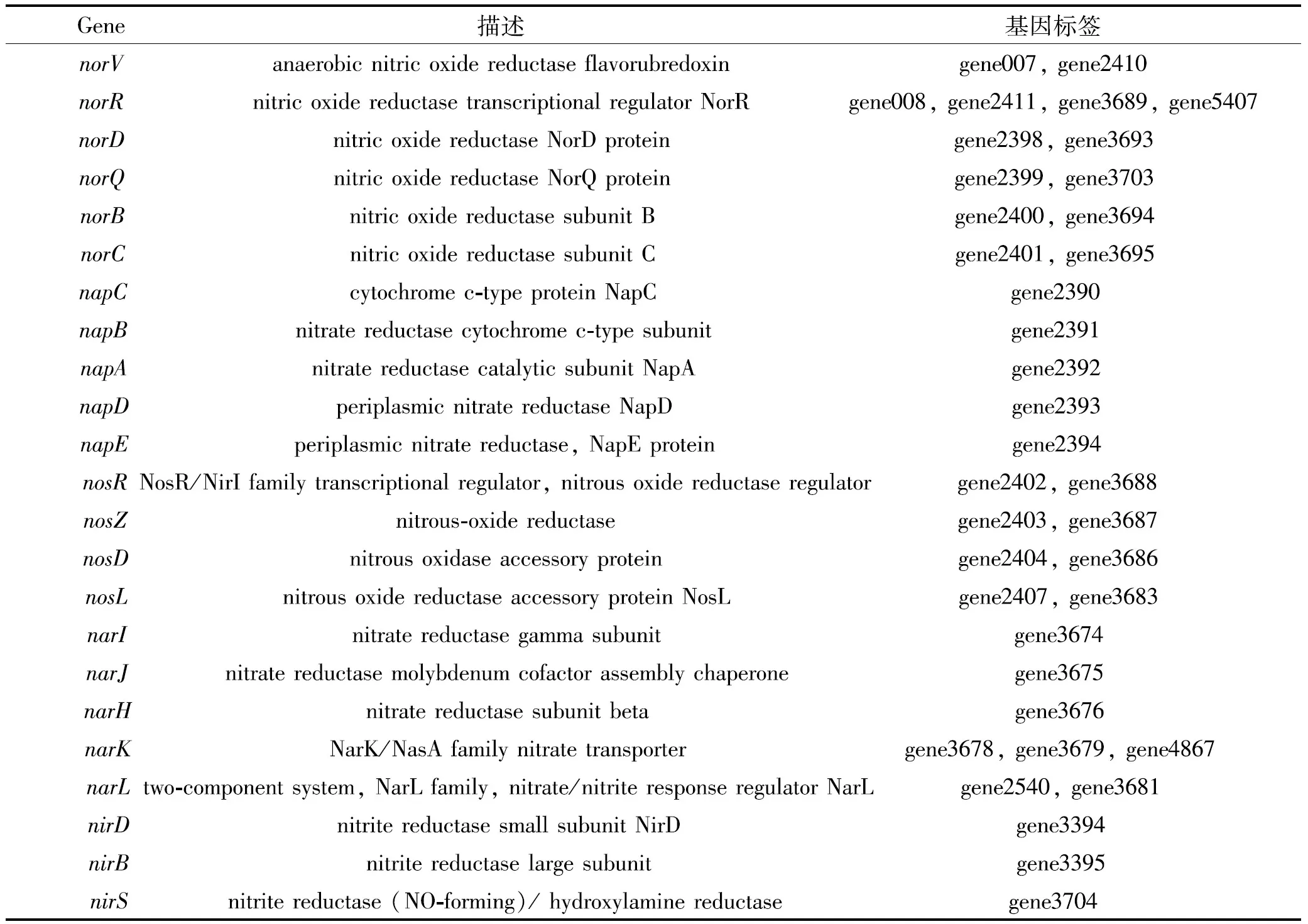
表1 氮代谢基因
2.4 比较基因组学分析
2.4.1 基因组基本特征
前期研究发现,菌株Y39-6的16S rRNA基因序列与PseudomonasveroniiCIP 104663T、Pseudomonasextremaustralis14-3T、PseudomonasgrimontiiCFML 97-514T和PseudomonasmarginalisATCC 10844T的相似度很高,超过99%。
菌株Y39-6与上述近缘模式菌株的基因组基本特征进行了比对分析,结果如表2所示。G+C含量是每个物种基因组的重要指标,不同的基因组具有不同的G+C含量,菌株Y39-6的G+C含量为60.75%,与PseudomonasveroniiCIP 104663T的G+C含量(60.70%)最接近,与其他3株菌株的G+C含量相差也不大,属于假单胞菌属范围[21],与16S rRNA分析结果一致;菌株Y39-6的rRNA数量为19个,高于以上4株菌株,表明菌株Y39-6在DNA转录方面的基因数量可能比这4株菌株多。

表2 菌株Y39-6和近缘模式菌株的基因组基本特征
2.4.2 基因组同源性分析
维恩图(Venn diagram)是一种非常直观、易懂,用于描述数据集合之间的交集、并集、补集等关系的图。菌株Y39-6与近缘模式菌株基因组同源关系如图4所示。菌株Y39-6、PseudomonasveroniiCIP 104663T、Pseudomonasextremaustralis14-3T、PseudomonasgrimontiiCFML 97-514T、PseudomonasmarginalisATCC 10844T5株菌株间共有的基因簇有4 021个,占总基因簇的72.06%。菌株Y39-6与PseudomonasveroniiCIP 104663T、Pseudomonasextremaustralis14-3T、PseudomonasgrimontiiCFML 97-514T、PseudomonasmarginalisATCC 10844T的共有基因分别有368、31、13、40个,菌株Y39-6与PseudomonasveroniiCIP 104663T的共有基因最多。菌株Y39-6、PseudomonasveroniiCIP 104663T、Pseudomonasextremaustralis14-3T、PseudomonasgrimontiiCFML 97-514T、PseudomonasmarginalisATCC 10844T的特有基因簇分别有15、7、55、11、41个。菌株Y39-6特有基因簇的功能主要表现在跨膜运输、DNA介导的转换、硫化合物代谢、肽酶活力、抗生素生物合成、脂肪酸分解和烷基磺酸盐单加氧酶7个方面。
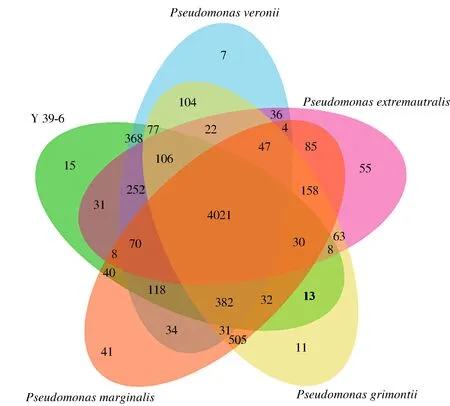
图4 菌株Y39-6基因组同源分析Fig.4 Genome homology analysis of strain Y39-6
2.5 生理生化特征分析
2.5.1 碳源利用
由BIOLOG检测结果可知,菌株Y39-6可利用的碳源有β-D-葡萄糖、1%乳酸钠、甘油、L-丙氨酸、L-精氨酸、L-谷氨酸、L-焦谷氨酸、四硫酸奎纳、D-葡萄糖酸、葡糖醛酰胺、乙酸、β-羟基-D,L-丁酸、γ-氨基丁酸、L-苹果酸、L-乳酸、α-氧化-谷氨酸、柠檬酸和奎宁酸;微弱利用的碳源有糊精、D-海藻糖、蔗糖、D-甘露糖、D-果糖、D-半乳糖、D-岩藻糖、L-岩藻糖、肌苷、D-丝氨酸、D-山梨醇、D-甘露醇、D-阿拉伯糖醇、肌醇、D-果糖-6-磷酸、D-丝氨酸、L-天冬氨酸、L-组氨酸、L-丝氨酸、盐酸胍、D-半乳糖醛酸、L-半乳糖酸内脂、粘液酸、D-糖质酸、β-羟基-丙乙酸、甲基丙酮酸、D-苹果酸、乙酰乙酸和丙酸;不能利用二糖和多糖;对醋竹桃霉素、利福霉素SV、万古霉素、林肯霉素、四唑蓝、四唑紫具有抗性;氯化锂、二甲胺四环素不抑制菌株生长;对夫西地酸、亚碲酸钾、溴琥珀酸、萘啶酮酸、氨曲南抗性较弱;不能水解明胶。
将菌株Y39-6和PseudomonasveroniiCIP 104663T[22]、Pseudomonasextremaustralis14-3T[23]、PseudomonasgrimontiiCFML 97-514T[24]和PseudomonasmarginalisATCC 10844T[25]的碳源利用情况进行总结,结果如表3所示。菌株Y39-6的生理生化特征与PseudomonasveroniiCIP 104663T的生理生化特征较为接近,两菌株的生长温度范围相似,最佳生长温度相似,并且均具有硝酸盐还原能力,更支持了菌株Y39-6与Pseudomonasveronii同源[26]。

表3 菌株Y39-6与近缘模式菌株生理生化特征的差异
2.5.2 脂肪酸含量
将菌株Y39-6的脂肪酸种类和含量与PseudomonasveroniiCIP 104663T、Pseudomonasextremaustralis14-3T、PseudomonasgrimontiiCFML 97-514T、PseudomonasmarginalisATCC 10844T的脂肪酸含量进行对比,如表4所示。由表4可知,菌株Y39-6检测到的脂肪酸种类有C10∶0 3OH、C12∶0、C12∶0 2OH 、C12∶0 3OH、C16∶0、C17∶0 cyclo、C18∶1 ω7c和C19∶0 cyclo ω8c 8种,PseudomonasveroniiCIP 104663T、Pseudomonasextremaustralis14-3T、PseudomonasgrimontiiCFML 97-514T和PseudomonasmarginalisATCC 10844T的脂肪酸种类分别为11种、11种、10种和10种,菌株Y39-6的脂肪酸种类比其他4株假单胞菌所含有的脂肪酸种类少,但这5株菌株的C16∶0饱和脂肪酸含量均较高,分别为22.05%、27.96%、29.49%、25.7%和32.4%。菌株Y39-6的主要脂肪酸为C16∶0和C17∶0 cyclo,含量分别为22.05%和21.25%。该菌株的C12∶0 2OH、C12∶0 3OH、C17∶0 cyclo和C19∶0 cyclo ω8c 4种饱和脂肪酸含量均高于其他4株菌株。菌株Y39-6属于Pseudomonasveronii物种,但两者在脂肪酸种类与含量方面存在差异,表明种间差异大,菌株Y39-6具有独特的特异性。
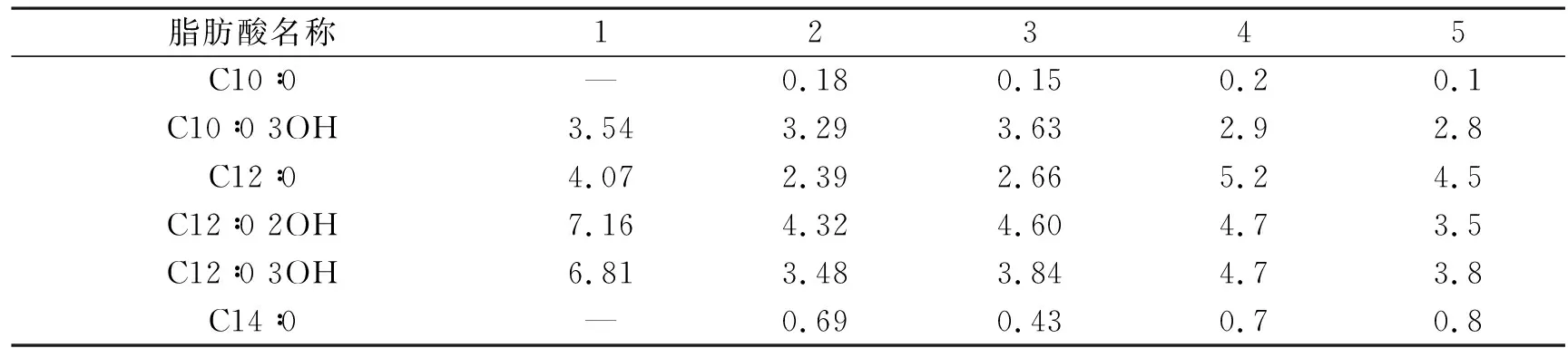
表4 菌株Y39-6与其近缘模式假单胞菌脂肪酸种类和含量的差异
3 结 论
通过对菌株Y39-6的基因组和生理生化特征进行分析,得到以下结论:
(1) 菌株Y39-6全基因组序列长度为7 021 566 bp,G+C含量为60.75%;COG注释的基因一共有5 378个,在氨基酸运输和代谢方面的基因数量最多,为497个;GO注释的基因一共有4 533个,在膜组成方面的基因数量最多,占GO注释基因总数的18.48%,以上基因组特征可能是菌株Y39-6在蛋白质合成、适应低温方面具有独特优势的主要原因。
(2) 在菌株Y39-6氮代谢基因中,norV、norR、norD、norQ、norC、norB、napC、napB、napA、napD、napE、nosR、nosZ、nosD、nosL、narI、narJ、narH、narK、narL和nirS参与异养反硝化过程,napC、napB、napA、napD、napE、nirD和nirB参与自养脱氮过程。
(3) 菌株Y39-6与PseudomonasveroniiCIP 104663T的基因组特征相似,G+C含量接近,共有基因数量最多,为368个,结合生理生化特征,证实菌株Y39-6属于Pseudomonasveronii菌种。
(4) 菌株Y39-6有15个特有基因,主要集中在跨膜运输、DNA介导的转换、硫化合物代谢、肽酶活力、抗生素生物合成、脂肪酸分解和烷基磺酸盐单加氧酶方面。
(5) 菌株Y39-6的细胞膜脂肪酸有C10∶0 3OH、C12∶0、C12∶0 2OH、C12∶0 3OH、C16∶0、C17∶0 cyclo、C18∶1 ω7c和C19∶0 cyclo ω8c 8种,主要脂肪酸为C16∶0和C17∶0 cyclo,含量分别为22.05%和21.25%,与Pseudomonasveronii菌种的其他菌株相比,存在明显的特异性。
