基于F2 和RIL 群体鉴定棉花抗黄萎病主效QTL
2023-07-06赵云雷王文菊陈伟王海红朱喜霞杨继华鲁宁宁赵佩桑晓慧崔艳利敦磊王红梅
赵云雷,王文菊,陈伟,王海红,朱喜霞,杨继华,鲁宁宁,赵佩,桑晓慧,崔艳利,敦磊,王红梅*
(1.中国农业科学院棉花研究所/ 棉花生物学国家重点实验室/ 棉花生物育种及产业技术国家工程研究中心,河南 安阳455000;2.安阳市殷都区农业农村局,河南 安阳 455000;3.山东众力棉业科技有限公司,山东 东营 257500)
棉花是世界上最重要的经济作物之一。棉花黄萎病是由大丽轮枝菌 (Verticillium dahliaeKleb.)引起的1 种真菌性病害,被称为“棉花的癌症”[1-2]。世界上几乎所有的棉花主产区都有发生,严重影响棉花的产量和纤维品质,其中籽棉产量损失可达7.9%[3]。实践证明,培育和推广抗病品种是控制棉花黄萎病危害最有效的途径。由于棉花的抗黄萎病机制相当复杂,常规育种提高抗性成效较小,而分子标记辅助选择育种能消除环境对选择效率的影响,加速育种进程。然而,当前鉴定到的、可以应用于育种实践的稳定性主效数量性状位点(quantitative trait locus,QTL)缺乏[3],导致分子标记辅助选择难以在育种中有效应用;另一方面,当前通过反向遗传学手段已克隆了大量的棉花抗黄萎病相关基因,但是这些基因并未在棉花抗病育种中成功应用。因此,发掘棉花抗黄萎病主效QTL,克隆关键抗病基因,对分子标记辅助选择育种和黄萎病抗性遗传改良具有重要的指导意义。
在棉花黄萎病抗性的遗传研究方面,国内外不同研究者所获得的研究结果不尽相同:一些研究中棉花黄萎病抗性是由显性单基因控制的质量性状[4],而另一些研究中棉花抗黄萎病性状是由2 个或2 个以上的显性基因控制的数量性状[5]。研究表明,在海岛棉(Gossypium barbadense)和少数陆地棉(G.hirsutum)中黄萎病抗性是质量性状遗传[3],然而利用传统的孟德尔分离定律分析却始终没有鉴定到抗黄萎病主效基因,并且截至目前在棉花中也没有鉴定到与毒性基因相对应的抗病基因[6]。因此,目前通常将棉花抗黄萎病性状看作是数量性状,采用传统的数量遗传学和基因组学研究方法开展棉花黄萎病抗性的遗传机制研究[3]。
研究者通过利用不同的分离群体已检测到400 多个棉花抗黄萎病QTL,这些QTL 在四倍体陆地棉基因组的26 条染色体上均有分布[6]。Palanga 等[7]利用陆地棉种内重组自交系(recombinant inbred line,RIL)群体在5 个环境条件下检测到119 个抗黄萎病QTL;Shi 等[8]利用海岛棉与陆地棉种间回交(backcross lines, BC)群体在人工病圃与大田病圃中进行鉴定,共检测到48 个抗黄萎病QTL;Zhang 等[6]利用11 个陆地棉亲本相互杂交配制的多亲本高级世代互交系(multiparent advanced generation inter-cross,MAGIC)群体,在温室鉴定条件下检测到25 个抗黄萎病QTL;Rashid 等[9]利用海岛棉与陆地棉种间染色体片段替换系 (chromosome segment substitution lines,CSSLs)群体在大田病圃鉴定下检测到40个抗黄萎病QTL;Abdelraheem 等[10]利用376 份美国陆地棉种质在温室鉴定的表型数据和Cotton-SNP63K 芯片杂交基因型数据,通过关联分析检测到15 个抗黄萎病QTL;此外,Zhang 等[11]利用401份陆地棉种质在温室鉴定的表型数据和深度重测序数据,通过关联分析检测到382 个与黄萎病抗性显著关联的单核苷酸多态性(singlenucleotidepolymorphism,SNP)位点。由于不同的作图群体在遗传背景、基因重组频率等方面存在差异,导致不同研究中QTL 的定位效率存在一定差异[12]。
通过采用转录组测序、蛋白组分析、代谢组分析等反向遗传学方法,已克隆了大量的抗黄萎病相关基因,其中既包括LysM、GhPR5等参与病原相关分子模式触发的免疫 (负责水平抗病性)的病菌激发子互作蛋白基因[13-14],又包括Gbvdr6、GhDSC1等参与效应子触发的免疫(专一的抗病蛋白识别病原菌特异的效应蛋白,负责垂直抗病性)的抗病基因[15-16]。其中,类受体蛋白(receptorlike protein, RLP)和类受体激酶(receptor-like ki nase,RLK)属于位于细胞表面的模式识别受体(pattern recognition receptor,PRR),两者均含有富含亮氨酸的重复(leucine rich repeat,LRR)结构域。研究表明,棉花RLK 蛋白GhWAK7A 能够参与识别真菌细胞壁中的几丁质成分,从而参与调控棉花对黄萎病病原菌和枯萎病病原菌的抗性反应[17],棉花RLP 基因Gbvdr6通过调节茉莉酸甲酯(methyl jasmonate,MeJA)/ 乙烯(ethylene,ET)信号通路和水杨酸(salicylic acid, SA)信号通路参与对黄萎病菌的抗性反应[15]。利用已经克隆的基因,获得了一些抗病性得到提高的转基因棉花植株,但是这些转基因植株并未在棉花抗病育种中成功应用,而且对于上述基因参与棉花抗黄萎病反应的分子机理还不清楚,有必要进一步开展棉花抗黄萎病基因的挖掘及鉴定工作。
本研究构建了中植棉2 号(抗)×冀棉11 号(感)的F2群体和RIL 群体,于2017 年和2019年对F2:3家系、2020 年和2021 年对RIL 群体进行了黄萎病抗性鉴定,并进行QTL 定位,以期获得能够在多个环境条件下被重复检测到的抗黄萎病主效QTL。对主效QTL 的定位区段进行基因注释和基因相对表达量分析,发掘棉花抗黄萎病相关候选基因,为后续抗病基因的图位克隆及功能解析奠定基础,并对棉花抗黄萎病分子标记辅助选择育种提供材料基础。
1 材料与方法
1.1 群体构建
2014 年以国家棉花品种区域试验中的抗黄萎病对照品种中植棉2 号和感黄萎病对照品种冀棉11 号为亲本进行杂交;2015 年经南繁加代,构建了由232 个单株组成的F2分离群体;进一步自交获得232 个F2:3家系。同时,利用单籽传法对F2分离群体进行连续自交,2020 年构建了由215个F2:6家系组成的RIL 群体。
1.2 材料种植及抗病性鉴定
将232 个F2:3家系及其双亲的种子分别于2017 年、2019 年种植于新疆温宿县青年农场棉花黄萎病重病地。该重病地连续多年种植陆地棉感病品种,每年通过秸秆还田将严重发病的棉花植株粉碎后与土壤进行混合,通过多年的田间测试,在该重病地种植的多数陆地棉品种具有黄萎病发病程度重、发病比较均匀的特点,因此该地可以作为抗病性鉴定的自然病圃。试验采用完全随机区组设计,每个小区2 行,行长3 m,行距45 cm,株距15 cm,3 次重复。以亲本中植棉2 号和冀棉11 号分别作为抗病对照和感病对照,每20 行连续种植2 行中植棉2 号和2 行冀棉11号。在2017 年7 月、8 月和2019 年8 月对群体进行黄萎病抗性鉴定。
将215 个F2:6家系及其双亲的种子分别于2020 年、2021 年种植于新疆温宿县青年农场棉花黄萎病重病地,种植模式与上述F2:3家系的相同。分别在2020 年7 月、8 月和2021 年7 月、8月进行黄萎病抗性鉴定。
采用国家棉花品种区域试验中黄萎病抗性鉴定方法[18],自7 月上中旬开始检测感病对照品种(冀棉11 号)的发病情况,待各个重复中冀棉11 号的平均病情指数达到40 时进行调查。首先采用5 级分级标准调查群体各个植株的病级[18];根据病级调查结果,参照如下公式计算病情指数。
病情指数=[∑(相应病级×每级的病株数)]/(调查总株数×4)×100。
1.3 群体基因型鉴定
采用十六烷基三甲基溴化铵法(cetyltrimethylammonium bromide,CTAB)法[19]提取亲本、F2群体和RIL 群体家系部分单株的基因组DNA,使用Nanodrop 2000 超微量紫外分光光度计和1%(质量分数,下同)琼脂糖凝胶电泳检测DNA 的浓度和质量。利用本实验室前期根据CottonGen网站(https://www.cottongen.org/)的序列信息合成的10 719 对简单重复序列(simple sequence repeat, SSR)引物 (包括SWU 系列8 208 对、CCRI 系列792 对、NAU 系列478 对、HAU 系列457 对、GH 系 列253 对、CGR 系 列107 对、MUSB 系列85 对、BNL 系列67 对、MUSS 系列62 对、DPL 系列60 对、TMB 系列57 对、JESPR系列40 对、CIR 系列39 对、MGHES 系列8 对、STV 系列6 对),基于亲本中植棉2 号和冀棉11号的基因组DNA 进行多态性标记的筛选;利用筛选到的扩增条带清晰的多态性引物对群体进行基因型检测。聚合酶链式反应(polymerase chain reaction,PCR)体系为10 μL,包含5 μL 的2×Taq Master Mix for PAGE(南京诺唯赞生物科技股份有限公司),模板DNA 50 ng,10 μmol·L-1的上/ 下游引物各0.5 μL,ddH2O 补足至10 μL。PCR 反应程序为94 ℃预变性5 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min,30 个循环;72 ℃延伸10 min;4 ℃保存。用10%聚丙烯酰胺凝胶电泳和银染技术对PCR 产物进行检测[20]。
1.4 遗传图谱构建及QTL 定位
用QTL IciMapping 4.1 软件的MAP 功能构建遗传连锁图谱,用MapChart 2.2 软件绘制遗传连锁图谱及物理图谱。利用QTL IciMapping 4.1软件的BIP 功能,选择完备复合区间作图法(inclusive composite interval mapping, ICIM)的加性效应模型ICIM-ADD,使用1 000 次排列检验分析表型数据,并设置显著性P=0.05,在对数优势比 (logarithm of the odd score,LOD)≥2.5 时,分别对各个环境条件下的群体的黄萎病抗性进行QTL 定位。
1.5 抗性QTL 聚合效应分析
本研究将置信区间相同或者互相重叠的QTL 确定为相同的QTL,能够在F2:3家系或者RIL 群体的多个环境下被检测到的QTL 确定为稳定的QTL,将表型变异解释率(phenotypic variation explained,PVE)在10%以上的稳定QTL 确定为主效QTL。
以鉴定到的稳定QTL 位点的2 个边界SSR标记分别对RIL 群体各个家系进行基因型分析,统计不同基因型RIL 家系的病情指数,并进行差异显著性分析。
1.6 主效QTL 区间内的基因注释及基因表达量分析
选择多环境稳定的主效QTL,根据遗传图谱与物理图谱的共线性分析结果,确定主效QTL的物理区间;针对参考基因组(G.hirsutum_genome_HAU_v1.0)中主效QTL 的物理区间内的序列信息,利用CottonGen(https://www.cottongen.org/)、UniProt(https://www.uniprot.org/)网 站进行基因注释。
将中植棉2 号和冀棉11 号的种子种植在人工气候培养箱中(湿度60%、25 ℃光照16 h/20 ℃黑暗8 h),当幼苗长至第一片真叶平展时(播种后大约21 d),采用无底纸钵定量蘸菌液法接种中等致病力的大丽轮枝菌Vd080(病原菌孢子浓度为1×107mL-1)。
在接菌后0 h、6 h、12 h、24 h 和48 h 取棉花叶片,使用天根生化科技(北京)有限公司的多糖多酚植物总RNA 提取试剂盒提取RNA;使用HiScript II Q Select RT SuperMix for qPCR(+gDNA wiper)反转录试剂盒(南京诺唯赞生物科技股份有限公司)获得第一链cDNA。根据注释基因的转录本序列,使用Primer Premier 5 软件设计特异引物序列(表1)。使用宝日医生物技术(北京)有限公司的SYBR Premix Ex TaqTMII 实时荧光定量试剂盒进行实时荧光定量聚合酶链式 反 应(quantitative real-time PCR,qRT-PCR),以GhACT14(GeneBank ID:AY305733)作为内参基因,采用2-ΔΔCt法计算目的基因的相对表达量。

表1 qRT-PCR 引物序列Table 1 Primer sequences for qRT-PCR
1.7 数据处理与分析
用Microsoft Excel 2010 软件整理调查数据,计算病情指数。相关性分析采用IBM SPSS Statistics 24 软件。使用Fisher 的最小显著差数法(least significant difference, LSD)进行多重比较。
2 结果与分析
2.1 F2:3 和RIL 群体黄萎病抗性分析
分别对F2:3家系和RIL 群体的病情指数进行调查,分析上述2 个群体在不同环境条件下的黄萎病抗性表现。结果表明,2 个群体在不同环境中的病情指数表现出广泛的差异。其中,F2:3家系在2017 年8 月的黄萎病发病程度更严重,其平均病情指数为49.51,病情指数最小值为27.31、最大值为75.52,均高于该家系在2017 年7 月和2019年8 月的病情指数。RIL 群体在2020 年8 月的黄萎病发病程度更为严重,其平均病情指数为48.06,病情指数最小值为25.34、最大值为62.94。所有环境中病情指数的峰度和偏度的绝对值均小于1,说明不同环境条件下的病情指数均符合正态分布,表明陆地棉抗黄萎病性状是以数量性状方式遗传的,可以进行QTL 定位(表2)。相关分析结果表明,F2:3家系及RIL 群体在不同环境条件下的病情指数均呈极显著正相关关系(表3)。
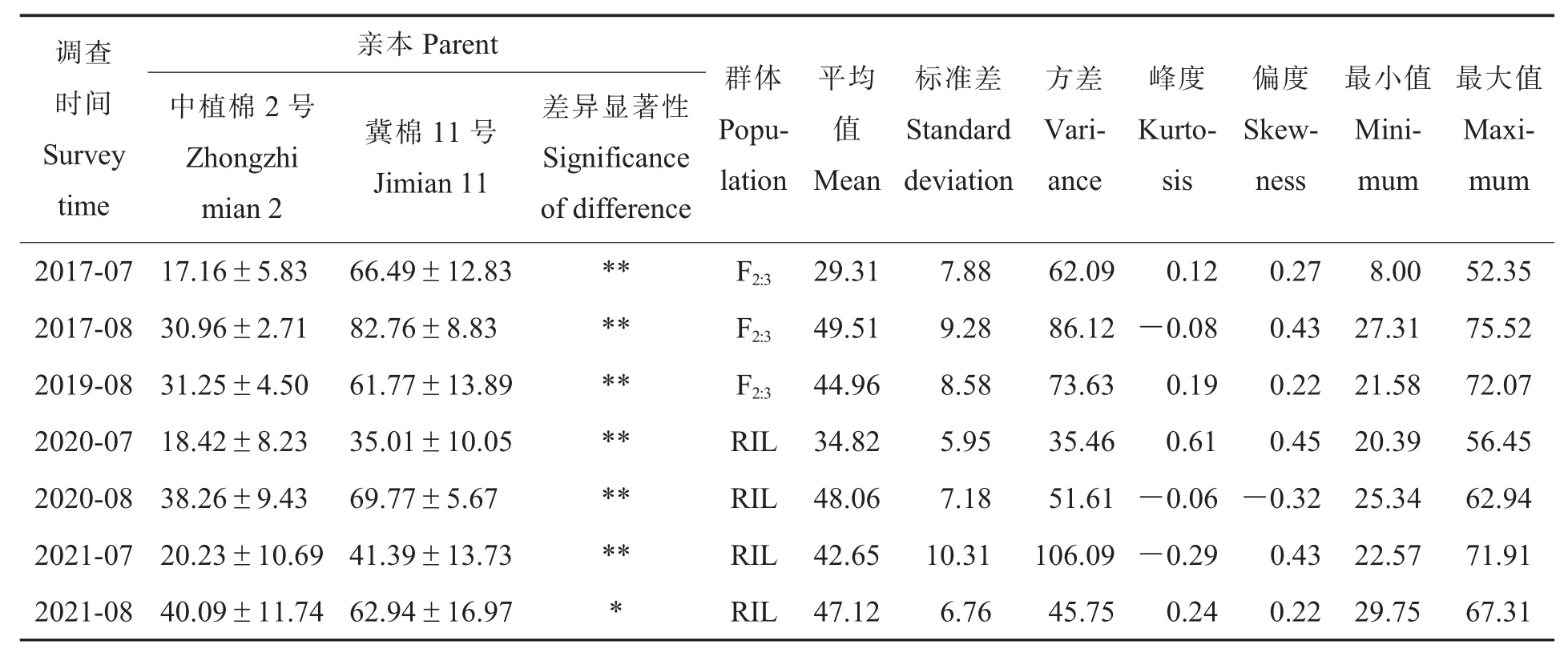
表2 F2:3 和RIL 群体及其亲本在不同环境条件下的病情指数统计分析Table 2 Descriptive statistics of disease index for the F2:3 population, RIL population and their parents identified in different environments

表3 F2:3 和RIL 群体在不同环境条件下的病情指数的相关性Table 3 Correlation analysis of disease index of the F2:3 and RIL populations identified in different environments
2.2 遗传连锁图谱的构建
利用覆盖全基因组的10 719 对SSR 引物以及亲本中植棉2 号与冀棉11 号的基因组DNA进行多态性分子标记筛选,获得133 对扩增条带清晰的多态性引物,利用这133 对多态性SSR 引物分别构建了F2:3群体及RIL 群体的遗传连锁图谱。其中,F2:3群体连锁图谱含有17 个连锁群,包含114 个多态性位点,全长共计1 873.45 cM(centiMorgan,厘摩),单个连锁群的距离为0.67~938.98 cM,单个连锁群的平均距离为110.20 cM,单个连锁群包含2~34 个SSR 标记(图1)。RIL群体连锁图谱含有18 个连锁群,包括71 个多态性位点,全长共计415.92 cM,单个连锁群的距离为0.00~56.67 cM,平均距离为23.11 cM,单个连锁群包含2~10 个SSR 标记(图2)。

图1 F2:3 群体遗传连锁图谱构建及QTL 定位Fig.1 Genetic linkage map construction and QTL mapping for the F2:3 population
2.3 棉花抗黄萎病性状QTL 定位
基于F2群体及RIL 群体的遗传连锁图谱,采用IciMapping 4.1 软件分别对F2:3家系及RIL群体进行QTL 定位。结果表明,在2017 年、2019年、2020 年和2021 年的环境条件下共检测到7个与棉花抗黄萎病性状相关的QTL (图1~2),最大定位区间为10.57 cM,最小定位区间为0.48 cM,并且RIL 群体检测到的QTL 定位区间均小于F2:3家系检测到的QTL 定位区间(表4~5)。

表4 F2:3 群体检测到的抗黄萎病QTLTable 4 QTL for Verticillium wilt resistance mapped by the F2:3 population
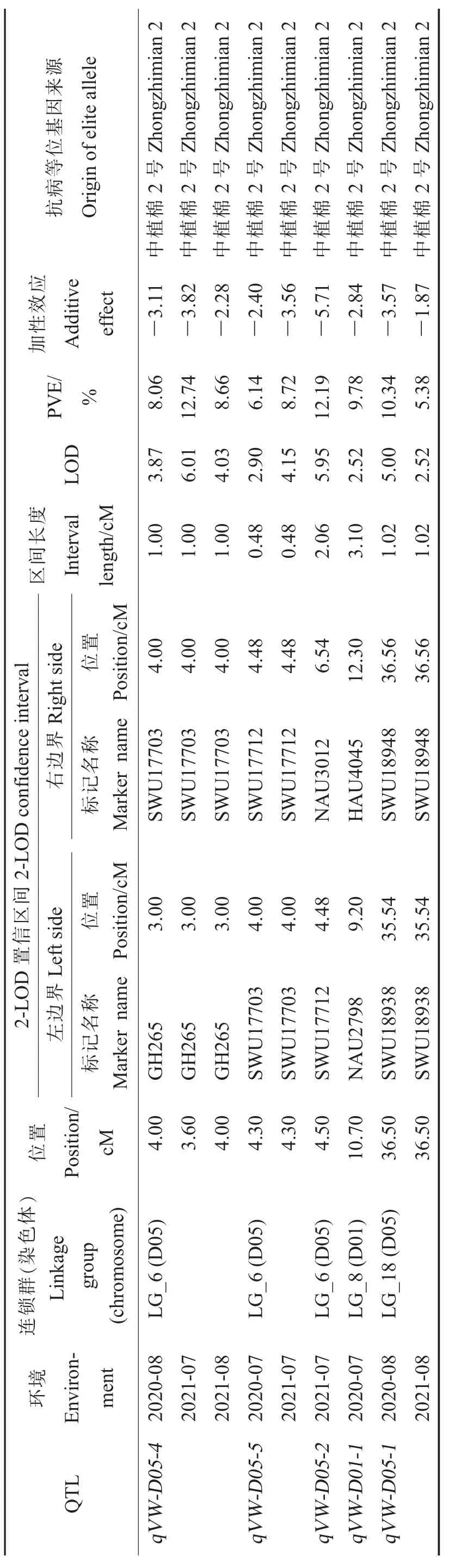
表5 RIL 群体检测到的抗黄萎病QTLTable 5 QTL of Verticillium wilt resistance mapped by the RIL population
本研究共检测到4 个多环境稳定的QTL:qVW-D05-1、qVW-D05-2、qVW-D05-4和qVWD05-5(表4~5)。其中,qVW-D05-1在2017 年、2019 年、2020 年和2021 年的环境条件下共被检测到5 次,其LOD 值范围为2.52~8.78,PVE 变化范围为5.38%~15.74%,该QTL 在3 个环境(2017 年8 月、2019 年8 月和2020 年8 月)中的PVE 均在10%以上,为抗黄萎病主效QTL;qVW-D05-2在2017 年、2019 年 和2021 年 的 环境条件下共被检测到3 次,其LOD 值范围为4.38~5.95,PVE 变化范围为7.42%~12.19%;qVW-D05-4在2020 年和2021 年环境条件下共被检测到3 次,其LOD 值范围为3.87~6.01,PVE 变化范围为8.06%~12.74%;qVW-D05-5在2020 年和2021 年环境条件下共被检测到2 次,其LOD 值范围为2.90~4.15,PVE 变化范围为6.14%~8.72%。上述4 个多环境稳定QTL 的加性效应值均为负值,说明其抗病等位基因来自母本中植棉2 号。
通过比对陆地棉参考基因组(G.hirsutum_genome_HAU_v1.0),上述4 个稳定QTL 均位于D05 染色体上,qVW-D05-2、qVW-D05-4和qVWD05-5的物理区间为2 293 776~3 205 058 bp,qVW-D05-1位于62 407 897~62 582 344 bp 区间。说明上述2 个染色体区段可能与黄萎病抗性密切相关。
2.4 抗性QTL 聚合对棉花黄萎病抗性的影响
为进一步探讨上述4 个QTL 与棉花抗黄萎病性状的相关性,对RIL 群体各个家系进行基因型分析,统计各个RIL 家系中含有的抗性QTL的数量。方差分析表明,整体来看,随着抗性QTL数量的增加,平均病情指数逐渐降低。与不含上述4 个QTL 的材料相比,聚合4 个抗性QTL 的RIL 家系材料在各个环境中的平均病情指数均显著降低,最大可降低13.76(表6)。
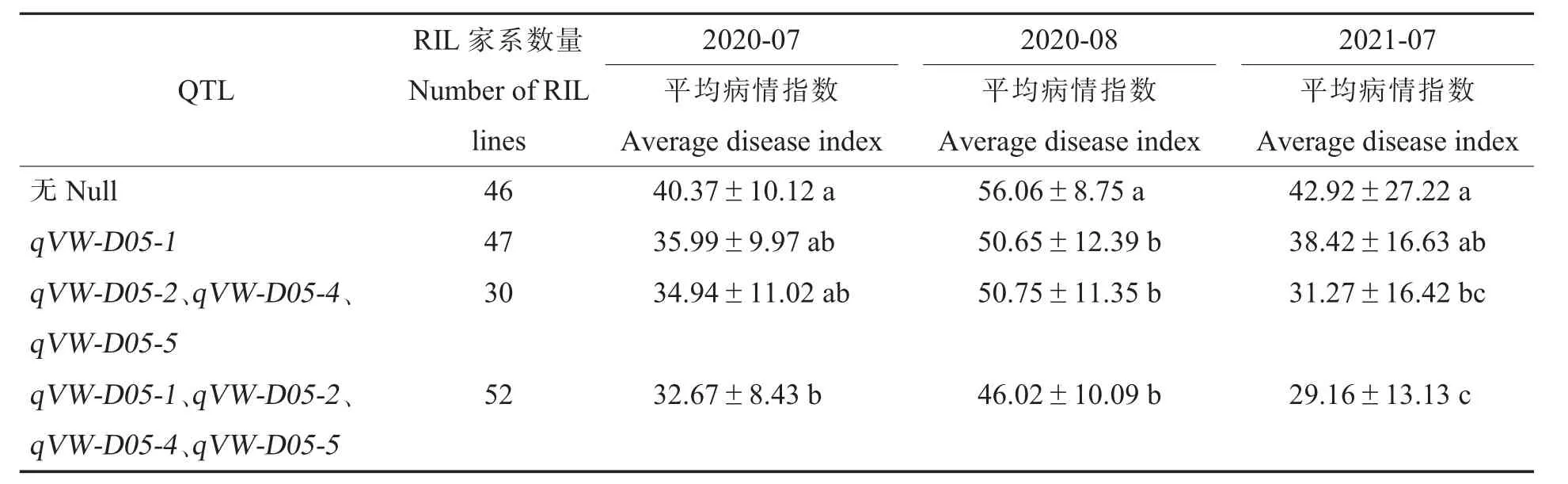
表6 抗性QTL 聚合对棉花黄萎病抗性的影响Table 6 Effects of polymerizing resistance QTL on Verticillium wilt resistance of cotton
2.5 qVW-D05-1 区间内的基因注释及候选抗性基因筛选
因主效QTLqVW-D05-1在2 个群体5 个环境条件下均被检测到,可靠性较高,并且该QTL在RIL 群体定位的置信区间仅为1.02 cM,对应于D05 染色体上174.447 kb(62 407 897 ~62 582 344 bp)的基因组区域,因此对该区间内的基因进行分析有可能鉴定到棉花抗黄萎病相关的关键候选基因。基因注释表明,qVW-D05-1的置信区间内共含有18 个基因(表7),其中1 个基因(Ghir_D05G039100)没有功能注释信息,可能为尚未报道的新基因。其余17 个基因中,8 个基因可能具有抗病功能,包括1 个丝氨酸羧肽酶类蛋白基因 (Ghir_D05G038980)、2 个 丝 氨 酸 /苏氨酸蛋白激酶基因(Ghir_D05G039000和Ghir_D05G039060)、1 个磷脂酰肌醇转运蛋白基因 (Ghir_D05G038990)、1 个RPM1 互作蛋白基因 (Ghir_D05G039030)、1 个 类 驱 动 蛋 白 基 因(Ghir_D05G039110)、1 个尿嘧啶激酶类蛋白基因(Ghir_D05G039120)和1 个NDR1/HIN1 类蛋白基因(Ghir_D05G039130)。另外,3 个连续的环烯醚萜苷葡萄糖基转移酶基因(Ghir_D05G039070、Ghir_D05G039080和Ghir_D05G039090)可能具有抗菌功能。

表7 主效QTL qVW-D05-1 区间内基因注释Table 7 Gene annotation of the major QTL qVW-D05-1
根据前期亲本中植棉2 号和冀棉11 号重测序(测序深度30×)的结果[36],对qVW-D05-1置信区间内的18 个注释基因进行SNP 分析。结果表明,10 个基因的外显子区含有SNP 变异,其中8 个基因(Ghir_D05G038990、Ghir_D05G039000、Ghir_D05G039050、Ghir_D05G039060、Ghir_D05G039080、Ghir_D05G039100、Ghir_D05G039110和Ghir_D05G039130)的外显子区发生了非同义突变(表8)。

表8 18 个注释基因在中植棉2 号和冀棉11 号间的SNP 变异分析Table 8 SNP variation between Zhongzhimian 2 and Jimian 11 for 18 annotated genes
为进一步筛选候选抗病基因,使用qRT-PCR对qVW-D05-1置信区间内的18 个注释基因在抗病和感病品种接种黄萎病菌后不同时间的表达量进行测定(图3)。结果显示,18 个基因主要分为3 类。第1 类为在黄萎病菌诱导后抗病品种和感病品种在部分时间点均能表现出优势表达的基因,包括Ghir_D05G038960、Ghir_D05G038980和Ghir_D05G039030共3 个基因;第2 类为黄萎病菌诱导后的部分时间点在感病品种中的表达量显著高于抗病品种的基因,包括Ghir_D05G039010、Ghir_D05G039050、Ghir_D05G039060和Ghir_D05G039110共4 个基因,这类基因被认为是黄萎病菌诱导后在感病品种中优势表达的基因;第3 类为黄萎病菌诱导后多个时间点在抗病品种中的表达量显著高于感病品种的基因,包括剩余的11 个基因,这类基因是黄萎病菌诱导后在抗病品种中优势表达的基因。

图3 主效QTL qVW-D05-1 区间内注释基因的表达模式分析Fig.3 Expression pattern of annotated genes in the major QTL qVW-D05-1
根据表达模式、SNP 变异和基因注释信息,初步推测Ghir_ D05G038990、Ghir_D05G039060、Ghir_D05G039100、Ghir_D05G039110和Ghir_D05G039130可能为控制棉花黄萎病抗性的候选基因。
3 讨论
3.1 RIL 群体比F2:3 群体具有更高的QTL 定位精度
本研究利用筛选获得的133 对多态性SSR引物同时对F2:3群体和RIL 群体进行遗传连锁图谱构建。由于RIL 群体是由F2:3家系的个体连续多代自交而得到的纯合家系,经历过数次的减数分裂及染色体的自由组合,极大地增加了连锁基因之间重新组合的概率,因此RIL 群可以有更多机会检测到连锁标记间的重组。本研究结果表明,F2群体的遗传连锁图谱全长1 873.45 cM,含有114 个多态性位点,位点间平均距离16.43 cM;获得的RIL 群体遗传连锁图谱全长415.92 cM,含有71 个多态性位点,位点间平均距离5.86 cM。此外,本研究中基于F2群体的连锁群出现了比对到多条染色体上的情况,推测可能与本研究的SSR 标记偏少、部分标记间的连锁距离过大有关。尽管RIL 群体检测到的多态性位点数目明显少于F2:3群体,但是RIL 群体遗传连锁图谱上位点间的平均距离远小于F2:3群体,说明RIL 群体的重组交换概率远高于F2:3群体。QTL 定位结果表明,基于F2:3家系检测到的QTL 的置信区间长度在5.57~10.57 cM;而基于RIL 群体检测到的QTL 的置信区间长度在0.48~3.10 cM,说明利用RIL 群体大大提高了QTL 定位精度。进一步通过共线性分析表明,基于F2:3家系在D05 染色体2 293 776~3 205 058 bp 基因组区域只检测到1 个多环境稳定的QTL(qVW-D05-2),而基于RIL群体在上述基因组区域检测到3 个多环境稳定的QTL(qVW-D05-2、qVW-D05-4和qVW-D05-5),说明利用RIL 群体不仅大大提高了QTL 定位精度,同时还可以提高QTL 的发掘效率。
3.2 棉花D05 染色体上存在抗黄萎病主效QTL
目前,尽管在棉花中检测到的抗黄萎病QTL已经超过了400 个,但是对于表型解析率较高(10%以上)、多环境稳定检测(至少在2 个以上环境检测到)的主效QTL 的发掘效率极低。例如,利用陆地棉种内重组自交系群体在5 个环境条件下检测到119 个抗黄萎病QTL,其中仅有7 个QTL 在至少2 个环境中被检测到[7];利用11 个陆地棉亲本相互杂交配制的MAGIC 群体在2 个环境条件下检测到25 个抗黄萎病QTL,其中仅有3 个QTL 可以重复检测到[6]。本研究利用F2:3家系和RIL 群体在多个环境条件下共检测到7 个抗黄萎病QTL,其中4 个QTL 可以重复检测到,共线性分析表明上述4 个多环境稳定的QTL 集中分布于D05 染色体上2 293 776~3 205 058 bp和62 407 897~62 582 344 bp 区间内。通过与前人的QTL 定位结果进行比较,发现主效QLTqVW-D05-1的置信区间与前人定位的抗黄萎病QTLqDR85T1-c19的置信区间重叠,qDR85T1-c19的表型解释率为10.64%[37];qVW-D05-5的置信区间与前人定位的抗黄萎病QTLqVL-A5-2BC1S2592和qVW-C5-2的置信区间重叠,其表型解释率分别为11%和13.59%[8,38];此外,qVWD05-2的置信区间与前期通过全基因组关联分析(genome wide association study,GWAS)鉴定的与棉花抗黄萎病性状显著关联的位点(NAU3828)重叠[39]。因此,本研究鉴定的位于D05 染色体上2 293 776~3 205 058 bp 和62 407 897~62 582 344 bp 2 个基因组区域内的抗黄萎病QTL 的表型解释率大多在10%以上,说明上述2 个区域可能存在抗黄萎病主效QTL。
尽管目前已在棉花中鉴定到部分抗黄萎病主效QTL,但是其置信区间长度大多在5 cM 以上,分子标记与主效QTL 的连锁不紧密,难以直接应用于分子标记辅助选择育种。本研究鉴定到的4 个多环境稳定的QTLqVW-D05-1、qVWD05-2、qVW-D05-4和qVW-D05-5在RIL 群 体中的置信区间长度分别为1.02 cM、2.06 cM、1.00 cM 和0.48 cM,分子标记与QTL 的遗传距离均在0.5 cM 以下,因此实现了分子标记与QTL 的紧密连锁。进一步分析表明,与不含有抗性QTL 的RIL 材料相比,含有上述抗性QTL 的材料的病情指数均降低,聚合4 个抗性QTL 的RIL 家系材料在各个环境中的平均病情指数均显著降低。因此,本研究所获得的主效QTL 可以有效应用于棉花抗黄萎病性状的分子标记辅助选择育种。
3.3 主效QTL 区间内候选抗病基因的挖掘
主效QTLqVW-D05-1在F2:3家系和RIL 群体的多个环境条件下均被检测到,可靠性较高。qVW-D05-1区间包含18 个基因,其中8 个基因可能具有抗病功能,结合qRT-PCR、SNP 变异分析和基因注释,最终将5 个基因确定为可能控制棉花黄萎病抗性的候选基因。在这些候选基因中,Ghir_D05G038990与磷脂酰肌醇/ 磷脂酰胆碱转运蛋白编码基因高度同源,该类转运蛋白在生物体内大量存在并高度保守,参与对脂质信号及植物发育相关信号的调控[24];在烟草中,该类转运蛋白在响应青枯病菌侵染、模式识别受体和效应分子处理后显著上调表达,说明其参与了植物的免疫反应[40]。Ghir_D05G039060与LRR 类受体丝氨酸/ 苏氨酸蛋白激酶编码基因RGI3高度同源,RGI3 蛋白属于肽激素RGF1 的受体蛋白,RGI3 蛋白可以与拟南芥模式识别受体FLS2 形成复合体,同时RGI3 可识别内生信号肽GLV2,从而增加FLS2 的数量进而控制免疫反应[41];在棉花中,LRR 类受体蛋白激酶GhRLK1 参与纤维次生壁形成的信号传递过程[42],RLK基因Gh-WAK7A正调控棉花对枯黄萎病病原菌侵染的反应[17]。Ghir_D05G039110与类驱动蛋白编码基因高度同源,在水稻中,类驱动蛋白KLP 通过与株型相关蛋白LPA1 互作从而增强水稻对纹枯病的抗性[33]。另外,驱动蛋白VdKin2 通过调控液泡形成,影响大丽轮枝菌菌丝生长和侵染结构的形成[43]。Ghir_D05G039130与类NDR1 蛋白编码基因高度同源,研究表明NDR1 含有卷曲螺旋结构域,是抗病蛋白激活所必需的,2 个拟南芥类NDR1 蛋白基因NHL25和NHL3在抗病反应中发挥潜在作用[35],而目前关于类NDR1 蛋白在棉花中的相关研究还未见报道。
4 结论
利用中植棉2 号(抗)×冀棉11 号(感)的F2:3群体和RIL 群体进行遗传连锁图谱构建和QTL定位,共检测到7 个抗黄萎病QTL,其中4 个QTL(qVW-D05-1、qVW-D05-2、qVW-D05-4和qVWD05-5)可以重复检测到。这4 个QTL 集中分布于D05 染 色 体 上2 293 776~3 205 058 bp 和62 407 897~62 582 344 bp 2 个基因组区域。上述4 个QTL 优势等位基因型聚合可以显著降低棉花的病情指数。通过对主效QTLqVW-D05-1区间内基因进行注释、表达模式分析和SNP 变异分析,鉴定到5 个可能与棉花抗黄萎病相关的候选基因Ghir_D05G038990、Ghir_D05G039060、Ghir_D05G039100、Ghir_D05G039110 和 Ghir_D05G039130。
