Pladienolide B对牛胚胎干细胞多能性相关基因表达及细胞活力的影响
2023-06-27赵芳丁强夏淑雯高运东兰国成林志平王慧利仲跻峰
赵芳,丁强,夏淑雯,高运东,兰国成,林志平,王慧利,仲跻峰
Pladienolide B对牛胚胎干细胞多能性相关基因表达及细胞活力的影响
赵芳1,丁强1,夏淑雯1,高运东2,兰国成3,林志平4,王慧利,仲跻峰
1江苏省农业科学院畜牧研究所/江苏省畜禽精准育种工程研究中心/农业农村部种养结合重点实验室,南京 210014;2山东奥克斯畜牧种业有限公司,济南 250100;3香港大学李嘉诚医学院,香港 999077;4江苏优源奶业产业研究院有限公司,南京 211100
【背景】牛胚胎干细胞(bovine embryonic stem cells,BESCs)因具备较高的多能性,在牛品种保存、品种选育及研究家畜胚胎发育调控机制方面具有重要的应用价值。然而,BESCs多能性维持及分化调控的研究相对缺乏,很多机制仍不清楚。【目的】探究不同浓度Pladienolide B(PlaB)对BESCs多能标记基因、全能标记基因、胚胎细胞谱系基因表达及细胞活力的影响,为提高BESCs多能性提供参考和理论依据。【方法】利用免疫荧光检测牛BESCs多能标记基因的表达,利用实时荧光定量PCR(real-time fluorescence quantitative PCR,RT-qPCR)检测不同浓度PlaB对BESCs剪接体、全能标记基因及胚胎细胞谱系基因表达的影响,利用RT-qPCR和Western Blot分别检测不同浓度PlaB对BESCs多能标记基因mRNA和蛋白表达的影响,利用CCK8和EDU染色检测不同浓度PlaB对BESCs增殖的影响。【结果】RT-qPCR 结果显示,PlaB浓度为0.5—1.5 nmol·L-1时显著下调EPSCM-BESCs中和的mRNA表达水平;PlaB浓度为1.5 nmol·L-1时,CTFR-BESCs中和的mRNA表达下降;PlaB浓度范围为0.5—1.5 nmol·L-1时,CTFR-BESCs和EPSCM-BESCs的和的mRNA表达水平呈剂量依赖性增加。此外,PlaB显著下调CTFR-BESCs中的mRNA表达。PlaB浓度为0.5—1.5 nmol·L-1时,EPSCM-BESCs和CTFR-BESCs中剪接体的mRNA表达显著下调。PlaB浓度为0.5—1.5 nmol·L-1时,显著下调CTFR-BESCs的mRNA表达水平;PlaB浓度为1—1.5 nmol·L-1时能显著下调BEPSCM-BESCs的mRNA表达水平。浓度为0.5—1.5 nmol·L-1范围内,PlaB均以剂量依赖性上调CTFR-BESCs和EPSCM-BESCs中多能标记基因、及的mRNA表达水平和蛋白水平。在PlaB浓度为0.5—1.5 nmol·L-1范围内,PlaB以剂量依赖性上调CTFR-BESCs和EPSCM-BESCs中全能标记基因、及的mRNA表达水平,而下调和的mRNA表达水平。CTFR-BESCs中添加PlaB均上调胚胎细胞谱系基因的表达,而EPSCM-BESCs中添加PlaB显著降低、、等胚胎细胞谱系基因的表达,但是对于没有显著影响。随着PlaB剂量增加和处理时间延长,CTFR-BESCs和EPSCM-BESCs细胞活力均呈下降趋势,但CTFR-BESCs比EPSCM-BESCs更敏感。【结论】PlaB显著上调CTFR-BESCs和EPSCM-BESCs中多能标记基因和部分全能标记基因的表达;降低EPSCM-BESCs中胚胎细胞谱系基因的表达和细胞活力。PlaB对两种BESCs发挥作用的浓度及对基因表达的影响并不完全一致。由于PlaB降低BESCs的细胞活力,仍需要进一步研究以优化培养体系。
牛胚胎干细胞;Pladienolide B;剪接体;多能性相关基因;细胞活力
0 引言
【研究意义】干细胞是一类具有自我更新和多向分化潜能的细胞群体,可在体外诱导条件下分化成为多种类型的细胞。在畜禽上,将多能干细胞与基因编辑、显微注射、核移植、胚胎移植等技术结合,可应用于畜禽育种、濒危动物保种、优良种质扩繁、疾病防治及生物反应器等方面,对畜禽生产具有重要的研究价值和应用意义[1]。提高干细胞的多能性有助于提高早期胚胎发育的能力,并有助于更有效地生产用于研究和器官移植的嵌合动物。【前人研究进展】在真核细胞内,大多数蛋白质编码基因的表达需要由DNA先转录为前体RNA(precursor mRNAs,pre-mRNA),再经过重要的加工才能产生成熟的mRNA。其中一个基本环节是通过剪接体将非编码序列(即内含子)从pre-mRNA中去除,并连接两侧的外显子以形成有功能的mRNA[2-3]。剪接体是一种多巨量核糖核蛋白(RNP)复合物,由5个小的核RNA(small nuclear RNAs,snRNAs)组成,分别是U1、U2、U4、U5和U6,每一个snRNA都与特定的蛋白质相关联,产生相应的小核核糖核蛋白复合物(small nuclear ribonucleoprotein complexes,snRNP)[4]。snRNPs依次结合到pre-mRNA中的顺式作用元件,包括5′和3′剪接位点、分支序列和多嘧啶束,通过E、A、B、B*和C预剪接体复合物组装成功能性的剪接体[3]。在细胞中,剪接体通过选择性剪接增加单个基因的表达数量,提高蛋白表达的复杂性和多样性,并在胚胎发育及器官功能行使中发挥至关重要的作用。大量研究表明pre-mRNA的异常剪接是造成许多人类疾病发生和恶化的基础[5],亦是早期胚胎发生滞育的主要原因[6-7]。近年来,人们在牛干细胞方面开展了大量研究,先后建立了牛胚胎多能干细胞[8]和牛胚胎扩展多能干细胞[9]。此外,在牛乳腺干细胞[10]、间充质干细胞[11]等成体干细胞及诱导多能干细胞研究方面也取得了一定的进展[12]。然而,建立能发育成完整胚胎的牛全能性干细胞仍然是一项极具挑战的工作。越来越多的研究表明剪接体参与干细胞多能性维持及分化调控[13-14]。近期的研究发现,在小鼠ESCs培养中添加剪接体抑制剂Pladienolide B(PlaB)能驱动小鼠ESCs多能性向全能性转变[15]。PlaB是从培养的链霉菌工程菌株Mer-11107中分离得到的一种由12个大环内酯组成的复合物[16],与SF3B1结合以抑制mRNA的剪接活性。然而,在牛多能干细胞多能性维持及分化上,剪接体抑制剂的功能尚缺乏研究报道。PlaB在牛胚胎干细胞中是否存在相似的驱动机制尚不清楚。【本研究切入点】本研究参考已报道的两种牛胚胎干细胞培养体系CTFR[8]和EPSCM[9],建立了两种不同的牛胚胎干细胞系,通过在培养液中添加不同浓度的剪接体抑制剂PlaB,分析PlaB对CTFR-BESCs和EPSCM-BESCs细胞中剪接体、全能因子、多能因子表达的影响,以及比较PlaB对CTFR-BESCs和EPSCM-BESCs细胞活力的影响。【拟解决的关键问题】从分子水平上揭示剪接体抑制剂PlaB对牛胚胎干细胞多能标记基因、全能标记基因及胚胎细胞谱系基因表达的影响,以期为提高牛胚胎干细胞多能性,优化培养体系提供依据和参考。
1 材料与方法
本试验于2021年上半年在江苏省农业科学院畜牧研究所农业农村部种养结合重点实验室完成。
1.1 试剂
Anti-Nanog(14-5761-80,Ebioscience),Anti-Sox2(sc17320,SantaCruz),Anti-Oct4(sc5279,SantaCruz),mLIF(HY-P7084,MCE),丝裂霉素-C(HY-13316,MCE),PD0325901(S1036,Selleck),CHIR99021(S1263,Selleck),WH-4-023(S7565,Selleck),IWR-1(S7086,Selleck),FBS(SH30396,Hyclone),Matrigel(354277,CORNING),β-巯基乙醇(M 6250,Sigma),Vitamin C(49752-100G,Sigma),Activin A(338-AC,R&D)。高糖DMEM(11965-092),DMEM-F12(11330-032),KnockOut Serum Replacement(A3181502),GlutaMAX(35050-061),非必需氨基酸(11140-050),AF(S006100),Penicilin-Streptomycin(15140-122),TrypLE™ Select(12563-029)购自Thermo Fisher Scientific公司;mTeSR™1购自STEMCELL公司;BCIP/NBT碱性磷酸酯酶显色试剂盒(C3206)购自碧云天生物公司。
1.2 试验方法
1.2.1 牛扩展多能胚胎干细胞培养基配制 牛扩展多能胚胎干细胞的培养基(expanded potential stem cells medium,EPSCM)以mTeSR™1为基础培养基[9],具体配制如下:470 mL mTeSR™1,25 mL KnockOut Serum Replacement,5 mL 100×penicillin-streptomycin,0.1 mmol·L-12-mercaptoethanol(Gibco),1 μmol·L-1CHIR99021,0.3 μmol·L-1WH-4-023,5 μmol·L-1IWR-1,50 μg·mL-1Vitamin C,20.0 ng·mL-1Activin A。
1.2.2 牛Primed胚胎干细胞培养基配制 牛Primed胚胎干细胞培养基(custom TeSR1 base medium supplemented with FGF2 and IWR1,CTFR)以mTeSR1 Medium Without Select Factors为基础培养基[17],具体配制如下:470 mL mTeSR™1,25 mL KnockOut Serum Replacement,5 mL 100×penicillin-streptomycin,0.979 mmol·L-1GABA,0.984 μmol·L-1Pipecolic Acid,0.98 mmol·L-1LiCl,20 ng·mL-1FGF2,2.5 μmol·L-1IWR-1。
1.2.3 牛胚胎干细胞分离及建系 参考Bogliotti YS的方法进行牛胚胎干细胞分离及建系方法[8]。具体为,取扩展的牛囊胚(如图1所示),利用酸性台式液处理2 min,脱掉透明带。用CTFR细胞培养液将剩余囊胚细胞清洗干净,将囊胚细胞置于铺好饲养层的细胞培养板中,添加适量含10 μmol·L-1Y-27632的CTFR培养液于37 ℃、5% CO2培养箱中培养,24 h后观察细胞贴附情况,对于48 h内未贴附细胞团,利用30 g无菌针头按压细胞团,辅助其贴附。待囊胚贴附并生成Outgrowth后,继续培养4—7 d,利用TrypLE™ Select消化3—4 min,添加含10% KSR的DMEM F12终止消化,将消化后的细胞悬液以1 000×离心5 min,弃上清,加入含10μmol·L-1Y-27632的CTFR培养液接种至含MEF饲养层的培养皿中,24 h后培养液更换为CTFR继续培养4—6 d,待细胞克隆密度达80%汇合度时,进行1﹕5传代。
1.2.4 饲养层制备 将传代三代以内的原代小鼠胎儿成纤维细胞(mouse embryo fibroblast,MEF)以106/mL密度接种到细胞培养板上,添加适量的DMEM培养基进行培养,等细胞单层覆盖培养板时用浓度为10 µg·mL-1的丝裂霉素C处理3 h,用PBS清洗3遍,加入适量DMEM培养液,置于37℃、5% CO2细胞培养箱中备用。
1.2.5 CCK8细胞活力检测 将BESCs消化成细胞悬液,用40 μmol·L-1细胞过滤器过滤后进行细胞计数,然后将细胞以每孔1 000个铺于96孔板中,分别添加PlaB浓度为0.5、1及1.5 nmol·L-1的EPSCM培养液和CTFR培养液,每组设置4个重复孔,同时设置对照组(含细胞、培养基、CCK-8 溶液,不含药物)和空白组(含培养基、CCK-8 溶液,不含细胞、药物),分别处理24和72 h。检测前每孔加入10 µL CCK-8,37 ℃细胞培养箱中孵育2 h;用酶标仪测定波长在450 nm处的吸光度(OD)值。细胞存活率=[(As-Ab) / (Ac-Ab)]×100%;As:试验孔吸光度(含细胞、培养基、CCK-8 溶液和药物溶液);Ac:对照孔吸光度(含细胞、培养基、CCK-8 溶液,不含药物);Ab:空白孔吸光度(含培养基、CCK-8 溶液,不含细胞、药物)。
1.2.6 荧光定量PCR 根据NCBI核酸数据库中相应基因mRNA的全序列,利用Primer premier 5.0软件设计引物,引物由擎科生物公司合成(引物序列见表1)。Trizol法提取细胞样品中的总RNA,紫外分光光度计检测RNA质量和浓度,进行反转录及荧光定量PCR扩增。被检样品均重复3次,取平均值;以GAPDH作为内参基因,利用2-△△Ct法计算各基因的相对表达量,△Ct = Ct目的基因-Ct内参基因,△△Ct= (Ct处理组目的基因-Ct处理组内参基因)-(Ct对照组目的基因-Ct对照组组内参基因),2-△△Ct表示处理组目的基因的表达相对于对照组的变化倍数。
1.2.7 碱性磷酸酶染色 弃掉细胞培养液,加入DPBS清洗细胞。加入4%多聚甲醛,室温固定15 min;弃掉多聚甲醛,用DPBS清洗两次,每孔加入500 µL的BCIP/NBT染色工作液染液,室温避光染色15 min至2 h,弃掉染色液;加入DPBS清洗,蒸馏水终止染色,倒置显微镜下观察拍照。
1.2.8 Western Blot 收集PlaB处理48 h 后的BESCs,加入RIPA蛋白裂解液(含1% PMSF)提取总蛋白,BCA 试剂盒测定蛋白浓度,将蛋白浓度进行均一化;加入上样缓冲液(4﹕1)于100℃进行变性。取电泳凝胶,将胶板装入垂直板电泳槽,拔掉梳子加入1×Tris-Gly 电泳缓冲液,点样,先80 V 电压电泳30 min,后调电压至 120 V,待样品中溴酚蓝迁移至凝胶最下端时,结束电泳;取出凝胶,提前用甲醛浸泡PVDF膜1—2 min,用电转液浸泡滤纸;使用 Bio-Rad 公司的电转仪进行转膜;转膜结束后,将PVDF膜放入 TBST 溶液洗10 min;加入封闭液室温封闭1 h,封闭结束后 TBST洗3次,5 min/次;用一抗稀释液将一抗稀释至相应比例:GAPDH(1﹕5000),OCT4(1﹕2000),SOX2(1﹕2000),NANOG(1﹕250),一抗4℃孵育PVDF膜过夜;TBST溶液漂洗膜3次,5 min/次;加入二抗室温下摇床上孵育膜1 h;TBST溶液漂洗膜3次,5 min/次;ECL化学发光试剂进行显影。
1.2.9 免疫荧光染色 用500 µL DPBS缓慢清洗细胞3次,每次5 min。加入200 µL的4%多聚甲醛固定细胞,室温放置15—30 min;用DPBS洗细胞3次,每次5 min;0.5%Triton X-100通透20 min,PBS洗3次,每次5 min;封闭液封闭细胞1 h;添加一抗(按照一抗稀释比例进行稀释),4 ℃避光过夜;然后,用PBS清洗3次,每次5 min;添加二抗(按照二抗稀释比例进行稀释),室温避光孵育1 h;PBS清洗细胞3次,每次5 min;DPBS清洗细胞1次,每次5 min;加200 µL的Hoechst33342避光染色5 min,PBS清洗细胞,在荧光显微镜下观察拍照。

表1 实时荧光定量 PCR分析所用引物
1.2.10 EDU染色 根据BeyoClickTMEdU-488细胞增殖检测试剂盒(碧云天,C0071S)操作说明书对不同处理组的BESCs进行增殖检测。试验步骤简要如下:将BESCs消化成细胞悬液,然后将细胞以每孔10 000个铺于24孔板中,分别添加PlaB浓度为0.5、1、1.5 nmol·L-1的EPSCM培养液和CTFR培养液处理24 h。用细胞培养液1﹕500稀释EdU(10 mmol·L-1)即可得到2X的EdU工作液,将37 ℃预热的2X的EdU工作液(20 μmol·L-1),与培养液以1﹕1的体积比加入24孔板中,使24孔板中的EdU终浓度变为1X,继续孵育细胞2 h;EdU标记细胞完成后,去除培养液,并加入1 mL 4%的多聚甲醛固定液,室温固定15 min;去除固定液,每孔用1 mL洗涤液洗涤细胞3次,每次3 min;去除洗涤液,每孔加入500 μL 0.3% Triton X-100,室温孵育10 min;去除通透液,每孔用1 mL洗涤液洗涤细胞2次,每次3 min。每孔加入0.5 mL Click反应液,轻轻摇晃培养板以确保反应混合物可以均匀覆盖样品,室温避光孵育30 min;吸除Click反应液,用洗涤液洗涤3次,每次3 min。吸除洗涤液后,每孔加1X Hoechst 33342溶液1 mL,室温避光孵育10 min;吸除1X Hoechst 33342溶液;用洗涤液洗涤3次,每次3 min;利用荧光显微镜进行检测。
1.2.11 数据统计与分析 试验中所有数据均进行3次重复性检验,数据以均值±标准差表示,用SPSS 20软件分析不同处理组间对比用One-Way ANOVA,以<0.05为差异有统计学意义;本研究数据采用GraphPad Prism 7 软件进行作图。
2 结果
2.1 牛胚胎干细胞建系
如图1所示,牛早期囊胚在MEF饲养层上贴附后(图1-A),1 d后长出Outgrowth(图1-B—D)。Outgrowth生长4 d后,进行消化传代,建立牛胚胎干细胞。免疫荧光染色检测BESCs多能因子的表达,结果表明BESCs中表达多能因子OCT4、SOX2及NANOG(图2-A)。对第26代BESCs进行畸胎瘤试验,结果表明BESCs具备生成三胚层的潜力(图2-B)。对第48代BESCs进行核型分析,结果表明BESCs具备正常核型(2n=60,32/40,80%。图2-C)。
将建立的牛胚胎干细胞分别加入CTFR培养液和EPSCM培养液中培养,两种培养液的成分差异见表2。结果表明,牛胚胎干细胞在不同的培养液中呈现出不同的形态特征。EPSCM-BESCs细胞排列紧密,呈集落状生长,克隆呈突起圆顶状,边缘遮光性强,与周围的饲养层细胞界限分明,形态类似Naïve-ESCs(图3-A,B),而CTFR-BESCs克隆呈扁平状,形态类似Primed-ESCs(图3-D,E),碱性磷酸酶染色均呈阳性(图3-C,F)。RT-qPCR检测CTFR-BESCs和EPSCM-BESCs中多能标记基因的mRNA表达,结果表明,CTFR-BESCs和EPSCM- BESCs中多能标记基因的mRNA表达显著高于BEF,且EPSCM- BESCs中的多能标记基因表达高于CTFR-BESCs(图3-G)。CTFR-BESCs和EPSCM- BESCs中胚胎细胞谱系基因的表达水平均较低,但CTFR-BESCs中胚胎细胞谱系基因的表达水平高于EPSCM-BESCs(图3-H)。

A. 贴附的囊胚;B. 第一天的Outgrowth;C. 第二天的Outgrowth;D. 第四天的Outgrowth
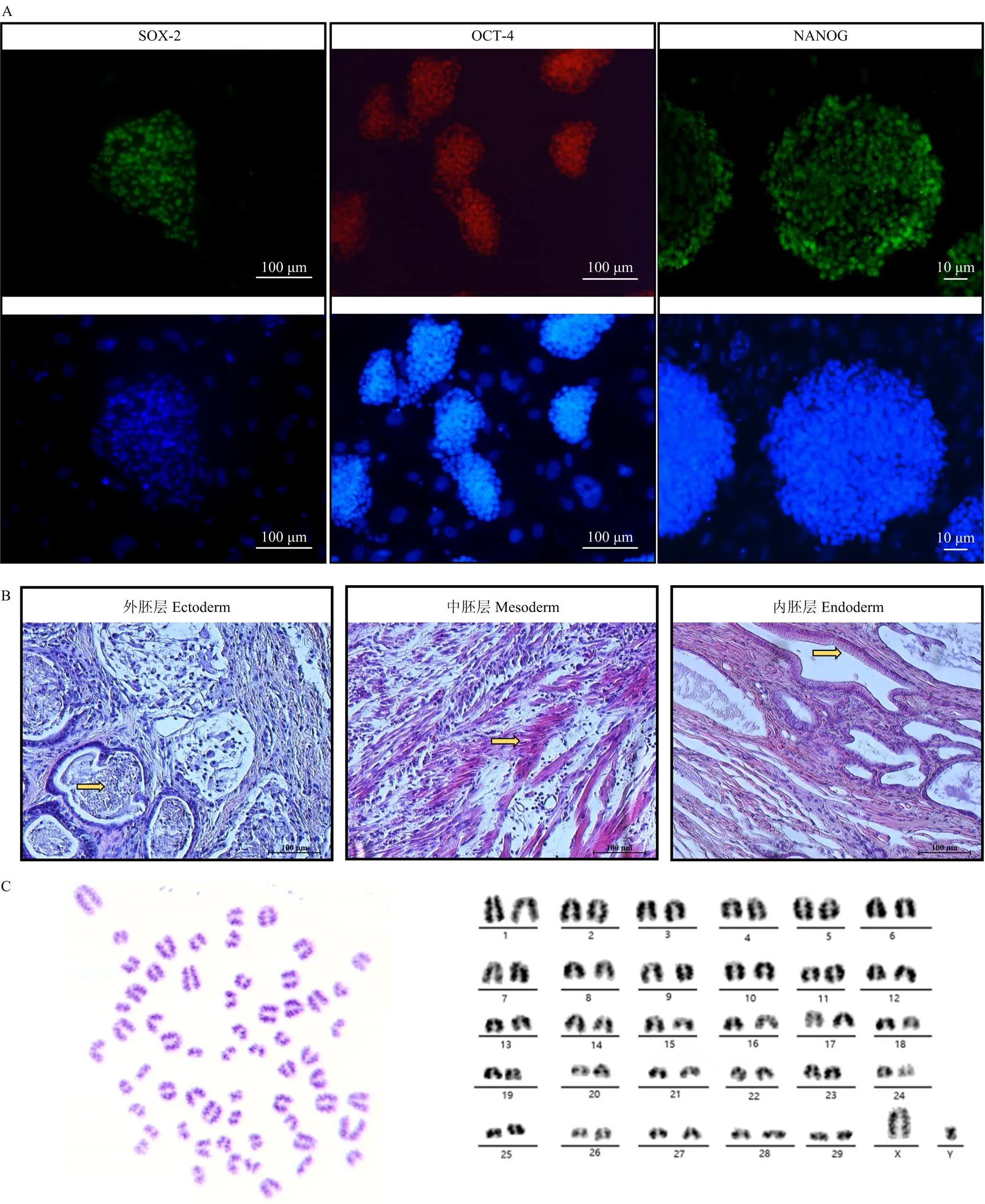
A. 免疫荧光染色鉴定BESCs多能因子表达;B. 传代26代的BESCs用以制备畸胎瘤,HE染色鉴定表明畸胎瘤组织切片包含外胚层(黄色箭头所指为有髓神经)、中胚层(黄色箭头所指为肌肉细胞)和内胚层(黄色箭头所指为柱状上皮细胞);C. 传代48代的BESCs细胞具备正常核型
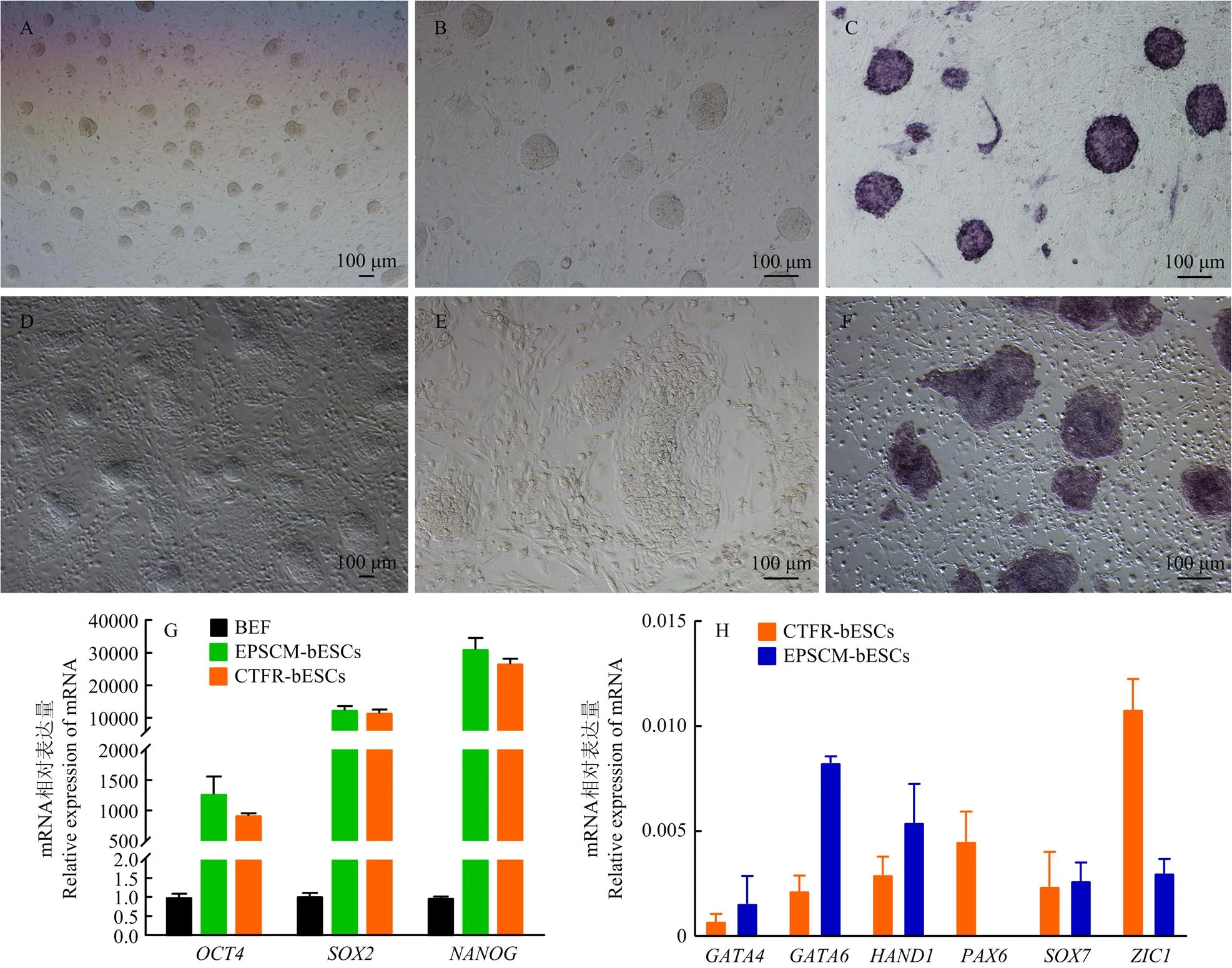
A. 4X镜下EPSCM-BESCs;B. 10X镜下EPSCM-BESCs;C. EPSCM -BESCs碱性磷酸酶染色;D. 4X镜下CTFR-BESCs;E. 10X镜下CTFR-BESCs;F. CTFR-BESCs碱性磷酸酶染色;G. 多能标记基因的mRNA表达情况;H. 胚胎细胞谱系基因的mRNA表达情况
2.2 PlaB对BESCs剪接体表达的影响
分别在培养液中添加不同浓度PlaB培养CTFR-BESCs和EPSCM-BESCs,荧光定量PCR检测各组BESCs中s各亚基的mRNA表达水平。结果如图4所示,PlaB浓度为0.5—1.5 nmol·L-1时显著下调EPSCM-BESCs中和的mRNA表达水平;PlaB浓度为1.5 nmol·L-1时能下调CTFR中的mRNA表达;在PlaB浓度范围为0.5—1.5 nmol·L-1时,CTFR-BESCs中的呈剂量依赖性上调;而在EPSCM-BESCs中,PlaB浓度为1 nmol·L-1时,显著下调,其他浓度则无显著变化。在PlaB浓度范围为0.5—1.5 nmol·L-1时,CTFR-BESCs和EPSCM-BESCs的和的 mRNA表达水平均呈现剂量依赖性增加。此外,PlaB浓度范围为0.5—1.5 nmol·L-1时,CTFR-BESCs中的mRNA表达均显著下调。
荧光定量PCR检测各组BESCs中剪接体的mRNA表达水平,结果如图5所示,PlaB浓度为0.5—1.5 nmol·L-1时,EPSCM-BESCs和CTFR-BESCs中剪接体的mRNA表达水平均显著下调。PlaB浓度为0.5—1.5 nmol·L-1时,能显著下调CTFR-BESCs的mRNA表达水平;PlaB的浓度为1—1.5 nmol·L-1时能显著下调EPSCM-BESCs的mRNA表达水平。PlaB浓度为0.5—1.5 nmol·L-1时,对EPSCM-BESCs和CTFR-BESCs中的和的mRNA表达均无显著影响。

* P<0.05,** P<0.01,*** P<0.001。下同 The same as below
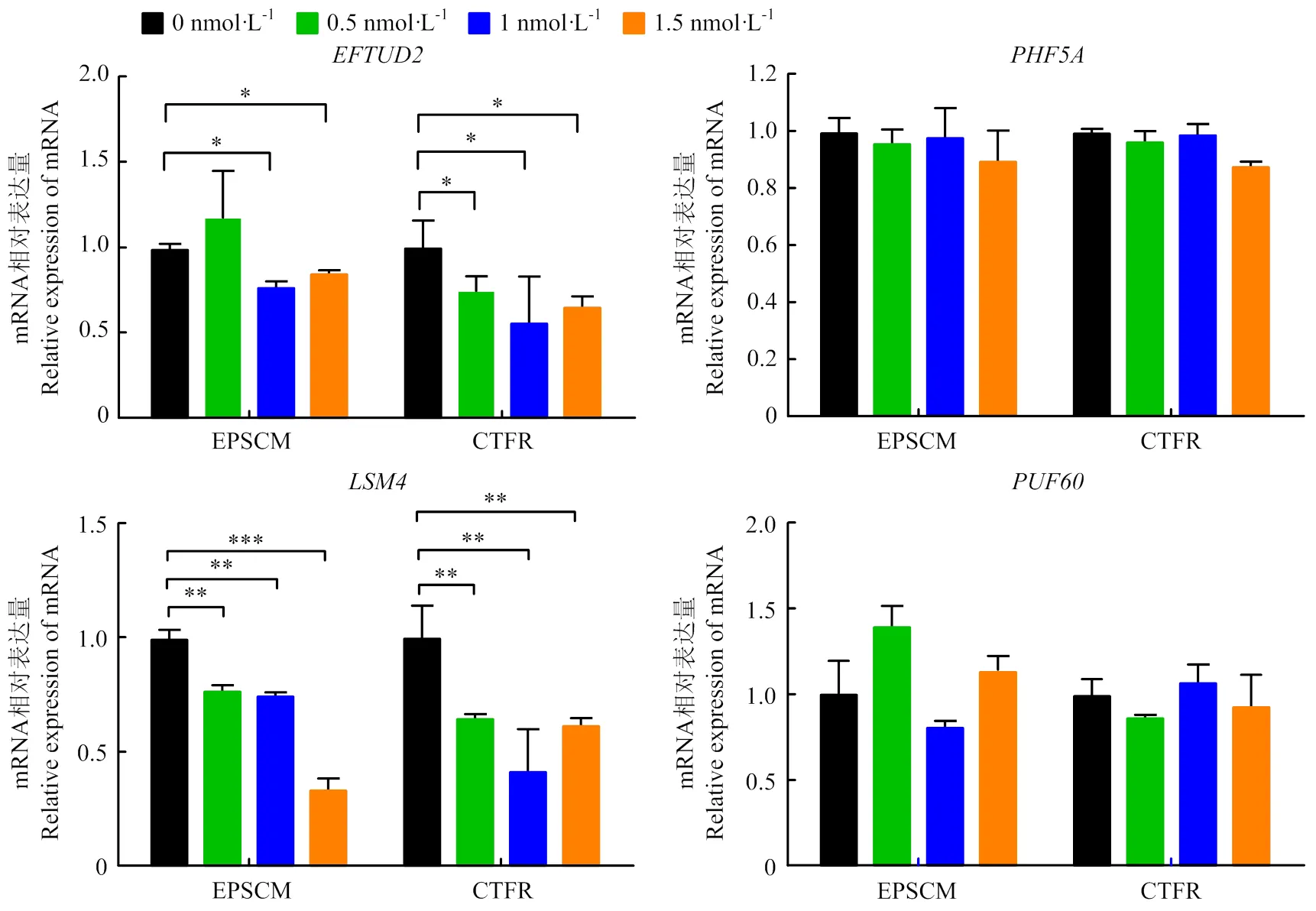
图5 PlaB对BESCs中剪接体 mRNA表达的影响
2.3 PlaB对BESCs多能标记基因表达的影响
在PlaB浓度为0.5—1.5 nmol·L-1范围内,PlaB均以剂量依赖性上调CTFR-BESCs和EPSCM-BESCs中多能标记基因、及的mRNA表达水平(图6-A—C);此外,PlaB能显著上调EPSCM-BESCs中多能标记基因的蛋白表达水平(图6-D、E)。
2.4 PlaB对BESCs全能标记基因表达的影响
在PlaB浓度为0.5—1.5 nmol·L-1范围内,PlaB均以剂量依赖性上调CTFR-BESCs和EPSCM-BESCs中全能标记基因、及的mRNA表达水平,而下调和的mRNA表达水平(图7)。
2.5 PlaB对BESCs胚胎细胞谱系基因表达的影响
如图8所示,CTFR-BESCs中添加PlaB均上调胚胎细胞谱系基因的表达,而EPSCM-BESCs中添加PlaB显著抑制、、等胚胎细胞谱系基因的表达,但是对于没有显著影响。
2.6 PlaB对BESCs细胞增殖的影响
添加不同浓度PlaB培养CTFR-BESCs和EPSCM-BESCs,分别于24和72 h后检测细胞活力,结果如图9所示。随着PlaB剂量增加和处理时间延长,CTFR-BESCs和EPSCM-BESCs细胞活力均呈现下降趋势,且PlaB处理时间延长,CTFR-BESCs和EPSCM- BESCs细胞活力下降显著。
3 讨论
本研究参考已报道的两种牛胚胎干细胞培养体系CTFR[8]和EPSCM[9],分别建立了CTFR-BESCs和EPSCM-BESCs两种牛胚胎干细胞系,通过在培养液中添加不同浓度的剪接体抑制剂PlaB,评估PlaB对CTFR-BESCs和EPSCM-BESCs细胞中剪接体、多能标记基因、全能标记基因、胚胎细胞谱系基因表达的影响,以及比较PlaB对CTFR-BESCs和EPSCM-BESCs细胞活力的影响。
3.1 PlaB对牛胚胎干细胞中剪接体表达的影响
真核生物细胞中,mRNA在细胞核中被转录合成为前体mRNA(pre-mRNA),再由剪接体完成选择性剪接,去除内含子。在pre-mRNA剪接过程中,剪接体根据所依赖的底物完成复合物组装、催化活化和活性部位重塑,表现出特殊组分和结构动力学,以达到既能准确识别又能灵活选择pre-mRNA中的反应性剪接位点[17-18]。有研究发现,选择性剪接是早期胚胎发育的关键调控机制,如U5核心组分EFTUD2在整个胚胎发育过程中都有表达[19],EFTUD2突变导致小鼠着床前阻滞[20]。然而,剪接体在牛胚胎干细胞中的作用缺少研究报道。本研究中,在培养基中添加了不同浓度的SF3B1抑制剂PlaB,发现PlaB不仅下调EPSCM-BESCs和CTFR-BESCs中的mRNA水平,还下调的mRNA表达水平,且的表达模式与相似。虽然PlaB下调了EPSCM- BESCs和CTFR-BESCs中的、表达,但却剂量依赖性地上调了和的mRNA表达。此外,PlaB显著下调CTFR-BESCs中的mRNA表达。

A—C. 不同浓度PlaB对BESCs多能标记因子mRNA表达的影响;D. 不同浓度PlaB对EPSCM-BESCs多能标记因子蛋白表达的影响;E. 多能标记因子蛋白表达量化结果

图7 PlaB对BESCs全能标记基因mRNA表达的影响

图8 PlaB对BESCs胚胎细胞谱系基因mRNA表达的影响
除了SF3Bs亚单元,本研究还检测了剪接体的其他功能基因的表达。LSMs蛋白是剪接体U6 snRNP的核心组成部分[21]。LSM4与LSM2、LSM3、LSM8直接参与U6 snRNA的3' 端识别,参与成熟U6 snRNP复合物的组装[22]。在本研究中,PlaB浓度为0.5—1.5 nmol·L-1时,bEPSCM-BESCs和CTFR-BESCs中的mRNA表达均显著下调。编码一个GTP酶,是剪接体U5 snRNP 组成部分的核心模块。的GDP/GTP状态和磷酸化状态都有助于剪接体的剪接和拆卸。本研究结果表明,PlaB的浓度为1—1.5 nmol·L-1时显著下调bEPSCM-BESCs的mRNA表达水平。PlaB浓度为0.5—1.5 nmol·L-1时,显著下调CTFR-BESCs的mRNA表达水平;而PlaB浓度为0.5—1.5 nmol·L-1时,对bEPSCM-BESCs和CTFR-BESCs中的和的mRNA表达均无显著影响。以上结果表明,相同培养体系下,PlaB对SF3B亚基和剪接体关键组分的mRNA表达影响并不一致。而不同培养体系下,PlaB对剪接体各组分关键基因mRNA表达的影响也不尽相同。

A—D. CCK8检测不同处理组的BESCs细胞增殖情况;E. EDU检测不同浓度PlaB处理CTFR-BESCs和EPSCM-BESCs 72 h后BESCs的细胞增殖情况
3.2 PlaB对牛胚胎干细胞多能性标记基因和胚胎细胞谱系基因表达的影响
胚胎干细胞具有多能潜能和自我更新能力,能够发育为不同胚层。这些多能性的维持取决于转录因子、和的表达水平。,又被命名为或,是第一个被鉴定为多能性调控因子的转录因子[23]。在建立的胚胎干细胞中,诱导缺失导致自我更新丧失和分化。也是ES细胞自我更新所必需的。在ES细胞中的失活导致滋养层形成,及表达缺失。的过表达使ES细胞易于分化。表达水平的升高可以在不依赖LIF的情况下维持小鼠胚胎干细胞的自我更新,并使人类胚胎干细胞无需饲养层细胞也能生长[24]。此外,已经被证明OCT4、SOX2和NANOG形成一个核心三联体,存在相互交叉调节,通过自我维持的正反馈来维持多能性状态。在小鼠ESCs培养中添加2.5 nmol·L-1PlaB显著降低小鼠ESCs多能因子的表达,导致表达缺失[13]。然而,在本研究中,PlaB浓度为0.5—1.5 nmol·L-1范围内均以剂量依赖性上调CTFR-BESCs和EPSCM-BESCs中多能因子、及的mRNA表达水平和蛋白水平。这一结果的不一致,一方面可能与小鼠ESCs中使用的浓度不同有关,另一方面也可能是牛ESCs转录后的剪接调控有别于小鼠ESCs。添加PlaB上调BESCs中多能因子表达的作用机制仍需进一步研究。
在本研究中,检测了胚胎细胞谱系基因的mRNA表达变化,发现CTFR-BESCs中添加PlaB均上调胚胎细胞谱系基因的表达,而EPSCM-BESCs中添加PlaB显著抑制、、等胚胎细胞谱系基因的表达。这一差异可能与不同培养体系下BESCs的多能性状态差异有关。这一结果提示,相比于CTFR,在EPSCM培养体系添加PlaB更有助于抑制BESCs的分化,该结果为后续深入优化BESCs培养体系提供了参考依据。
3.3 PlaB对牛胚胎干细胞全能标记因子表达及细胞活力的影响
全能性细胞具有最高的发育潜力,通常指哺乳动物体内的受精卵、2细胞和4细胞的卵裂球[25]。一直以来,分离和培养能够产生整个胚胎的全能性干细胞是一项非常具有挑战性的工作。在小鼠ESCs培养中添加2.5 nmol·L-1PlaB显著上调小鼠ESCs全能因子、及的mRNA表达[15]。与其一致的是,在本研究中,PlaB浓度为0.5—1.5 nmol·L-1范围内,PlaB以剂量依赖性上调CTFR-BESCs和EPSCM-BESCs中、及的mRNA表达水平;但不同的是,和的mRNA表达水平下调。在本研究中,随着PlaB剂量增加和处理时间延长,CTFR-BESCs和EPSCM-BESCs细胞活力均呈现下降趋势,且PlaB处理时间延长,CTFR- BESCs和EPSCM-BESCs细胞活力下降显著。这一结果提示,PlaB在时间、剂量和给药时间表上对两种牛胚胎干细胞系均有细胞毒性作用,但CTFR-BESCs比EPSCM-BESCs更敏感。有研究表明,突变导致的mRNA表达上调[26]。在本研究中,添加PlaB后,的表达下降而的mRNA表达上调,这与上述研究结果一致。MDM2靶向结合肿瘤抑制因子p53,抑制p53活性,参与细胞周期调节、凋亡、分化和基因组稳定性调节[27-28]。近期的研究表明,小鼠的缺失导致胚胎致死[29]。BTG2是p53的下游因子,作为肿瘤抑制因子,BTG2过表达导致细胞生长抑制[30]。与以上研究一致,在本研究中,PlaB导致BESCs中、表达上调,而促增殖因子和下调可能是影响细胞活力的原因之一。
4 结论
提高多能干细胞发育潜能有助于提高早期胚胎发育的能力,并有助于更有效地生产用于研究和器官移植的嵌合动物。在本研究中,添加PlaB能有效抑制和的mRNA表达,并影响剪接体其他组分关键基因的mRNA表达,显著上调多能标记基因表达和部分全能标记基因表达,下调促增殖因子表达,此外,CTFR-BESCs中添加PlaB均上调胚胎细胞谱系基因的表达,而EPSCM-BESCs中添加PlaB显著抑制、、等胚胎细胞谱系基因的表达。这一结果提示,相比于CTFR,在EPSCM培养体系添加PlaB更有助于抑制BESCs的分化,然而,由于PlaB影响BESCs的细胞活力,因此,仍需进一步研究以优化培养体系。
[1] 王冰源, 牟玉莲, 李奎, 刘志国. 农业动物干细胞研究进展. 遗传, 2020, 42(11): 1073-1080.
WANG B Y, MU Y L, LI K, LIU Z G. Research progress of stem cells in agricultural animals. Hereditas(Beijing), 2020, 42(11): 1073-1080. (in Chinese)
[2] HOSKINS A A, RODGERS M L, FRIEDMAN L J, GELLES J, MOORE M J. Single molecule analysis reveals reversible and irreversible steps during spliceosome activation. eLife, 2016, 5: e14166.
[3] JURICA M S, MOORE M J. Pre-mRNA splicing: awash in a sea of proteins. Molecular Cell, 2003, 12(1): 5-14.
[4] WILL C L, LUHRMANN R. Spliceosome structure and function. Cold Spring Harbor Perspectives in Biology, 2011, 3(7): a003707.
[5] LÓPEZ-CÁNOVAS J L, DEL RIO-MORENO M, GARCÍA- FERNANDEZ H, JIMÉNEZ-VACAS J M, MORENO-MONTILLA M T, SÁNCHEZ-FRIAS M E, AMADO V, L-LÓPEZ F, FONDEVILA M F, CIRIA R, GÓMEZ-LUQUE I, BRICEÑO J, NOGUEIRAS R, DE LA MATA M, CASTAÑO J P, RODRIGUEZ- PERÁLVAREZ M, LUQUE R M, GAHETE M D. Splicing factor SF3B1 is overexpressed and implicated in the aggressiveness and survival of hepatocellular carcinoma. Cancer Letters, 2021, 496: 72-83.
[6] GÓMEZ-REDONDO I, RAMOS-IBEAS P, PERICUESTA E, FERNÁNDEZ-GONZÁLEZ R, LAGUNA-BARRAZA R, GUTIÉRREZ- ADÁN A. Minor splicing factors Zrsr1 and Zrsr2 are essential for early embryo development and 2-cell-like conversion. International Journal of Molecular Sciences, 2020, 21(11): 4115.
[7] LÖB S, VATTAI A, KUHN C, SCHMOECKEL E, MAHNER S, WÖCKEL A, KOLBEN T, SZEKERES-BARTHO J, JESCHKE U, VILSMAIER T. Spliceosome protein EFTUD2 is upregulated in the trophoblast of spontaneous miscarriage and hydatidiform mole. Journal of reproductive immunology, 2020(140): 103149.
[8] BOGLIOTTI Y S, WU J, VILARINO M, OKAMURA D, SOTO D A, ZHONG C Q, SAKURAI M, SAMPAIO R V, SUZUKI K, IZPISUA BELMONTE J C, ROSS P J. Efficient derivation of stable primed pluripotent embryonic stem cells from bovine blastocysts. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(9): 2090-2095.
[9] ZHAO L X, GAO X F, ZHENG Y X, WANG Z X, ZHAO G P, REN J, ZHANG J, WU J, WU B J, CHEN Y L, SUN W, LI Y X, SU J, DING Y L, GAO Y, LIU M N, BAI X C, SUN L Z, CAO G F, TANG F C, BAO S Q, LIU P T, LI X H. Establishment of bovine expanded potential stem cells. Proceedings of the National Academy of Sciences of the United States of America, 2021(118): e2018505118.
[10] CAPUCO A V, CHOUDHARY R K, DANIELS K M, LI R W, EVOCK-CLOVER C M. Bovine mammary stem cells: Cell biology meets production agriculture. Animal, 2012, 6(3): 382-393.
[11] GUGJOO M B, AMARPAL, CHANDRA V, WANI M Y, DHAMA K, SHARMA G T. Mesenchymal stem cell research in veterinary medicine. Current Stem Cell Research & Therapy, 2018, 13(8): 645-657.
[12] ZHAO L X, WANG Z X, ZHANG J D, YANG J, GAO X F, WU B J, ZHAO G P, BAO S Q, HU S X, LIU P T, LI X H. Characterization of the single-cell derived bovine induced pluripotent stem cells. Tissue and Cell, 2017, 49(5): 521-527.
[13] PARK J W, FU S Y, HUANG B R, XU R H. Alternative splicing in mesenchymal stem cell differentiation. Stem Cells, 2020, 38(10): 1229-1240.
[14] ZHOU Z J, QU J, HE L, PENG H, CHEN P, ZHOU Y. α6-Integrin alternative splicing: Distinct cytoplasmic variants in stem cell fate specification and niche interaction. Stem Cell Research & Therapy, 2018, 9(1): 122.
[15] SHEN H, YANG M, LI S Y, ZHANG J, PENG B, WANG C H, CHANG Z, ONG J, DU P. Mouse totipotent stem cells captured and maintained through spliceosomal repression. Cell, 2021, 184(11): 2843-2859.e20.
[16] GHOSH A K, ANDERSON D D. Enantioselective total synthesis of pladienolide B: A potent spliceosome inhibitor. Organic Letters, 2012, 14(18): 4730-4733.
[17] YAN C Y, WAN R X, SHI Y G. Molecular mechanisms of pre-mRNA splicing through structural biology of the spliceosome. Cold Spring Harbor Perspectives in Biology, 2019, 11(1): a032409.
[18] WAHL M C, WILL C L, LÜHRMANN R. The spliceosome: Design principles of a dynamic RNP machine. Cell, 2009, 136(4): 701-718.
[19] SMALL E C, LEGGETT S R, WINANS A A, STALEY J P. The EF-G-like GTPase Snu114p regulates spliceosome dynamics mediated by Brr2p, a DExD/H box ATPase. Molecular Cell, 2006, 23(3): 389-399.
[20] BEAUCHAMP M C, DJEDID A, DAUPIN K, CLOKIE K, KUMAR S, MAJEWSKI J, JEROME-MAJEWSKA L A. Loss of function mutation of Eftud2, the gene responsible for mandibulofacial dysostosis with microcephaly (MFDM), leads to pre-implantation arrest in mouse. PLoS ONE, 2019, 14(7): e0219280.
[21] MATERA A G, WANG Z F. A day in the life of the spliceosome. Nature Reviews Molecular Cell Biology, 2014, 15(2): 108-121. doi:10.1038/nrm3742.
[22] ZHOU L J, HANG J, ZHOU Y L, WAN R X, LU G F, YIN P, YAN C Y, SHI Y G. Crystal structures of the Lsm complex bound to the 3' end sequence of U6 small nuclear RNA. Nature, 2014, 506(7486): 116-120.
[23] OKAMOTO K, OKAZAWA H, OKUDA A, SAKAI M, MURAMATSU M, HAMADA H. A novel octamer binding transcription factor is differentially expressed in mouse embryonic cells. Cell, 1990, 60(3): 461-472.
[24] PAN G J, THOMSON J A. Nanog and transcriptional networks in embryonic stem cell pluripotency. Cell Research, 2007, 17(1): 42-49.
[25] FU X D, WU X J, DJEKIDEL M N, ZHANG Y. Myc and Dnmt1 impede the pluripotent to totipotent state transition in embryonic stem cells. Nature Cell Biology, 2019, 21(7): 835-844.
[26] BEAUCHAMP M C, DJEDID A, BAREKE E, MERKURI F, ABER R, TAM A S, LINES M A, BOYCOTT K M, STIRLING P C, FISH J L, MAJEWSKI J, JEROME-MAJEWSKA L A. Mutation in Eftud2 causes craniofacial defects in mice via mis-splicing of Mdm2 and increased P53. Human Molecular Genetics, 2021, 30(9): 739-757.
[27] WIENKEN M, MOLL U M, DOBBELSTEIN M. Mdm2 as a chromatin modifier. Journal of Molecular Cell Biology, 2016, 9(1): 74-80.
[28] HAUPT Y, MAYA R, KAZAZ A, OREN M. Mdm2 promotes the rapid degradation of p53. Nature, 1997, 387(6630): 296-299.
[29] HUMPTON T J, NOMURA K, WEBER J, MAGNUSSEN H M, HOCK A K, NIXON C, DHAYADE S, STEVENSON D, HUANG D T, STRATHDEE D, BLYTH K, VOUSDEN K H. Differential requirements for MDM2 E3 activity during embryogenesis and in adult mice. Genes & Development, 2021, 35(1/2): 117-132.
[30] TSUI K H, CHIANG K C, LIN Y H, CHANG K S, FENG T H, JUANG H H.is a tumor suppressor gene upregulated by p53 andin human bladder carcinoma cells. Cancer Medicine, 2018, 7(1): 184-195.
Effects of Pladienolide B on Expression of Pluripotency Related Genes and Cell Viability of Bovine Embryonic Stem Cells
ZHAO Fang1, DING Qiang1, XIA ShuWen1, GAO YunDong2, LAN GuoCheng3, LIN ZhiPing4, WANG HuiLi, ZHONG JiFeng
1Institute of Animal Science, Jiangsu Academy of Agricultural Sciences/Jiangsu Province Engineering Research Center for Precision Animal Breeding/Key Laboratory of Crop and Animal Integrated Farming, Ministry of Agriculture and Rural Affairs, Nanjing 210014;2Shandong OX Livestock Breeding Co., Ltd, Jinan 250100;3Li Ka Shing Faculty of Medicine, The University of Hong Kong, Hong Kong 999077;4Jiangsu Youyuan Dairy Industry Research Institute Co. Ltd, Nanjing 211100
【Background】 Due to high pluripotency of bovine embryonic stem cells (BESCs), they have important application values in cattle breed conservation, breed selection, and regulation mechanism study of livestock embryo development. However, the studies on the maintenance of pluripotency and differentiation of BESCs are limited, and the regulative mechanism remains unclear.【Objective】The aim of this study was to investigate the effects of different concentrations of Pladienolide B (PlaB) on the expression of pluripotent markers, totipotent markers and embryonic cell-lineage genes as well as the cell viability of BESCs,so as to provide the reference and theoretical basis for improving the developmental potency of BESCs.【Method】Immunofluorescence was used to detect the expression of pluripotent markers of bovine BESCs, and real-time fluorescence quantitative PCR (RT-qPCR) was used to detect the effects of different concentrations of PlaB on the expression of spliceosome, totipotent markers and embryonic cell-lineage genes of BESCs. RT-qPCR and Western Blot were used to detect the effects of different concentrations of PlaB on both mRNA and protein expression of pluripotent markers of BESCs. CCK8 and EDU staining was used to detect the effects of different concentrations of PlaB on the proliferation of BESCs. 【Result】RT-qPCR results showed that the mRNA expression levels ofandnmol·L-1to 1.5 nmol·L-1PlaB; when the PlaB concentration was 1.5 nmol·L-1, the mRNA expressions ofandnmol·L-1to 1.5 nmol·L-1, the mRNA expression levels ofandnmol·L-1to 1.5 nmol·L-1, the mRNA expression of spliceosomeboth in EPSCM-BESCs and CTFR-BESCs were significantly down-regulated. The concentration from 0.5 nmol·L-1to 1.5 nmol·L-1PlaB significantly down-regulated the expression levels ofmRNA in CTFR-BESCs; the mRNA expression ofmRNA was significantly down-regulated in BEPSCM-BESCs with 1 nmol·L-1and 1.5 nmol·L-1PlaB while PlaB concentration from 0.5 to 1.5 nmol·L-1, both the mRNA expression and protein levels of the pluripotent markers,andin CTFR-BESCs and EPSCM-BESCs were up-regulated in a dose-dependent manner. By the concentration range from 0.5 to 1.5 nmol·L-1, PlaB dose-dependently up-regulated the mRNA levels of totipotent markers such as,andandwere down-regulated. The mRNA expression of embryonic cell lineage genes in the CTFR-BESCs were up-regulated while the PlaB was added. The addition of PlaB in EPSCM-BESCs significantly reduced the expression of,,and other embryonic cell lineage genes, but had no significant effect on. The cell viability of CTFR-BESCs and EPSCM-BESCs showed a downward trend with increasing of PlaB dose and treatment time, while CTFR-BESCs was more sensitive than EPSCM-BESCs. 【Conclusion】PlaB significantly up-regulated the expression of pluripotent markers and partial totipotent markers in CTFR-BESCs and EPSCM-BESCs, and the expression of gene lineages and cell viability in EPSCM-BESCs were decreased. The effective concentration and effects on gene expression of PlaB in the two types BESCs were not completely consistent. Due to the inhibiting effect of PlaB on cell viability of BESCs, the further studies were needed to optimize the culture system.
bovine embryonic stem cells; pladienolide B; spliceosome; pluripotency related genes; cell viability
2022-01-20;
2022-09-07
国家重点研发计划(2021YFD1200404)、江苏省自然科学基金面上项目(BK20221433)、江苏省农业科学院探索性颠覆性创新计划项目(ZX(21)1214)、江苏省农业科技自主创新资金(CX(19)2037)
赵芳,E-mail:zanzi006@163.com。通信作者王慧利,E-mail:wanghuili318@163.com。通信作者仲跻峰,E-mail:zhongjifeng64@sina.cn
10.3864/j.issn.0578-1752.2023.11.012
(责任编辑 林鉴非)
