基于生物信息学研究分析弥漫大B细胞淋巴瘤的潜在靶标与挖掘治疗药物
2023-06-04梅汉玮何国平赵轩竹王华庆
梅汉玮,何国平,赵轩竹,王华庆
(1.天津中医药大学 中西医结合学院,天津 301617; 2.天津市人民医院 肿瘤诊疗中心,天津 300122; 3.南开大学人民医院 转化医学研究院,天津 300122)
0 引言
弥漫性大B细胞淋巴瘤是最常见的非奇霍金淋巴瘤类型,约占所有非霍奇金淋巴瘤的31%。不同类型的DLBCL具有不同分子特征和预后特点[1],尽管临床基于R-CHOP的化疗可治愈大多数患者,但仍有30%~40%的患者会出现复发难治等问题。研究驱动DLBCL的发病机制,预测寻找治疗DLBCL的化学药物具有重要的实践和理论价值。
1 研究方法
1.1 实验集数据获取
从GEO数据库中获取DLBCL基因芯片GSE56315测序基因集,从ICGC数据库中获取DLBCL基因数据,通过R语言软件筛选出各数据集中具有统计学意义的差异基因取交集。将两者数据共同差异基因芯片与GSE180161芯片数据中暴露风险一致的基因再次取交集,获得实验集基因差异表达基因。
1.2 关键基因筛选
将实验集DEGs基因输入STRING11.5数据库中,绘制出蛋白互作网络图;将PPI数据输出保存,采用Cytoscape 3.9.1软件实现可视化,基于CytoHubba插件中的MCC算法,筛选出PPI蛋白互作中排名前10的hub基因。
1.3 化学药物筛选
将预测Top10靶标作为治疗靶点,在Cmap数据库中筛选对Top10具有生物学效应的化学药物。
1.4 关键基因验证
为进一步验证Top10关键基因对DLBCL患者预后情况的影响,采用带有预后的芯片GSE180161作为验证集数据,验证DEGs中Top10关键基因的预后生存情况。
1.5 靶点-药物分子对接
根据分析结果选择具有生存意义的hub基因与预测的治疗药物进行分子对接,通过Pymol软件构建靶点-药物可视化结果。
2 研究结果
2.1 差异基因数据分析
通过对GEO数据库汇总DLBCL组织芯片GSE56315进行R语言分析,得到差异表达基因,上调基因1 336个,下调基因1 257个(图1);在ICGC数据库中获取的DLBCL差异基因中,上调基因3 483个,下调基因368个(图2)。用R语言软件取交集,共获取上调基因456个,下调基因38个。将高表达与低表达差异基因分别与芯片GSE180161数据集中风险因素一致的基因做交集,使用R语言软件分析芯片GSE180161数据集与高表达基因,取交集共获得61个DEGs基因。将芯片GSE180161数据集中的基因(HR<1.0)与低表达基因取交集,得到13个DEGs基因。
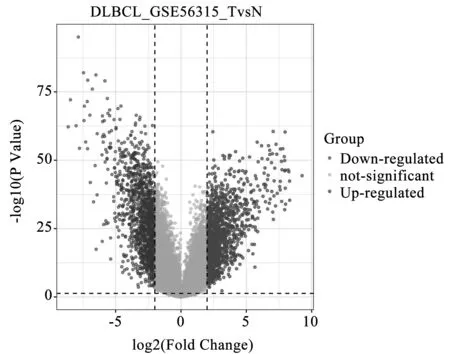
图1 组织芯片GSE56315(55个淋巴瘤组织与33个扁桃腺组织)差异基因Fig.1 Differential genes of tissue microarray GSE56315 (55 lymphoma tissues and 33 tonsil tissues)
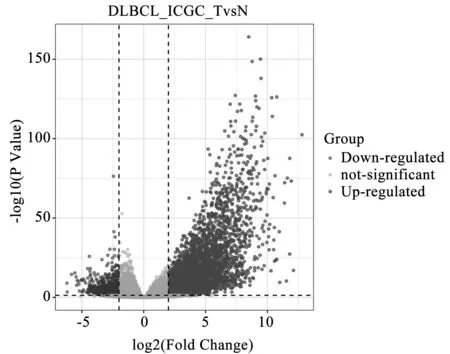
图2 ICGC数据库(27个DLBCL组织样本和7个扁桃腺组织样本)差异基因Fig.2 Differential genes of ICGC database (27 DLBCL tissue samples and 7 tonsil tissue samples)
2.2 PPI筛选关键基因
将上述DEGs基因导入STRING 11.5数据库中,以图片形式和数据格式输出PPI关系结果。利用Cytoscape 3.9.1 软件分析PPI数据,利用插件中的MCC算法计算出前10位hub基因,即TLR2、IL10、CCRL2、CD163、S100A9、C1QA、SIGLEC1、C5AR1、VSIG4、S100A8。
2.3 治疗DLBCL的化学药物筛选
将与DLBCL发生发展密切相关的Top 10靶标导入Cmap数据库中,从中筛选出治疗DLBCL最优的靶标化学药物Palomid-529。
2.4 Top10基因预后生存情况
为进一步验证Top10基因是否对DLBCL发生发展及预后生存具有临床意义,基于芯片GSE180161数据集验证Top10基因预后生存情况,发现CCRL2、SIGLEC1、VSIG4具有显著生存意义,而CD163基因的预后生存差异值为(P=0.05),因此将CCRL2、SIGLEC1、VSIG4、CD163靶点作为治疗DLBCL疾病的关键靶标。
2.5 靶标-药物分子对接
基于Cmap数据库筛选出治疗DLBCL的最优化学物质,利用Auto dock软件,对化学药物与关键的4个靶点进行治疗性模拟对接,发现对接结Palomid-529能有效地与靶标结合,CD163、CCRL2、VSIG4、SIGLEC1的能量值分别为-6.79 KJ/mol、-8.99 KJ/mol、-6.12 KJ/mol、-5.17 KJ/mol。
3 讨论
弥漫大B细胞淋巴瘤临床治疗基于R-CHOP方案可治愈大多数患者,但仍有部分患者发生复发转移,因此迫切需要寻找有效、特异性高、敏感的分子靶标及治疗药物。对GEO与ICGC数据库进行分析发现,CCRL2、SIGLEC1、VSIG4、CD163基因是可能驱动弥漫大B细胞淋巴瘤疾病发生发展的关键基因。CCRL2又名为趋化因子诱饵受体2,是一种跨膜G蛋白偶联受体,在人体组织和器官中广泛分布并参与了肿瘤炎症浸润与免疫反应。研究表明,敲除CCRL2基因可抑制骨髓增生异常综合征的发生发展[2]。VSIG4又名V-set和免疫球蛋白结构域包含4,是I型膜蛋白的一种,临床上一直被认为是致癌基因。研究发现,VSIG4基因在肝癌、肺癌、卵巢癌等癌种中出现过表达且抑制招募T细胞淋巴细胞激活,从而促进肿瘤发生发展。Jin Roh等对多发性骨髓瘤患者进行研究发现,VSIG4高表达组的总生存期明显低于VSIG4低表达组,说明多发性骨髓瘤中的VSIG4基因表达可作为预后不良的独立指标。本研究发现,在DLBCL患者中VSIG4高表达组的总生存期明显低于VSIG4低表达组,由此猜想VSIG4 基因表达可作为DLBCL患者预后不良的独立指标,VSIG4靶点可作为治疗靶标。CD163和VSIG4均是I型膜蛋白,研究表明,CD163的表达与DLBCL不良预后有关系。研究发现,CD163阳性乳腺癌患者明显出现较差的总生存期和较短的无病生存率且高表达预后情况差,因此CD163基因有望作为治疗DLBCL的一个重要靶标。SIGLEC1是唾液酸黏附素,又名CD169。SIGLECS蛋白广泛表达分布于免疫细胞的表面,在维持平衡免疫与炎症反应中起着关键作用。研究发现,肿瘤细胞能够通过唾液酸化配体与抑制性SIGLECS结合,实现肿瘤免疫逃逸,这是肿瘤治疗出现复发难治的重要原因[3]。而淋巴瘤出现复发难治问题很可能是因为DLBCL患者存在SIGLEC1基因的高表达情况。
通过对GEO数据和ICGC数据库中DLBCL的基因表达谱研究发现上调基因61个DEGs、下调基因13个DEGs,将其作为靶标蛋白得到蛋白互作网络,运用Cytoscape软件中的算法,分析了Top10靶点(TLR2、IL10、CCRL2、CD163、S100A9、C1QA、SIGLEC1、C5AR1、VSIG4、S100A8)并对其预后生存情况进行分析,发现CCRL2、SIGLEC1、VSIG4、CD163对弥漫大B细胞淋巴瘤患者的预后有一定的临床价值,有望成为评估预后的新指标和治疗靶点。基于Cmap数据分析筛选发现,Palomid-529可能会成为新的靶向治疗药物。通过分子-靶标对接结果发现,Palomid-529能与CCRL2、SIGLEC1、VSIG4、CD163关键靶标结合良好,可能成为治疗DLBCL的新的靶向药物,为治疗提供新思路。
