向日葵AGO和DCL基因家族的鉴定和表达分析
2023-06-02张红员世宇王文浩刘文俊何丽芬闫玉星王彦尊王鹏冬郑洪元张鑫
张红, 员世宇, 王文浩, 刘文俊, 何丽芬, 闫玉星, 王彦尊, 王鹏冬, 郑洪元, 张鑫,2
向日葵和基因家族的鉴定和表达分析
张红1, 员世宇1, 王文浩1, 刘文俊1, 何丽芬1, 闫玉星1, 王彦尊1, 王鹏冬1, 郑洪元1*, 张鑫1,2*
(1. 山西农业大学经济作物研究所,太原 030031;2. 省部共建有机旱作农业国家重点实验室(筹),太原 030031)
为了解向日葵()的Argonaute (AGO)和Dicer-like (DCL)的功能,利用拟南芥()的和基因序列在向日葵基因组数据库中进行同源比对,对向日葵AGO和DCL家族成员进行生物信息学分析。结果表明,从向日葵中鉴定到15个和5个家族成员;这2类基因在染色体上的分布均不均匀。系统发育分析表明,与拟南芥相似,家族成员可分为3个分支,可分为4个分支;所有的都具有保守的N domain、DUF1785、PAZ和PIWI结构域,家族成员都含有PAZ和RIBOc结构域。表达分析表明,和在茎和花序中高度表达;亚细胞定位表明HaAGO多定位于细胞核。这表明向日葵中可能存在典型的RNAi干扰机制,并可能参与了协调向日葵的生长发育过程。
RNA干扰;AGO家族;DCL家族;向日葵;基因表达
RNAi (RNA interference)即RNA干扰,是在生物体内普遍存在的一种古老的生物学现象,是一种高度保守的基因表达调节机制。典型的RNAi是指在Dicer-like (DCL)和Argonaute (AGO)作用下,利用双链RNA (double-stranded RNA, dsRNA)诱导形成的小RNA (small RNA, sRNA),对目标序列的mRNA进行降解从而特异性地阻断或抑制相应基因表达的过程[1]。DCL和AGO分别参与了RNAi过程中的启动阶段和效应阶段。具有RNaseIII型活性DCL蛋白可将dsRNA加工成为包括siRNAs (small interfering RNAs)、miRNAs (microRNAs)和piRNAs (piwiRNA)等,长度约为20~30个核苷酸的sRNA (small RNA, sRNA)[2–3],进而启动RNAi。AGO蛋白则分别与miRNAs、siRNAs和piRNAs等不同类型的小非编码RNA (small non-coding RNA)结合,在这些小RNA的引导下,AGO蛋白特异地停留在与小RNA互补的靶基因mRNA上,通过AGO蛋白自身的内切酶活性对目标基因mRNA进行切割或翻译抑制等以引起靶基因的沉默[4–5]。
DCL是植物中一种高度保守的核糖核酸酶,属于RNase Ⅲ家族,是RNAi途径中至关重要的组分[6]。DCL蛋白是加工dsRNA前体生成小分子RNA的关键酶,可通过剪切dsRNA产生sRNA,启动RNAi途径。AGO是一个进化上高度保守、成员数量较多的家族[7]。该基因最初由Bohmert等[8]在拟南芥中发现,并在生物界中广泛存在。在动、植物中,AGO与小RNA共同参与维持基因组的稳定性。两者介导的RNAi沉默机制在调控植物个体的生长发育水平及生物和非生物胁迫的反应等重要的生理活动中发挥了关键作用[9–10]。目前对RNAi途径及其关键成分的研究主要集中在拟南芥和水稻等模式植物上,在其他作物中的研究还较少。
向日葵()是重要的油料作物之一,在盐碱和贫瘠地区广泛种植,具有抗旱和耐盐碱特性,是优异的抗逆基因资源[11]。向日葵生长发育过程中会受到许多逆境影响,培育和科学利用抗逆品种是提高向日葵产量最经济有效的方式。RNAi机制在植物中普遍存在,解析RNAi通路关键基因,从RNAi分子机制的角度分析向日葵基因,对提高向日葵的抗逆性、抗病性、产量及品质等有重要意义。本研究利用生物信息学方法,结合向日葵基因组测序数据,鉴定了向日葵和基因家族,分析了AGO和DCL的理化性质、系统进化树、蛋白保守结构域、三级结构和亚细胞定位,以及基因的染色体定位和组织特异性表达等,进而对和基因功能进行预测,以期为深入研究向日葵RNAi沉默机制提供依据,为向日葵抗逆分子育种奠定理论基础。
1 材料和方法
1.1 基因的搜索与确认
在拟南芥数据库TAIR (http://www.arabidopsis. org)以关键词“AGO”和“DCL”搜索全部和家族,得到10个和4个基因的核酸序列及其对应的氨基酸序列。
参考和家族的序列信息,以向日葵()自交系XRQ为研究对象, 在Phytozome13向日葵基因组r1.2数据库(https://phyto zome.jgi.doe.gov/pz/portal.html)中用拟南芥同源序列比对得到向日葵和基因家族所有候选成员的氨基酸序列,先删除掉氨基酸数量过少的序列,再将剩余蛋白的氨基酸序列提交到蛋白质结构域预测网站PFAM (http://pfam.sanger.ac.uk/)及SMART (http://smart.embl-heidelberg.de/),根据基因家族典型结构域PAZ与PIWI,基因家族结构域PAZ和RIBOc,筛除不具有上述结构域的基因,得到向日葵相应基因家族的所有成员和,进而得到相应的CDS序列、氨基酸序列、基因登录号和染色体定位信息。
1.2 生物信息学分析
使用在线软件ExPASy (http://www.expasy.org./ tools/protparam.html)分析和基因家族成员的理化特性。使用在线软件NCBI (https:// blast.ncbi.nlm.nih.gov/Blast.cgi)获得基因外显子数量。使用GSDS 2.0 [Gene Structure Display Server 2.0 (gao-lab.org)]获得基因内含子数量。
1.3 系统发育树建立
使用Phylogeny (http://phylogeny.lirmm.fr/)软件构建向日葵和拟南芥的AGO和DCL蛋白的系统发育树。MUSCLE参数设置为Run mode:full processing mode;Gblocks参数设置为Min. seq. for flank pos.: 85%、Max. contig. nonconserved pos.:8、Min. block length:10、Gaps in final blocks:no;Phy ML参数设置为Model: Default、Statistical test: alrt、Number of categories: 4、Gamma: estimated、Invariable sites: estimated; Tree Dyn参数设置为Conformation: rec- tangular、Branch annotation: bootstra。
1.4 多序列比对
用Clustal Omega (https://www.ebi.ac.uk/Tools/ msa/clustalo/)和Mview (https://www.ebi.ac.uk/Tools/ msa/mview/)多重比对工具进行保守残基可视化和结构域的显示。参数设置为Output Format: Mview、Html Markup: Head、Alignment Width: 120、Con- sensus: OFF、Congaps: On。
1.5 蛋白质和基因结构域的搜索及绘制
用SMART和PFAM进行结构域搜索,为保证氨基酸序列的准确性,去除在SMART中结构域残缺的氨基酸序列,确定所需每个结构域的具体位置, 并使用IBS 1.0 (Illustrator for Biological Sequences)进行可视化绘图。SMART具体参数设置为Outlier homologues and homologues of known structure、PFAM domains、signal peptides、internal repeats。
1.6 三维结构预测
用SWISS-MODEL (https://swissmodel.expasy. org/)对HaAGO蛋白进行三维结构同源性建模。首先通过SWISS-MODEL模板库进行BLAST和HHBlits模板搜索,其次根据检索结果筛选QMEAN (-4~0)和GMQE (0~1)最大值的模板,并在SAVES v 6.0 (http:// saves.mbi.ucla.edu)中进行鉴定,最后通过PyMOL (The PyMOL Molecular Graphics System)软件对预测的三维结构模型进行可视化。
1.7 植物亚细胞定位预测
利用PSI (http://bis.zju.edu.cn/psi/)在线工具对HaAGOs和HaDCLs蛋白进行亚细胞定位,设置- values<0.01。
1.8 组织特异性表达分析
将和基因的ID号输入eFP- Browser数据库(http://bar.utoronto.ca/)中查询,分析其相对表达模式,参数设置为:Use Local Max。
2 结果和分析
2.1 向日葵AGO和DCL基因的筛选及其编码蛋白的理化性质分析
利用拟南芥中的10个和4个基因进行同源比对分析,最终确定向日葵中有15个和5个基因家族成员(表1)。HaAGOs蛋白由869~1 387氨基酸组成,最多的是HaAGO1b, 最少的是HaAGO4b;等电点为8.98~ 9.57,均为碱性;分子量为97.77~155.88 kDa;的外显子和内含子数量均比较少,只有2~4个,而其他s成员的外显子数量均较多。HaAGOs蛋白的亲水性值均为负值,表明均为亲水蛋白,最小值是-0.924, 为HaAGO1b, 亲水性最强; 除HaAGO1b外, HaAGOs蛋白的脂溶性指数都较大,平均73.00~83.97。HaDCLs有1 272~1 710氨基酸组成,最多的为HaDCL1, 最少为HaDCL3a;等电点为6.10~6.46,均为酸性; 分子量为142.26~191.99 kDa,脂溶性指数最大的是HaDCL2,亲水性值最大的也是HaDCL2,且HaDCLs亲水性值均为负值,预测均为亲水蛋白。
本研究分析了和基因的染色体定位,结果表明,在14号染色体上有5个基因,分别是和;在16号染色体上有3个,分别是和;其他染色体上分布的较少,大多为1~2个。2、4、7、8、9、15号染色体上无和基因分布,表明向日葵和基因在染色体上分布不均匀。
2.2 HaAGOs和HaDCLs的系统进化分析
基于拟南芥和向日葵和基因编码蛋白的氨基酸序列,采用最大似然法构建系统进化树。从图1: A可见,25个AGO家族成员可分为3类,第I类分支中有6个成员,其中3个HaAGOs家族成员,分别是HaAGO2a、HaAGO2b和HaAGO7, 编码该类成员的基因外显子数目较少(表1); 第II类分支有9个成员,其中6个HaAGOs家族成员,分别是HaAGO1a、HaAGO1b、HaAGO1c、HaAGO5a、HaAGO5b和HaAGO10; 第III类分支有10个成员,其中6个HaAGOs家族成员,分别是HaAGO4a、HaAGO4b、HaAGO8a、HaAGO8b、HaAGO8c和HaAGO8d。相比第I类分支,编码第II和第III类成员的基因外显子数目明显较多(表1)。

表1 向日葵AGO和DCL基因家族成员信息
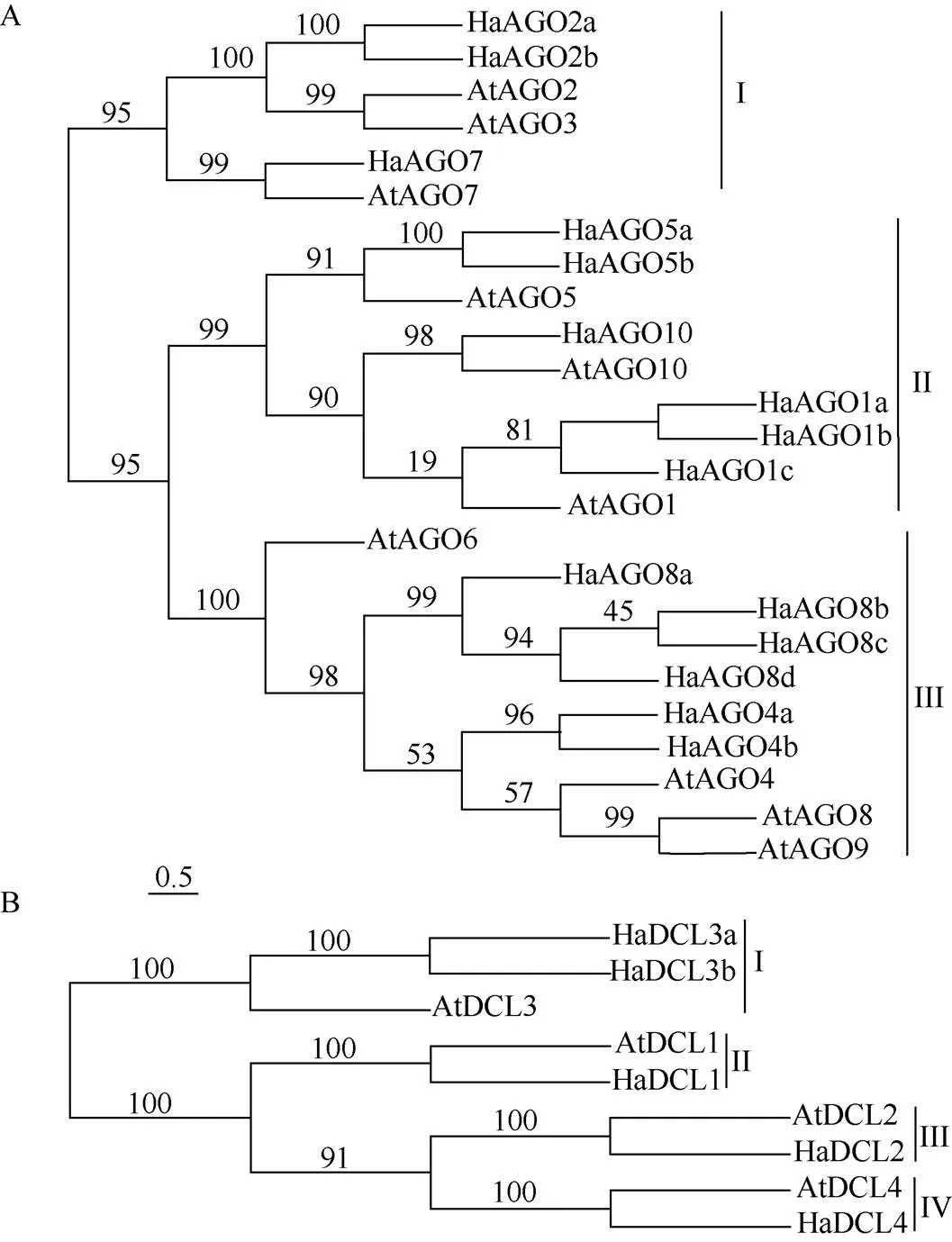
图1 向日葵和拟南芥AGO (A)和DCL (B)家族蛋白的系统进化树。分支上的数字为1 000次复制的支持值。
从图1: B可见,5个HaDCL蛋白可分为4个分支, AtDCL1与HaDCL1、AtDCL2与HaDCL2、AtDCL3与HaDCL3a和HaDCL3b、AtDCL4与HaDCL4分别聚在同一分支中。
2.3 HaAGOs和HaDCLs蛋白的保守结构域预测
本研究使用SMART进行了蛋白结构域搜索,结果表明,15个HaAGOs蛋白都包含PAZ和PIWI结构域(图2: A)。位于进化树第I分支的HaAGO2a、HaAGO2b、HaAGO7和第III分支的HaAGO8a、HaAGO8B没有预测到MID结构域。位于进化树第II分支的HaAGO1a、HaAGO1b、HaAGO1c、HaAGO5a、HaAGO10和第III分支的HaAGO4a、HaAGO4b、HaAGO8c、HaAGO8d均有保守结构域N domain、DUF1785、PAZ、L2、MID和PIWI。在HaAGO1a中有1个富含甘氨酸(A Gly-Rich)的特异性结构域。
与拟南芥的DCL相似,HaDCLs家族也包含DEXDc、HELICc、Dicer_dimer、PAZ、RIBOc和DSRM等保守结构域(图2: B)。位于进化树第I分支的HaDCL3b没有预测到DEXDc结构域, HaDCL3a和HaDCL3b没有预测到HELICc、Dicer_dimer和DSRM结构域。位于进化树第II分支的HaDCL1和第IV分支的HaDCL4没有预测到上述结构域。位于第III分支的HaDCL2没有预测到DSRM结构域。而HaDCL1和HaDCL4在N末端有1个额外的DSRM结构域。

图2 HaAGOs (A)和HaDCLs (B)蛋白的保守结构域
2.4 HaAGOs蛋白PIWI结构域的多重序列比对
15个HaAGOs中有8个(第I分支的HaAGO2a、HaAGO2b、HaAGO7和第II分支的HaAGO1a、HaAGO1c、HaAGO5a、HaAGO5b、HaAGO10)具有保守的H798位点,而位于第II分支的HaAGO1b的H798位点则被P (脯氨酸)替换。位于第III分支的HaAGO4a和HaAGO4b的H798位点也被P替换,HaAGO8a和HaAGO8b的H798位点被S (丝氨酸)替换,HaAGO8c和HaAGO8d的H798位点被A (丙氨酸)替换,预示这些被替换的蛋白酶活性功能可能丧失。除了第I分支的HaAGO2a和HaAGO2b有DEDD基序外,其余HaAGOs蛋白均为DEDH基序(图3)。
2.5 HaAGOs的三级结构预测
结合系统发育关系和蛋白质结构域可以看出,向日葵和拟南芥的AGO蛋白三维模型总体上都是带有底部凹槽的新月形结构,但各自又有所不同(图4)。第I分支的HaAGO2a、HaAGO2b和HaAGO7 和第II分支都没有预测到MID结构域(红色区域),而第III分支仅部分成员有MID结构域。
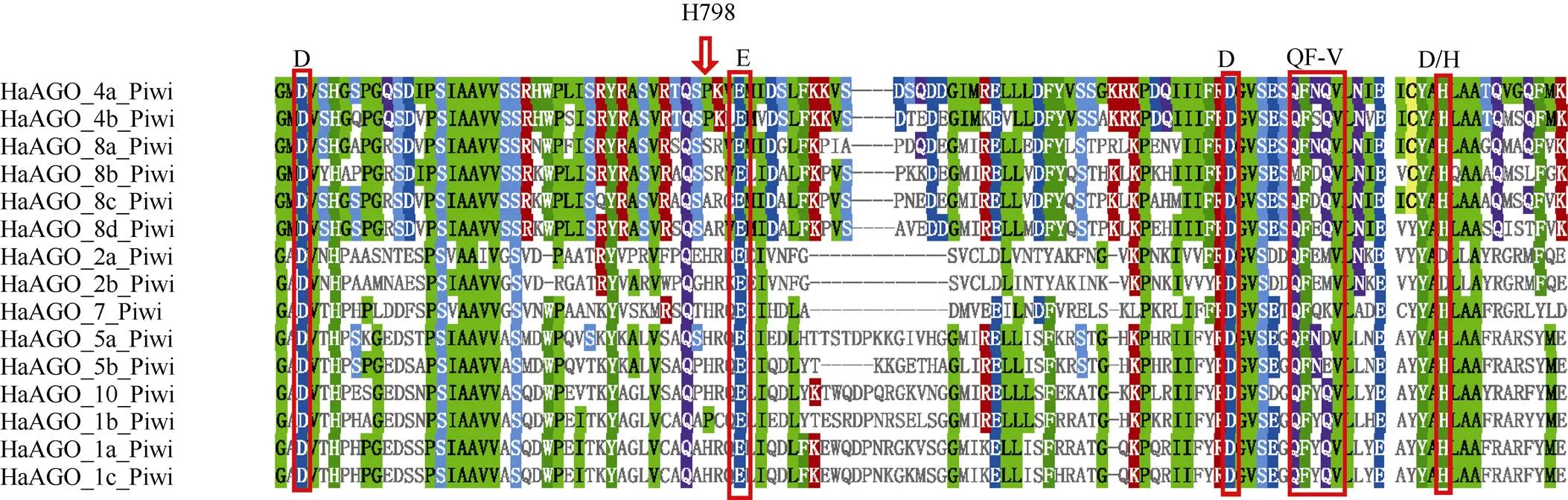
图3 向日葵AGO蛋白PIWI结构域的多重序列比对。红色框: DEDD/H和QF-F基序。
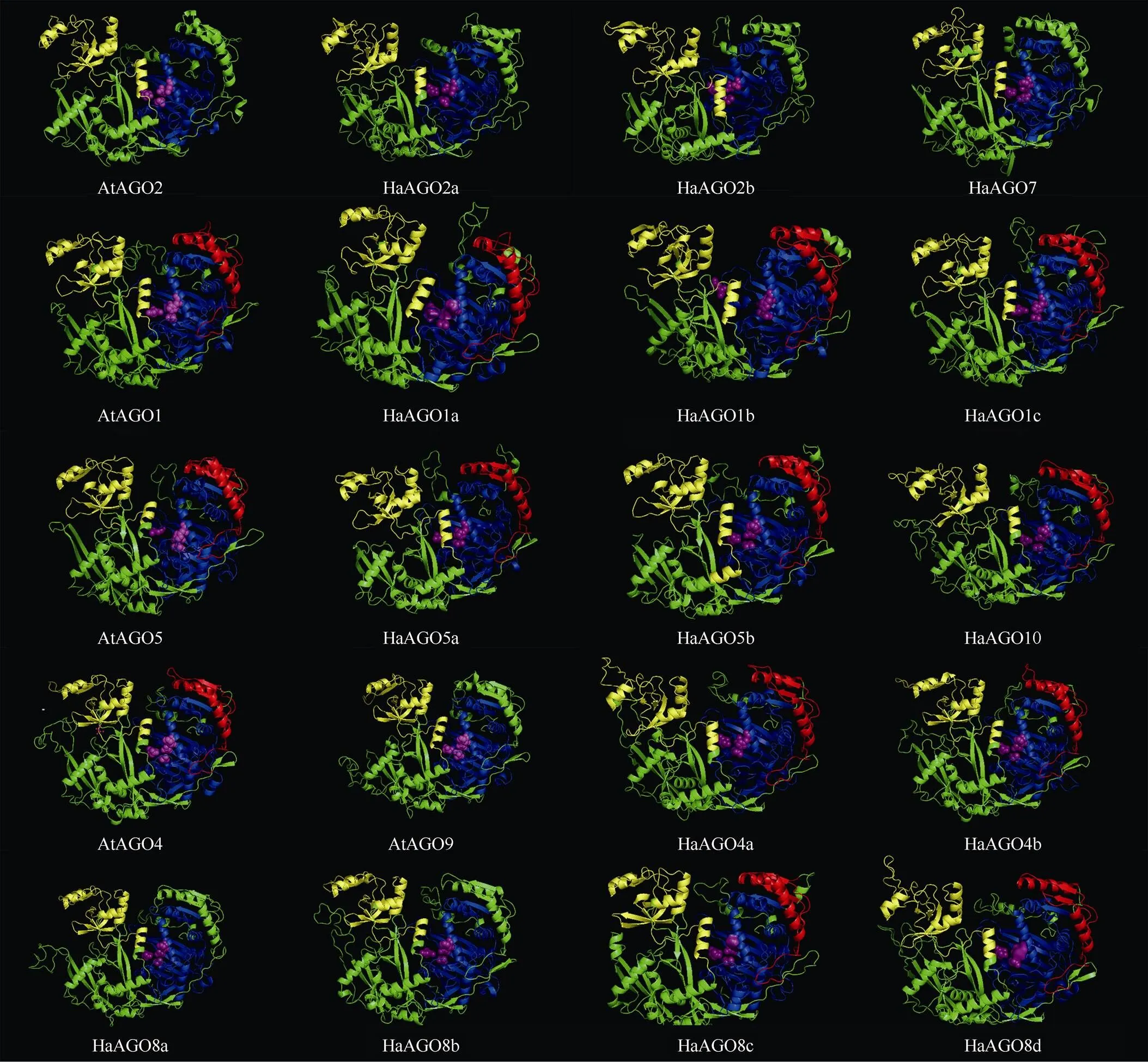
图4 HaAGOs蛋白的三级结构。黄色: PAZ; 蓝色: PIWI; 红色: MID; 紫红色: DEDD/H。
2.6 HaAGOs和HaDCLs的表达模式和亚细胞定位
利用eFP-Brower对和在花序、茎、叶和种子4种组织中的特异性表达进行了分析,结果表明(表2),和在不同组织中的表达差异明显。其中和在花序中高表达,和分别在种子和叶中高表达。值得注意的是,和在4个组织中均不表达。5个基因在各组织中均有不同程度的表达,和在茎和花序中高表达。
亚细胞定位分析结果表明(表3),HaAGO1b、HaAGO2a和HaAGO8c定位于细胞核,HaAGO1c和HaAGO2b定位于细胞核和细胞质,HaAGO7定位于细胞核和线粒体,HaAGO1a、HaAGO4a、HaAGO4b和HaAGO10定位于线粒体,HaAGO8a定位于高尔基体和质体。

表2 向日葵AGO和DCL家族的组织特异性表达
3 结论和讨论
RNAi是真核生物调节基因表达的一种保守机制, 在植物生长发育、表观遗传修饰、对非生物和生物胁迫的反应和防御等多个过程中发挥着重要作用[12–14]。由AGO和DCL介导的RNAi是一个重要的生理调控过程,二者协同作用,参与了不同种类小RNA的产生、靶基因的沉默和生物途径的调节。在拟南芥中,和分别在抗病毒防御和表观遗传途径中发挥重要作用,并且这两个基因编码的氨基酸序列表现出高度的同源性[15], AtAGOs蛋白还包括介导mRNA裂解的AtAGO1、指导DNA甲基化的AtAGO4、调控植株发育的AtAGO7等[16]。拟南芥AtDCL1主要参与miRNA的生物生成[17],AtDCL2主要负责切割dsRNA从而产生22nt siRNA,对病毒起抵抗作用[18],AtDCL3参与RNA指导的DNA甲基化和染色体修饰过程, AtDCL4与ta-siRNA的产生有关,并在转录后水平的基因沉默发挥作用[19]。但是在向日葵中RNAi途径的关键基因尚未见报道。
本研究共挖掘出向日葵15个和5个家族成员,分析并注释了这些成员的氨基酸数量、分子量及基因登录号等,并通过对20个蛋白的理化性质分析以及染色体定位预测,以确定不同基因间的进化关系。与拟南芥类似,系统发育树分析表明基因包含3个分支, 并且基因的内、外显子数量、大小在不同分支内保持较高的一致性, 这些与拟南芥中基因家族的结构和特征一致,说明他们的基因起源甚至功能有一定的关联性。每个分支都有来自向日葵和拟南芥的基因,推断向日葵的祖先种类含有至少3个基因,也说明向日葵在进化上相对保守。有趣的是,向日葵家族成员数量高于拟南芥,第Ⅲ分支且位于14号染色体上的,以及第I分支且位于16号染色体的出现在同一基因区间,且不超过1个干预基因,推测基因的串联复制导致了向日葵基因的多样性。
AGO和DCL蛋白成员结构域存在差异, HaAGO家族包含7种结构域,15个HaAGOs都有PAZ和PIWI结构域,有研究表明PAZ有助于siRNA 3′末端的结合,而PIWI则将siRNA 5′末端与靶RNA结合[20],暗示所有的HaAGO具有与小RNA结合的功能。在HaAGO1a中有1个富含甘氨酸(Gly-rich)的特异性结构域,这与拟南芥和咖啡等植物中AGO1富含甘氨酸(Gly-rich)的结构域类似,具体功能未知,有待进一步研究。PIWI会形成一种类似于RNase H酶的折叠结构,具有内源核酸酶的活性[21],可将siRNA的5′末端与靶RNA结合,是核心AGO蛋白家族最主要的功能域。H798 (组氨酸)位点、QF-V (谷氨酰胺-苯丙氨酸-缬氨酸)和DEDD/H基序则是PIWI功能域的核心元件,有研究表明H798是AtAGO1蛋白行使体外内切酶活性的重要位点[22],QF-V是sRNA双重识别和分类中所必需的高度保守的基序[23],DEDD/H是PIWI行使切割功能不可或缺的保守基序[24]。本研究中的PIWI大部分都包含了上述主要的功能元件,也有部分残基被取代, 说明了向日葵AGO在功能上与拟南芥存在一定的差异。三维建模是解析蛋白功能的有效方式,用SWISS-MODEL对向日葵AGO蛋白的氨基酸序列进行蛋白质三维结构同源性建模[25]。SWISS-MODEL是迄今为止AGO蛋白的首选建模工具[26],并通过PyMOL分子进行可视化显示。HaAGO家族三级结构模型更明显的显示向日葵与拟南芥AGO结构相似但有细微差别。HaDCLs家族成员都含有PAZ、RIBOc结构域,氨基酸数量较多的HaDCL1和HaDCL4包含有DEXDc、HELICc、Dicer_dimer、PAZ、RIBOc和DSRM等6种结构域;而HaDCL3a和HaDCL3b没有预测到Dicer_dimer结构域, 有研究表明AtDCL3也没有预测到Dicer_dimer结构域,而拟南芥中Dicer_dimer结构域参与介导AtDCL4的异源二聚作用[27],因此没有预测到Dicer_dimer可能会导致HaDCL3a和HaDCL3b相应功能的丧失。综上所述,与拟南芥相比,向日葵在进化的过程中既有保守性又有自身的特点。
和组织特异性表达分析表明,在花序和种子中表达量最高,而在拟南芥的花和种子的各发育阶段,也有较高的表达[28],这可能是由于积极参与植物分生组织活动的结果。和基因在花序和茎中的表达量比其他组织高,推测其在花序和茎的生长发育中发挥重要的调节作用。
本研究对向日葵的AGO和DCL家族进行了挖掘和鉴定,共发掘出15个和5个家族成员,并对其理化性质、染色体定位、系统进化树、蛋白保守结构域、三级结构、亚细胞定位及组织特异性表达等进行了分析,结果表明向日葵中可能存在典型的RNAi干扰机制,向日葵和基因其功能在进化上是保守的,但有其自己的特殊性,并且在花序和茎的生长发育中发挥着关键作用,但具体的功能还需要更多试验数据去佐证,以便全面解析向日葵RNAi途径及其介导的沉默机制。这为揭示向日葵AGO和DCL蛋白功能、发掘向日葵的抗逆育种靶向基因资源提供了一定的理论依据。
[1] ZHANG S W, DENG X, WANG L, et al. Identification and analysis of RNA interference-related enzyme gene families in[J]. Pratac Sci, 2021, 38(7): 1380–1392. [张司雯, 邓欣, 王龙, 等. 谷子RNA干扰相关酶类基因家族的鉴定与分析 [J]. 草业科学, 2021, 38 (7): 1380–1392. doi: 10.11829/j.issn.1001-0629.2021-0024.]
[2] VALENCIA-SANCHEZ M A, LIU J D, HANNON G J, et al. Control of translation and mRNA degradation by miRNAs and siRNAs [J]. Genes Dev, 2006, 20(5): 515–524. doi: 10.1101/gad.1399806.
[3] AXTELL M J. Classification and comparison of small RNAs from plants [J]. Annu Rev Plant Biol, 2013, 64: 137–159. doi: 10.1146/ annurev-arplant-050312-120043.
[4] TOLIA N H, JOSHUA-TOR L. Slicer and the argonautes [J]. Nat Chem Biol, 2007, 3(1): 36–43. doi: 10.1038/nchembio848.
[5] GHILDIYAL M, ZAMORE P D. Small silencing RNAs: An expanding universe [J]. Nat Rev Genet, 2009, 10(2): 94–108. doi: 10. 1038/nrg2504.
[6] MARGIS R, FUSARO A F, SMITH N A, et al. The evolution and diversification of dicers in plants [J]. FEBS Lett, 2006, 580(10): 2442–2450. doi: 10.1016/j.febslet.2006.03.072.
[7] XIANG Y, XIAN Z Q, LIN D B, et al. Cloning and expression analysis ofin tomato [J]. Chin J Trop Crops, 2013, 34(4): 641–647. [向娅, 先志强, 林冬波, 等. 番茄基因的克隆及表达分析 [J]. 热带作物学报, 2013, 34(4): 641–647. doi: 10.3969/j. issn.1000-2561.2013.04.011.]
[8] BOHMERT K, CAMUS I, BELLINI C, et al.defines a novel locus ofcontrolling leaf development [J]. EMBO J, 1998, 17(1): 170–180. doi: 10.1093/emboj/17.1.170.
[9] CAI J Y, WU X Y, GUO Y S, et al. Bioinformatics analysis of argonaute proteins in genome of tobacco [J]. Acta Agric Jiangxi, 2017, 29(2): 1–8. [蔡健宇, 武晓云, 郭玉双, 等. 烟草基因组中AGO蛋白的生物信息学分析 [J]. 江西农业学报, 2017, 29(2): 1–8. doi: 10. 19386/j.cnki.jxnyxb.2017.02.01.]
[10] LIU Q P, FENG Y, ZHU Z J. Dicer-like (DCL) proteins in plants [J]. Funct Integr Genom, 2009, 9(3): 277–286. doi: 10.1007/s10142-009- 0111-5.
[11] SUN R F, ZHANG Y F, NIE L Z, et al. Gene cloning offrom sunflower () and its responses to biotic and abiotic stress [J]. J Agric Biotechnol, 2021, 29(5): 900–914. [孙瑞芬, 张艳芳, 聂利珍, 等. 向日葵基因克隆及其对生物和非生物胁迫的响应 [J]. 农业生物技术学报, 2021, 29(5): 900–914. doi: 10.3969/j.issn.1674-7968.2021.05.007.]
[12] ZHANG Z H, LIU X Y, GUO X W, et al.AGO3 predo- minantly recruits 24-nt small RNAs to regulate epigenetic silencing [J]. Nat Plants, 2016, 2(5): 16049. doi: 10.1038/nplants.2016.49.
[13] MALLORY A C, ELMAYAN T, VAUCHERET H. MicroRNA maturation and action-the expanding roles of ARGONAUTEs [J]. Curr Opin Plant Biol, 2008, 11(5): 560–566. doi: 10.1016/j.pbi.2008.06. 008.
[14] MALLORY A, VAUCHERET H. Form, function, and regulation of ARGONAUTE proteins [J]. Plant Cell, 2010, 22(12): 3879–3889. doi: 10.1105/tpc.110.080671.
[15] YE R Q, WANG W, IKI T, et al. Cytoplasmic assembly and selective nuclear import ofARGONAUTE4/siRNA complexes [J]. Mol Cell, 2012, 46(6): 859–870. doi: 10.1016/j.molcel.2012.04.013.
[16] NAGANO H, FUKUDOME A, HIRAGURI A, et al. Distinct substrate specificities ofDCL3 and DCL4 [J]. Nucl Acids Res, 2014, 42(3): 1845–1856. doi: 10.1093/nar/gkt1077.
[17] XIA E H, TONG W, WU Q, et al. Tea plant genomics: Achievements, challenges and perspectives [J]. Hort Res, 2020, 7: 7. doi: 10.1038/s 41438-019-0225-4.
[18] MOISSIARD G, PARIZOTTO E A, HIMBER C, et al. Transitivity incan be primed, requires the redundant action of the antiviral Dicer-like 4 and Dicer-like 2, and is compromised by viral- encoded suppressor proteins [J]. RNA, 2007, 13(8): 1268–1278. doi: 10.1261/rna.541307.
[19] PIKAARD C S. Cell biology of thenuclear siRNA pathway for RNA-directed chromatin modification [J]. Cold Spring Harb Symp Quant Biol, 2006, 71: 473–480. doi: 10.1101/sqb.2006. 71.046.
[20] RIVAS F V, TOLIA N H, SONG J J, et al. Purified argonaute2 and a siRNA form recombinant human RISC [J]. Nat Struct Mol Biol, 2005, 12(4): 340-349. doi:10.1038/nsmb918.
[21] YUAN Y R, PEI Y, MA J B, et al. Crystal structure ofargonaute, a site-specific DNA-guided endoribonuclease, provides insights into RISC-mediated mRNA cleavage [J]. Mol Cell, 2005, 19 (3): 405–419. doi: 10.1016/j.molcel.2005.07.011.
[22] BAUMBERGER N, BAULCOMBE D C.ARGONAUTE1 is an RNA slicer that selectively recruits microRNAs and short inter- fering RNAs [J]. Proc Natl Acad Sci USA, 2005, 102(33): 11928– 11933. doi: 10.1073/pnas.0505461102.
[23] ZHANG X M, NIU D D, CARBONELL A, et al. ARGONAUTE PIWI domain and microRNA duplex structure regulate small RNA sorting in[J]. Nat Commun, 2014, 5: 5468. doi: 10.1038/ ncomms6468.
[24] XU D D. Cloning and functional identification of maizegene [D]. Beijing: Chinese Academy of Agricultural Sciences, 2014. [徐东东. 玉米基因分离与功能鉴定 [D]. 北京: 中国农业科学院, 2014.]
[25] SCHWEDE T, KOPP J, GUEX N, et al. SWISS-MODEL: An automated protein homology-modeling server [J]. Nucl Acid Res, 2003, 31(13): 3381–3385. doi: 10.1093/nar/gkg520.
[26] ŠEČIĆ E, ZANINI S, KOGEL K H. Further elucidation of the argonaute and dicer protein families in the model grass species[J]. Front Plant Sci, 2019, 10: 1332. doi: 10. 3389/fpls.2019.01332.
[27] QIN H N, CHEN F D, HUAN X L, et al. Structure of theDCL4 DUF283 domain reveals a noncanonical double- stranded RNA-binding fold for protein-protein interaction [J]. RNA, 2010, 16(3): 474–481. doi: 10.1261/rna.1965310.
[28] YUE L M, SONG J B, XU X F, et al. Bioinformatical and experi- mental analysis ofgenes in response to salt stress [J]. J Shenzhen Univ (Sci Eng), 2017, 34(4): 352–357. [岳路明, 宋剑波, 徐晓峰, 等.拟南芥基因家族分析及盐胁迫下的表达验证 [J]. 深圳大学学报(理工版), 2017, 34(4): 352–357. doi: 10.3724/SP.J.1249.2017. 04352.]
Identification and Expression Analysis ofandGene Families in Sunflower
ZHANG Hong1, YUAN Shiyu1, WANG Wenhao1, LIU Wenjun1, HE Lifen1, YAN Yuxing1, WANG Yanzun1, WANG Pengdong1, ZHENG Hongyuan1*, ZHANG Xin1,2*
(1. Institute of Industrial Crops, Shanxi Agricultural University,Taiyuan 030031, China; 2.State Key Laboratory of Sustainable Dryland Agriculture (in preparation), Taiyuan 030031, China)
Ribonucleic acid interference (RNAi) is a biological process in which small RNAs regulate gene silencing at the transcriptional or post-transcriptional level. The argonaute (AGO) and dicer-like (DCL) proteins are two of the key components in the RNAi machinery of eukaryotes. In order to understand the function of AGO and DCL in sunflower (), the sequence ofandinwere used for homology alignment in the sunflower database. The bioinformatics of AGO and DCL family members in sunflower were analysed. The results showed that there were 15and 5members obtained, which were unevenly distributed on chromosomes. Themembers could be cluster into 3 clades, containing conserved domains of N domain, DUF1785, PAZ and PIWI, whereas those ofcould be cluster into 4 clades, containing conserved domains of PAZ and RIBOc. The expressions ofandin the steam and peduncle were stronger than those in other tissues. The subcellular localization showed that HaAGO was mostly localized in the nucleus. Therefore, it was suggested that a typical RNAi mechanism might exist in sunflower, and involve in coordinating growth and development in sunflower.
RNAi; AGO; DCL;; Gene expression
10.11926/jtsb.4612
2022-01-17
2022-05-02
山西省重点研发计划项目(201903D221088);国家特色油料产业技术体系项目(CARS-14-2-05);山西农业大学生物育种工程项目(YZGC047);山西省现代农业产业技术体系建设专项资金(2022-05)资助
This work was supported by the Project for Key Research and Development in Shanxi (Grant No. 201903D221088), the Project of National Characteristic Oil Industry Technology System (Grant No. CARS-14-2-05), the Project for Biological Breeding Engineering in Shanxi Agricultural University (Grant No. YZGC047), and the special Project for Modern Agricultural Industry Technology Research System in Shanxi (Grant No. 2022-05).
张红(1984年生),女,助理研究员,主要从事向日葵栽培与育种。E-mail: foogoohoo@126.com
. E-mail: 18935439365@163.com; 15177178@qq.com
