加工和消化后蘑菇DNA提取与分析
2023-05-31程兢业吴鸿亿赵力王婧
程兢业 吴鸿亿 赵力 王婧

摘 要:蘑菇进入食用和消化环节后难以辨认,本试验探究加工与消化对蘑菇DNA提取的影响,改良DNA提取方法,为毒蘑菇中毒物种鉴别奠定基础。结果表明,改良CTAB法将OD260/OD280值提升至1.97~2.00,浓度由559.00~1 131.68 μg·mL-1提升至877.33~1 219.33 μg·mL-1;对油脂含量高的样品进行除油处理,除油后DNA浓度达1 339.33 μg·mL-1;改良CTAB法可提取所有样品DNA,且提取的DNA较为完整;改良CTAB法与试剂盒法相比在DNA提取过程中可操作性较高,提取成本低,提取的DNA浓度高,平均浓度达到1 291.05 μg·mL-1,OD值较为稳定,均在1.8~2.0。
关键词:蘑菇;加工;消化;DNA提取
Abstract: This experiment investigates the effect of processing and digestion on the extraction of mushroom DNA to improve the DNA extraction method and to lay the foundation for the identification of poisonous mushroom species. The results showed that the modified CTAB method increased the OD260/OD280 values to 1.97~2.00 and concentrations from 559.00~1 131.68 μg·mL-1 to 877.33~1 219.33 μg·mL-1. The samples with high oil content were de-oiled and the DNA concentration reached 1 339.33 μg·mL-1 after de-oiling. The modified CTAB method was more operable and less costly than the kit method in the DNA extraction process, and the extracted DNA concentration was higher, with an average concentration of 1 291.05 μg·mL-1, and the OD values were more stable, ranging from 1.8~2.0.
Keywords: mushrooms; processing; digesting; DNA extraction
在我國,误食毒蘑菇而引发的中毒事件时有发生,毒蘑菇在进入食用和消化环节后难以用传统形态学方法辨认,近年来DNA分子标记技术在真菌物种鉴别中发展迅速,而高质量基因组DNA提取为其奠定了基础。目前国内外已有一些关于蘑菇基因组DNA提取方法的报道,但由于蘑菇组织中含有大量的多糖、蛋白质和色素等物质,以及加工和消化过程中DNA有所降解,导致后续相关分子实验受到影响[1-4]。因此,笔者在前人的研究基础上,采用试剂盒法和CTAB法提取加工和消化后的蘑菇DNA,且对经典的CTAB法进行改良,利用琼脂糖凝胶电泳、紫外分光光度和PCR扩增检测DNA质量,对提取方法、加工方式及消化对DNA的影响进行初步的研究,同时也为毒蘑菇DNA的提取提供参考。
1 材料和方法
1.1 试验材料
食用菌杏鲍菇、香菇购于当地超市。
CTAB缓冲液(2% CTAB、100 mmol·L-1 Tris-HCl、25 mmol·L-1 EDTA、1.5 mol·L-1 NaCl);2-巯基乙醇;蛋白酶K(20 mg·mL-1);RNase A;酚∶氯仿∶异戊醇(25∶24∶1);氯仿∶异戊醇(24∶1);胃蛋白酶;甲醇∶氯仿∶水(1.0∶2.0∶0.8)。
1.2 试验方法
1.2.1 加工方式
加工方式采用炒、水煮、烘干和冻干的方法。①炒:平底锅烧热后倒入食用油将蘑菇炒至熟透。②水煮:水沸腾后放入蘑菇,分别在水中煮至5 min、10 min、15 min、30 min、45 min和60 min。③烘干:将蘑菇切成薄片,60 ℃烘箱烘干6~7 h备用。④冻干:将蘑菇切成薄片于-80 ℃冰箱放置一晚后真空冷冻干燥48 h,取出密封备用。以上样品均分为两份,一份消化实验备用,一份作为对照。
1.2.2 消化试验
人工胃液配制及消化处理参考GAUSTERER等的试验[5]。
1.2.3 DNA提取
本研究参照原有CTAB法[6-7],对其进行改良,提高DNA质量和提取成功率。改良后CTAB法操作步骤如下。称取1 g蘑菇组织研磨后放入10 mL灭菌离心管中,加入5 mL CTAB缓冲液,500 ?L 2-巯基乙醇,80 ?L蛋白酶K,置于65 ℃的恒温水浴锅中消化1 h。取上清液至新的离心管中,加入等体积的酚∶氯仿∶异戊醇(25∶24∶1)溶液,振荡至乳白色,12 000 r·min-1离心10 min。加入1% RNase A溶液,37 ℃水浴30 min。加入等体积的氯仿/异戊醇(24∶1),振荡至乳白色,12 000 r·min-1离心10 min。取上清液至新的离心管中,加入等体积预冷异丙醇,-20 ℃放置30 min后12 000 r·min-1离心10 min。弃上清液,加入800 ?L 70%乙醇洗涤两次,无水乙醇洗涤一次,弃去乙醇,室温晾干或超净工作台吹干,加入100 ?L灭菌双蒸水,-20 ℃下保存。
真菌基因组DNA提取试剂盒购自索莱宝生物技术有限公司,具体操作方法参照试剂盒说明书。
1.2.4 DNA質量分析
(1)紫外检测。取5 μL DNA样品,加入双蒸水或TE缓冲液稀释10倍,利用紫外分光光度计测量DNA浓度及OD260/OD280。
(2)PCR扩增。采用引物ITS4/ITS5和LROR/LR5,PCR反应体系总体积为20 μL,其中包含2 μL 10×PCR buffer,1.6 μL dNTP(2.5 mmol·L-1),1.2 μL Mg2+(25 mmol·L-1),0.4 μL Taq酶(2.5 U),上下引物各1 μL,DNA模板1 μL,ddH2O补足至20 μL。PCR扩增程序为94 ℃预变性5 min;94 ℃变性30 s,58 ℃退火40 s,72 ℃延伸1 min,27次循环;72 ℃延伸10 min。
(3)琼脂糖凝胶电泳检测。取DNA样品5 μL,6×Loading buffer 1 μL混匀,在1%的琼脂糖凝胶点样孔中上样,置于0.5×TAE缓冲液中,120 V电泳35 min,采用Gel-Pro analyzer软件拍照并保存。
2 结果与分析
2.1 紫外分析结果
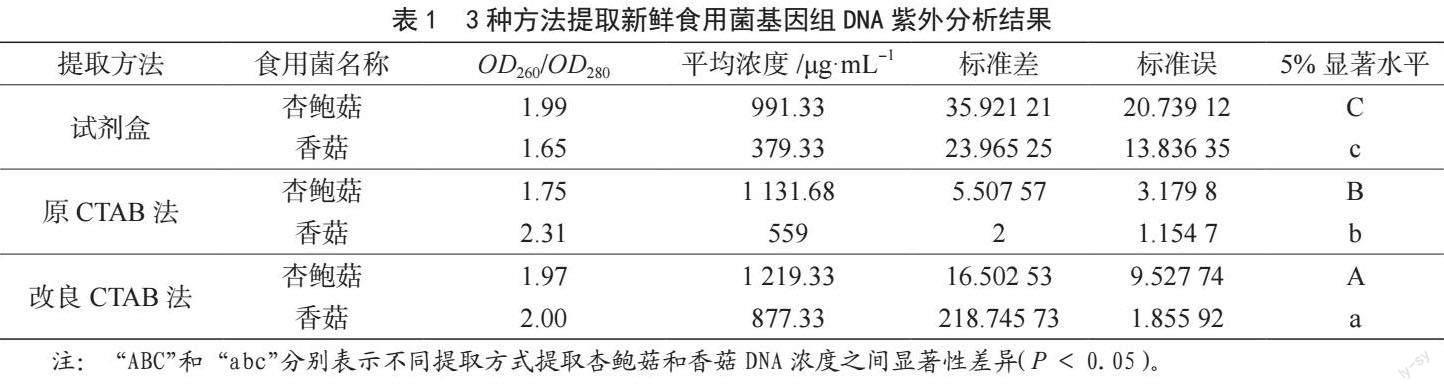
2.1.1 不同提取方法紫外分析结果
3种方法提取新鲜食用菌基因组DNA紫外分析结果见表1,试剂盒法、CTAB法、改良CTAB法均能提取杏鲍菇和香菇子实体中的DNA,3种提取方法存在显著性差异(P<0.05)。改良CTAB法的OD260/OD280值在1.8~2.0,说明蛋白质和酚类物质已去除干净,无RNA污染,得到的DNA纯度最好。CTAB法提取的杏鲍菇OD260/OD280值小于1.8,存在蛋白质和酚类物质,香菇OD260/OD280值大于2.0,存在RNA污染,且CTAB法在提取DNA过程中,出现较多黏稠胶状物质,上清液为黄褐色,抽提后DNA沉淀仍有色素残留。试剂盒法提取香菇DNA的OD260/OD280值小于1.8,说明受到蛋白质及酚类物质污染较严重。3种方法相比,改良CTAB法得到的DNA浓度较高,其次是CTAB法,试剂盒法得到的DNA浓度最低。
2.1.2 加工和胃消化样品紫外分析结果
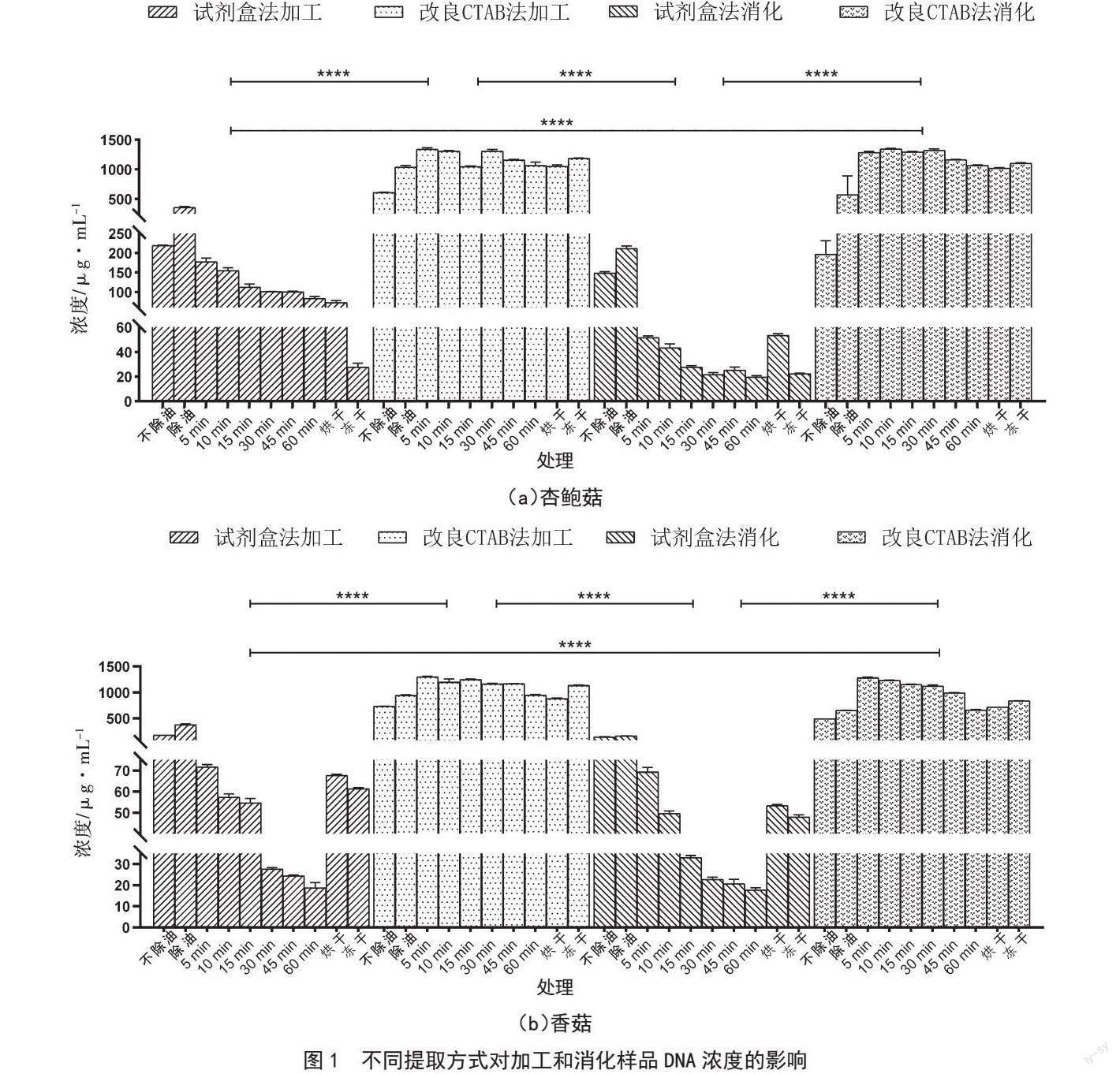
试剂盒和改良CTAB两种方法DNA紫外分析结果见表2和3,两种方法所提取的除油和未除油样品DNA浓度均存在显著差异性(P<0.05),未除油样品DNA的OD260/OD280值小于1.8,说明蛋白质、酚类污染严重,DNA溶液中杂质较多。除油后样品DNA质量有所提高,OD260/OD280值均在1.8~2.0,且DNA浓度高于除油前,浓度达1 339.33 μg·mL-1。因此,可在DNA提取前对炒制样品对进行预处理以消除样品中油脂对DNA的影响。改良CTAB法提取杏鲍菇水煮5 min、10 min和15 min之间,45 min和60 min之间DNA浓度变化无显著性差异,消化组样品在水煮5~30 min时DNA浓度变化无显著性差异。
试剂盒法提取的水煮样品中,杏鲍菇水煮15~45 min时DNA浓度无明显变化,其余水煮组内样品浓度均存在差异,浓度由51.67~177.67 μg·mL-1逐渐降低至19.67~83.33 μg·mL-1。随着水煮时间的延长,水煮样品DNA浓度整体呈现下降趋势。改良CTAB法提取的干制样品DNA存在显著性差异(P<0.05),烘干样品浓度均大于冻干样品浓度,两种提取方法均表明消化后干制样品DNA浓度明显低于消化前。
试剂盒法和改良CTAB法提取效果比较见图1,两种DNA提取方法之间存在显著性差异(P<0.05),改良CTAB法所提取DNA浓度均高于试剂盒法,平均浓度达到1 291.05 μg·mL-1,DNA质量较高,
OD值均在1.8~2.0。因此所建立的DNA提取方法用于消化和加工等复杂样品DNA提取,且可应用于毒蘑菇中毒案例中,提取蘑菇加工样品及胃消化残留物等,对毒蘑菇的中毒与防治具有重大意义。
2.2 电泳分析结果
采用试剂盒法提取加工和消化样品DNA电泳结果见图2,新鲜样品DNA作为对照。DNA易溶于水,随着高温与水煮时间的延长,很难从水煮样品中提取完整DNA片段。所有加工样品成功扩增出ITS片段,消化后水煮杏鲍菇45 min、60 min和炒制杏鲍菇未能扩增出ITS片段,消化后杏鲍菇水煮60 min和未除油未能扩增出LSU片段。水煮杏鲍菇45 min、水煮香菇30~60 min和消化后炒制香菇LSU条带较暗,香菇未除油ITS条带较暗,说明DNA提取成功但含量较低。对炒制样品进行除油处理后,存在弥散片段,干制样品基因组DNA片段完整主带清晰,烘干香菇和消化后冻干香菇未能扩增出LSU片段。所有消化样品条带均变暗,说明与新鲜和加工样品相比,消化样品DNA有所降解,DNA得率较低。引物ITS扩增成功率为86.5%,引物LSU扩增成功率为84.6%,两种引物结合使用可将扩增成功率提高至88.5%。
新鲜食用菌DNA作为对照,改良CTAB法提取加工和消化样品DNA电泳结果见图2,水煮5~10 min样品基因组DNA条带清晰,消化后水煮样品无完整条带,所有加工样品成功扩增出ITS和LSU片段,水煮香菇30 min的LSU条带和水煮香菇15 min的ITS条带较暗,消化后的水煮香菇5~10 min扩增出ITS非特异性片段,片段大小在250 bp左右,消化后水煮杏鲍菇15 min样品LSU条带较暗,其余消化后样品均成功扩增出ITS片段。炒制样品未进行除油处理时基因组DNA无明显条带,进行除油处理后可见其主带清晰完整,且无拖尾现象。干制样品基因组DNA主带不完整,存在大量弥散DNA片段条带。改良CTAB法提取样品DNA引物ITS扩增成功率为86.5%,LSU扩增成功率为82.7%,两种引物结合使用可将扩增成功率提高至100%,因此可将ITS和LUS两种引物联合使用以保证PCR扩增成功率。
3 结论与讨论
由于进入加工和消化阶段的蘑菇很难通过形态学鉴定其物种,对于误食毒蘑菇中毒案例,针对性治疗便难以进行,因此本试验通过研究加工条件和DNA提取方法,提高DNA提取成功率,为后续DNA分子鉴别实验奠定基础。食用菌中含有较多酚类、多糖及色素等物质,这些物质会形成胶黏状物并裹挟着DNA[8]。1995年Borroso.G.提出了CTAB法,但CTAB法最早应用于植物基因组DNA的提取,随后此方法开始应用于蘑菇DNA的提取。韩利刚等[9]在这种方法的基础上进行改良来提取丝状真菌的DNA且取得了很好的效果。同年王春晖等[10]将CTAB法加以改进,使此法更简单、易操作。江玉姬等[11]将CTAB法与其他方法加以比较,发现CTAB法是一种优良的蘑菇DNA提取方法,DNA产量高,多糖和蛋白质含量低,符合分子生物学实验要求。
本试验在对CTAB法进行改良时,采用加入蛋白酶K以去除蘑菇组织中的蛋白质,加入2-巯基乙醇以去除多酚类物质,加入RNase A以去除RNA。由于RNA酶会影响DNA质量,于是选择将RNase A溶液在使用酚/氯仿/异戊醇抽提液抽提一次后加入,在进行第二次抽提时可将RNA酶一起去除。油脂的存在对DNA质量存在一定的影响,于是在进行DNA提取前对炒制样品进行除油处理,所得DNA质量与除油前相比具有明显的提升。DNA为水溶性极强的物质,利用水煮的加工方式较难提取完整DNA片段。本试验设计水煮时间梯度探究水煮时间对DNA提取的影响,结果显示随着加工时间延长,所得样品DNA浓度逐渐减少,试剂盒法未能成功提取所有样品DNA,而改良CTAB法则成功提取所有样品DNA。引物ITS结合引物LSU便能将扩增成功率提高至100%,改良CTAB法能提取水煮5~10 min样品较为完整的DNA片段。综上所述,改良CTAB法适用于加工和消化样品高质量DNA的提取。
参考文献
[1]陈国明,魏海莲,李志强,等.平菇DNA不同提取方法比较[J].广西林业科学,2014,43(1):69-74.
[2]杨振华,张靠稳,马爱瑛.贺兰山紫蘑菇基因组DNA提取方法研究[J].安徽农业科学,2010,38(20):10557-10559.
[3]MAETA K,OCHI T,TOKIMOTO K,et al.Rapid species identification of cooked poisonous mushrooms by using real-time PCR[J].Appl Environ Microbiol,2008,74(10):3306-3309.
[4]WANG S,LIU Y,XU J.Comparison of different drying methods for recovery of mushroom DNA[J].Sci Rep,2017,7(1):3008.
[5]GAUSTERER C,PENKER M,KRISAI-GREILHUBER I,et al.Rapid genetic detection of ingested Amanita phalloides[J].Forensic Sci Int Genet,2014,9:66-71.
[6]李剛,李宝健.一种简单高效提取食用菌总DNA的方法[J].中山大学学报(自然科学版),2000(增刊3):56-58.
[7]张红,秦莲花,谭琦,等.用改进的CTAB法提取香菇基因组DNA[J].上海大学学报(自然科学版),2006(5):547-550.
[8]王艺琴,郭豪,张素勤,等.脆木耳基因组DNA提取方法优化[J].中国食用菌,2022,41(4):48-52.
[9]韩利刚,袁毅,王罡.丝状真菌组织DNA的提取[J].生物技术,1999(6):38-41.
[10]王春晖,刘文彬,邓洁姣.一种提取食用菌DNA的有效方法[J].食用菌,1999(1):9-10.
[11]江玉姬,谢宝贵,陈文校.草菇DNA提取方法初探[J].福建农业学报,2000(2):61-64.
