基于e-PCR的小麦、山羊草属SSR电子指纹图谱的开发
2023-05-30尹誉蓉王长彪任永康牛瑜琦唐朝晖
杨 晟,尹誉蓉,王长彪,任永康,牛瑜琦,韩 斌,唐朝晖
(1.山西农业大学农学院,山西晋中 030801; 2.山西农业大学生命科学学院,山西太原 030031)
品种鉴定工作是小麦生产环节中必不可少的关键步骤,同时也是保障优良品种优异遗传性状充分发挥、防止小麦品种混杂退化的重要举措[1]。山羊草属植物富含抗病虫害相关基因,通过远缘杂交技术可将山羊草属中的优异抗性基因转移至小麦属植物中,进而提高小麦的抗性[2]。因此,对于山羊草属植物的区分、鉴定工作会加强小麦远缘杂交育种工作的目的性和针对性。品种的真实性鉴定,究其根本是品种的基因型鉴定。DNA分子标记作为品种鉴定的有力工具,是基于不同品种DNA序列多态性,通过展示不同品种在基因水平上的差异,来实现品种的真实性鉴定[3]。
简单重复序列(simple sequence repeat,SSR)是基因组内以1~6个核苷酸为重复单元组成的串联重复序列[4-6],具有丰富的多态性。相较于其他标记,SSR标记还具有重复性高、覆盖面极广等优点[4-5, 7],在小麦品种标准DNA指纹图谱的构建方面具有巨大潜力[1]。朱岩芳[1]指出,利用分子标记进行作物品种鉴定首先应满足稳定性好、操作简单、鉴定快速和经济的要求。随着分子标记技术的不断改进,指纹图谱的构建工作进展快速。赵 宇等[8]筛选出13个在21份黄瓜材料中显示多态性的SSR标记,其中mtSSR4标记可对南水3号商品种子进行高精度的品种鉴定。为提高指纹图谱的直观性,梁 燕等[9]利用15个SSR分子标记构建了17份山东核桃种质的DNA指纹图谱,创新生成了品种分子二维码、条形码。张 颖等[10]从20个SSR分子标记中筛选出5个标记,构建了48份美洲南瓜种质的数字指纹图谱,并进一步转化为可直观展示等位基因缺失与否的指纹图谱。
电子PCR(electronic PCR , e-PCR)是一种常用的核苷酸序列电子分析工具,在SSR引物辅助设计方面发挥着重大作用[11]。传统的SSR引物开发十分繁琐,耗时、费力且伴有很大的随机性。熊登坤等[12]通过真实PCR验证,表明利用电子PCR可以提高SSR多态性引物的筛选效率。此外,前人采用e-PCR辅助设计野生花生[13]、玉米[14]、高丹草[15]基因组的SSR引物,发现有清晰扩增条带的SSR引物占比均高于80%,也表明e-PCR有助于提高SSR引物的筛选效率。
前人在DNA指纹图谱的构建方面进展飞速,但在鉴定的标准化方面进展缓慢,且同时着手小麦属、山羊草属植物的叶绿体和线粒体DNA指纹图谱的构建也未见有报道。本研究以小麦属、山羊草属共61个叶绿体和13个线粒体的全基因组序列为研究对象,基于e-PCR筛选多态性SSR标记,对部分引物进行真实PCR验证,并绘制电子指纹图谱,以期为分子标记鉴定标准化提供新思路。
1 材料与分析
1.1 SSR引物的搜集及SSR引物的设计
从美国国家生物技术信息中心(NCBI)的GenBank数据库(https://www.ncbi.nlm.nih.gov/nuccore)下载小麦属、山羊草属共61个叶绿体和13个线粒体的全基因组序列(表1)。用MicroSAtellite(MISA)软件对叶绿体、线粒体SSR标记进行搜索、预测。叶绿体和线粒体的预测标准一致。预测标准参数设置为:重复基序1~6 bp,且一到六核苷酸基序的最小重复次数分别为10、5、4、3、2、2;复合型SSR中间碱基间隔长度不大于100 bp。根据所要鉴定的SSR侧翼序列,将MISA的结果文件导入到Primer 3软件进行SSR引物的设计。核心参数设置为:引物长度范围18~22 bp,最适长度20 bp,PCR预期扩增产物大小 50~1 000 bp,退火温度50~65 ℃,最适退火温度57 ℃,GC含量45%~50%。

表1 小麦属和山羊草属61个叶绿体和13个线粒体基因组的GenBank序列号和全长Table 1 GenBank serial numbers and full length of 61 chloroplasts and 13 mitochondrias genomes in Triticum L.and Aegilops L.

(续表1 Continued table 1)
1.2 基于e-PCR进行电子指纹图谱的绘制
用e-PCR对SSR引物进一步筛选,扩增产物长度设置为50~1 000 bp,剔除重复的、无明显多态性的引物,用Powermarker软件计算引物的杂合度和多态性信息量(polymorphism information content,PIC),挑选PIC值大于0.5的多态性SSR引物;将初筛引物扩增数据再次导入到MapChart中,通过模拟电泳筛选多态性SSR引物。并用MapChart软件绘制电子指纹图谱。
2 结果与分析
2.1 小麦属、山羊属叶绿体和线粒体全基因组SSR位点的数量和种类
从NCBI下载小麦属、山羊草属共61个叶绿体和13个线粒体的全基因组序列。将山羊草属叶绿体全基因组按倍性及所含基因型分为CP.A.D(所包含物种编号:1~21)、CP.A.M(22和23)、CP.A.S(24~41)、CP.A.U(42和43)、CP.A.DC(44和45)、CP.A.UM(46和47)、CP.A.US(48和49);将小麦属叶绿体全基因组按倍性分为:CP.T.2(50~53)、CP.T.4(54)、CP.T.6(55~61);将13个线粒体全基因组按照属及倍性分为:MT.A(62)、MT.T.4(63和64)、MT.T.6(65~74)。按以上分组顺序对叶绿体、线粒体全基因组的SSR位点频率进行统计,以上13个分组中,平均每Mb所含SSR位点数目为3 139~4 150,两个SSR位点之间的平均距离为0.269~0.313 kb(表1)。7个山羊草属叶绿体全基因组分类中,CP.A.UM的SSR位点出现频率最高,平均每Mb所含SSR位点数目为3 714个,两个SSR位点的平均距离为0.269 kb;CP.A.D的SSR位点出现频率最低,平均每Mb所含SSR位点数目为3 402个,两个SSR位点的平均距离为0.294 kb。3个小麦属叶绿体全基因组分类中,CP.T.2的SSR位点出现频率最高,平均每Mb所含SSR位点数目为3 652个,两个SSR位点的平均距离为0.274 kb;CP.T.4的SSR位点出现频率最低,平均每Mb所含SSR位点数目为3 406个,两个SSR位点的平均距离为0.294 kb。
本研究中相同属的SSR位点数量与物种的倍数并无太大的关系,主要与基因组长度有关,对于同一属的物种,在相同参数设置下,物种的基因组越长,每Mb所含SSR位点的数目就越大。在山羊草属的二倍体物种中,CP.A.M组叶绿体全基因组序列中SSR位点的出现频率均高于CP.A.D、CP.A.S和CP.A.U三组;在山羊草属的四倍体物种中,CP.A.UM组叶绿体全基因组序列中SSR位点的出现频率也最高,说明在山羊草D、M、S、U四个基因组中,SSR位点在M基因组中的出现频率最高,同时也说明SSR位点数目可能与基因组的类型相关。将拟斯卑尔脱山羊草叶绿体全基因组(编号27~32)与线粒体全基因组(编号62)、普通小麦叶绿体全基因组(编号55~60)与线粒体全基因组(编号65~68)、中国春叶绿体全基因组(编号61)与线粒体全基因组(编号69)序列中SSR位点的出现频率进行比对后发现,同一物种中,线粒体全基因组序列中SSR位点的出现频率要远小于叶绿体全基因组序列,进一步印证了植物线粒体在植物三套遗传体系(线粒体基因组、叶绿体基因组和核基因组)中的保守性最高。
2.2 叶绿体、线粒体基因组中SSR基序重复特征和不同基序重复次数分布
分析发现,除二核苷酸重复基序外,单核苷酸至六核苷酸重复基序在叶绿体基因组和线粒体基因组中均有分布。每个物种的单核苷酸至六核苷酸重复基序都有其优势重复序列,在所有的叶绿体、线粒体全基因组中,五核苷酸重复基序在整个SSR位点中占比最高,在13个分组中所占比例为57.32%~74.32% ;六核苷酸重复基序在整个SSR位点中所占比例次之,介于19.81%~ 34.06%之间,一核苷酸至四核苷酸重复基序在整个SSR位点中共占比5.02%~10.93%。CP.A.S、CP.T.6、MT.A和MT.T.6分组中的SSR基序重复特征见表2。从图1可以看出,小麦属和山羊草属的叶绿体、线粒体SSR标记主要以2次重复的五核苷酸和六核苷酸重复基序为主。所以,小麦属和山羊草属的进化水平或突变频率相差不大。

表2 CP.A.S、CP.T.6、MT.A、MT.T.6组不同全基因组序列中SSR位点的重复基序数目Table 2 Number of SSR repeat motifs in different whole genome sequences of CP.A.S, CP.T.6, MT.A and MT.T.6
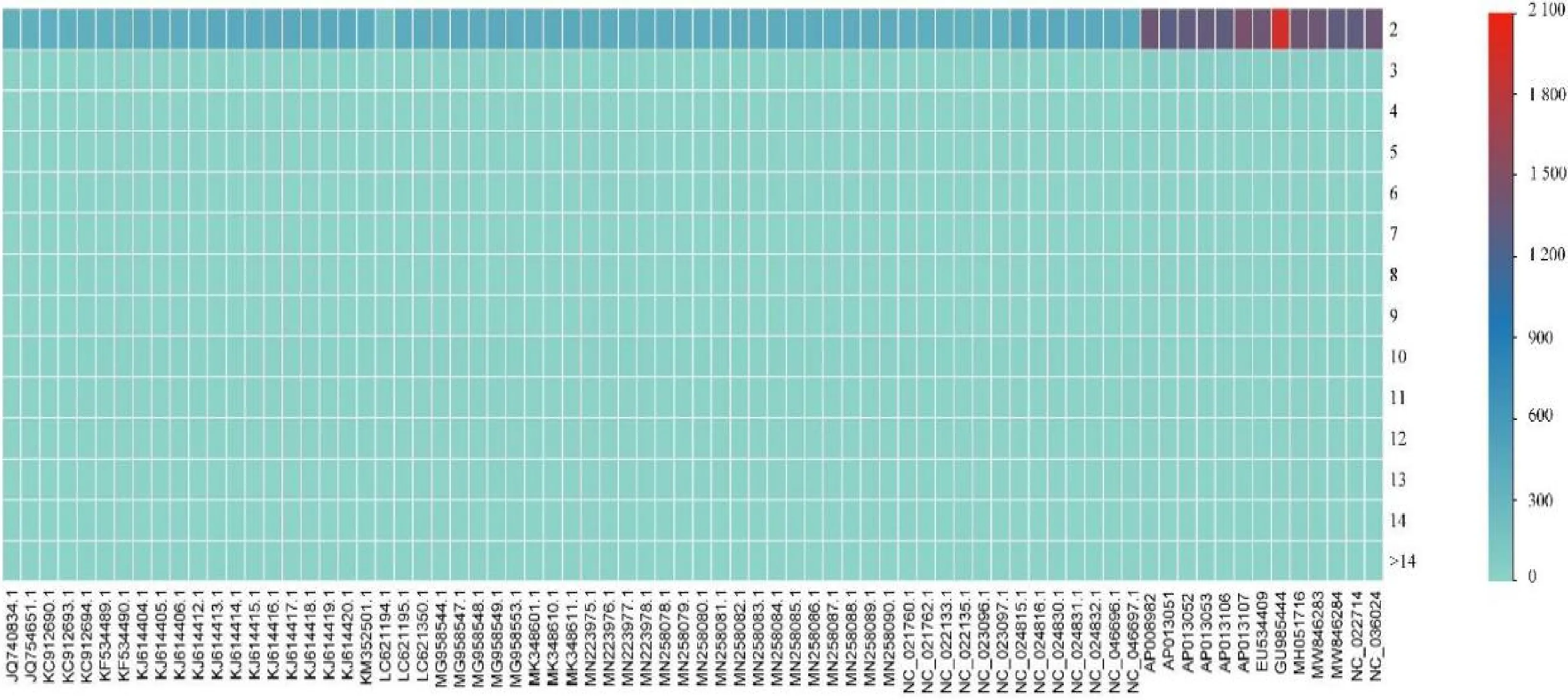
图1 基于74个全基因组不同基序重复次数热图Fig.1 Heat maps based on different motif repeats of 74 whole genomes
2.3 e-PCR对SSR引物的进一步筛选结果
将e-PCR产物长度参数设置为50~1 000 bp,对Primer 3软件设计的28 129对叶绿体全基因组引物、18 487对线粒体全基因组引物进行扩增产物大小、扩增产物数目预测,基于e-PCR的结果,剔除一些重复的、无明显多态性的引物。将初步筛选到的277对引物扩增产物大小数据格式调整为 Boulder-IO,然后导入到Powermarker软件,计算引物的杂合度和PIC值,最终挑选出PIC值大于0.5的11对理想多态性SSR引物(表3);将初筛引物扩增数据再次导入到MapChart软件中,通过模拟电泳,再次筛选出19对多态性SSR引物。两次共筛选出30对多态性引物(表4)。其中,来源于二倍体和四倍体山羊草叶绿体全基因组的引物分别有16和2对;来源于四倍体和六倍体小麦叶绿体全基因组的引物分别有5和2对;来源于六倍体小麦线粒体全基因组的引物有5对。选取引物CP-T和CP-10对13个材料进行真实PCR验证,发现引物扩增条带清晰,多态性显著(图2),说明e-PCR在引物筛选、多态性验证方面具有重要作用。

表3 PIC值大于0.5的11对SSR引物的基本信息Table 4 Basic information of 11 SSR primers with PIC value greater than 0.5

表4 筛选到的30对多态性SSR引物的序列Table 4 Sequences of 30 polymorphic SSR primers screened
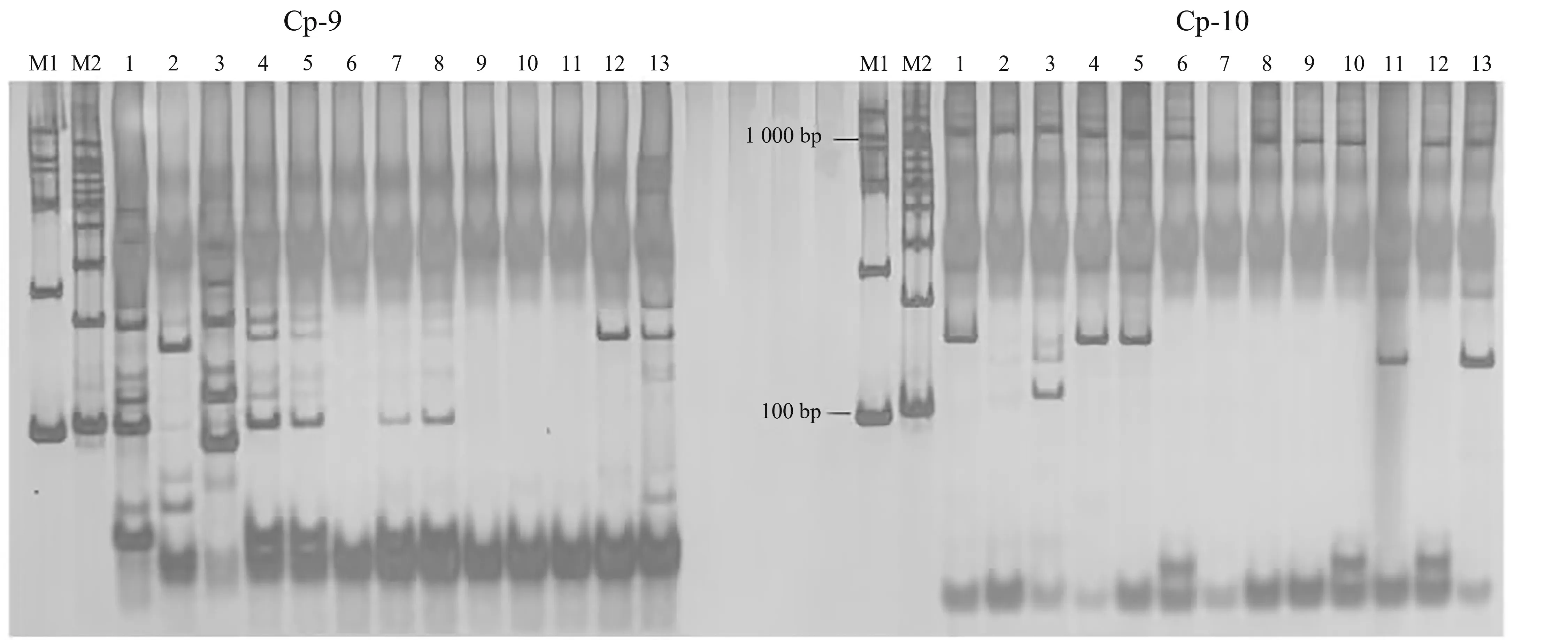
M1和M2:Marker; 1:硬粒小麦(AABB);2:一粒小麦(AA);3:小黑麦(AAGG);4:豫麦47;5:豫麦21;6:山羊草(DD);7:中国春;8:并麦1号;9:AE510;10:AE502;11:AE508;12:RM206;13:RM241。M1和M2:Marker;1:Durum wheat(AABB);2:Einkorn wheat(AA);3:Triticale(AAGG);4:Yumai 47;5:Yumai 21;6:Aegilops(DD);7:Chinese Spring;8:Bingmai 1;9:AE510;10:AE502;11:AE508;12:RM206;13:RM241.图2 SSR标记CP-9和CP-10的扩增结果Fig.2 Amplifiication results of SSR markers CP-9 and CP-10
2.4 基于e-PCR绘制的e-指纹图谱
e-指纹图谱,就是利用软件将e-PCR的引物扩增结果进行可视化。以mt-1、cp-2 SSR引物为例,引物mt-1可以在13个线粒体全基因组中特异性识别出GenBank编号为AP013107的线粒体全基因组,也可以识别出GenBank编号为AP013106和MW846284的线粒体全基因组,进一步通过其他引物检测,便能将二者区分开来(图3);引物cp-2可以在61个叶绿体全基因组中,特异型的识别出GenBank编号为MK348611.1的叶绿体全基因组(图4)。引物的扩增数据则可以通过二维码的方式附加在电子指纹图谱的后面,如mt-1、cp-2的扩增信息通过图5可进行扫描查看,进一步丰富电子指纹图谱的内容。

M: Marker; 1: AP008982; 2: AP013051; 3: AP013052; 4: AP013053; 5: AP013106; 6: AP013107; 7: EU534409; 8: GU985444; 9: MH051716; 10: MW846283; 11: MW846284; 12: NC_022714; 13: NC_036024.图3 mt-1引物在13个线粒体全基因组中的电子指纹图谱Fig.3 Electronic fingerprint of mt-1 primer in 13 mitochondrial whole genomes

M: Marker; 1: JQ740834.1; 2: JQ754651.1; 3: KC912690.1; 4: KC912693.1; 5: KC912694.1; 6: KF534489.1; 7: KF534490.1; 8: KJ614404.1; 9: KJ614405.1; 10: KJ614406.1;11: KJ614412.1; 12: KJ614413.1; 13: KJ614414.1; 14: KJ614415.1; 15: KJ614416.1; 16: KJ614417.1; 17: KJ614418.1; 18: KJ614419.1; 19: KJ614420.1; 20: KM352501.1; 21: LC621194.1; 22: LC621195.1; 23: LC621350.1; 24: MG958544.1; 25: MG958547.1; 26: MG958548.1; 27: MG958549.1; 28: MG958553.1; 29: MK348601.1;30: MK348610.1; 31: MK348611.1; 32: MN223975.1; 33: MN223976.1; 34: MN223977.1; 35: MN223978.1; 36: MN258078.1; 37: MN258079.1; 38: MN258080.1; 39: MN258081.1; 40: MN258082.1; 41: MN258083.1; 42: MN258084.1; 43: MN258085.1; 44: MN258086.1; 45: MN258087.1; 46: MN258088.1; 47: MN258089.1; 48: MN258090.1; 49: NC_021760.1; 50: NC_021762.1; 51: NC_022133.1; 52: NC_022135.1; 53: NC_023096.1; 54: NC_023097.1; 55: NC_024815.1; 56: NC_024816.1; 57: NC_024830.1; 58: NC_024831.1; 59: NC_024832.1; 60: NC_046696.1; 61: NC_046697.1 .图4 cp-2引物在61个叶绿体全基因组中的电子指纹图谱Fig.4 Electronic fingerprint of cp-2 primer in 61 chloroplast whole genomes

图5 mt-1、cp-2引物的扩增信息二维码Fig.5 Mplification information QR codes of mt-1 and cp-2 primers
3 讨 论
SSR分子标记具有低成本、速度快、准确性高等优点,是作物品种鉴定较为理想的分子标记之一[5]。传统SSR分子标记的开发不仅浪费物力财力,且具有一定的盲目性。针对这一难题,史通麟[16]首先构建了苦荞基因组的(CT)n和(GT)n富集文库,以此为基础开发了苦荞SSR引物;郑玉莹等[17]基于转录组测序数据开发了SSR分子标记;熊登坤等[12]通过e-PCR对SSR引物进行筛选,并证实e-PCR对SSR分子标记的开发具有促进作用。以上三种方法均在一定程度上降低了SSR分子标记的开发难度,其中e-PCR的作用最明显,但经过e-PCR的筛选,部分引物在电泳时仍会出现条带模糊的现象,原因之一可能小麦基因组较为复杂,且使用的小麦材料与小麦基因组数据之间有一定的差异[18]。总体来说,e-PCR具有广泛的适用性和可期的前景。
本研究对小麦属、山羊草属共61个叶绿体和13个线粒体的全基因组进行了SSR位点信息分析,结果显示,同属物种在同一参数设置下,基因组越长,SSR位点的数目就越大;SSR位点数目可能与基因组的类型相关,在山羊草D、M、S、U四个基因组中,M基因组的SSR位点出现频率最高;同一物种中,线粒体全基因组中SSR位点的出现频率要远小于叶绿体全基因组,进一步证实了植物线粒体在植物三套遗传体系(线粒体基因组、叶绿体基因组和核基因组)中的保守性最高;植物线粒体、叶绿体基因组均以2次重复的五核苷酸和六核苷酸重复基序为主,同一种类型基序的 SSR 位点, 基序重复次数与SSR数目成反比。这与仇静静[13]的研究结论一致。本研究中由于所设引物大小固定,所以并未对“SSR引物的长度越长,可筛选多态性SSR引物的可能性越高”这一规律进行验证。在中国,农作物DNA指纹数据库构建所采用的技术、标准、软件等各不相同,同一农作物存在若干不同的DNA指纹数据库,且大多数据库之间存在不能兼容的现状[19]。
本研究所开发的电子指纹图谱中,Marker最下端为0 bp,相邻条带间相差100 bp,通过Marker可以快速鉴定一些扩增产物大小差别较大的作物品种。电子指纹图谱与传统指纹图谱和数字指纹图谱相比,对Marker进行了统一,对扩增信息进行了富集,使检索更加方便、快捷,更符合农作物品种快速、精确鉴定的要求。科研人员可通过电子指纹图谱所提供的完备的引物扩增信息,来相互使用所需引物,选择适宜的电泳体系。本研究基于e-PCR分析结果,进行了电子指纹图谱的绘制,并通过真实PCR检测,构建了可视化SSR电子指纹图谱,不仅可提升引物的流通性,减少各个团队之间因引物的重复开发而导致的资源浪费,而且可改变DNA指纹数据库不兼容的现状。
