剪接因子SQD在家蚕中的表达定位与功能分析
2023-05-27蔡敏玲邓惠敏朱子丹
蔡敏玲,张 康,张 纯,邓惠敏*,朱子丹*
(1. 华南师范大学生命科学学院,广东省昆虫发育生物学与应用技术重点实验室,广州 510631;2. 广东省植物保护新技术重点实验室,广州 510640)
遗传信息从DNA转移到RNA的过程称为转录(transcription)。在真核生物中,最初转录生成的RNA称为核内不均一RNA(heterogeneous nuclear RNA,hnRNA)。hnRNA是mRNA的未成熟前体,包括各种基因的转录产物及其成为mRNA前的各中间阶段分子。大部分hnRNA在核内与各种特异蛋白质形成复合体即核内不均一核糖核蛋白(heterogeneous nuclear ribonucleoprotein,hnRNP)。hnRNP家族(hnRNPs)是一类与mRNA生物学功能密切相关的RNA结合蛋白,主要结构包括氨基端(N端)的两个RNA结合区域(RNA-binding domain,RBD I和RBD II)与羧基端(C端)富含甘氨酸的区域,其与前体mRNA(pre-mRNA)结合形成复合体并参与一系列重要的生命功能如mRNA转运、代谢及剪接等过程的调控(邵羊阳等,2017)。hnRNP具有辅助RNA加工的功能,除至少包含有一个RNA结合区域,其还包含辅助区域。
RNA识别结构域(RNA recognition motif,RRM),也称RBD或核糖核蛋白结构域(ribonucleoprotein domain,RNP),在真核生物中是含量最丰富的蛋白质结构域之一。RRM RNA结合蛋白的显著特征是包含一个或数个明显的RRM,可参与pre-mRNA剪接、RNA细胞定位及RNA稳定性等多种转录后调控过程(杜光伟等,1999)。通常RRM中包含2个高度保守的共有序列,称为RNP1和RNP2。从RRM的三维结构可以看出,RRM保守氨基酸RNP区域存在两个作用:(1)RNP1和RNP2的带电和芳香族侧链暴露在外侧溶剂中,可通过氢键和环的堆积力直接与RNA结合;(2)位于RNP1最后位置的芳香族侧链指向折叠的结构域内部和两个α螺旋的高度保守疏水氨基酸形成结构域的疏水核心(Kenan, 1991)。从典型的RRM序列比对显示,RNP1位于RRM的β3链上,其共有序列为(Lys/Arg)-(Gly)-(Phe/Tyr)-(Gly/Ala)-(Phe/Tyr)-(Val/Ile/Leu)-X-(Phe/Tyr);RNP2则位于RRM的β1链上, 共有序列是(Ile/Val/Leu)-(Phe/Tyr)-(Ile/Val/Leu)-X-Asp-Leu(X表示任意氨基酸)(Marisetal., 2005; Katherineetal., 2017)。
家蚕Bombyxmori是一种重要的经济昆虫,也是鳞翅目的模式昆虫。Squid(SQD)在家蚕中通过RNA的选择性剪接编码不同的hnRNP亚型,每个亚型氨基酸残基共同的氨基端有两个共同的RRM,这些亚型是hnRNP复合物的主要成分(霍锦霞,2010)。果蝇中的Squid/SQD蛋白hrp40为一类hnRNPs,可作为剪接抑制因子和hrp38共同与poly ADP-ribose(pADPr)结合,调控Hsrw-RC的选择性剪接(Ji and Tulin, 2009)。因此,推测Squid(SQD)是一种hnRNPs。
对家蚕剪接因子SQD的研究,有助于阐释核不均一核糖核蛋白SQD在家蚕甚至昆虫生命活动中的功能作用。本研究制备了BmSQD的多克隆抗体,并对该蛋白在各发育时期与各组织中的表达与定位进行了分析。研究结果为进一步探究SQD在家蚕乃至昆虫中的功能及其在昆虫基因选择性剪接的调控机制方面提供了参考。
1 材料与方法
1.1 实验材料与主要试剂
采用的家蚕品种为P50T大造。蚕卵与桑叶均由广东省农业科学院蚕业与农产品加工研究所提供,幼虫置于27℃,相对湿度65%~75%,光周期L∶D=12 h∶12 h条件下饲养至成虫;原核蛋白表达载体pET-32a及大肠杆菌DH5α与BL21为本实验室保存;质粒提取试剂盒购自天根生化科技有限公司;克隆试剂盒One step Cloning Kit购自Vazyme公司;逆转录酶M-MLV(RNase free)、dNTP(10 mM)、Recombinant RNase Inhibitor、RNase free Water、Recombinant DNase I(RNase free)及Trizol Isolate抽提试剂盒均购于TaKaRa公司;实时荧光定量PCR试剂盒EastepRqPCR Master Mix购于上海普洛麦格公司;弗氏完全佐剂和弗氏不完全佐剂购自Sigma公司;实验涉及的引物合成和相关DNA测序委托华大基因公司完成。
1.2 生物信息学分析
使用DNAMAN软件对BmSQD的CDS(Coding Sequence)核苷酸序列进行氨基酸序列翻译;利用在线工具Protparam软件对BmSQD基本特性进行分析;使用ProtScale在线软件分析蛋白的亲/疏水性;采用NetPhos 2.0 Server工具在线预测蛋白的磷酸化位点;利用SOPMA在线工具对BmSQD的二级结构进行预测与分析;采用SMART网站分析蛋白的保守结构域;运用SWISS-MODEL数据库在线分析BmSQD蛋白的三维结构;使用DNAstar软件中的Megalign工具和MEGA6软件对SQD的序列进行比对与保守性分析。
1.3 总RNA提取与cDNA合成
分别选取5龄期(第4、6天)、游走期第1、2天及预蛹期翅原基,及蛹期第3、5天的蛹翅和游走期第2天(W2)不同组织,包括翅原基、中肠、表皮、丝腺及头部;胚胎组织按照家蚕成虫交配产卵后每24 h为一天的时间及胚胎发育时期 2 h(卵受精期)、18 h(原胚期)、30 h(原肠胚期)、72 h(器官形成期)、120 h(反转期)、168 h(点青期)收集材料;将各组织在液氮中速冻后研磨。按照TaKaRa公司Trizol Isolate试剂盒的方法提取各组织的总RNA,再按照M-MLV RNase cDNA Synthesis Kit试剂盒说明书进行cDNA的合成。
1.4 引物设计与合成
在NCBI网址(https://www.ncbi.nlm.nih. gov/)搜索下载家蚕Bmsqd的mRNA序列,根据该基因的ORF序列,利用Primer 5.0软件设计特异性引物,设计出Bmsqd-1F和Bmsqd-1R基因克隆引物和荧光定量PCR引物RT-Bmsqd-F和RT-Bmsqd-R;同时利用DNAMAN软件分析选取BamH I/SalI为酶切位点,设计带有酶切位点的引物Bmsqd-2R和Bmsqd-2F。

表1 本实验所用引物
1.5 家蚕BmSQD的基因克隆及蛋白表达载体构建
以逆转录的家蚕幼虫翅原基cDNA为模板,利用上述特异性引物Bmsqd-1F和Bmsqd-1R进行PCR扩增Bmsqd的开放阅读框(Open Reading Frame,ORF)区域。反应条件为:95℃预变性5 min,94℃变性30 s,55℃退火30 s,72℃延伸30 s,32个循环,最后72℃延伸10 min。反应结束后,PCR产物用1%琼脂糖凝胶检测,利用凝胶回收试剂盒进行PCR产物的回收。回收的DNA片段与质粒pMD-18T载体进行连接反应,转化后筛选测序确认阳性克隆。以重组的pMD-18T-SQD质粒为模板,在上述扩增条件下扩增带有酶切位点的DNA。在琼脂糖凝胶检测后同样用凝胶回收试剂盒回收PCR产物。用BamH I和SalI双酶切pET-32a质粒,反应体系为10 μL,质粒2 μg,BamH I酶1 μL,SalI酶1 μL,10×T Buffer 3 μL,反应条件为37℃ 3 h,酶切产物用1%琼脂糖凝胶检测并回收目的DNA片段。将线性化pET-32a载体和带有酶切位点的DNA片段用同源重组试剂盒进行连接,反应体系为:线性化载体4 μL,目的DNA片段4 μL,连接酶1 μL,Buffer 2 μL,ddH2O 9 μL,37℃反应30 min。连接产物用大肠杆菌BL21感受态细胞进行转化反应,涂板后37℃培养16 h,挑取菌落并进行PCR反应后送公司测序筛选阳性克隆。
1.6 诱导重组SQD蛋白表达及纯化与抗体制备检测
将保存的重组质粒菌液接入5 mL含氨苄的LB液体培养基(50 μg/mL),37℃ 220 rpm过夜培养,隔天菌液按1∶100(v∶v)的比例接入新的含氨苄LB液体培养基,以同样的条件扩大培养;之后加入IPTG(0.1 mM),37℃ 220 rpm继续培养4~5 h。将诱导的菌液于4℃ 12 000 rpm离心10 min收集菌体。以不含目的基因的pET-32a菌体为对照,将菌体进行超声破碎,分别收集沉淀和上清液,用12%凝胶进行SDS-PAGE检测。按照Ni-NTA His·BindRResin(NovagenR)试剂盒的方法进行蛋白纯化,取1 mL Ni2+柱于管中,使用两倍体积蒸馏水洗2次,两倍体积1×Charge(50 mM NiSO4)buffer洗3次,两倍体积1×Binding buffer(0.5 M NaCl,20 mM Tris-HCl,5 mM Imidazole,pH7.9)洗2次,将上清液用0.45 μM规格的滤膜过滤后加入管中,用10倍体积的1×Binding buffer、6倍体积的1×Washing buffer(0.5 M NaCl,20 mM Tris-HCl,60 mM Imidazole,pH7.9)和6倍体积的1×Elution buffer(0.5 M NaCl,20 mM Tris-HCl,1 M Imidazole,pH7.9)分别洗脱柱子,离心收集纯化蛋白上清。用SDS-PAGE检查蛋白纯化效果。
新西兰雄兔稳定饲养4 d,禁食24 h后于耳缘动脉抽取3~5 mL血液,37℃放置2 h后4℃沉淀过夜,次日12 000 rpm离心,取上清作为对照组,-80℃保存。将纯化后的重组蛋白与Sigma弗氏不完全佐剂等体积混合,确保蛋白量为2 mg,蛋白与佐剂混合后震荡过夜,使抗原充分乳化,次日在兔子的腹部和四肢腋下等部位进行皮下注射;7 d后将纯化后的重组蛋白与Sigma弗氏完全佐剂等体积混合,确保蛋白量为2 mg,蛋白与佐剂混合后震荡过夜,使抗原充分乳化,次日在兔子的腹部和四肢腋下等部位进行皮下注射;7 d后,再次免疫注射2次;4次免疫后收集血液制备抗体血清,之后Western blot检测抗体效价。
1.7 Western blotting
分别选取5龄期(第1、3和5天)、游走期第1与2天翅原基及蛹期第0天和3天的蛹翅和游走期第1天(W1)不同组织,包括卵巢、中肠、脂肪体、精巢;胚胎组织按照家蚕成虫交配产卵后每24 h为一天的时间,于第3~7天收集材料;将各组织在预冷的PBS中研磨,取总蛋白用于Western blotting实验。
配制12% SDS-PAGE分离胶及5%浓缩胶,之后进行样品蛋白凝胶电泳。把配制好的蛋白胶置于电泳槽,按顺序上样,上样量为20~60 μg。先用80V低电压电泳浓缩蛋白,30 min后采用100 V电压进行蛋白分离,时间为1~2 h。电泳结束后,利用电转移系统将SDS-PAGE凝胶的蛋白转移至硝酸纤维素膜或聚偏二氟乙烯膜(PVDF膜)上,用3% BSA in TBST浸泡置于4℃封闭过夜,以1∶1 000稀释比例的BmSQD或Bmβ-tublin抗体(鼎国生物,中国)于37℃孵育2 h,去除抗体之后用TBST洗脱液洗膜3次,每次10 min去除未结合的抗体。接着加入1∶10 000稀释比例的碱性磷酸酶或辣根过氧化物酶标记的山羊抗兔IgG酶标抗体(威佳,中国),在37℃孵育1 h,去除二抗后,用TBST洗脱液洗膜4次,每次5 min,再用TBS洗脱液洗膜3次,每次10 min。最后用337.5 μg/mL四唑硝基蓝(NBT)和175 μg/mL 5-溴-4-氯-3-吲哚基-磷酸盐(BCIP)混合液或ECL试剂盒进行显色。
1.8 免疫组化定位
将5龄第5天家蚕幼虫的翅原基放置于固定液中,在4℃中慢速旋转过夜。在室温下加入3% BSA封闭1~2 h,再用PBT(0.1% Triton×100 in PBS)洗3次,每次5 min(以下皆是);接着用BmSQD抗体(1∶400)孵育2 h,PBT洗3次;之后用二抗(1∶400)(鼎国生物,中国)孵育1~2 h,PBT洗3次;4′,6-二脒基-2-苯基吲哚DAPI(4′,6-Diamidino-2-phenylindole dihydrochloride)(鼎国生物,中国)孵育30 min,PBT洗2次;样品于共聚焦扫描显微镜(奥林巴斯FV3000)下观察拍照。
将家蚕胚胎发育72 h的蚕卵经过软化固定后用石蜡包埋做切片,再经过二甲苯和乙醇对切片脱蜡,PBS清洗3次,每次5 min(以下皆是);3%甲醇-H2O2溶液浸泡3 min消除内源性过氧化物酶的活性,PBS清洗1次;每个切片滴加 150 μL 10 μg/mL Proteinase K PBS溶液并用封口膜覆盖,37℃,20 min,PBS清洗1次;冷的Gly/PBS (2 mg/mL)溶液在冰上清洗10 min,PBS清洗1次;滴加3% BSA/PBS于37℃封闭1 h;加BmSQD抗体(1∶500),37℃孵育2 h,PBS清洗3次;加入荧光标记的二抗(1∶500)和DAPI,避光,37℃孵育30 min,PBS清洗3次;之后用激光共聚焦扫描显微镜(OLYMPUS,日本)观察拍照。
1.9 实时荧光定量PCR
用稀释至100 μL的cDNA样品定量检测目的基因和内参基因的扩增效率是否符合定量PCR标准,之后进行实时定量PCR(Quantitative Real-time PCR,qRT-PCR)检测,采用20 μL体系:10 μL qPCR Master Mix(2×),上、下游引物(RT-Bmsqd-F和RT-Bmsqd-R)各0.4 μL,2 μL DNA模板,7.2 μL ddH2O。PCR标准扩增程序:95℃变性2 min,40个循环,循环条件为95℃ 15 s,60℃ 40 s。每个样品设置3组生物学重复。数据的分析和作图采用GraphPad Prism软件。
2 结果与分析
2.1 BmSQD理化性质与结构特性分析
家蚕Bmsqd(Accession number: NW_00458 1760.1)cDNA全长5 636 bp,ORF为975 bp,其编码的BmSQD蛋白由324个氨基酸组成,分子质量约34.671 kDa,等电点pI为8.37,在氨基端(N端)存在2个保守的RRM结构域,在羧基端(C端)含有2个富含甘氨酸(G)的低复杂度区域(Low Complexity Region,LCR)(图1)。RRM结构域能够识别结合RNA特定序列,且通常含有RRM结构域的RNA结合蛋白参与前体RNA的剪接(杜光伟等,1999)。hnRNPs的主要结构包括N端的两个RNA结合区域(RBD I和RBD II,RRM1和RRM2)及C端的富含甘氨酸区域(邵羊阳等,2017)。由此,推测BmSQD是一类具有识别结合特定基因RNA序列并参与剪接调控的hnRNPs。
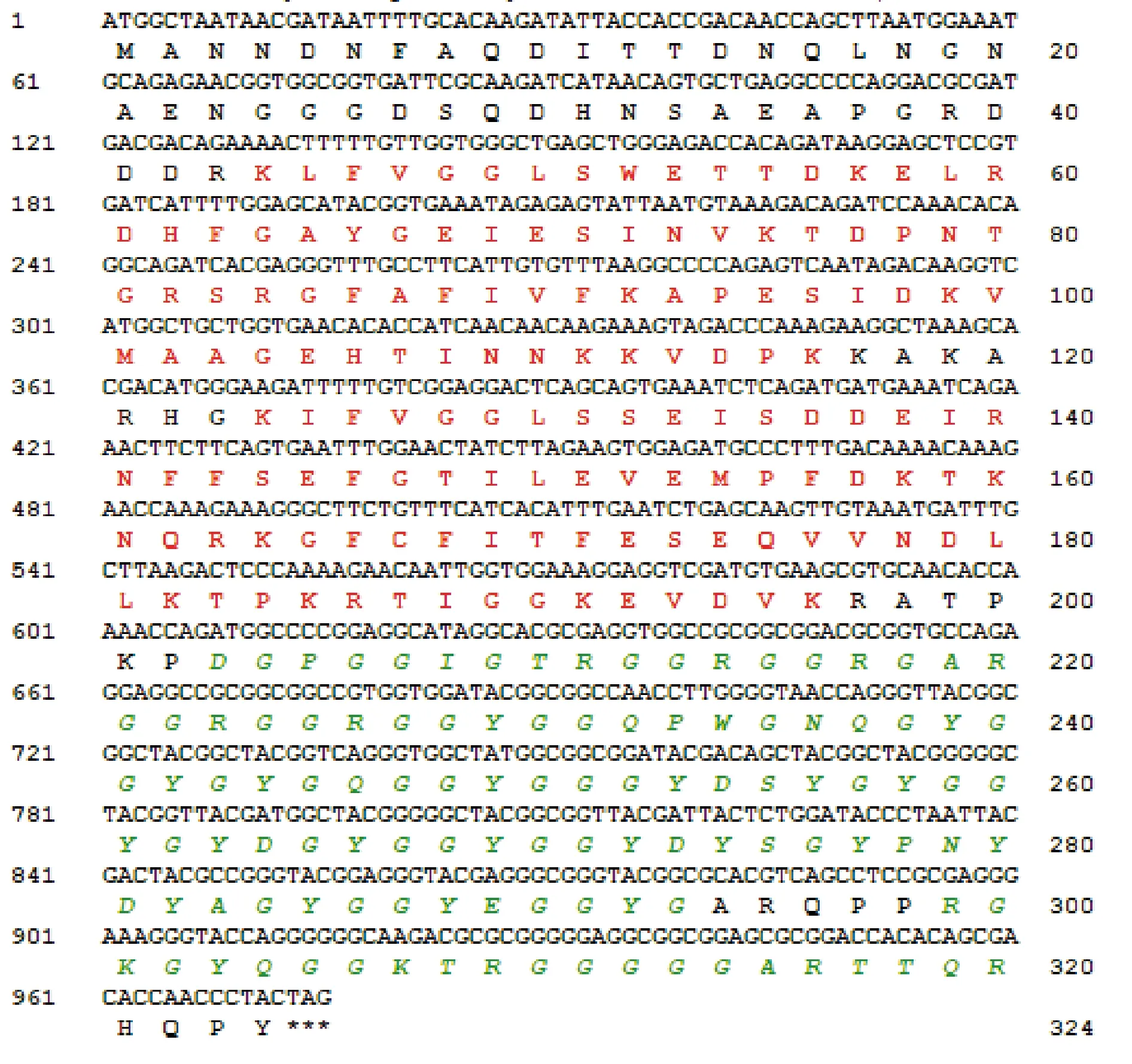
图1 BmSQD CDS的核苷酸与氨基酸序列Fig.1 Nucleotide and amino acid sequences of BmSQD CDS注:左边数字表示核苷酸序列,右边数字表示氨基酸序列;总氨基酸数,324;分子质量,34.671 kDa;终止密码子以*标记,BmSQD的RRM结构域用红色字体标示,RRM1位于44-116 aa,RRM2位于124-196 aa;低复杂度区域用绿色斜体标示。Note: Numbers on the left indicated nucleotide sequences, and numbers on the right indicated amino acid sequences. Total amino acid count, 324; Molecular weight, 34.671 kDa; Termination codon was marked with *, and the RRM domains of BmSQD were marked in red, RRM1 was located at 44-116 aa, and RRM2 was located at 124-196 aa. Low Complexity Regions were marked in green italics.
使用NetPhos 2.0 Server在线预测蛋白的磷酸化位点,在BmSQD的氨基酸序列中,磷酸化位点主要在于酪氨酸和苏氨酸,其次是丝氨酸,其中酪氨酸的磷酸化位点最多,且集中在序列后半部分,暗示BmSQD蛋白可能需要这些氨基酸位点的磷酸化以行使功能(图2)。
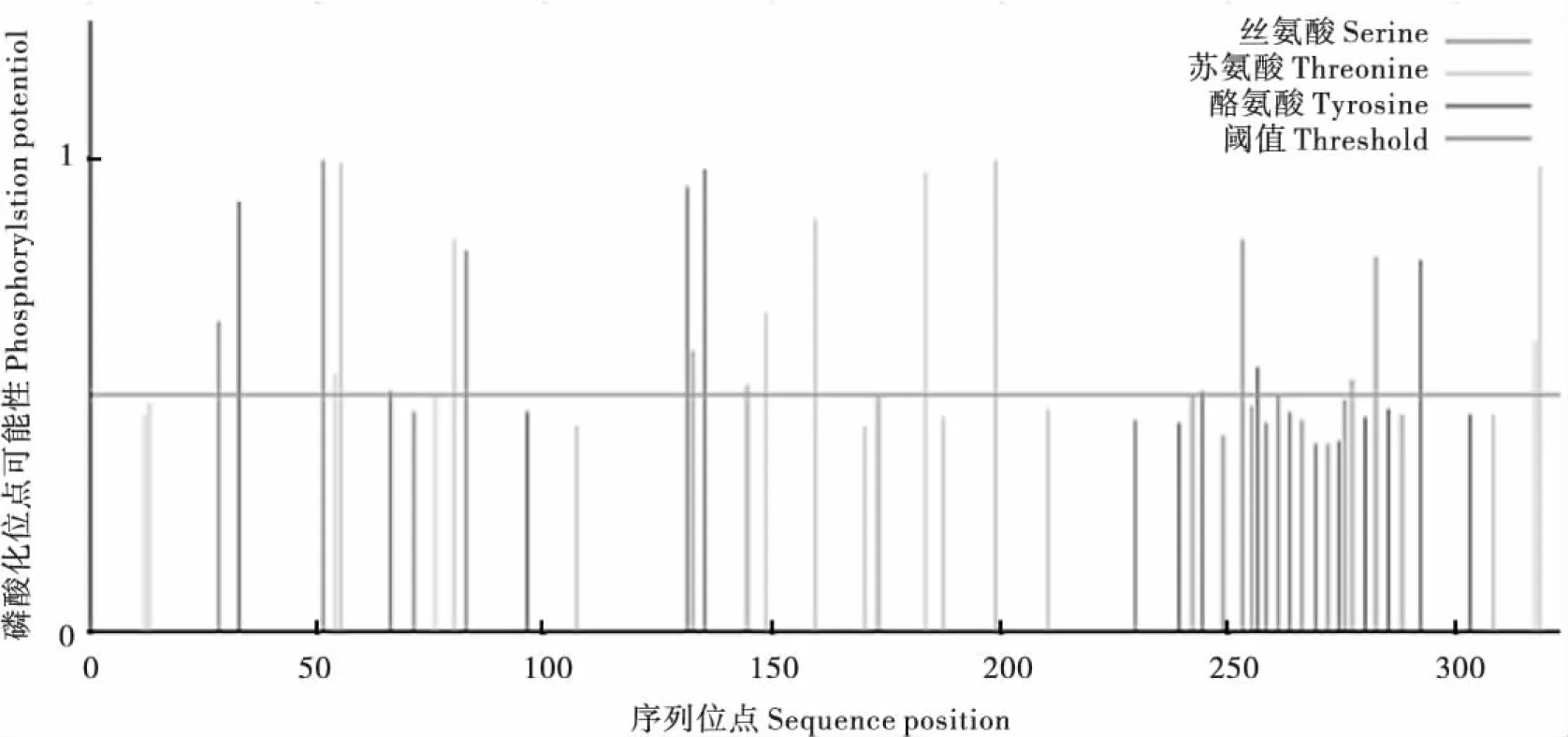
图2 BmSQD的磷酸化位点预测Fig.2 Phosphorylation site prediction of BmSQD
进一步利用SOPMA在线工具对BmSQD的二级结构进行预测与分析。结果显示,在BmSQD的二级结构中,α螺旋占13.58%,β折叠占8.33%,延伸链结构(Extended strand)占21.6%,比例最多为无规则卷曲(Random coli)高达56.48%(图3)。其中α螺旋和β折叠氨基酸序列通过自身盘绕折叠构成发挥RNA结合功能的RRM结构区域,而延伸链结构和无规则卷曲形成α螺旋和β折叠的多肽片段,构成蛋白主体部分,是蛋白功能实施和构象的重要区域。

图3 BmSQD的二级结构预测Fig.3 Secondary structure prediction of BmSQD
采用SMART网站分析蛋白的保守结构域,BmSQD具有两个保守的RRM结构域和两个低复杂度区域,暗示BmSQD具有结合RNA的能力(图4)。
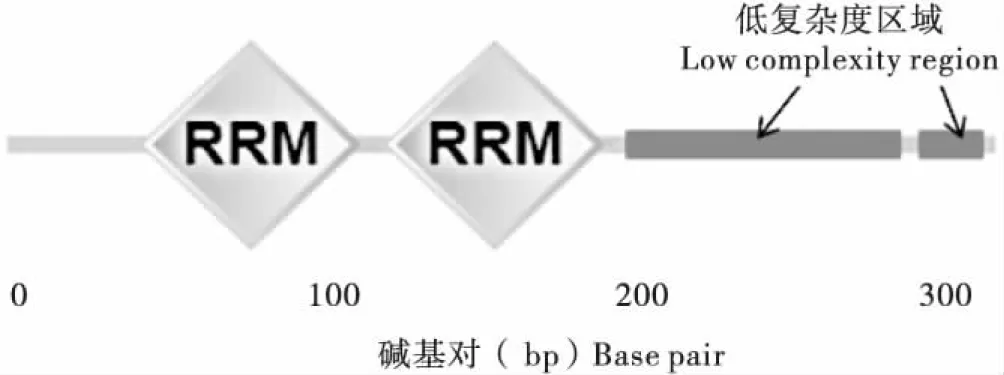
图4 BmSQD保守结构域图示Fig.4 Schema of the conserved domain of BmSQD
使用SWISS-MODEL数据库在线分析蛋白的三维结构,预测的BmSQD三维结构主要是两个RRM结构域组成的骨架结构,从而行使结合RNA和发挥相应作用的功能(图5)。
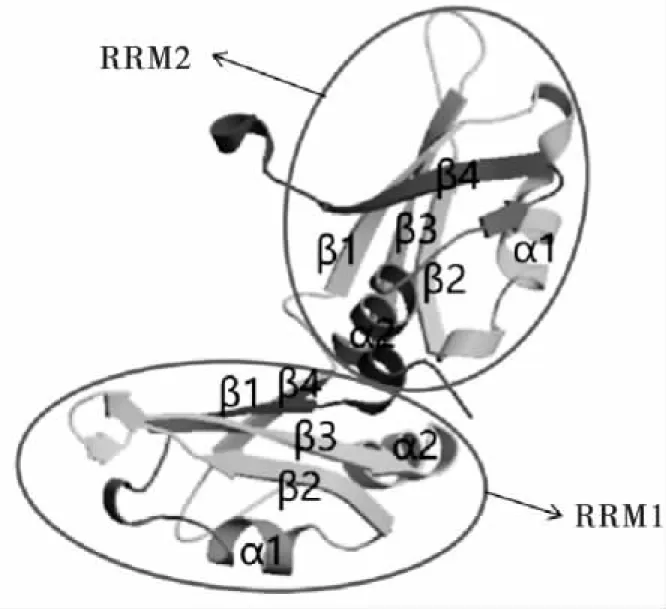
图5 BmSQD的三维模式预测Fig.5 3D model prediction of BmSQD
2.2 SQD保守性分析
为探究昆虫中SQD的功能是否保守,对不同昆虫目如鳞翅目、膜翅目、鞘翅目、双翅目、半翅目中不同物种的SQD进行聚类分析和氨基酸序列比对。结果显示,各类目中的SQD是可汇聚溯源的(图6-A),且从氨基酸序列比对看出,SQD的两个RRM结构域是高度相似保守的(图6-B)。进一步对不同昆虫SQD的RRM1和RRM2结构域氨基酸序列进行比对分析,发现两个RRM结构域序列比对总体匹配度高,且都存在两个高度保守的氨基酸区域RNP1和RNP2,分别位于RRM中的β3和β1区域(图7)。这些结果表明,SQD蛋白尤其是其RRM结构域在昆虫进化中是高度保守的,暗示SQD在昆虫中与RNA结合并行使剪接调节功能的高度保守性。
2.3 诱导重组SQD蛋白表达及纯化与抗体制备检测
将含有重组蛋白表达的菌体经过超声破碎后检测重组蛋白的可溶性,结果显示,重组BmSQD在上清液中,这表明重组蛋白是可溶的(图8-A);利用镍柱进行纯化,获得了纯化的重组蛋白(图8-B)。利用纯化蛋白进行抗体制备后获得了相应的多克隆抗体。Western-blot检测结果显示,BmSQD蛋白的多克隆抗体能较好地识别重组蛋白,而免疫前血清未能识别重组蛋白(图8-C),这表明制备的BmSQD多克隆抗体特异性较好,可以用于后续实验。
2.4 家蚕BmSQD的mRNA和蛋白表达水平
Bmsqd在家蚕幼虫期的翅原基和蛹期的翅中均有表达,通过实时荧光定量PCR可以分析其mRNA水平的表达情况;经过数据处理分析发现,在蛹期第3天(P3)Bmsqd表达量与其它时期比较具有显著差异,较之前显著升高,在蛹期第5天(P5)达到峰值(图9-A)。在对家蚕胚胎不同发育时期的表达量分析可以看出,Bmsqd的mRNA表达量在胚胎组织的第1天即受精后的24 h最高,在第2天后表达降低,在第4天表达量又逐渐增加,第5天表达量上升到一定量后于第6天下降,接着第7、8和9天表达量都较低(图9-B)。细化胚胎分化与发育时间,进一步分析Bmsqd在卵受精期、原胚期、原肠胚期、器官形成期、反转期、点青期的表达情况。结果显示,在胚胎发育受精卵分裂时期即原胚期,Bmsqd表达量较高,而后在原肠胚时期表达量有所下降,到器官形成期时表达量大致不变,但在反转期表达量大幅上升至原胚期水平,之后在点青期下降至原来下降的水平(图9-C)。
对家蚕不同时期的组织进行BmSQD蛋白表达水平分析,发现在翅原基或翅发育的不同时期,BmSQD均有表达,其中5龄第5天(L5D5)、游走期第1天(W1)、游走期第2天(W2)表达量较高(图10-A);由于家蚕胚胎发育第1、2天的卵黄原蛋白较多,第8、9天黑色素沉积,对目的蛋白的抗体孵育造成影响,故取胚胎发育第3天到第7天进行蛋白表达检测,从结果看出,在胚胎发育第4、5天蛋白表达量较高(图10-B),处于器官形成期和胚胎反转期,与mRNA表达水平相符合。此外,SQD抗体可检测到3条目的蛋白条带(图10),推测分子量较大的蛋白条带可能是磷酸化修饰后的蛋白条带,这与生物信息学分析预测BmSQD存在丝氨酸、苏氨酸、酪氨酸等磷酸化位点一致(图6),分子量较小的两条蛋白条带可能是由于SQD不同剪接形式而形成的两种同源异构体蛋白,详细情况有待进一步实验验证。

图6 昆虫不同目物种SQD的聚类分析与氨基酸序列比对Fig.6 Clustering analysis of SQD and amino acid alignment among different species of different insect orders注:A,SQD的聚类分析;B,SQD氨基酸序列比对(红色下划线为RRM1和RRM2区域)。Note: A, Clustering analysis of SQD; B, SQD amino acid alignment (RRM1 and RRM2 regionswere underlined in red).

图7 部分昆虫SQD-RRM结构域氨基酸序列比对分析Fig.7 Alignment and analysis of SQD-RRM domains amino acid sequence in some insects
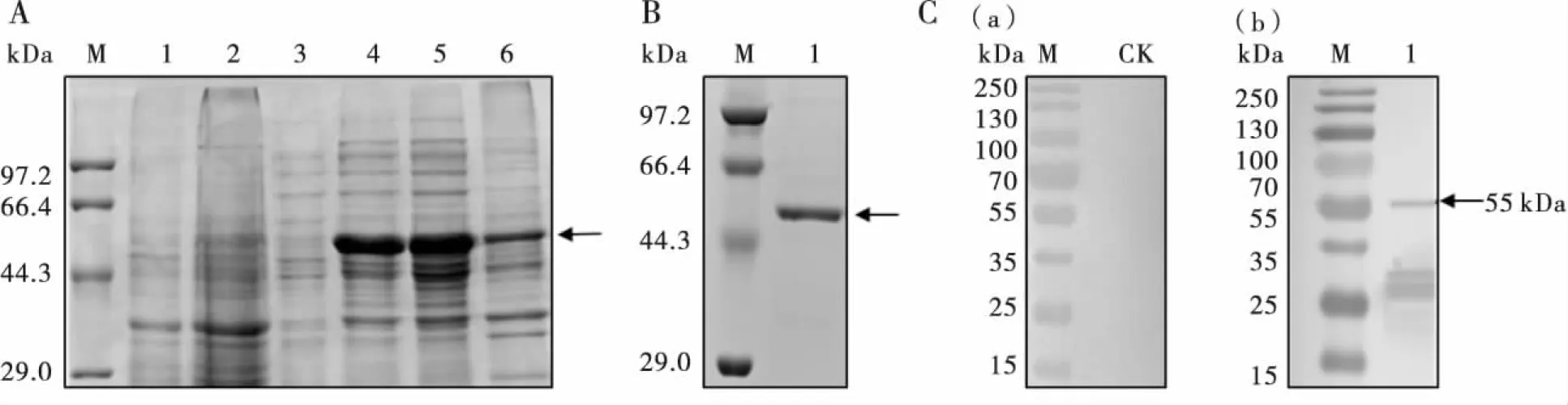
图8 BmSQD的SDS-PAGE和Western-blot结果分析Fig.8 SDS-PAGE and Western-blot analysisof BmSQD注:A,重组蛋白pET-32a-SQD的诱导表达检测;M,蛋白质分子量标准;1,pET-32a空白对照;2,pET-32a IPTG诱导;3,重组pET-32a-SQD;4,重组pET-32a-SQD IPTG诱导;5,重组pET-32a-SQD IPTG诱导上清;6,重组pET-32a-SQD IPTG诱导沉淀;B,经过镍柱纯化后的重组蛋白SDS-PAGE图,箭头所指为目的条带;C,Western-blot;(a),免疫前的血清;CK,免疫前的血清作为对照;(b),重组蛋白多克隆抗体。Note: A, Detection of induced expression of recombinant protein pET-32a-SQD; M, Protein molecular weight standard; 1, pET-32a blank control; 2, IPTG induction of pET-32a; 3, Recombinant pET-32a-SQD; 4, IPTG induction of recombinant pET-32a-SQD; 5, Supernatant of recombinant pET-32a-SQD induced byIPTG ; 6, Precipitation of recombinant pET-32a-SQD induced by IPTG; B, SDS-PAGE diagram of the recombinant protein after purification by nickel column, the arrow indicated the target band; C, Western-blot; (a), Pre-immunization serum; CK, Pre-immunization serum as control; (b), Polyclonal antibody against recombinant protein.
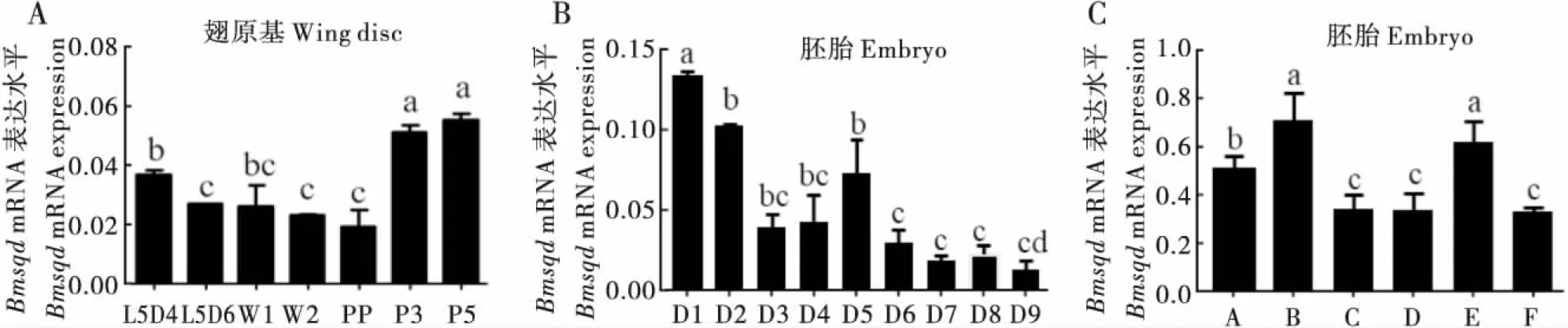
图9 BmSQD的mRNA表达水平分析Fig.9 mRNA expression level analysis of BmSQD注:A,Bmsqd在家蚕翅原基或蛹翅中不同时期的mRNA水平表达;L5D4,5龄第4天;L5D6,5龄第6天;W1,游走期第1天;W2,游走期第2天;PP,预蛹期;P3,蛹期第3天;P5,蛹期第5天;B,家蚕胚胎不同发育时期的Bmsqd mRNA表达水平;D1~D9,胚胎发育第1~9天(发育24 h为1天);C,胚胎发育各阶段的Bmsqd mRNA表达水平;A~F,卵受精期;原胚期;原肠胚期;器官形成期;反转期;点青期;图中数据为平均值±标准差;不同字母表示不同时期表达量存在显著差异(P<0.05)。Note: A, mRNA expression levels of Bmsqd in silkworm wing disc or pupal wing at different stages; L5D4, the 4th day of the 5th instar; L5D6, the 6th day of the 5th instar; W1, the 1st day of wandering period; W2, the 2nd day of wandering period; PP, pre-pupal stage; P3, the 3rd day of pupal stage; P5, the 5th day of pupal stage; B, Bmsqd mRNA expression levels at different day of embryonic development stages; D1~D9, Day 1~9 of embryonic development (24 hours of development count as one day); C, Bmsqd mRNA expression levels at different developmental stages of silkworm embryos; A~F, zygote stage; proembryo stage; progut embryo stage; organ formation stage; reversal stage; and dot green stage; Data in the figure were mean ± standard deviation; Different letters indicated significant differences in expression levels in different periods (P<0.05).
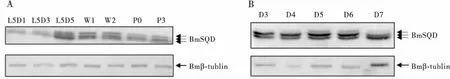
图10 不同组织BmSQD蛋白表达量Western blot结果Fig.10 Western blot results of BmSQD protein expression in different tissues注:A,不同时期家蚕幼虫翅原基或蛹翅;L5D1,5龄第1天;L5D3,5龄第3天;L5D5,5龄第5天;W1,游走期第1天;W2,游走期第2天;P0,刚进入蛹期;P3,蛹期第3天;B,家蚕各发育时期胚胎组织;D3~D7,发育至第3天~第7天的胚胎组织。Note: A, Wing discs or pupal wing of silkworm larvae at different stages; L5D1, the 1st day of the 5th instar; L5D3, the 3rd day of the 5th instar; L5D5, the 5th day of the 5th instar; W1, day 1 of the wandering stage; W2, day 2 of the wandering stage; P0, just entering the pupal stage; P3, day 3 of the pupal stage; B, Embryonic tissue at various stages of silkworm embryonic development; D3~D7, Tissue from day 3 to day 7.
2.5 BmSQD在翅原基和胚胎组织的定位
利用免疫组化分析BmSQD蛋白在家蚕5龄第5天幼虫翅原基和发育72 h的胚胎中的组织定位,结果显示,BmSQD蛋白在家蚕翅原基和胚胎组织中表达,且BmSQD蛋白与染细胞核的DAPI信号重叠在一起(图11,图12),这表明BmSQD蛋白定位于组织细胞的细胞核中,推测BmSQD蛋白可进入细胞核中,参与RNA剪接的相关调控。
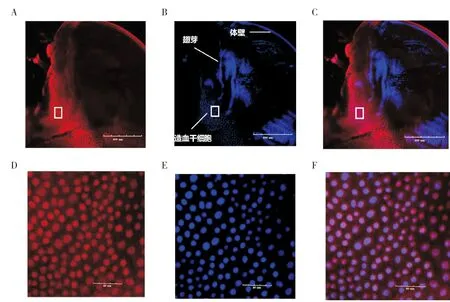
图11 BmSQD在5龄第5天幼虫翅原基中的免疫组化定位分析Fig.11 Immunohistochemical localization of BmSQD in the wing disc of larvae at the 5th day of the 5th instar larva注:A~C比例尺,400 μm;D~F比例尺,40 μm;A和D,BmSQD抗体;B和E,DAPI染色;C和F,重叠图像;D、E、F分别为图A、B、C中方框的放大图。Note: A~C scale, 400 μm; D~F scale, 40 μm; A and D, BmSQD antibodies; B and E, DAPI staining; C and F, overlapping images; D, E and F were enlargements of the boxes in Fig.A, B and C, respectively.
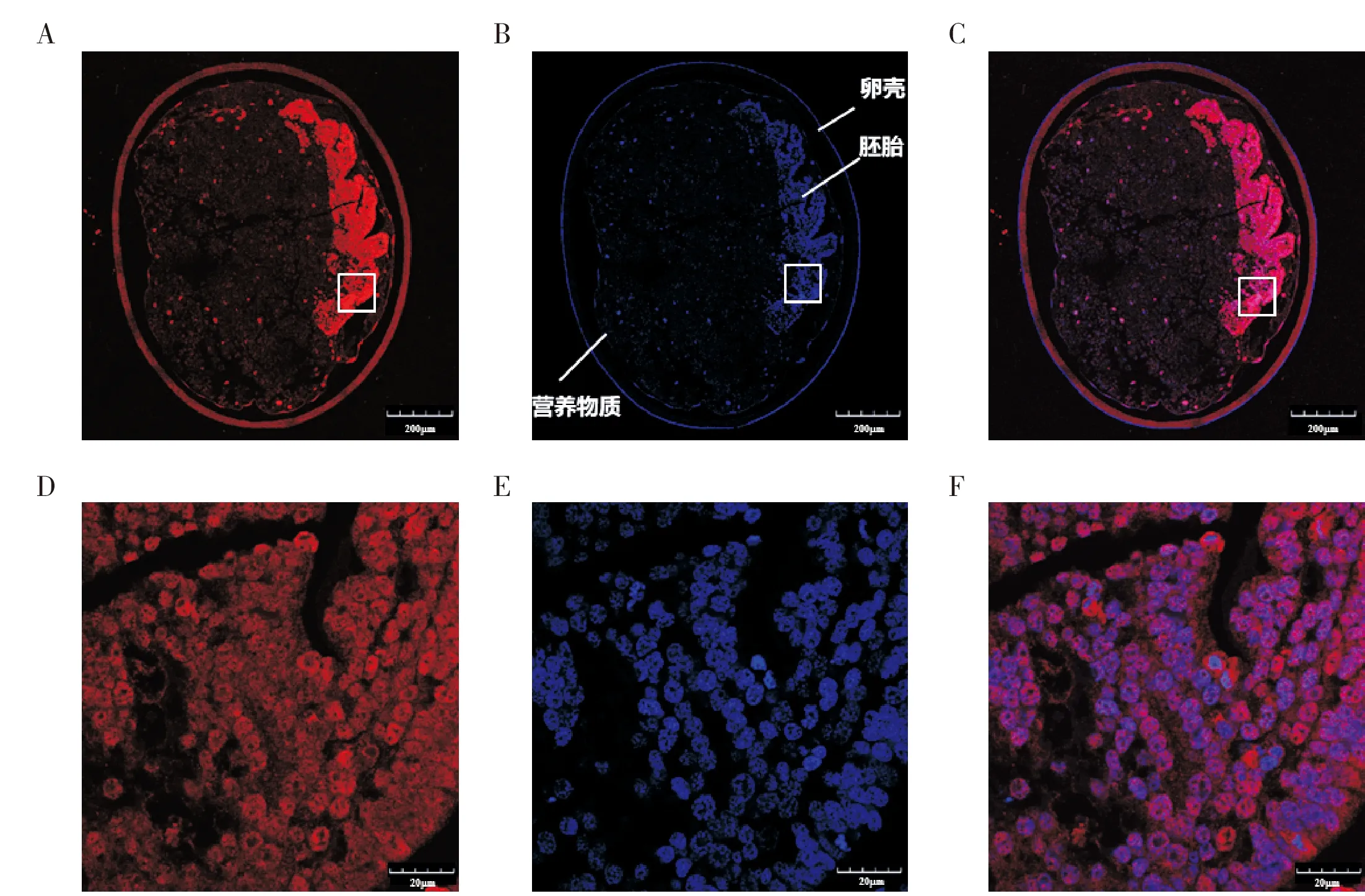
图12 BmSQD在发育72 h的胚胎中的免疫组化定位分析Fig.12 Immunohistochemical localization of BmSQD in embryos at 72 hafter oviposition注:A~C比例尺,200 μm;D~F比例尺,20 μm;A和D,BmSQD抗体;B和E,DAPI染色;C和F,重叠图像;D、E、F分别为图A、B、C中方框的放大图。Note: A~C scale, 200 μm; D~F scale, 20 μm; A and D, BmSQD antibodies; B and E, DAPI staining; C and F, overlapping images; D, E and F were enlargements of the boxes in Fig. A, B and C, respectively.
3 结论与讨论
BmSQD包含两个位于N端且高度保守的RRM结构域和两个位于C端的富含甘氨酸的低复杂度区域。对昆虫的SQD的序列与结构分析发现,相同昆虫目的SQD可聚为同源的一支且不同目的SQD含有高度保守的RRM结构域,特别是RRM中的RNP1和RNP2区域(图6,图7)。含有一个或数个RRM是RRM RNA结合蛋白的显著特征且RRM具备与RNA结合的能力(杜光伟等,1999)。故BmSQD蛋白属于一类hnRNPs。有研究表明,hnRNPs的两个RRM一般都参与这类蛋白与RNA的结合,如果hnRNP A1两个RRM结构域之间的相互作用被中断或任一个RRM的RNA结合能力丧失均会削弱其剪接抑制作用(Moursyetal., 2017),BmSQD包含两个位于N端且高度保守的RRM结构域,表明其具有作为剪接因子的共同结构域,也具备剪接因子的功能。RNP1和RNP2的带电和芳香族侧链可能通过分子间氢键与环的堆积力直接和RNA结合,而RNP1也可能通过疏水作用与RNA接触(Kenan, 1991);因此,RNP特定的氨基酸序列与结构位置是保证RRM具有结合RNA并发挥相应功能活性的重点。本研究发现SQD在昆虫中高度保守(图6),其中序列基本一致的RNP区域是保证SQD在昆虫进化中结合RNA功能高度保守一致的关键。RRM中高度保守的RNP又是保证该类蛋白与RNA结合活性所必需的,β1-α1-β2-β3-α2-β4空间顺序排列是高度保守的(Marisetal., 2005),BmSQD两个RRM都按照β1-α1-β2-β3-α2-β4的顺序进行空间结构排列且具有位置序列一致的RNP1和RNP2区域(图7);故两个RRM且RRM中的RNPs是保证BmSQD与RNA结合的关键基序,与上述已报道的研究结果一致,这些条件都表明BmSQD可能作为重要的剪接因子参与家蚕的胚胎发育和翅原基变态发育过程。
除N端两个高度保守的RRM结构域外,BmSQD的C端还包含两个富含甘氨酸的低复杂度区域。低复杂度区域在蛋白序列中很常见,通常具有低保守性和结构灵活性,在翻译后修饰调控的蛋白质互作中发挥作用(Kastanoetal., 2021)。低复杂度区域不仅可结合RNA,还可以与对RNA起剪接作用的酶直接结合,例如核异质核糖核蛋白G蛋白通过其N端的RRM和C端的Arg-Gly-Gly基序结合RNA,并且在低复杂性区域使用Arg-Gly-Gly基序直接结合RNA聚合酶II的磷酸化羧基末端结构域(Zhouetal., 2019)。剪接因子hnRNP A通过其RRMs与外显子剪接沉默子结合,但C端富含甘氨酸的低复杂度区域才是抑制外显子被剪接的效应区域(Gatto-Konczak, 1999);Western blot的结果显示,BmSQD在卵巢、精巢、翅原基和胚胎组织中存在3条蛋白条带(图10),推测较小的两个条带可能是BmSQD的两种同源异构体,较大的蛋白条带是经过磷酸化修饰的SQD(姜铮等,2009);生信分析结果显示,BmSQD的低复杂度区域所在的250-300 aa包含众多潜在的酪氨酸磷酸化位点(图1,图2),暗示低复杂度区域可能受到磷酸化作用后发挥功能。因此推测,位于C端富含某类氨基酸且经过磷酸化后的低复杂度区域可能是hnRNP类剪接因子促进或抑制pre-mRNA剪接的效应结构域,即BmSQDC端富含甘氨酸的低复杂度区域可能是主要发挥促进或抑制pre-mRNA剪接作用的效应区域。
hnRNPs是一类与mRNA生物学功能密切相关的RNA结合蛋白,它能与pre-mRNA结合形成复合体并参与一系列重要的生命功能如mRNA转运、代谢、剪接等过程的调控(邵羊阳等,2017)。BmSQD在L5D4和L5D6幼虫时期的翅原基中表达水平也较高(图9-A),在L5D3和L5D5时期蛋白水平也较高,推测在幼虫的生长发育过程中,SQD作为一类hnRNPs参与了重要的生命功能,如通过mRNA转运、代谢、剪接等过程,调控幼虫的正常进食行为和常规发育过程。其中的hnRNPA1在真核细胞hnRNP复合物中含量十分丰富,是最早发现作用于前体mRNA剪接过程的。在家蚕中,BmhnRNPA1具有两个RNA结合结构域,属于hnRNPs家族,定位于细胞核内,表明其可能参与mRNA的选择性剪接作用(张康,2019)。hnRNPs蛋白家族与人体健康密切相关,参与了多种疾病的发生过程,如病毒疾病、肿瘤疾病、自身免疫性疾病等,也参与特异基因可变剪切的调节(陈尧等,2018)。本研究表明,作为一类hnRNPs蛋白的BmSQD在5龄第5天幼虫翅原基中定位于细胞核中(图11),其也可能存在相应的剪接调控功能,参与调节许多生命活动,对家蚕的生长发育有至关重要的作用。
果蝇中,hrp40是由Squid基因编码的,作为一类hnRNPs,参与了果蝇发育过程中的选择性剪接过程的调控(Marcoetal., 2009);hrp40是卵形成过程中背部腹侧轴形成所必需的,来自Squid纯合子亲本的卵和胚胎严重背化,完全缺失Squid基因会导致胚胎致死(Matunisetal., 1994)。更有研究表明RNA结合蛋白Squid是果蝇卵母细胞建立前后极性所必需的(Steinhauer and Kalderon, 2005);在果蝇卵发生过程中,Squid不仅是oskarmRNA有效定位于卵后部的必要条件(Norvelletal., 2005),还介导了果蝇中未定位gurken(grk)mRNA的翻译抑制,其中gurken(grk)mRNA与蛋白的正确定位是果蝇卵子和未来胚胎背腹轴建立的必要条件(Clouseetal., 2007)。综上表明,SQD在果蝇中对卵子形成和胚胎发育具有重要意义,基于SQD在昆虫进化上的高度相似保守性,推测BmSQD在家蚕卵子形成和胚胎发育方面也具有重要作用。在本研究结果中,BmSQD在产卵后72 h处于发育阶段的胚胎中有高表达,主要定位在细胞核内(图12),且SQD在家蚕主要生长组织均有表达,其中卵巢、精巢、表皮和中肠表达量较高,暗示BmSQD在家蚕生长发育过程中有重要作用;推测定位在细胞核中的BmSQD具有进入细胞核并参与RNA转录剪接调控的功能,可通过对卵巢、精巢、表皮、中肠和翅原基等组织器官发育的剪接调控进而调控家蚕胚胎发育和变态发育过程。
