基于OpenMP的ANGSD软件优化设计和可行性研究
2023-05-23李金光唐友李丹
李金光 唐友 李丹
基金项目:吉林省科技发展计划项目;项目名称:基于数据挖掘技术的全基因组选择方法研发及云计算平台体系构建;项目编号:YDZJ202201ZYTS692。
作者简介:李金光(1999— ),男,河北廊坊人,硕士研究生;研究方向:先进控制技术与控制系统集成。
*通信作者:李丹(1980— ),男,吉林吉林人,副教授,博士研究生;研究方向:电气工程。
摘要:近年来,多核技术在基因测序方面有着广泛的应用。因此,基因测序的并行化成为目前国内热点。在基因测序领域,随着高通量测序技术的迅猛发展和应用越加广泛,其无时无刻不在产生大量的数据。对此,串行程序进行单个程序运行已经无法满足社会对基因测序的需求。为了同时分析数千个样本,测序需要快速、灵活和内存高效地实现。文章认为,对一个名为ANGSD的程序进行OpenMP的程序并行的软件优化设计和可行性研究非常必要。
关键词:OpenMP;下一代测序;多线程
中图分类号:TP39 文献标志码:A
0 引言
测序技术经过多年发展拥有了3个阶段成果,分别是第一代测序技术、下一代测序技术(NGS)和最新的第三代单分子测序技术。下一代测序技术(Next Generation Sequencing,NGS)又被叫作高通量测序技术,此技术一次测序可以检测几十万到几百万条序列。由于此技术具有高通量、强稳定、高准确度等优点,因此在商业上被广泛应用[1]。
现有的通用多样本NGS分析程序的例子有单线程SAMtools (C)和多线程GATK(Java)以及ANGSD。这3个程序之间有许多不同之处,但ANGSD的关键优势在于:(1)允许与原始测序数据直接相关的多种输入数据类型(文本堆积、二进制基因型可能性文件、VCF文件);(2)允许用户选择多种方法中间分析(例如计算GL的不同方法);(3)包括一组在任何其他软件中都没有实现的下游分析的实现。ANGSD中很多方法都是基于GLs。ANGSD支持4种不同的模型来计算GLs:(1)重新校准的SOAPsnp模型;(2)原GATK模型;(3)SAMtools 1.16 +修改Maq模型;(4)特定类型的错误模型。这些GL模型中的排序错误率要么是固定的(由qscores获得),要么是从数据中估计出来的。4个实现的GL模型假设为二倍体样本。工作流程分为2个步骤:(1)ANGSD生成特定的分析输入数据。(2)根据ANGSD输出,使用二级关联程序进行下游分析。对于简单的测试,例如ABBA-BABA/D-statistic,次要程序可以是简单的Rscript;对于计算密集型的方法,次要程序可以是多线程的c/c++程序[2]。OpenMP是多核系统最流行的并行编程之一。因此,本文就OpenMP并行在ANGSD软件的应用可行性做展开论述。
1 研究意义
20世纪70年代,Walter Gilbert和Frederick Sanger发明了人类史上第一台测序仪,并通过测序仪探测到第一个基因组序列噬菌体X174,全长拥有5 375个碱基。
在Sanger基因测序技术的催动下,人们对生命的本质产生极大的兴趣。在此动力下,人们对生命科学的研究逐渐步入了基因组学的时代。40多年來,人们通过不断努力已将测序技术发展到了可观的境界,已经由第一代发展到了第三代测序技术。
由Sanger发明的最初的测序方法被人们称为第一代测序技术,虽然是第一代技术,但至今仍在被广泛应用。同时,它也有着无法掩盖的缺陷。它进行一次测序只能得到一条长度为700~1 000个碱基的序列,这个缺陷成为在世界舞台上更进一步的掣肘,无法满足现代科学发展所需要的庞大生物基因序列。
高通量测序(High-Throughput Sequencing,HTS)弥补了第一代基因测序的缺陷,运行一次便可以同时获得几十万至几百万条核酸分子的序列,因为在Sanger基因测序技术的基础上进行了突破,所以高通量测序也被称为新一代测序 (Next Generation Sequencing,NGS)或第二代测序。
第二代测序技术虽然对各个领域产生了极大的影响,但是也有缺点。第二代测序技术获得单条序列长度很短,如果想得到更为准确的基因序列信息,就不得不依赖于较高的测序覆盖度和更准确的序列拼接技术。由于技术问题,最终得到的结果中会存在一定量的错误信息,误导研究人员的判断。
为了防止结果中出现错误信息扰乱人的认知,科研人员在保证高通量测序的基础上,发明了第三代测序技术,也称为单分子测序技术(Single-molecule sequencing technology)。该技术在上一代测序技术上做出了突破,解决了单条序列长度过短的问题,如今能够直接得到长度在数万个碱基的核酸序列信息。
目前,基因测序技术在包括胚胎的植入前遗传学诊断研究、法医学的少量DNA测序、表观遗传学和考古学、物种进化演替过程等众多领域得到广泛应用。
就当前的市场势态而言,第二代短读长序列检测技术在世界测序技术市场上一直占据着绝对的优势地位,而第三代测序技术也在近年来实验中迅速发展。
若要进行ANGSD软件的简化提速,就需要对ANGSD软件运行的流程进行分析:可以通过程序运行来确定ANGSD读取数据、运算以及输出数据的时间占比;还可以通过Pthread和OpenMP的两种并行语言的对比进行分析,以此来判断ANGSD软件是否拥有简化的可能性。
2 ANGSD数据处理流程
ANGSD是一个多线程程序。该程序可以计算各种汇总统计,利用下一代测序数据中的完整信息,通过直接处理原始测序数据或使用基因型可能性,进行关联映射和群体遗传分析。该程序不仅支持多种输入格式,包括BAM和beagle基因型概率文件,而且允许用户在现有方法的组合之间进行选择,并可以执行其他地方没有实现的分析。此篇文章便对输入为BAM基因型概率的文件进行研究。
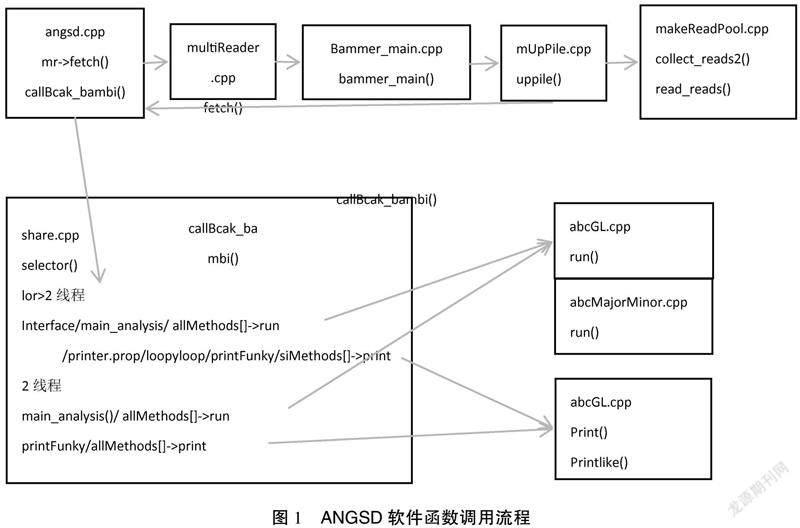
本文通过利用gdb调试工具,来判断程序函数的调用关系,得出图1为ANGSD软件的函数调用流程。命令如下:
gdb ./angsd
set args-GL 1-doGlf 2-b bam2.filelist-doMajorMinor 1 -doMaf 1
b angsd.cpp:main
b shared.cpp:main_analysis
set print pretty on
r
以上已经得出ANGSD软件运行处理的流程,接下来为了更直观地表现出哪一部分在软件中可以进行简化更改,本文对程序选取不同规格的Bam文件进行测序,观测在虚拟机以及服务器的运行速度。
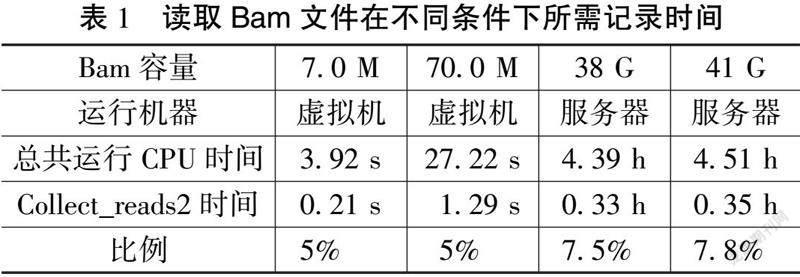
从表1可以发现不同容量Bam文件分别在reads运行时间占CPU总时间的5%,5%,7.5%,7.8%,所以可以得出结论读取Bam文件reads记录时间约为总时间的7%。
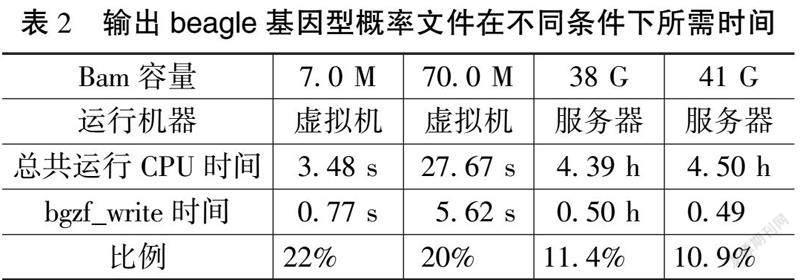
从表2可以发现不同容量Bam文件分别在输出beagle基因型概率文件时间占CPU总时间的22%,20%,11.4%,10.9%,所以可以得出结论输出beagle基因型概率文件约为总时间的10%。
从表3可以发现不同容量Bam文件分别在计算基因型概率时间占CPU总时间的7%,10%,16%,19%,所以可以得出结论:计算基因型概率时间约为总时间的20%。
通过上述数据可以发现ANGSD软件拥有进一步提速的可能性,接下来本文将对其软件加速的方案进行探讨分析。
3 两种并行方式简介
3.1 OpenMP并行
并行计算机可简单分成共享内存和分布式存储器。共享内存是指几个内核之间共用的一条存储器。而对只注重计算、只需要对线程间关系做出最基本控制的应用而言,OpenMP十分适用。OpenMP属于半自动多核并行,它介于手工并行和全自动并行之间。它通过两方面实现并行化:(1)依靠编译器来实现多核并行化;(2)需要程序员指定并行的代码段或分配的粒度。
通常情况下,人工介入的部分只需要加上注释或是标注,相比手工并行,半自动并行更加通俗易懂[3] 。
OpenMP是一个应用程序接口(API),由一组主要的计算机硬件和软件供应商联合定义。OpenMP为共享内存并行应用程序的开发人员提供了一个可移植、可伸缩的模型。该API在多种体系结构上支持C/C++和Fortran。
OpenMP还给出了对并行算法的高层抽象说明,更适用于在多核CPU计算机上的并行程序实现。它在多核心CPU的机器中采用OpenMP程序方式实现了并行计算,编译时通过在程序中附加的pragma命令,可以自动地对程序并行处理,使OpenMP减少了对并行程序实现的困难度和复杂性[4]。
3.2 Pthreads并行
POSIX线程(POSIX Threads,常被缩写为Pthr-eads)是POSIX的线程标准,定义了创建和操纵线程的一套API。Pthreads作为手工多核并行,程序员需要在程序中手动调用API来实现并行化。
学术界将符合POSIX 线程标准的库称为Pthreads,一般应用于Unix-likePOSIX 系统,例如Linux系统。但是Windows上的实现也存在,例如直接使用Windows API实现的第三方库pthreads-w32;而利用Windows的SFU/SUA子系统,则可以使用微软提供的一部分原生POSIX API 。
Pthreads API中大致共有100个函数调用,全都以“pthread_”开头,并可以分为4类:
(1)线程管理,例如创建线程、等待(join)线程、查询线程状态等。
(2)互斥锁(Mutex):创建、摧毁、锁定、解锁、设置属性等操作。
(3)条件变量(Condition Variable):创建、摧毁、等待、通知、设置与查询属性等操作。
(4)使用了互斥锁的线程间的同步管理。
不但如此,Pthreads还拥有一套贴近底层的API,可以用来实现对线程的精准控制,例如创建/取消/同步某一个线程,设置各个线程的属性,操作互斥锁和条件变量,进行线程间同步等功能。
3.3 OpenMP和Pthreads共用模型
Pthreads和OpenMP都是多線程编程模型,这两种并行语言通常采用如图2所示的fork-join执行。fork递归地将任务分解为较小的独立子任务,直到它们足够简单以便异步执行。join将所有子任务的结果递归地连接成单个结果,或者在返回void的任务的情况下,程序只是等待每个子任务执行完毕。
4 ANGSD软件两种并行方式区别
ANGSD作为高通量测序的一个软件在基因测序领域有着不可忽视的作用。该程序使用的是Pthreads并行,而本文进行对比的是OpenMP并行,虽然都是针对共享内存而编写的API,但是这两者存在着本质上的区别。Pthreads要求程序员直接表明对一个线程的执行情况;而OpenMP则相对而言要求宽松很多,它允许程序员只需简短地说明哪条程序需要并行执行,届时将由编译器和所执行时的操作系统来决定具体的线程去执行某个任务。这种差异也证明了为什么共享内存程序同时具有两种完全不同的API。不过在Pthreads所有线程的行为中的细节都必须由程序员确定。与此相反,OpenMP最主要的优势之一,便是它的设计使编写者能够逐步地并行现有的串联型程式,而不会再从零开始写并行程序。这可以极大地简化编程烦琐的程序,因此把Pthreads更换为OpenMP并行进行简化是可行的[5] 。
5 结语
本文探讨的内容对ANGSD软件加速的可能性进行了初步的分析,ANGSD软件执行的流程图以及Bam文件在虚拟机以及服务器的运行也证明了其软件是有加速的可行性的。对比Pthreads和OpenMP的优缺点也可以说明OpenMP相对Pthreads而言具有更为简化的编程方式,因此接下来的工作就可以对ANGSD软件进行程序的更改,提高其运行的速度。
參考文献
[1]黄芝准.组学大数据环境下的基因信息并行处理与分析方法研究[D].合肥:中国科学技术大学,2017.
[2]DURVASULA A,HOFFMAN P J,KENT T V,et al. Angsd-wrapper:utilities for analysing next-generation sequencing data.[J]. Molecular ecology resources,2016(6):1449-1450.
[3]戴晨,陈鹏,杨冬蕾,等.面向多核的并行编程和优化研究[J].计算机应用与软件,2013(12):198-202,279.
[4]产院东.基于多核和众核平台的并行DNA序列比对算法[D].济南:山东大学,2019.
[5]段皞一.MPI、OpenMP、Taichi并行编程语言探究[J].电子元器件与信息技术,2022(4):123-134.
(编辑 王永超)
Abstract: In recent years, multi-core technology has been widely used in gene sequencing, so the parallelization of gene sequencing has become a hot topic in China. In the field of gene sequencing, with the rapid development and application of high-throughput sequencing technology, a large amount of data is generated all the time, and the single program operation of this serial program cannot meet the needs of society for gene sequencing. In order to analyze thousands of samples simultaneously, we need fast, flexible and memory-efficient implementations. OpenMP program parallelism for a program called ANGSD is very necessary.
Key words: OpenMP; next-generation sequencing; multithreading
