基于基因组微卫星的团头鲂养殖群体遗传多样性分析和指纹图谱构建
2023-05-23张芹王延晖王冰柯屈长义刘英杰
张芹 王延晖 王冰柯 屈长义 刘英杰


摘要 利用基因組微卫星检测2个团头鲂(Megalobrama amblycephala)养殖群体的遗传多样性,18个位点共检测到126个等位基因,大小在135~362 bp。各位点观测等位基因数(Na)在3~15个,平均为7个;有效等位基因数(N)在1.250~6.979,平均为3.603。各位点检测到的香农信息指数(I)在0.417~2.213,平均为1.384。观测杂合度(H)在0.219~0.856,平均值为0.647;期望杂合度(H)在0.200~0.857,平均值为0.665。多态信息含量指数(PIC)在0.190~0.843,平均值为0.624。群体间的遗传分化指数(F)为0.070,群体间的基因交流(N)为1.59,说明HH和YH 2个群体之间有一定程度基因流。2个位点检测到的近交系数(F)>0,其他16个位点近交系数均<0。研究表明,HH和YH两个人工养殖群体中均有较高的遗传多样性,群体间有一定程度的基因交流,指纹图谱的构建可以用来进行团头鲂种质资源品种间的鉴定以及品种内的亲缘关系研究,尤其是可以区分没有亲缘关系数据背景的群体,可以指导没有家系构建能力的苗种繁育场进行繁殖策略的升级。
关键词团头鲂;基因组;SSR;多态性
中图分类号S917文献标识码A
文章编号0517-6611(2023)08-0099-06
doi:10.3969/j.issn.0517-6611.2023.08.023开放科学(资源服务)标识码(OSID):
Genetic Diversity Analysis and Fingerprint Construction for Two Cultured Populations of Megalobrama amblycephala on Genomic Microsatellites
ZHANG Qin,WANG Yan-hui,WANG Bing-ke et al(Henan Academy of Fishery Sciences,Zhengzhou,Henan 450044)
AbstractIn this study,the genetic diversity for two populations of Megalobrama amblycephala was detected using genomic SSR.The results showed that 126 alleles were detected in 18 microsatellite loci,ranging from 135 bp to 362 bp.The observed heterozygosity ranged from 3 to 15,with mean value of 7.The expected heterozygosity ranged from 1.250 to 6.979,with mean value of 3.603.The Shannon information index was between 0.417 and 2.213,with an average of 1.384.The observed heterozygosity was between 0.219 and 0.856,with an average of 0.647.The expected heterozygosity was between 0.200 and 0.857,with an average value of 0.665.The polymorphism information content ranged from 0.190 to 0.843,with mean value of 0.624.The genetic differentiation index between the populations was 0.07,and the gene flow was 1.59,indicating that there was a certain degree of gene flow between the two populations.The inbreeding coefficients at two loci were greater than zero,and at the other 16 loci were all less than zero.In summary,the genetic diversity among the two pupulaitons was mid-high,and there was a certain degree of gene exchange between the two populations,which showed that all the artificial selection breeding strategies for the two cultural populations could maintain population diversity.There was a large genetic difference between wild individuals and cultured populations,which could be used as an effective supplement for the genetic diversity of Megalobrama amblycephala in later breeding process.Using these tags,the fingerprint was constructed for two Megalobrama amblycephala group.These microsatellite loci can be used for identification and genetic relationship research of megalobrama amblycephala germplasm resources.Especially,these tags can be used to distinguish pupulations with no pedigree data background and then to guide strategies for breeding ground without family building capacity.
Key wordsMegalobrama amblycephala;Genome;SSR;Polymorphism
团头鲂(Megalobrama amblycephala)为我国特有的淡水经济鱼类,因其生长快、成活率高、易捕捞、肉质好等优点,在全国各地均有养殖。团头鲂“华海1号”水产新品种经第五届全国水产原种和良种审定委员会审定通过,具有遗传性状稳定、生长快、成活率高等优良特征[1]。“伊河鲂”在分类上属于鲂属团头鲂,是近年来开发黄河流域土著鱼类品种时,选育出来的一个优良新品系[2]。微卫星标记(microsatellite)由于重复性好,多态性高,广泛应用于水生动物的种质资源鉴定以及遗传多样性研究[3-4],尤其是利用SSR的多态性对水生生物进行亲子鉴定的较多[5-7]。李宝玉等[8]利用微卫星标记对团头鲂二倍体和三倍体群体进行遗传特性分析,Xiong等[9]利用微卫星技术对团头鲂群体不同家系的肌间刺数量进行研究,而有关团头鲂基因组微卫星研究鲜见报道。笔者对团头鲂基因组SSR序列进行分析,筛选得到若干多态性微卫星SSR标记,笔者利用这些标记,对2个团头鲂养殖群体进行遗传多样性研究和指纹图谱的构建,旨在为团头鲂种质资源品种间的鉴定以及品种内的亲缘关系研究提供科学依据。
1材料与方法
1.1材料在河南省嵩县陆浑水库伊河鲂鱼种厂采集团头鲂伊河群体100尾,以下简称YH群体;从湖北省鄂州国家级团头鲂原种场采集团头鲂“华海1号”群体100尾,以下简称HH群体;在湖北省鄂州市梁子湖采集野生团头鲂个体2尾,以下简称LZH个体。所有个体测量体长、体重,剪尾鳍用无水乙醇保存备用。
1.2方法
1.2.1团头鲂DNA的提取。团头鲂DNA用磁珠法基因组DNA提取试剂盒(天根生化)提取,经 1% 琼脂糖凝胶电泳检测,用超微量分光光度计(NanoDrop One spectrophotometer)检测 DNA 浓度和纯度,置于 -20 ℃低温保存。
1.2.2PCR扩增及SSR检测。挑选出18对微卫星引物[10],使用 M13 通用接头序列(TGTAAAACGACGGCC AGT)加到每对引物的 F 引物的 5′方向,并合成带不同荧光基团的 M13 接头序列。
PCR 反应体系(15 μL):2×Taq PCR Master Mix 7.5 μL,Mix primer 2.0 μL,基因组DNA模板 1.0 μL(50~200ng),dd HO 4.5 μL。PCR扩增条件:96 ℃ 预变性 3 min(96 ℃ 变性 30 s,59~60 ℃退火 30 s,72 ℃ 延伸1 min)共 30个循环,72 ℃ 终延伸 10 min,12 ℃保存。
1.2.3荧光毛细管电泳检测。取3 μL荧光PCR产物进行琼脂糖凝胶电泳鉴定,检测PCR条带是否单一、片段大小是否与预期一致。条带单一且大小相符的,对照DNA Marker的浓度进行定量,将所有产物稀释至相同的浓度范围,取1 μL PCR稀释产物加10 μL甲酰胺(含0.5μL内标)变性后上DNA测序仪ABI 3730xl进行毛细管荧光电泳检测。
1.2.4结果整理。将毛细管电泳检测结果导入GeneMarkerV2.2.0 分析软件中对原始数据进行条带分型,每对引物导出PDF峰图文件,结果汇总统计后进行分析。
各位点等位基因数(N)、有效等位基因数(N)、观测杂合度(H)、期望杂合度(H)等遗传多样性参数使用GenAIex软件计算,各位點的多态信息含量指数(PIC)由Cervus软件计算,群体Hardy-Weinberg 平衡偏离用固定指数F衡量,使用GenAIex软件进行群体的哈代温伯格平衡检验。群体间的聚类分析运用 Phylip 软件对其进行 UPGMA方法构建系统发生树。群体间遗传分化系数(F)、地方群体内近交系数(F)、基因差异分化系数(G)、分子方差、PCA分析由GenAIex软件进行运算,热点图为R语言绘制。连锁不平衡使用R包poppr的pair.ia方法进行分析,图用R语言绘制。
2结果与分析
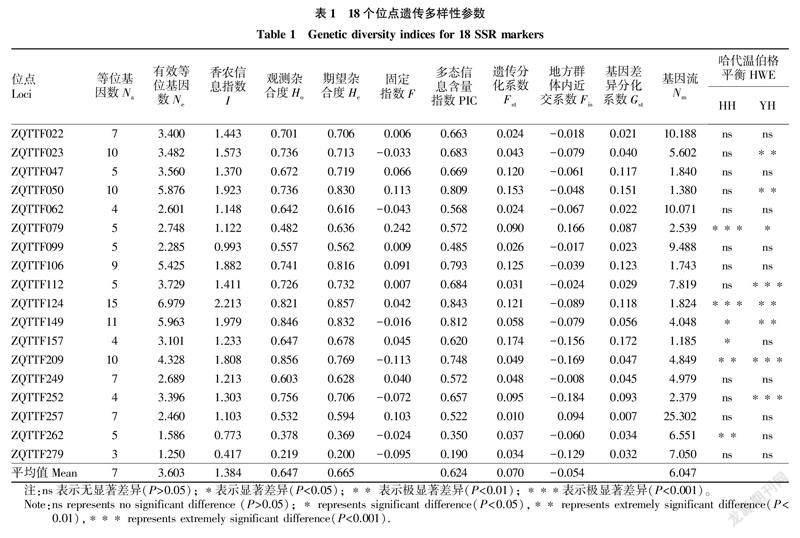
2.1各位点遗传多样性参数由表1可知,采用的基因组微卫星位点均为多态性位点,18个位点共检测到126个等位基因,大小在135~362 bp。各位点观测等位基因数目(N)在3~15个,平均数为7个;有效等位基因数(N)在1.250~6.979,平均值为3.603。各位点检测到的香农信息指数(I)在0.417~2.213,平均值为1.384。观测杂合度(H)在0.219~0.856,平均值为0.647;期望杂合度(He)在0.200~0.857,平均值为0.665。多态信息含量指数(PIC)在0.190~0.843,平均值为0.624。各位点检测到的遗传分化指数(F)值0.010~0.174,平均值为0.070。在ZQTTF079和ZQTTF257两个位点检测到的近交系数(F)>0,其他位点近交系数均<0。基因流(N)为1.185~25.302,平均值为6.047。HH群体在ZQTTF079、ZQTTF124、ZQTTF149、ZQTTF157、ZQTTF209和ZQTTF262位点上显著偏离哈代温伯格平衡,YH群体在ZQTTF023、ZQTTF050、ZQTTF079、ZQTTF112、ZQTTF124、ZQTTF149、ZQTTF209、ZQTTF252位点上显著偏离哈代温伯格平衡,其中ZQTTF079、ZQTTF124、ZQTTF149、ZQTTF209 位点在2个群体中都显著偏离哈代温伯格平衡。
2.22个群体各位点的遗传多样性参数由表2可知,HH群体检测到的观测等位基因数目平均值为5.889,有效等位基因数平均值为2.827,香农信息指数平均值为1.212,观测杂合度平均值为0.624,期望杂合度平均值为0.612;YH群体检测到的观测等位基因数目平均值为5.222,有效等位基因数平均值为3.057,香农信息指数平均值为1.202,观测杂合度平均值为0.671,期望杂合度平均值为0.616。
2.3群体遗传结构HH和YH 2个群体之间的F为0.07,说明二者间存在中等程度的分化。2个群体间的基因流(N)为1.59,说明二者间有一定程度基因交流。
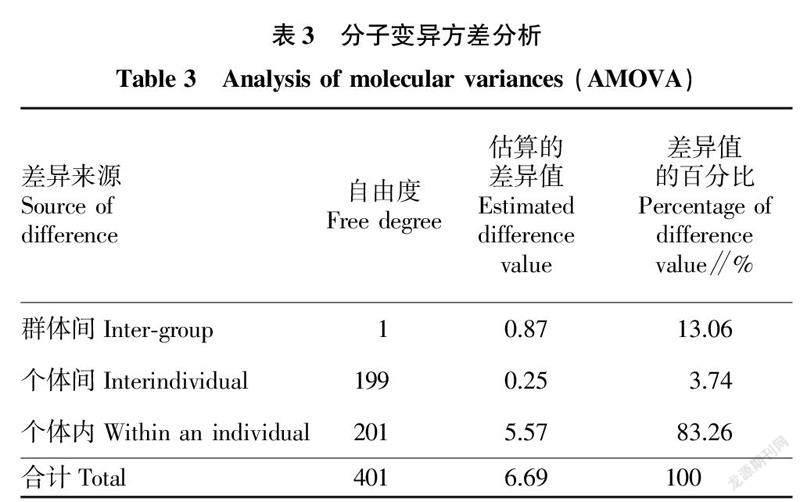
团头鲂群体的分子方差分析结果(表3)显示,群体间的差异占差异来源的13.06%,个体间的差异较小,差异的主要来源在个体内部,占总差异的83.26%。个体内部的差异是指由杂合等位基因引起的遗传差异,大小与个体杂合位点数相关,即个体的遗传多样性,由此可知2个养殖群体的个体内部存在着丰富的遗传多样性。
根据遗传距离绘制的群体间系统发生树(图1),YH群体和HH群体首先聚合在一起,然后再与LZH群体聚合在一起,说明YH群体和HH群体之间的遗传距离更近,而2个养殖群体离LZH群体之间的遗传距离更远。
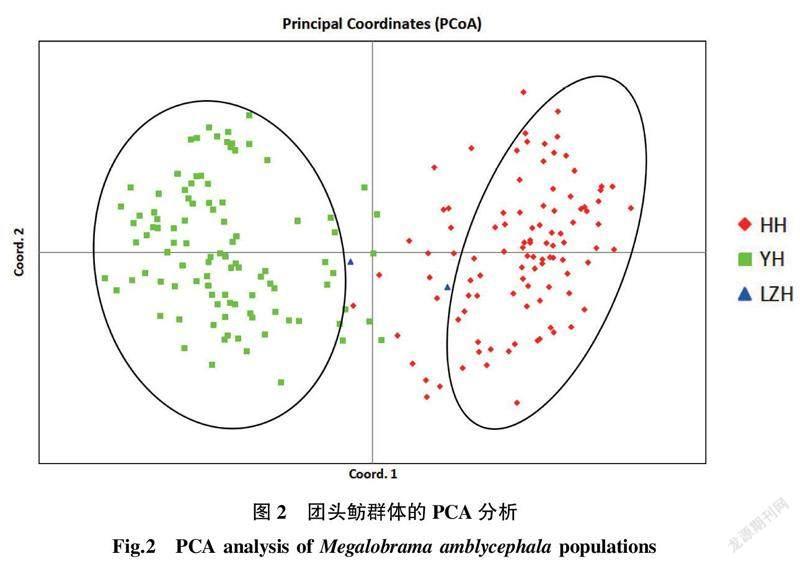
PCA(Principal co-ordinates analysis)用来研究样本群体组成的相似性或相异性(图2),HH群体的个体几乎集中分布在同一个区域,YH群体内的个体也是集中分布在另一个区域,而LZH的个体离2个集中分布的区域均较远,单独分布在一个区域。说明这3个群体之间存在一定程度的分化,个体之间有较明显的遗传差异。
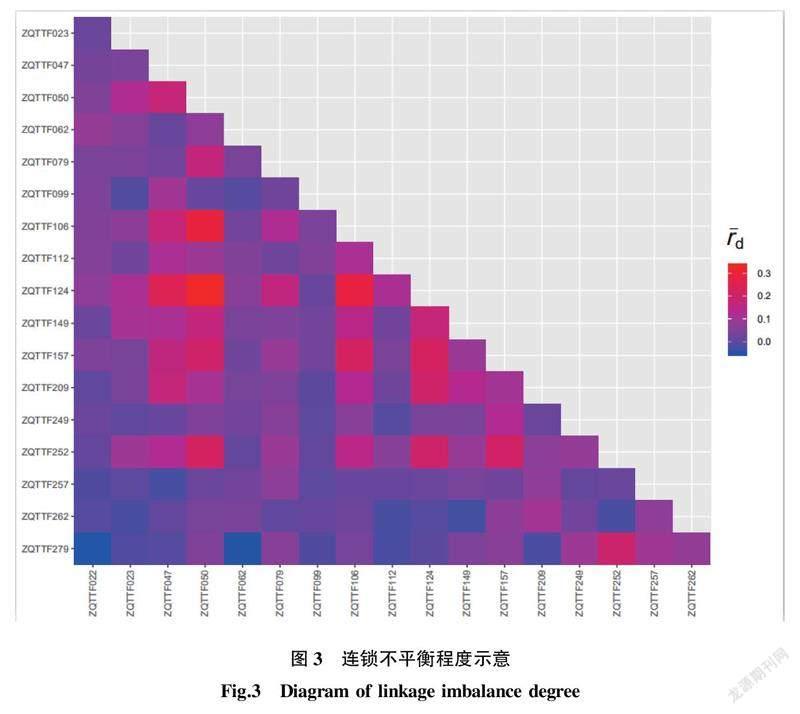
在连锁不平衡中,一般通过r值平方来表征连锁不平衡程度,18个位点两两之间r值平方在0.02~0.34(图3),说明位点之间存在一定程度的连锁不平衡。
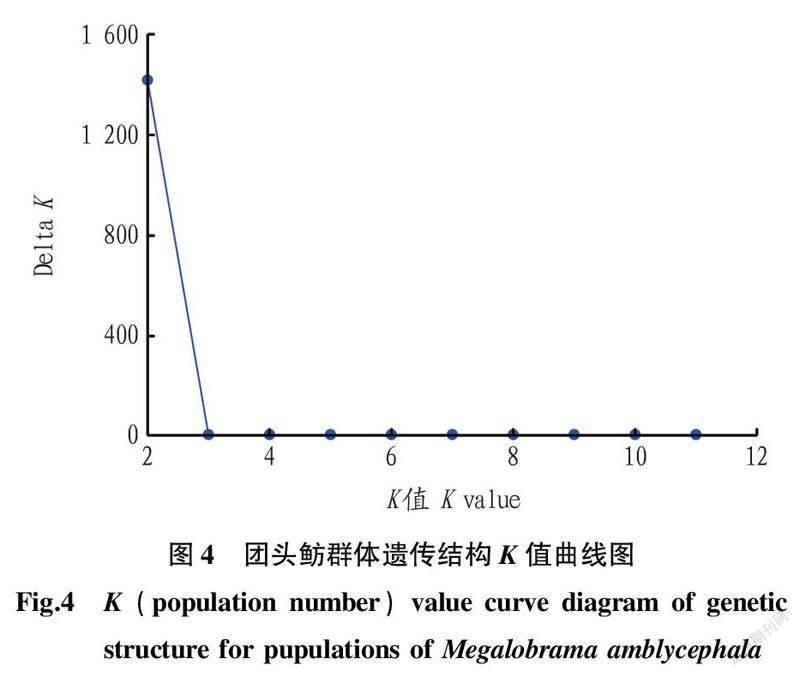
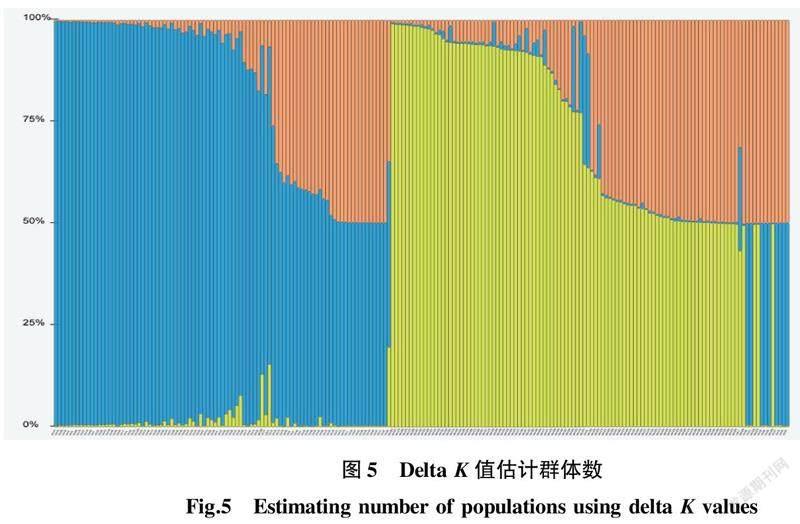
利用在线工具STRUCTURE HARVESTER 进行群体结构分析,参数设置如下:混合模型(admixture model);Burn-in值为10 000,MC值为100 000;K值设置为3~10,每个K值重复20次。软件对每个K值模拟的结果,都会对应产生最大似然值(likelihood),最大似然值是取自然对数后输出的数值(ln likelihood)。ln likelihood越大,说明K值越接近于真实情况。一般随着K值升高,ln likelihood值也会不断升高,但会慢慢进入平台期,选择最优K值的目标是要找到拐点。该研究最佳K值为3(图4),根据最佳K值估计群体数为3(图5),说明试验所检测的样品最佳可划分为3个群体。
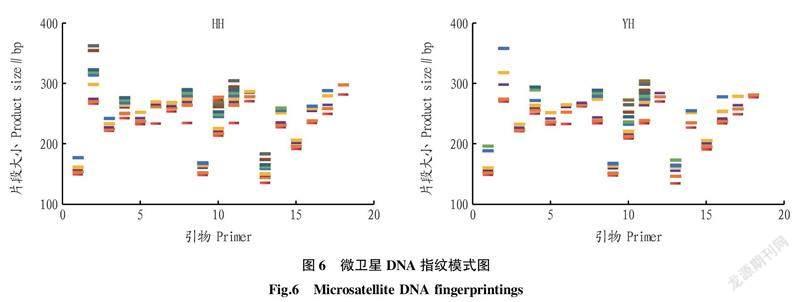
2.4利用DNA 指纹图谱鉴别2个团头鲂群体 根据2个养殖群体在 18 个微卫星位点上的扩增结果,按照表1中的编号顺序,利用EXCEL 作图软件构建这2个群体的 DNA 指纹模式图(图 6)。
从指纹图谱库中可以筛选出 6个特异的微卫星 DNA 标记(ZQTTF124、149、050、106、209和023)用以鉴别这 2 个群体,每个标记至少在其中一个群体中可以扩增出独有的条带,从而将这2个群体区分开。而将这6个标记配合使用,则可以将群体中99%的个体区分开来,这 6 个特异微卫星标记产生的多态位点可作为团头鲂育种过程中种质鉴定的依据。
3讨论
3.1群体遗传多样性分析该研究观测等位基因数和有效等位基因数差别较大,则等位基因在2个群体中分布不均匀。团头鲂养殖群体检测到的平均等位基因数目高于团头鲂二倍体和三倍体群体[8]、鳜鱼[11]、鲂鲌杂交及回交F2群体[12]、三角鲂×翘嘴鲌、团头鲂×翘嘴鲌2種杂交后代[13]、团头鲂耐低氧新品系雌核发育群体[14]和海蜇群体[15];观测等位基因数和有效等位基因数低于大口黑鲈[16]和刺参群体[17],高于花鲈群体[18]。等位基因情况反映出团头鲂2个养殖群体与其他水产养殖品种相比处于较高水平,一方面说明2个养殖群体保持了较高的遗传多样性,另一方面说明选择得到的基因组微卫星位点具有较高的遗传多样性,能够反映出团头鲂群体的遗传背景。
该研究团头鲂养殖群体检测到的观测杂合度和期望杂合度均低于团头鲂群体[9]和鳜鱼群体[11],高于三角鲂×翘嘴鲌、团头鲂×翘嘴鲌2种杂交后代[13]、海蜇群体[15]和花鲈群体[18]。观测杂合度高于刺参群体[17],低于团头鲂耐低氧新品系雌核发育群体[14];期望杂合度低于刺参群体[17],高于大口黑鲈群体[16]和团头鲂耐低氧新品系雌核发育群体[14]。观测杂合度和期望杂合度与鲂鲌杂交及回交F2群体[12]相比处于中等水平。综合来看,该研究团头鲂养殖群体的观测杂合度和期望杂合度与其他水产养殖品种相比处于中等水平。
通过固定指数可以衡量种群中基因型的实际频率是否偏离Hardy-Weinberg平衡[19],刺参[17]和团头鲂耐低氧新品系[14]中大量位点表现为杂合子过剩。HH群体在6个位点上显著偏离哈代温伯格平衡,YH群体在8个位点上显著偏离哈代温伯格平衡,说明人工选择育种过程给养殖群体造成了一定的选择压力。其中,4个位点在2个群体中都显著偏离哈代温伯格平衡,可见这4个位点在不同的选育策略中都承受了较大的选择压力,可在今后的选育过程中予以特别关注,以期进一步了解与位点相关的性状以及选育过程对其带来的影响。
该研究中微卫星位点检测到的多态信息含量指数(PIC)在0.190~0.843,平均值为0.624,高于鳜鱼群体[11]、三角鲂×翘嘴鲌、团头鲂×翘嘴鲌2种杂交后代[12]、团头鲂耐低氧新品系[14]和花鲈群体[18],低于团头鲂群体[9]和刺参群体[17],与鲂鲌杂交及回交群体[12]接近。2个团头鲂养殖群体的多态性较高,与等位基因衡量的结果一致。18个微卫星位点中有16个位点的PIC值均大于0.5,其中大于0.7的位点有5个,说明试验中采用的微卫星位点是高度多态的[20],能够为群体遗传信息监测提供丰富、高质量的数据。
3.2群体遗传结构分析遗传分化指数(F)是反映群体间遗传差异的重要参数[21],各位点检测到的遗传分化指数(F)值0.010~0.174,平均为0.070;在检测的18个位点中,9个位点属于轻度分化位点(0<F≤0.05),7个位点属于中度分化位点(0.05<F≤0.15),2个位点属于高度分化位点(0.15<F≤0.25)。群体间的F为0.070,说明2个群体属于中度分化群体[22],群体间遗传变异不显著。HH和YH 2个群体间的基因流(N)为1.59,说明2个养殖群体之间有一定程度基因交流。
基因差异分化系数(Gst)可以不必考虑地方群体形成历史、迁移方式、群体中等位基因数和基因型频率,只需将整个群体中杂合体频率分解为地方群体间变异和地方群体内变异[23]。各位点检测到的Gst范围在0.007~0.172,群体间有一定的遗传分化。
地方群体内近交系数(F)对地方群体内的繁育系统具有指示意义,在ZQTTF079和ZQTTF257 2个微卫星位点检测到的近交系数F>0,其他16个位点近交系数F<0,2个群体近交现象很少,2个群体在18个位点的F值的范围在-0.184~0.166,接近于0,說明群体比较接近于随机交配群体。各位点检测到的近交系数说明2个养殖群体在选育种过程中参与繁殖的亲本较多,群体保持较好的遗传多样性。
基因流(N)均大于1,就能有效抑制由遗传漂变而引起的遗传分化[24]。群体间基因流为1.59,群体间存在一定程度基因流,18个位点检测到的基因流(N)大于1,其中8个位点基因流大于5。推测原因可能是伊河鲂群体选育时间较短,河南地区养殖的团头鲂苗种较多来源于湖北地区,养殖群体逃逸及放流的个体给伊河流域的团头鲂种群带来了一定程度的基因交流。
群体最佳K值即为最佳群体分层情况[25-26],张智等[27]对西昌华吸鳅进行种群结构分析,计算得出K=2是比较理想的种群亚型类群数目,遗传分化分析结果相一致。高玉坤等[28]对马铃薯品种(系)的群体遗传结构进行分析, 65 个品种(系)划分为3个群体。该研究最佳K值为3,样品可划分为3个群体,与实际团头鲂采样群体情况一致,与群体间聚类结果也一致。
3.3团头鲂指纹图谱数据库的应用荧光标记SSR可以区分亲缘关系远的品种,也能将亲缘关系很近的个体区分开[29]。笔者利用 18 个微卫星标记,绘制了 2 个团头鲂养殖群体的微卫星 DNA 指纹图谱,筛选到 6 个能特异性鉴别这2个养殖群体的微卫星标记,将这6个标记配合使用,可以有效区分群体内的个体。该研究采集的样品没有遗传背景数据,筛选出的标记可以对这些个体进行准确区分,利用这些特异性谱带可以鉴定未知样本,是群体种质纯度鉴定的有效手段,可以用来指导亲本遗传背景不清晰或者没有家系构建能力的良种繁育场进行繁殖策略的升级。
4结论
HH和YH 2个人工养殖群体中的个体都有较高的遗传多样性,表明所采取的人工选择育种策略都能够保持群体的多样性。YH群体保持较大的亲本选育群体是检测到低水平近交的主要原因。由于长江禁渔,仅采集到很少量的野生个体,野生个体与养殖群体存在较大的遗传差异,可以作为后期育种过程中遗传多样性的有效补充。利用荧光标记SSR构建DNA指纹图谱,可以精确区分同一群体内的不同个体,在团头鲂土著品种进行抢救性选育过程中,可以建立有效的繁育体系,并对子代进行精准鉴定。
参考文献
[1] 王卫民,高泽霞,刘红,等.团头鲂“华海1号”[J].中国水产,2018(5):65-70.
[2] 张芹,孔令军,屈长义,等.伊河团头鲂2 种同工酶组织特异性分析[J].中国渔业质量与标准,2020,10(5):37-43.
[3] NAPORA-RUTKOWSKI ,RAKUS K,NOWAK Z,et al.Genetic diversity of common carp (Cyprinus carpio L.) strains breed in Poland based on microsatellite,AFLP,and mtDNA genotype data[J].Aquaculture,2017,473:433-442.
[4] DAS MAHAPATRA K,SAHOOL,SAHA J N,et al.Establishment of base population for selective breeding of catla (Catla catla) depending on phenotypic and microsatellite marker information[J].Journal of genetics,2018,97(5):1327-1337.
[5] LIU Y,CHEN Y Y,GONG Q,et al.Paternity assignment in the polyploid Acipenser dabryanus based on a novel microsatellite marker system[J].PLoS One,2017,12(9):1-15.
[6] XU X J,WANG G Z,ZENG C S,et al.Parentage determination of the mud crab Scylla paramamosain using microsatellite markers[J].Aquaculture research,2018,49(1):217-221.
[7] WANG P P,ZHAO C,CHEN S Q,et al.Parentage identification of Odontobutis potamophlia based on microsatellite DNA markers[J].Journal of genetics,2018,97(2):563-568.
[8] 李宝玉,郑国栋,崔文涛,等.团头鲂三倍体的微卫星遗传特征及生长性能分析[J].水产科学,2022,41(2):173-182.
[9] XIONG X M,ROBINSON N A,ZHOU J J,et al.Genetic parameter estimates for intermuscular bone in blunt snout bream (Megalobrama amblycephala) based on a microsatellite-based pedigree [J].Aquaculture,2019,502:371-377.
[10] 张芹,王延晖,王冰柯,等.团头鲂基因组微卫星特征及SSR位点开发[J].江苏农业科学,2022,50(3):79-85.
[11] 周梦晨,徐东坡,方弟安,等.6个不同群体鳜基于SSR的遗传多样性初步研究[J].中国农学通报,2020,36(26):147-152.
[12] 崔文涛,郑国栋,苏晓磊,等.鲂鲌杂交及回交F2群体的微卫星遗传结构及其生长性能分析[J].中國水产科学,2020,27(6):613-623.
[13] 苏晓磊,郑国栋,蒋霞云,等.三角鲂×翘嘴鲌、团头鲂×翘嘴鲌两种杂交后代微卫星遗传结构分析[J].水生生物学报,2019,43(2):264-271.
[14] 徐湛宁,李福贵,郑国栋,等.团头鲂耐低氧新品系雌核发育群体遗传结构的微卫星分析[J].水产学报,2017,41(3):330-338.
[15] 狄明玉,周遵春,李云峰,等.海蜇4个自然群体遗传多样性的微卫星标记分析[J].水产科学,2018,37(6):762-768.
[16] 苏胜彦,张林兵,李海洋,等.基于微卫星标记的大口黑鲈(Micropterus salmoides)原种和养殖群体遗传多样性和结构分析[J].浙江大学学报(农业与生命科学版),2020,46(6):687-698.
[17] 廖梅杰,王锦锦,李彬,等.基于SSR 标记的刺参不同地理群体的遗传结构分析及指纹图谱构建[J].渔业科学进展,2021,42(1):165-176.
[18] 黄皓,范嗣刚,王鹏飞,等.基于微卫星标记对6 个花鲈群体的遗传多样性分析[J].南方水产科学,2022,18(1):99-106.
[19] BOTSTEIN D,WHITE R L,SKOLNICK M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J].American journal of human genetics,1980,32(3):314-331.
[20] PURVIS I W,FRANKLIN I R.Major genes and QTL influencing wool production and quality:A review [J].Genetics selection evolution,2005,37 (S1):S97-S107.
[21] BALLOUX F,LUGON-MOULIN N.The estimation of population differentiation with microsatellite markers[J].Molecular ecology,2002,11(2):155-165.
[22] NEI M.Genetic distance between populations [J].The American naturalist,1972,106(949):283-292.
[23] NEI M.Analysis of gene diversity in subdivided populations [J].Proceedings of the national academy of sciences of the United States of American,1973,70(12):3321-3323.
[24] WRIGH T S.Evolution in Mendelian populations[J].Genetics,1931,16(2):97-159.
[25] PRITCHARD J K,STEPHENS M,DONNELLY P.Inference of population structure using multilocus genotype data[J].Genetics,2000,155(2):945-959.
[26] 刘清文,宋跃,李甲明,等.利用核心简单重复序列(SSR)标记分析西洋梨品种资源遗传多样性[J].农业生物技术学报,2015,23(5):579-587.
[27] 张智,俞丹,刘飞,等.西昌华吸鳅的微卫星引物筛选及赤水河四个地理种群的遗传多样性分析[J].水生生物学报,2019,43(6):1224-1230.
[28] 高玉坤,崔江慧,项晓冬,等.65个马铃薯品种(系)指纹图谱构建和遗传多样性分析[J].农业生物技术学报,2020,28(8):1363-1378.
[29] 陶乃奇,张斌,刘信凯,等.利用荧光标记SSR鉴别21个茶花新品种[J].植物学报,2019,54(1):37-45.
