外源褪黑素诱导下Hippo信号通路调控山羊绒生长mRNA和lncRNA的生物信息学分析
2023-05-11贾纯琰付绍印张文广
贾纯琰 付绍印 丽 春 张文广
(1.内蒙古农业大学 动物科学学院/内蒙古自治区动物遗传育种与繁殖重点实验室, 呼和浩特 010018;2.内蒙古农业大学 职业技术学院, 内蒙古 包头 014109;3.内蒙古农牧业科学院, 呼和浩特 010031;4.内蒙古民族大学 动物科技学院, 内蒙古 通辽028000;5.内蒙古农业基因组大数据工程研究中心, 呼和浩特 010000)
绒山羊基因组中潜藏着大量不能翻译成蛋白质,但可转录成RNA并在RNA水平行使调控功能的非编码RNA。目前对褪黑素调控山羊绒生长的非编码RNA的研究逐渐增多。褪黑素通过调节miRNA的表达诱导内蒙古绒山羊二次生绒[16];通过调节lncRNA MTC的表达激活NF-κB信号通路,从而调控辽宁绒山羊毛囊发育与羊绒生长[17];通过改变let-7家族重要成员的表达影响相应靶基因的表达,进而调控内蒙古绒山羊皮肤毛囊的周期性生长[18];与lncRNA lnc552通过Wnt信号通路调控陕北白绒山羊毛囊干细胞增殖与分化[19]。
在以往的褪黑素促山羊绒生长的研究中,更多关注能编码蛋白的mRNA功能,对不参与蛋白编码的lncRNA的功能研究较少;且以往的研究模式多为单个基因,很少从整体的、系统的角度研究所有基因间的互作;而以往研究调控山羊绒生长的信号通路中很少聚焦Hippo信号通路。Hippo信号通路对平衡哺乳动物皮肤生长和分化,调节毛囊形态发生至关重要[20],也是哺乳动物组织生长发育和分化过程中整合激素的一个关键元素。褪黑素信号和Hippo信号通路之间存在潜在的串扰[21]。本研究旨在从转录组学层面全面挖掘褪黑素作用下调控山羊绒生长的mRNA和lncRNA,揭示调控山羊绒生长的关键信号通路,可视化受Hippo信号通路调控的山羊绒生长候选基因集的mRNA和lncRNA网络调控关系,以期为深入探索褪黑素促绒生长的机理提供新的理论依据。
1 材料与方法
1.1 材料
6只成年试验母羊选自内蒙古罕山白绒山羊种羊场,随机分为对照组(C1、C2、C3)和褪黑素处理组(M1、M2、M3)各3个生物学重复。在阿鲁科尔沁旗牧户家放牧饲养。收牧后每天进行补饲,粗饲料以苜蓿草、玉米秸秆和当地草地上的杂草为主,精料主要是玉米。以自然年(完整的绒毛生长周期)为试验周期,每隔一个月按照每千克体重2 mg的剂量,使用兽用皮下埋植器(江苏泰兴市医用器械厂)在试验羊(M1、M2、M3)耳后皮下埋植褪黑素(中国农业科学院左家特产研究所研制)。每月用剪毛剪刀在试验羊肩胛骨后缘接近体中线处贴近皮肤剪下1 cm×1 cm 面积的全毛,并用剃刀将皮肤上残留的绒毛刮干净,酒精擦拭、碘酒消毒,用手术刀片割取1 cm×1 cm 面积的皮肤样本(伤口同时迅速涂抹云南白药粉止血),迅速放入5 mL无菌冻存管中,立即保存于液氮罐带回实验室,再转入-80 ℃冰箱保存。对照组和褪黑素处理组全年12个月的样本分别记为C1~C12和M1~M12。
1.2 方法
1.2.1 RNA制备、文库构建、上机测序
使用TRIZOL(Invitrogen)对72份皮肤组织样本进行RNA提取。RNA Nano 6 000试剂盒(Agilent)检测RNA样品的完整性和浓度,Ribo-ZeroTM试剂盒去除rRNA,RNA文库制备试剂盒(NEB)构建cDNA文库,Illumina HiSeq 2500测序仪进行双端测序。
1.2.2 质控、组装转录组
对日光温室茄子栽培技术进行研究,特别是在育苗、定植、施肥、浇水以及后期的收获和管理中要科学分析,并借助生物防治以及化学防治的方式防治病虫害,避免大面积的病虫害感染影响茄子的产量和质量。
使用FASTQC软件对RNA-seq产生的原始下机序列(Raw Reads)质控得到高质量序列(Clean Reads)。Bowtie2(v2.2.3)程序建立参考基因组的索引,山羊参考基因组(CHIR_2.0)及基因组gtf注释文件从NCBI下载(https:∥www.ncbi.nlm.nih.gov/genome/?term=goat),Tophat2(v2.1.1)程序将双端测序的 clean reads比对到参考基因组[22]。Cufflinks (v2.2.1) 程序依托参考基因组的gtf注释文件组装出转录组[23]。
1.2.3 筛选差异表达mRNA和lncRNA
使用Cuffdiff (v2.2.1) 程序分析绒山羊皮肤中自然年每月对照组和埋植组的差异表达mRNA(Differentially expressed mRNA, DE-mRNA)和转录本(FDR<0.05)[24],Cuffcompare (v2.2.1) 程序对差异表达转录本进行分类,筛选类别代码是“i”、“j”、“o”、“u”、“x”的作为新转录本的候选。CPAT (v1.2.2)程序和在线CPC (http:∥cpc.cbi.pku.edu.cn)识别差异表达lncRNA(Differentially expressed lncRNA, DE-lncRNA)。
1.2.4 DE-mRNA和DE-lncRNA的PCA及WGCNA分析
2 024个DE-mRNA和329个DE-lncRNA表达量数据的主成分分析(Principal component analysis, PCA)由R(v3.4.0)软件完成。依据WGCNA官网教程(http:∥www.genetics.ucla.edu/labs/horvath/CoexpressionNetwork/Rpackages/WGCNA)由R软件(v3.4.0)完成DE-mRNA和DE-lncRNA的WGCNA分析[25]。函数PickSoftThreshold进行无尺度拓扑分析,选择软阈值β=7调用函数adjacency计算基因间的邻接性,函数TOMdist将邻接矩阵转换为拓扑重叠矩阵(Topological overlap matrix, TOM)[26]。基于TOM矩阵调用函数hclust绘制基因聚类树状图,聚类树的分枝是表达模式相似的一组基因即基因模块[27]。函数ModuleEigengenes计算模块的特征基因值,通过模块特征基因值的相关性对模块聚类以研究模块之间的关系。采用基于Java运行环境的Cytoscape(v3.6.0)软件插件ClueGo(v3.4.2)进行基因模块的功能富集分析[28]。选取平衡哺乳动物皮肤生长和分化、调节哺乳动物毛囊形态发生的Hippo信号通路基因模块进行深入分析[29]。
1.2.5 可视化调控山羊绒生长的候选基因集
由R软件 (v3.4.0) 导出单个目标模块的全部加权网络关系,即对照组品红色(Magenta)基因模块,埋植组深蓝色(Mid-night-blue)、棕褐色(Tan)和橙色(Salmon)基因模块。Excel(v2016)筛选对照组品红色(Magenta)基因模块边(edges)的信息文件中,与富集到Hippo信号通路的17个基因、皮肤发育的18个基因、细胞周期的14个基因、中间纤维的14个基因中的每一个基因Weight值(即TOM值)排名Top10的基因。埋植组操作同上。
1.2.6 调控山羊绒生长候选基因集的功能透视
由R软件(v 4.1.2)AnnotationHub程序包构建山羊OrgDb,clusterProfiler程序包获取山羊GO数据库里GO.BP皮肤发育和GO.CC中间纤维的gene list[30]。KEGGREST程序包获取山羊Hippo信号通路chx04390、细胞周期chx04110、氧化磷酸化chx00190、Wnt信号通路chx04310的gene list。dplyr程序包制作gmt文件给GSEA(Gene set enrichment analysis)软件(v 4.1.0)进行分析。R软件(v4.1.2)pheatmap程序包完成候选基因集与样本的关联分析。候选基因集中转录因子和转录辅因子的注释基于动物转录因子数据库(http:∥bioinfo.life.hust.edu.cn/AnimalTFDB)[31]。
2 结果与分析
2.1 受外源褪黑素诱导的绒山羊皮肤中DE-mRNA和DE-lncRNA
分析埋植组和对照组的全年绒山羊皮肤组织样本共筛选得到2 024个DE-mRNA和329个DE-lncRNA(FDR<0.05)。DE-mRNA(图1(a))和DE-lncRNA(图1(b))火山图的每个点分别代表1个mRNA和1个lncRNA。与对照组相比,受外源褪黑素影响,表达量显著上调2倍及以上的mRNA有522个(图1(a)红点)、lncRNA有128个(图1(b)红点),表达量显著下调2倍及以上的mRNA有400个(图1(a)绿点)、lncRNA有81个(图1(b)绿点),表达量上调及下调小于2倍的mRNA有1 102个(图1(a)灰点)、lncRNA有120个(图1(b)灰点)。

图1(a)和图1(b)红色点表示上调2倍及以上的mRNA和lncRNA即log2FC≥1;绿色点表示下调2倍及以上的mRNA和lncRNA即log2FC≤-1;log2FC介于-1和1之间的mRNA和lncRNA用灰色点表示。The red dots in figures 1(a) and 1(b) indicated that mRNA和lncRNA were up-regulated by two times or more, i.e. log2FC≥1; the green dots indicated that mRNA和lncRNA were down-regulated by two times or more, i.e. log2FC≤-1; the mRNA和lncRNA with log2FC between -1 and 1 were represented by gray dots.图1 受外源褪黑素诱导的绒山羊皮肤中差异表达mRNA(a)和差异表达lncRNA(b)的火山图Fig.1 Volcano plot of DE-mRNA (a) and DE-lncRNA (b) induced by exogenous melatonin in cashmere goat skin
2.2 受外源褪黑素诱导的绒山羊皮肤中DE-mRNA和DE-lncRNA的PCA分析
基于2 024个DE-mRNA和329个DE-lncRNA的RNA-Seq表达量数据共同加权得到主成分1(Dim1)和主成分2(Dim2),埋植组和对照组的全年24个样本聚类的二维PCA散点图(图2)。Dim1 和Dim2分别占总体方差变异的39.9%和12.8%,Dim1+Dim2占总体方差的50%以上,既实现了降低数据维度为二维,又保证了信息损失最小。与埋植组相比,对照组样本更离散(红色散点),对照组来自正常生理状态下绒山羊皮肤组织,对次级毛囊生长周期的变化更敏感,5~7月(生长前期)、8~9月(生长期)、10~12月(生长旺盛期)、1~2月(退行期)、3~4月(休止期)聚集在一起,这应答了次级毛囊的正常生长周期。相比之下,埋植组的样本处于相同的状态(外源褪黑素诱导下)聚类在一起(绿色散点),4~7月(产夏季绒)、8~12月(产冬季绒)、1~3月(休止期)聚在一起,这应答了褪黑素埋植组的产绒周期。此结果表明DE-mRNA和DE-lncRNA应答了绒山羊皮肤次级毛囊的生长周期。

对照组和埋植组全年12个月的样本分别以C1~C12和M1~M12表示。The 12-month samples of the control and implanted group were expressed as C1-C12 and M1-M12, respectively.图2 受外源褪黑素诱导的绒山羊皮肤中差异表达mRNA和差异表达lncRNA的PCA散点图Fig.2 PCA scatter plot of DE-mRNA and DE-lncRNA induced by exogenous melatonin in cashmere goat skin
2.3 受外源褪黑素诱导的绒山羊皮肤中DE-mRNA和DE-lncRNA的WGCNA分析
2 024个DE-mRNA和329个DE-lncRNA经WGCNA分析,对照组和埋植组分别得到7个和9个高度关联的基因模块,每个基因模块都有唯一的颜色标识,其余关联较差的基因被归入灰色模块结果见表1。表1的基因模块经ClueGo功能富集分析,选取富集到Hippo信号通路的对照组品红色(Magenta)模块和埋植组深蓝色(Mid-night-blue)模块进行深入分析,目标模块显著富集到的GO条目或KEGG通路结果见表2。
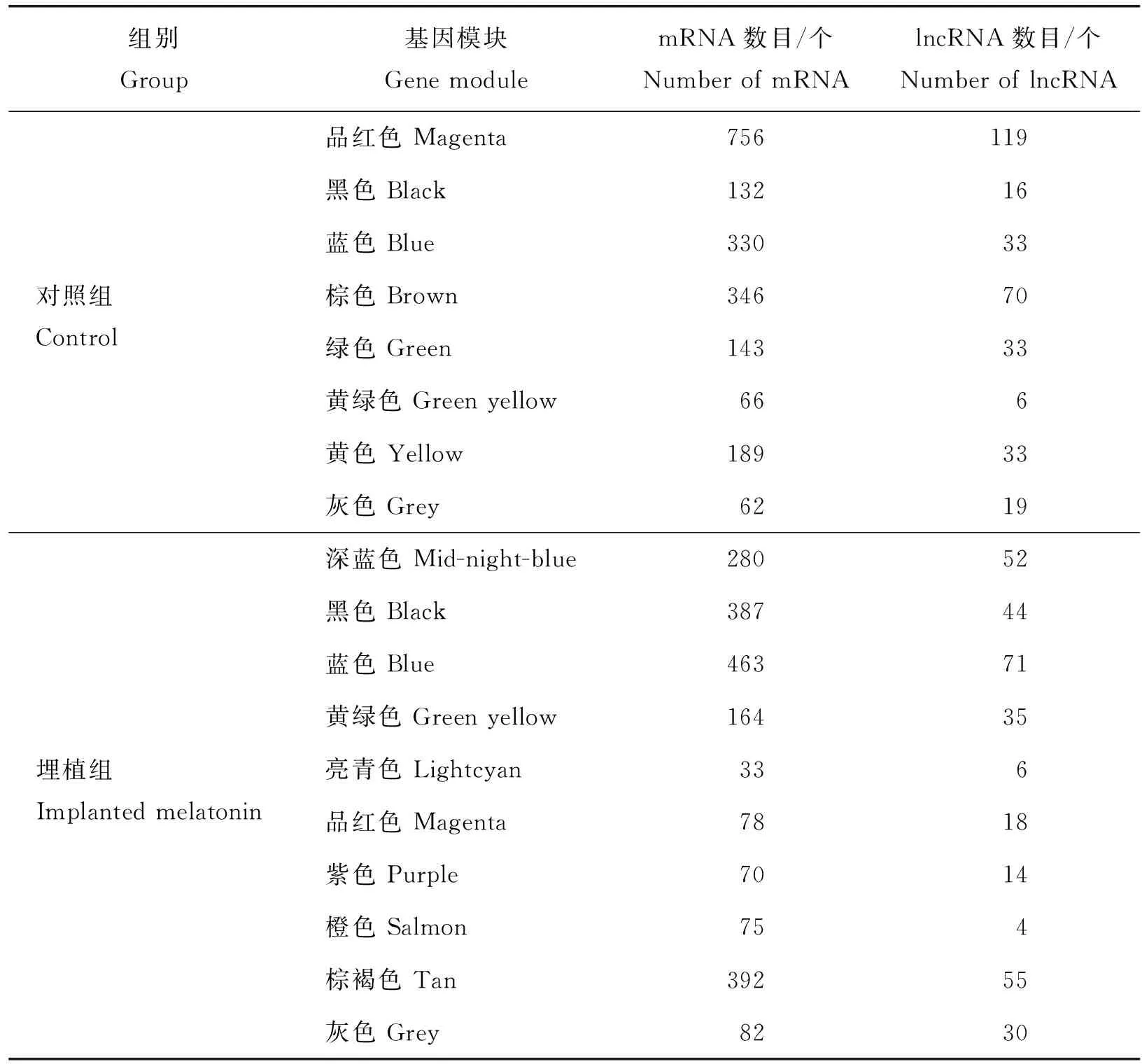
表1 受外源褪黑素诱导的绒山羊皮肤中mRNA和lncRNA在各基因模块的分布Table 1 Assignment of mRNA和lncRNA in each gene module induced byexogenous melatonin in cashmere goat skin

表2 受外源褪黑素诱导的绒山羊皮肤中目标基因模块的功能注释Table 2 Functional annotations enriched in target modules induced by exogenous melatonin in cashmere goat skin
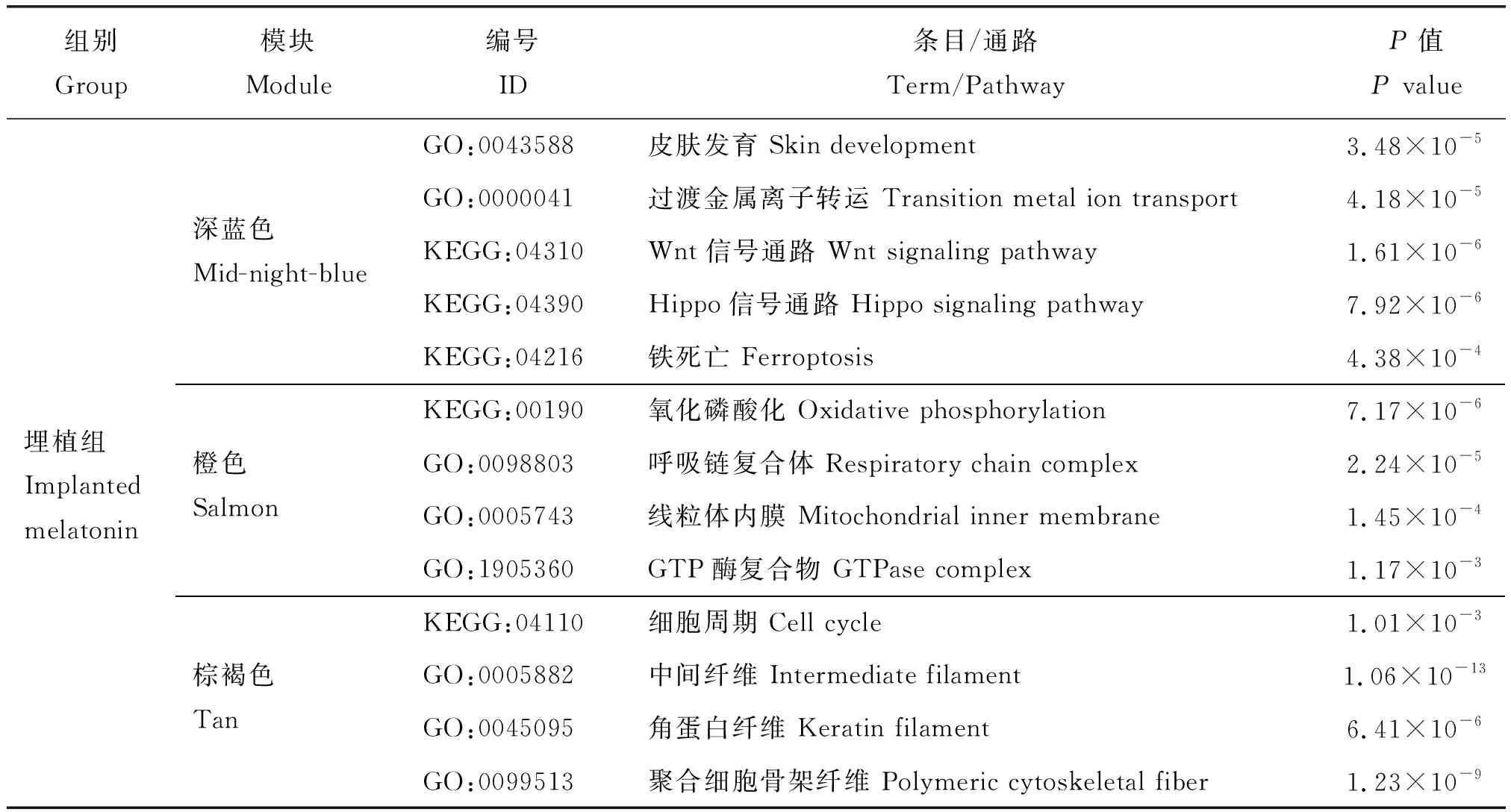
表2(续)
对照组基因模块的特征基因树状图(图3(a))与基因模块间的邻接热图(图3(a))品红色(Magenta)Hippo目标基因模块是独立发挥作用,不与其他基因模块互作。外源褪黑素改变了基因模块间的相互作用模式,埋植组基因模块的特征基因树状图(图3(b))与基因模块间的邻接热图(图3(b))深蓝色(Mid-night-blue)Hippo目标基因模块与橙色(Salmon)和棕褐色(Tan)基因模块存在互作,且基因模块间特征基因相关度在0.5以上。
此结果表明深蓝色(Mid-night-blue)Hippo基因模块是与橙色(Salmon)氧化磷酸化基因模块和棕褐色(Tan)细胞周期基因模块协同发挥作用。
2.4 可视化褪黑素调控山羊绒生长的候选基因集
褪黑素调控山羊绒生长候选基因集的mRNA和lncRNA共表达调控网络见图4,由3个独立的同心圆网络组成,同心圆从左至右分别对应图3(b)的深蓝色(Mid-night-blue)Hippo基因模块、棕褐色(Tan)细胞周期基因模块与橙色(Salmon)氧化磷酸化基因模块。居左的同心圆是82个mRNA和20个lncRNA的共表达调控网络,由内而外,第一环紫色V形节点是目标生物学过程皮肤发育、目标通路Hippo信号通路;第二环橙色菱形节点是对照组和埋植组共有的7个lncRNA;第三环浅绿色菱形节点是埋植组特有的13个lncRNA;第四环红色圆形节点是对照组和埋植组共有的51个mRNA;第五环绿色圆形节点是埋植组特有的31个mRNA。居中的同心圆是87个mRNA和27个lncRNA的共表达调控网络,由内而外,第一环紫色V形节点是目标细胞组分中间纤维、目标通路细胞周期;第二环橙色菱形节点是对照组和埋植组共有的7个lncRNA;第三环浅绿色菱形节点是埋植组特有的20个lncRNA;第四环红色圆形节点是对照组和埋植组共有的46个mRNA;第五环绿色圆形节点是埋植组特有的41个mRNA。居右的同心圆是22个mRNA和2个lncRNA的共表达调控网络,由内而外,第一环紫色V形节点是目标通路氧化磷酸化;第二环浅绿色菱形节点是埋植组特有的2个lncRNA;第三环绿色圆形节点是埋植组特有的22个mRNA。此结果表明外源褪黑素诱导下,这191个mRNA和49个lncRNA由Hippo信号通路、细胞周期和氧化磷酸化过程介导,协调互作调控山羊绒生长的共表达网络关系。
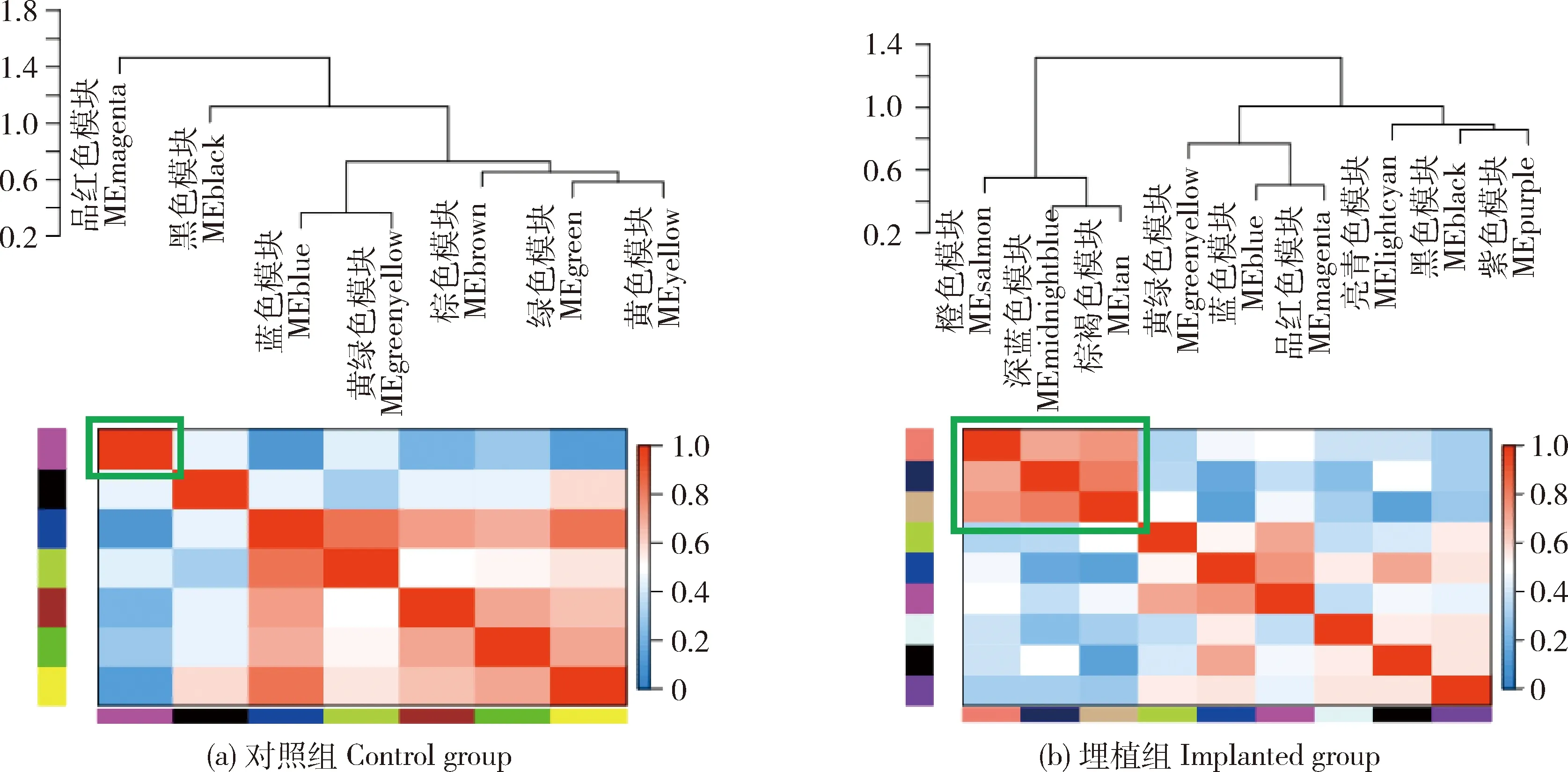
图3 不同组基因模块的特征基因树状图与基因模块间的邻接热图Fig.3 Eigengene dendrogramand adjacency heatmap of gene module of different groups
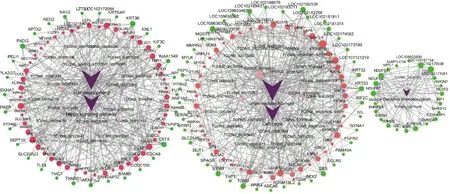
紫色V形节点表示通路、生物学过程、细胞组分;红色和橙色节点分别表示在对照组和埋植组共有的mRNA和lncRNA;绿色节点和浅绿色节点分别表示在埋植组特有的mRNA和lncRNA。圆形节点表示mRNA;菱形节点表示lncRNA。节点的大小表示基因的连通度,节点越小基因的连通度越低。The purple V-shaped nodes indicated pathways, biological processes and cell components; The common mRNA and lncRNA in the control group and the implanted melatonin group were indicated by red and orange nodes respectively; The exclusive mRNA and lncRNA in the implanted melatonin group were indicated by green and light green nodes respectively. The round nodes indicated mRNA; The diamond nodes indicated lncRNA. Node size indicated the degree of gene, low degree map to small node.图4 褪黑素调控山羊绒生长候选基因集mRNA和lncRNA的共表达网络Fig.4 Co-expression networks of mRNA and lncRNA induced by melatonin in the candidate gene set that regulated cashmere growth
2.5 调控山羊绒生长候选基因集的功能透视
2.5.1 候选基因集的GSEA分析
调控山羊绒生长候选基因集的GSEA分析结果见表3,与埋植组相比,对照组更加富集于皮肤发育生物学过程和中间纤维细胞组分;与对照组相比,埋植组更加富集于Hippo信号通路、Wnt信号通路、细胞周期、氧化磷酸化和呼吸链复合体细胞组分。
皮肤发育和中间纤维的富集评分(Enrichment score,ES)为正值,这2个基因集的成员在对照组呈上调趋势。候选基因集在对照组的表达与皮肤发育生物学过程显著关联(|NES|>1且FDR<25%),与中间纤维细胞组分关联(|NES|>1且FDR=0.41)(表3)。此结果表明这2个基因集成员对候选基因集在正常生理状态下的山羊绒生长有贡献。
Hippo信号通路、Wnt信号通路、细胞周期、氧化磷酸化和呼吸链复合体的ES为负值,这5个基因集的成员在埋植组呈上调趋势。候选基因集在埋植组的表达与Hippo信号通路(|NES|>1且FDR<25%)、Wnt信号通路(|NES|>1且FDR<25%)、细胞周期(|NES|>1且FDR<25%)显著关联,与氧化磷酸化(|NES|>1且FDR=0.30)和呼吸链复合体细胞组分(|NES|>1且FDR=0.38)关联(表3)。此结果表明这5个基因集成员对候选基因集在外源褪黑素诱导下的山羊绒生长有贡献。
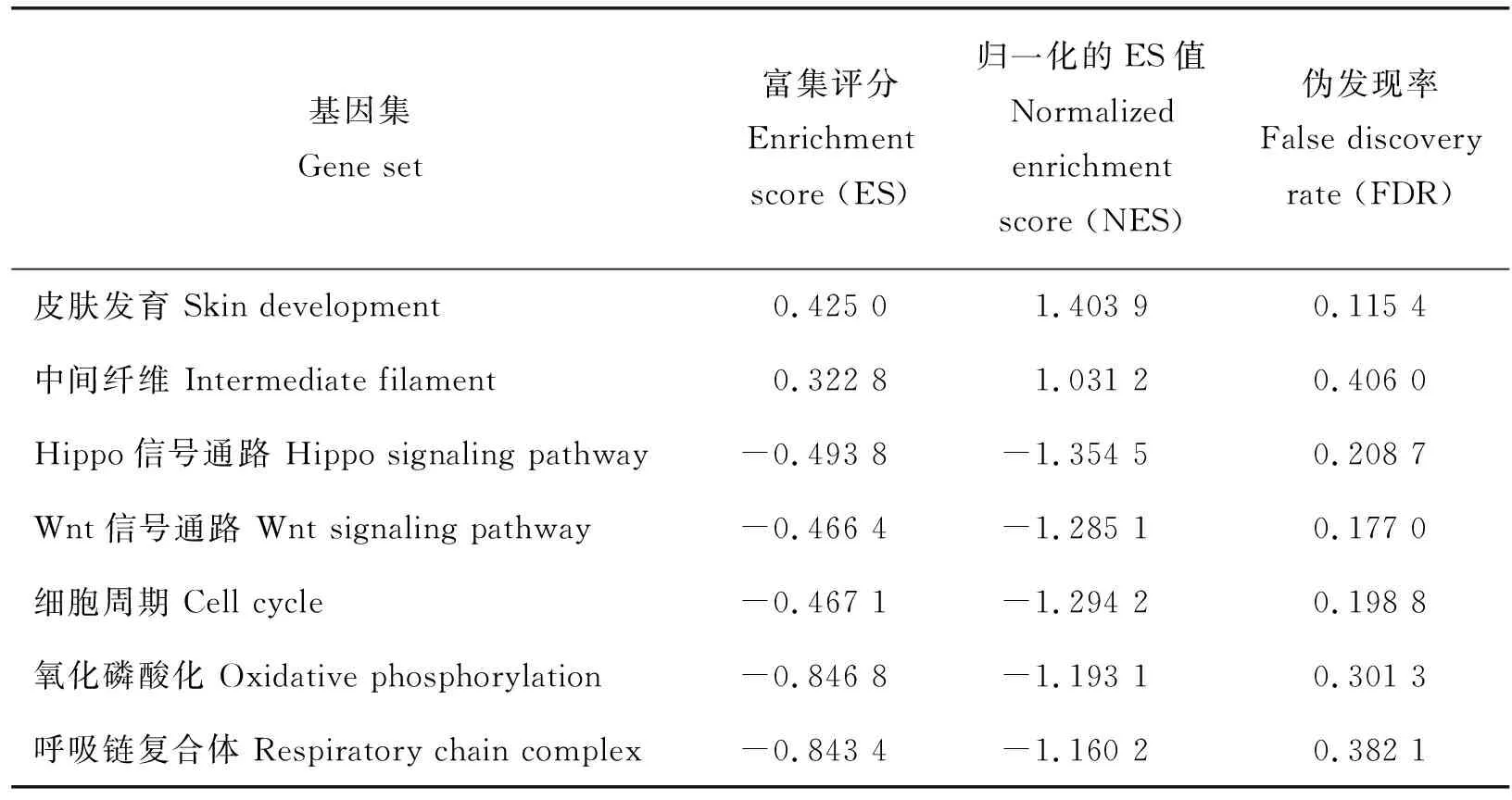
表3 候选基因集GSEA富集评分Table 3 GSEA enrichment score of candidate gene set
2.5.2 定量候选基因集与样本的关联
调控山羊绒生长候选基因集的253个mRNA和59个lncRNA与样本的关联热图,及候选基因集的核心基因、转录因子、转录辅因子之间的聚类模式见图5。对照组和埋植组候选基因集的全部312个基因被聚成两类(图5),一类为212个mRNA和50个lncRNA与正常山羊绒生长期(8~12月)的样本呈正相关,这262个基因促进正常生理状态下的山羊绒生长;另一类为41个mRNA和9个lncRNA与正常山羊绒生长期(8~12月)的样本呈负相关,这50个基因负调控山羊绒生长。对照组绒山羊8月开始长绒,8~12月皮肤次级毛囊处于生长期及生长旺盛期。结果表明,候选基因集与对照组样本的关联结果与生理性状一致,候选基因集参与调控正常生理状态下绒山羊产绒期的绒生长。绒山羊皮肤埋植褪黑素后,候选基因集的312个基因间的互作模式发生了明显改变,这些基因以不同于对照组的方式被聚成两类(图5),协调互作与非生绒期(4~7月)及正常生绒期(8~12月)的样本呈正相关,在外源褪黑素诱导下促进非产绒期和正常产绒期的绒生长。埋植褪黑素后改变了羊绒的生长模式,除正常生绒期外,在4~7月(非产绒期)也生长了一批羊绒。结果表明,候选基因集与埋植组样本的关联结果与生理性状一致,证明外源褪黑素诱导下候选基因集参与调控非产绒期和正常产绒期的绒生长。
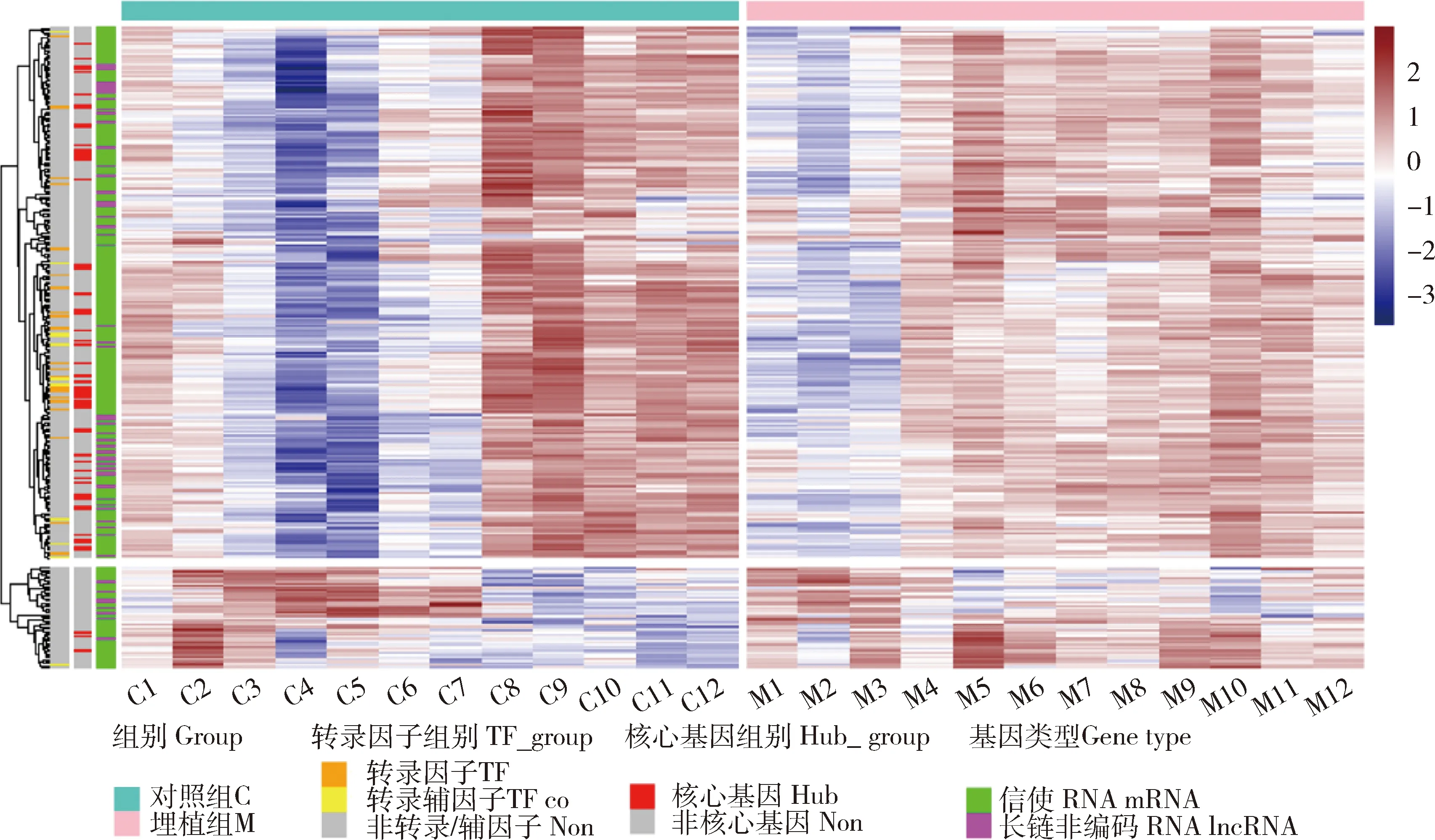
C表示对照组;M表示埋植组;Hub表示核心基因;TF表示转录因子;TF co表示转录辅因子。每一行对应一个基因,每一列对应一个样本。热图的每个单元格根据图例颜色展示基因与样本的相关性。C represented the control group; M represented the implant group; Hub represented hub gene; TF represented transcription factor; TF co(TF cofactor) represented transcription cofactor. Each row corresponded to a gene, column to a sample. Each cell of heat map was color-coded by correlation according to the color legend.图5 调控山羊绒生长候选基因集与样本的关联热图Fig.5 The heat map between the gene set that regulated cashmere growth and samples
3 讨 论
褪黑素是由皮肤产生并可在皮肤中进行代谢的胺类激素,可促进山羊绒生长。lncRNA在调节生物体内的生物学过程中发挥着重要作用[32]。同为研究褪黑素介导lncRNA对山羊绒生长的影响,Jin等[17]研究的皮肤样本取自山羊绒生长期,而本研究不仅对非羊绒生产期的山羊皮肤进行了试验,还将试验范围扩大到非羊绒生产期之前和之后的整个羊绒生长周期。与10~3月相比,4~9月的DE-mRNA和DE-lncRNA数量较多, 其中5月最多(DE-mRNA共计1 178个,DE-lncRNA共计191个),其余11个月(除5月)的DE-mRNA共2 293个和DE-lncRNA共331个,其余11个月(除5月)DE-mRNA的平均数和标准差分别为208.45和200.93,1 178大于811.24(μ±3σ),P<0.01,其余11个月(除5月)DE-lncRNA的平均数和标准差分别为30.09和23.97,191大于101.99(μ±3σ),P<0.01,根据Z检验5月与其余11个月相比差异极显著。这说明与对照组相比,受外源褪黑素影响绒山羊皮肤组织在5月发生了极其重要的生理事件。在正常生理状态下,罕山白绒山羊5月脱绒,7月体表开始长出过冬绒毛。埋植褪黑素导致提前一个月脱绒,5月体表已经长出夏季绒毛。发现DE-lncRNA和DE-mRNA在全年每月分布的变化趋势相似,推测受外源褪黑素影响lncRNA和mRNA协同发挥作用调控山羊绒生长,这有助于更深入了解lncRNA在绒山羊皮肤中的功能。分析发现基因间lncRNA的数量最多,其他类型lncRNA较少。基因间lncRNA的转录激活机制与mRNA类似,以组织特异性的方式表达[33],比蛋白编码基因更具组织特异性,在多种脊椎动物中都是保守的。这表明了基因间lncRNA功能的重要性。在外源褪黑素诱导下,有176个基因间lncRNA在绒山羊皮肤组织特异性表达,提示其可能以保守稳定地方式参与调控山羊绒生长。
同为研究褪黑素介导mRNA和lncRNA对山羊绒生长的影响,Ge等[34]利用STRING数据库(https:∥string-db.org)分析mRNA和lncRNA的相互作用网络,STRING数据库基于文献研究报道的试验验证的直接物理互作或生物信息学方法推断的间接功能相关来检索基因间可能的相互作用,本研究使用WGCNA分析方法基于基因的加权相关系数,将具有相似表达模式的基因归为同一个模块,为研究mRNA和lncRNA之间的共表达网络调控关系提供了新途径,相似表达模式的基因在功能上具有相关性,这有助于挖掘山羊产绒这一复杂性状相关的弱效应mRNA和lncRNA,也为注释家畜lncRNA提供了新思路。Zhang等[20]和Watt等[35]提出Hippo信号通路调节哺乳动物毛囊形态发生、器官大小和发育,Federica等[21]提出Hippo信号通路和褪黑素信号存在串扰,本研究结果证明了褪黑素和Hippo信号通路间存在互作,推测褪黑素可能通过Hippo信号通路调节绒山羊皮肤次级毛囊的大小和发育,从而调控羊绒在非生绒期的生长。山羊绒生长的生物学过程和调节网络如此复杂,传统的基因表达分析策略侧重于识别在两种生物学状态之间表现出差异的单个基因,无法检测到代谢途径、转录程序和应激反应等生物过程,本研究使用GSEA在基因集的水平上分析调控山羊绒生长的候选数据,考虑检测到的基因数量、它们的相对排名和注释到感兴趣通路的基因数量,统计检验显著差异的通路,将大的候选基因列表总结为一个较小的更容易解释的通路列表[36]。而不是仅考虑那些变化倍数或显著性高于临界值的基因,保留了基因间相关性,提供了更准确的模型,有助于将先验知识与新生成的数据联系起来,发现基因型与表型的关系,揭示正常生理状态下和外源褪黑素诱导下基因的集体行为,验证了候选基因集调控山羊绒生长的功能。属于同一基因集或通路的基因更可能以协调的方式表达,并表现出一定程度的相关性。包含相关基因的基因集更一致,会比不一致、不相关的基因集提供更高的生物信号。候选基因集在正常生理状态下和外源褪黑素诱导下与样本的关联结果与山羊绒生长周期一致,同样验证了候选基因集调控山羊绒生长的功能。在候选基因集里发现了16个已经被报道的毛囊发育和绒毛生长的调节因子:FZD1、FZD7、FZD10、TCF7L2[37],WNT11、WNT2B[38]、LEF1[39]、FGFR2[40]、BMP2、BMP4[41]、FOXN1[42]、HOXC13[43]、SOX9[44]、GPRC5D、PRR9、LRRC15[45],其中TCF7L2、LEF1、FOXN1、HOXC13和SOX9是转录因子,FGFR2是转录辅因子,LEF1、GPRC5D、LRRC15、FOXN1、HOXC13和WNT11是核心基因,这也证实了分析方法的可靠性。基因对生物遗传性状的控制具有时间和空间复杂性,在生命体内生物学通路之间存在相互作用的复杂关系网,而探索褪黑素促绒生长的更多基因、更多pathway依赖于研究技术和研究方法的不断创新与完善。
4 结 论
本研究筛选在褪黑素影响下全年每月绒山羊皮肤组织样本共得到2 024个DE-mRNA和329个DE-lncRNA,从转录组水平对绒山羊特有的产绒性状,在基因集层面揭示了在外源褪黑素诱导下,Hippo信号通路调控山羊绒生长的关键lncRNA及mRNA和lncRNA多基因之间的互作关系,从2个不同角度透视了候选基因集调控山羊绒生长的生物学功能,为进一步研究外源褪黑素促山羊绒生长的机制提供了参考。
