兰科植物遗传图谱与QTL定位研究进展
2023-05-11陈和明蔡文杰吕复兵肖文芳朱根发
陈和明 蔡文杰 吕复兵 肖文芳 李 佐 朱根发
(1.广东省农业科学院 环境园艺研究所/广东省园林花卉种质创新综合利用重点实验室,广州 510640;2.台湾成功大学 热带植物与微生物科学研究所,中国 台南 701)
兰科植物(Orchidaceae)有5个亚科801个属28 237个种,是单子叶植物第一大科,约占所有被子植物种类的10%[1-2],其物种分布于世界各地,主要生长于热带及亚热带雨林,亦有部分生存于温带地区。我国兰花资源十分丰富,分布于5个亚科约有190个属1 600个种以及大量的变种,但以云南、台湾和海南最为丰富[3-4]。目前我国兰花品种的选育仍以选择育种和杂交育种为主,但常规育种存在育种年限长、工作量大和育种效率低,以及易受外界环境条件影响等实际问题[2],导致我国兰花品种选育较为缓慢,不能满足日益增长的市场需求。伴随着现代生物技术的发展,构建高密度遗传图谱,挖掘与目标性状紧密连锁的分子标记进行早期间接选择,能够大幅缩短育种周期,加快育种进程[5]。但有关兰科植物遗传图谱构建和QTL定位的研究相对较少,且多数兰科植物遗传组成杂合性高,遗传背景复杂,许多重要观赏性状多为数量性状基因所控制,易受环境条件的影响,一定程度上制约了兰花分子标记辅助育种的进程[6-7]。因此,本研究拟对有关兰科植物遗传图谱的构建与重要性状的QTL定位的研究进展进行综述,旨在为兰科植物遗传图谱的加密、重要性状的基因定位和分子标记辅助育种等提供参考。
1 遗传图谱及相应的分子标记
遗传图谱是指以基因或遗传标记间重组频率为基础的染色体或基因位点的相对位置线性排列图[8],通常以厘摩(Centi-morgan,cM)表示标记间的距离[9]。1994年Peltier等[10]以矮牵牛构建了第一张基于RAPD标记和形态学标记的花卉连锁遗传图谱。早期研究主要通过扩增片段长度多态性(Amplification fragment lengthpolymorphism,AFLP)和随机扩增多态性(Randomamplified polymorphic DNA,RAPD)等分子标记构建遗传图谱,但所构建的遗传图谱质量较低,只能通过多个群体和多次作图才能将图谱加密;而微卫星(Simple sequence repeat,SSR)标记是一种共显性标记,广泛应用于不同的群体研究,但易受到标记数量限制且标记密度较低。随着技术的发展,目前出现了单核苷酸多态性(Single nucleotide polymorphism,SNP)标记,它是在基因组水平上由单个核苷酸变异引起的DNA序列多态性,变异类型和变异频率丰富,其标记的数量多且密度高,在染色体上的分布也很均匀[11]。当前随着高通量测序技术的快速发展和成本的不断降低,SNP标记已广泛运用于高密度遗传图谱的构建、重要性状的QTL定位和基因挖掘等方面的研究[12-13]。
2 兰科植物遗传图谱的构建
2.1 亲本组合
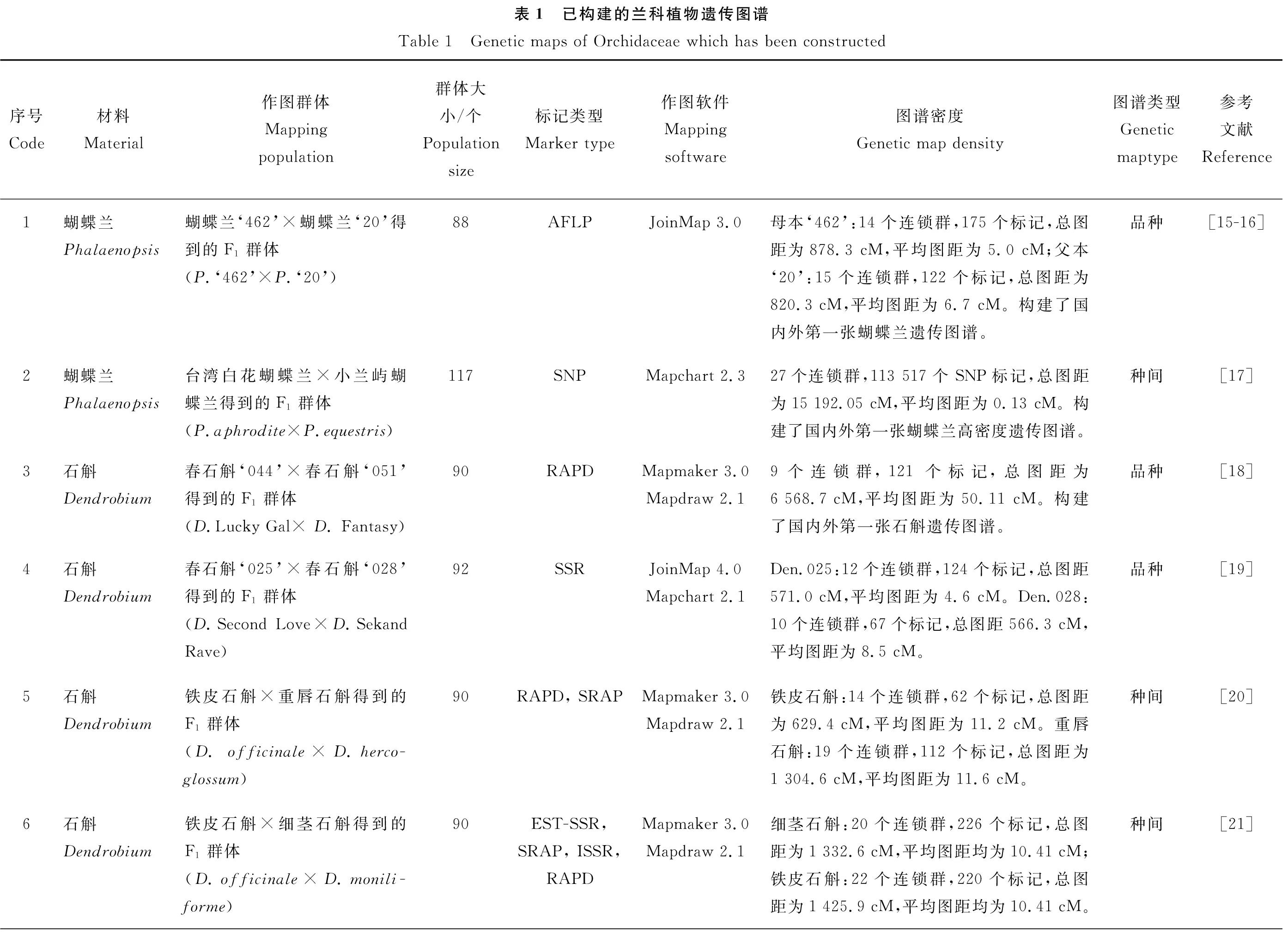
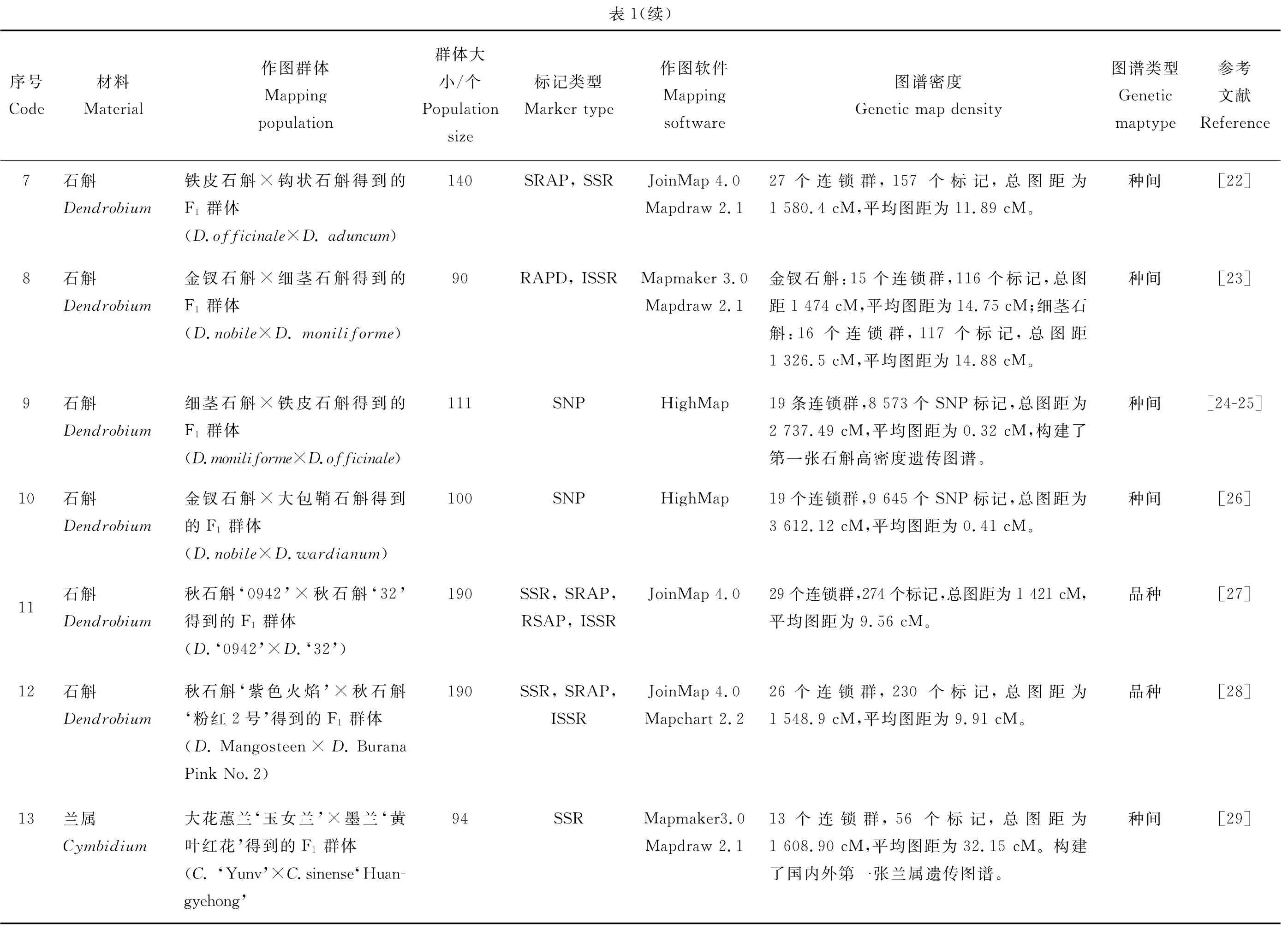
杂交组合亲本的选择对遗传图谱的构建具有决定性意义,应选择亲缘关系较远、遗传差异较大的亲本进行杂交,以便获得更多分离位点的F1代,进而提高图谱密度。但双亲间的遗传差异也不能太大,过大会抑制杂种染色体间的正常配对与重组,出现严重的偏分离现象,并造成杂种可育性下降或不育,影响所建图谱的准确性和可靠性;双亲间的遗传差异也不能过小,太小则亲本间多态性水平低,导致连锁图相对饱和度不高[5,14]。因此,亲本选择在遗传差异性和杂种后代可育性都应兼顾。从表1可以看出,所构建的兰科植物遗传图谱亲本选择均在品种或种间,其中蝴蝶兰‘462’(Phalaenopsis‘462’)和蝴蝶兰‘20’(P.‘20’)是蝴蝶兰品种亲本[15-16],台湾白花蝴蝶兰(P.aphrodite)和小兰屿蝴蝶兰(P.equestris)是蝴蝶兰种间亲本[17],春石斛‘044’(DendrobiumLucky Gal)和春石斛‘051’(D.Fantasy)[18]、春石斛‘025’(D.Second Love)和春石斛‘028’(D.Sekand Rave)[19]均是春石斛品种亲本,铁皮石斛(D.officinale)和重唇石斛(D.hercoglossum)[20]、铁皮石斛(D.officinale)和细茎石斛(D.moniliforme)[21]、铁皮石斛(D.officinale)和钩状石斛(D.aduncum)[22]、金钗石斛(D.nobile)和细茎石斛(D.moniliforme)[23]、细茎石斛(D.moniliforme)和铁皮石斛(D.officinale)[24-25]、金钗石斛(D.nobile)和大包鞘石斛(D.wardianum)[26]均是石斛种间亲本,秋石斛‘0942’(D.‘0942’)和秋石斛‘32’(D.‘32’)[27]、秋石斛‘紫色火焰’(D.Mangosteen)和秋石斛‘粉红2号’(D.Burana Pink No.2)[28]均是秋石斛品种亲本,而大花蕙兰‘玉女兰’(Cymbidium‘Yunv’)和墨兰‘黄叶红花’(C.sinense‘Huangyehong’)[29]是兰属(Cymbidium)不同种间亲本。
2.2 作图群体
包括兰科植物在内的观赏植物由于遗传背景较复杂、杂合性高和世代周期长等特点,作图群体的构建比较困难。而拟测交或双假测交(Two-way pseudo-testcross)理论[30]的提出使得高度杂合的亲本之间杂交获得的F1群体也能够构建遗传图谱。兰花的种子极为细小,呈粉末状,由种胚和种皮构成,不含贮存组织胚乳,缺乏营养物质,在自然条件下很难萌发[31],只能通过人工授粉杂交的方式获得种子并采用组织培养无菌播种的方法繁殖种苗[32-33],从而获得杂种F1代,即作图群体。遗传图谱的精度是由作图群体的大小所决定。当构建的群体越小,则遗传图谱精度越低且图距越大,基因定位时很难检测到重组事件,并引起标记偏分离现象的产生;当作图群体越大,则遗传图谱精度越高且图距越小,有利于紧密连锁位点的区分,但作图费用和工作量增大。因此,构建兰花遗传图谱时,应根据亲本的遗传差异、群体分离情况、标记类型以及作图目标等确定作图群体大小,但从已有的兰科植物作图群体来看,群体的数量在88~190株之间(表1),与林木的遗传图谱基本一致[34]。
2.3 作图软件
在构建遗传图谱的过程中需要利用相关的计算机软件进行作图处理。作图的算法大多采用Allard的计算公式和表格法[35],目前主要的作图软件有Mapmaker[36-37]、JionMap[38]和HighMap[39]等。其中,Mapmaker软件应用最为广泛,但缺乏对原始数据的检查与分析,无法得到连锁图图型文件,影响作图的精度,而MapDraw很大程度上弥补了Mapmaker在绘图方面的不足[40];JionMap软件操作简便,功能强大,可用于标记数量较多的遗传图谱的构建,主要适用于F2、BC、RIL、DH和CP等类型的分离群体[40];HighMap软件能够处理更多、甚至上万的标记进行构图,主要包括连锁分群、标记排序、基因型纠错和图谱评估等部分,是基于高通量测序构建高密度遗传图谱的理想软件[24],其运算速度、标记的排序和图谱质量等方面远胜其他作图软件。从已构建的兰科植物遗传图谱来看,2007—2017年其作图软件主要采用Mapmaker 3.0和Mapdraw 2.1、JoinMap 3.0和JoinMap 4.0,相对应的分子标记主要是SRAP、ISSR、RAPD、EST-SSR和SSR等,而2018—2022年主要采用HighMap软件,相对应的分子标记是SNP,表明兰科植物作图精度越来越高(表1)。
3 兰科植物遗传图谱研究现状
兰科植物遗传图谱的研究较晚。2007年黄少玲[18]以春石斛‘044’(D.Lucky Gal)ב051’(D.Fantsy)的90株F1代个体作为作图群体,利用RAPD标记构建了国内外第一张兰科春石斛遗传图谱,该图谱产生了121个RAPD标记,分布于9个连锁群上,覆盖基因组总长度为6 568.7 cM,平均图距为50.11 cM,为兰科遗传图谱构建提供了基础。但该图谱标记较少,图距较大,不能用于后续QTL定位研究。为了构建更好的石斛遗传图谱,Xue等[20]、赵红燕[21]和Feng等[23]增加了标记类型,赵丹[19]、Lu等[22]、任羽[27]和潘宏兵[28]不但增多了标记类型还扩大了作图群体,他们所构建石斛图谱的图距相对于黄少玲[18]的较小,精度有所提高,但仍然存在标记较少,图距较大,饱和度不高的问题(表1)。为了构建更高密度的石斛遗传图谱,2018年刘玉洋[24]及Lu等[25]以细茎石斛(D.moniliforme)作为母本,铁皮石斛(D.officinale)作为父本构建了111株F1代的作图群体,利用SLAF-seq技术对石斛双亲及其杂交后代进行简化基因组高通量测序,开发了大量石斛全基因组多态性SNP标记,绘制了第一张石斛高密度遗传图谱。该图谱包含8 573个SNP标记,分布于19条连锁群,总图距为2 737.49 cM,平均图距为0.32 cM。2019年Li等[26]利用石斛(D.noble)×大苞鞘石斛(D.wardianum)的100株个体的F1群体进行作图,采用RNA测序(RNA-seq)技术进行高通量测序,绘制了另一张石斛高密度遗传图谱,该图谱包含9 645个SNP标记,同样分布于19个连锁群,总长度为3 612.12 cM,平均标记间隔为0.41 cM。
除石斛外,在兰属和蝴蝶兰方面也构建了遗传图谱。陈起馨[29]以大花蕙兰‘玉女兰’(Cymbidium‘Yunv’)×墨兰‘黄叶红花’(C.sinense‘Huangyehong’)的F1群体94个个体为作图群体,利用SSR标记从墨兰转录组和大花蕙兰转录组中获得78个SSR标记,采用Mapmaker/EXP 3.0作图软件构建出包含13个连锁群和56个SSR标记的遗传连锁图谱,总图距为1 608.90 cM,平均图距为32.15 cM,构建了第一张兰属SSR分子标记遗传图谱。许申平[15]及Xu等[16]报道了第一张蝴蝶兰遗传连锁图谱,利用AFLP标记对蝴蝶兰‘462’×蝴蝶兰‘20’的88个F1个体植株进行连锁作图,其中蝴蝶兰‘462’连锁图产生了175个AFLP标记,分布于14个连锁群,总图距为878.3 cM,平均图距为5.0 cM;蝴蝶兰‘20’连锁图产生了122个AFLP标记,分布于15个连锁群,总图距为820.3 cM,平均图距为6.7 cM。陈起馨[29]构建的兰属遗传图谱图距较大,而Xu等[16]构建的蝴蝶兰图谱饱和度不高,与刘玉洋[24]和Li等[26]采用SNP标记构建的高密度石斛遗传图谱相比,还需要更多的标记和更大的群体来构建更全面的遗传图谱。因此,2022年Hsu等[17]利用台湾白花蝴蝶兰(P.aphrodite)×小兰屿蝴蝶兰(P.equestris)的117株F1群体进行作图,采用全基因组测序技术(GWAS)进行高通量测序,绘制了第一张蝴蝶兰高密度遗传图谱。该图谱包含113 517个SNP标记,分布于27个连锁群,总长度为15 192.05 cM,平均图距为0.13 cM(表1)。
4 兰科植物重要性状的QTL定位
4.1 叶片相关性状的QTL定位
叶片是绿色植物积累有机物质的重要场所,对植株的生长发育具有十分重要的作用。据报道蝴蝶兰叶长与叶面积具有很高的相关性,其开花率与叶面积呈正相关[41-42]。在叶片相关性状的基因定位方面,许申平等[43]以蝴蝶兰杂交组合P.‘462’×P.‘20’后代为材料,对88个单株F1的叶长、叶宽和株幅3个性状进行了初步的QTL定位研究(表2)。结果表明,在父本中共检测到60个QTL,分布于8个连锁群上,包括控制叶长性状的20个QTL,控制叶宽性状19个QTL,控制株幅性状21个QTL;在母本中共检测到28个QTL,分布于5个连锁群上,包括控制叶长性状10个QTL,控制叶宽性状8个QTL,控制株幅性状10个QTL;且统计分析表明蝴蝶兰叶长与株幅间具有显著相关性,2个性状调控基因位点在MLG-1、MLG-2、MLG-3、MLG-8、MLG-9、FLG-3和FLG-6连锁群上都有QTL重叠现象,呈现出同一或相近连锁群的现象,即性状相关度高的QTL大多聚集在同一连锁群的相近或相同区域内,而且具有相关功能的基因成簇分布的现象[44-46]。这是蝴蝶兰的首个QTL研究,为今后蝴蝶兰基因克隆与分子标记辅助育种等提供依据。

表2 兰科植物相关性状QTL定位Table 2 QTL mapping for related characters in Orchidaceae
4.2 花色相关性状的QTL定位
花色是兰科植物重要的观赏性状之一[47]。花色调控是一个复杂的过程,影响花色的因素很多:植物品种[48]、糖转运[49]、环境应激[50]、逆转录转座子激活[51]、类胡萝卜素和花青素生物合成途径[52]以及调控基因如MYB家族[53]和MADS-box家族[54]等。Hsu等[17]在对NCBI nr数据库进行BLAST搜索后,从10个QTL中鉴定出唇瓣黄色(LipYel)、唇瓣红色(LipMag)、花瓣红色(PetalMag)和花瓣红色区(PetalMagArea)等4个不同颜色相关性状的35个候选基因。其中,6个QTL中的26个候选基因与3个不同性状的PetalMagArea、PetalMag和LipMag相关,这些基因分别包括与PetalMagArea性状显著相关的SNP S2_195281745和S5_43813578周围的MYB52基因和MYB11启动子区。MYB家族中的不同基因具有通过调节花青素生物合成来确定红色花部组织的功能。并在S2_195281745、S5_45647571和S5_45345022 的QTL中分别鉴定了MADS-box家族基因AGL65、MADS5和MADS4,这些基因可能不仅调节花形态发生,而且参与花青素生物合成途径。同时,位于LipYel性状的4个QTL中的9个候选基因包括黄酮3-羟化酶(F3H)基因和磺基喹诺糖基转移酶SQD-2基因,以及在PetalMag性状的QTL S5_45647571中也发现了F3H的一个短片段。因此,F3H可能参与蝴蝶兰红色和黄色的调控。这是蝴蝶兰遗传图谱和花色相关QTL的首次报道。
4.3 花朵萼片相关性状的QTL定位
花朵数量遗传学研究揭示了自然选择是如何在数量性状QTL位点上运作[55-57]。任羽等[58]对石斛兰花萼相关的4个性状(中萼片长、中萼片宽、侧萼片长和侧萼片宽)进行了QTL定位,在母本的遗传连锁图谱上检测到9个QTL,其中中萼片长1个、中萼片宽3个、侧萼片长2个和侧萼片宽3个,获得紧密连锁的标记2个(M10E3-146和M8E8-284);在父本的遗传连锁图谱上检测到6个QTL位点,其中中萼片长2个、中萼片宽1个、侧萼片长2个和侧萼片宽1个。同时还发现4个性状之间在连锁群存在共区域的现象,在母本图谱上检测到的侧萼片宽Lsw3位点与中萼片长Dsl位点和侧萼片长Lsl2位点位于相邻区段;中萼片长Dsl位点和侧萼片长Lsl2位点在16连锁群上位于R6R10-1121-M10E5-96同一区段内;在父本图谱上检测到的中萼片长的2个位点和侧萼片长的2个位点都位于同一区段内。这与已经报道的花器官大小QTL的共区域现象是一致的[59-61]。
4.4 石斛茎及多糖相关性状的QTL定位
石斛的茎是石斛主要药用和食用部位。Li等[26]利用高密度遗传图谱鉴定了3个与茎长和茎粗相关的eQTL(Expression quantitative trait locus,一类能够影响基因表达量的遗传位点),其中2个eQTL与茎长相关,分别位于LG3和LG19上,共有33个标记;另一个eQTL与LG13的茎粗相关。这3个eQTL分布在不同的连锁群上,每个eQTL解释的表型变异在11.74%~19.85%。刘玉洋[24]及Lu等[25]利用构建的高密度遗传图谱检测到11个与石斛茎总多糖含量(STPC)相关的QTL,表型贡献率在8.10%~11.80%,平均贡献率为9.15%,其中STPC-9表型贡献率最高,是主效QTL。11个QTL分别位于6个连锁群上,其中LG2连锁群上有4个,LG15和LG19连锁群上分别有2个,LG5、LG11和LG13连锁群上分别有1个。石斛茎及多糖含量STPC相关的QTL定位,为以后石斛碱、黄酮和酚类等石斛重要活性成分相关的QTL定位奠定了基础。
5 展 望
目前已构建的兰科植物遗传图谱,总图距越来越高且相邻分子标记间的平均图距越来越小,为图谱的实际应用奠定了基础,但作图群体较小始终是个问题。在构建遗传图谱时,为了能够检测到减数分裂时的重组事件,需要一定数量的群体。徐云碧等[62]认为构建图谱的理论群体数量应>150个,当需要检测到的重组率达到0.3时,则群体数量至少需300个[63-64]。目前已构建的兰科植物遗传图谱群体数量在88~190之间,其中<150的有11个,<300 有2个。一般认为标记间的平均距离<10 cM,则图谱能够用于QTL定位;而标记间的平均图距<1 cM,则图谱能够用于基因精细定位或基因克隆[65]。当前已构建的13个兰科植物遗传图谱,其中7个可以用于QTL定位,仅有3张图谱可用于精细定位或基因克隆,分别是春石斛[24-26]和蝴蝶兰[17]。在重要性状的QTL定位方面,虽然在蝴蝶兰叶片、花色,石斛兰萼片大小及石斛茎及多糖含量等相关性状进行了基因定位,但还有很多性状如花朵大小、花朵数、花香、花型、花斑和抗寒耐热性等方面均未涉及。因此,基于已构建的兰科植物遗传图谱,今后应加强以下3个方面的科研工作:1)已构建的作图群体偏小,大部分群体数量小于150个,为构建更饱和及分布更均匀的遗传图谱,必须扩大作图群体,才能够满足兰科植物的深入研究;2)已构建的遗传图谱标记间的平均距离多数较大,且均匀性不够好,给基因定位、基因克隆和分子标记辅助育种等带来较大困难,应完善图谱,使其更加饱和;3)观赏价值高的性状,如花数、花香和花型等方面应深入研究,结合遗传图谱加快育种进程。