基于BSA-seq技术的马铃薯块茎蛋白含量基因定位与分子标记开发
2023-05-05史可昕张朝澍
史可昕,石 瑛,张朝澍
(1.东北农业大学农学院,哈尔滨 150030;2.寒地粮食作物品种改良与生理生态教育部重点实验室,哈尔滨 150030)
【研究意义】马铃薯是世界第四大粮食作物,第一大非谷类粮食作物,在工业、农业生产中占有重要地位。随着马铃薯利用程度不断加深,我国高品质马铃薯产品的消费需求不断增加。马铃薯块茎蛋白是一种优质蛋白,含有较高的必需氨基酸,约占蛋白总量的47.90%。蛋白的氨基酸组成研究表明[1],马铃薯富含其他粮食作物所缺乏的赖氨酸,具有较高的生物价值。但是,马铃薯栽培种大多蛋白含量较低,因此,选育高蛋白的优质马铃薯品种,是提高马铃薯品种竞争力的重要途径。在以往的研究中,马铃薯复杂的四倍体遗传特性增加了研究遗传规律的难度[2],加之马铃薯基因组参考序列为二倍体序列,使马铃薯块茎高蛋白含量育种工作一直停滞不前。马铃薯传统育种周期长、筛选效率低、群体基础量大,限制了马铃薯育种的发展。分子标记辅助育种可加速育种进程[3],因此,开发与四倍体马铃薯蛋白含量相关的分子标记对马铃薯育种研究意义重大,但四倍体马铃薯又有着遗传背景狭窄、基因库匮乏、遗传重组率高、自交衰退等问题,开发分子标记难度较大[4-5]。【前人研究进展】目前,在二倍体马铃薯中已开发数十个与性状紧密连锁的标记,如:抗病毒病标记、抗晚疫病标记[6]、抗冻性状标记[3]、炸片颜色标记[7]等。而将上述开发的二倍体马铃薯标记应用于四倍体马铃薯品种中,复杂的遗传方式导致目的性状与标记产生分离,筛选准确率降低。到目前为止,尽管已有关于马铃薯块茎蛋白基因定位等研究的报道,但大多集中在二倍体群体的研究中,用于育种实践的分子标记较少。如邬信康等[8]将马铃薯蛋白多基因簇定位在第8染色体1.4 Mb的DNA片段中;将苋属作物蛋白合成关键基因转入马铃薯中,可以提高35%块茎蛋白含量。高通量测序技术的不断发展为快速挖掘基因、开发功能标记提供了有效手段。BSA-seq技术可在不构建遗传图谱的情况下快速挖掘目的基因,该技术已广泛应用于多种作物的研究中,成功定位出如瓠瓜果形、甜瓜抗霜霉病、黄褐棉纤维长度、水稻耐陈化、芝麻株高等基因[9-13]。【本研究切入点】本文以‘大西洋’为母本、‘定薯1号’为父本构建F1分离群体,以块茎蛋白质含量为目标性状,利用BSA-seq技术筛选与目标性状相关联的染色体区域,针对目标区域开发引物,并用F1分离群体及四倍体马铃薯品种对引物进行检测,获得与蛋白质含量紧密连锁的SSR标记。【拟解决的关键问题】开发的标记可加快马铃薯蛋白含量育种进程,为分子育种及品种选育提供有用工具,也为控制马铃薯块茎蛋白含量基因研究奠定基础。
1 材料与方法
1.1 试验材料
本研究以高蛋白含量马铃薯品种‘大西洋’(块茎平均蛋白含量2.3%)为母本,低蛋白含量马铃薯品种‘定薯1号’(块茎平均蛋白含量1.32%)为父本,杂交构建含有173份家系的F1分离群体。2019年和2020年在东北农业大学向阳农场实验基地种植亲本及F1代分离群体,采取完全随机区组设计,2次重复,单行种植,行长5.0 m,行距0.8 m,每行种植20株,正常田间管理。收获后各小区随机选取10个马铃薯块茎用于蛋白含量测定。选取48个马铃薯栽培品种用于检测开发与蛋白含量紧密连锁的分子标记,各马铃薯品种及蛋白含量如表1所示。供试材料由东北农业大学马铃薯课题组提供。

表1 马铃薯品种信息Table 1 Information of potato varieties
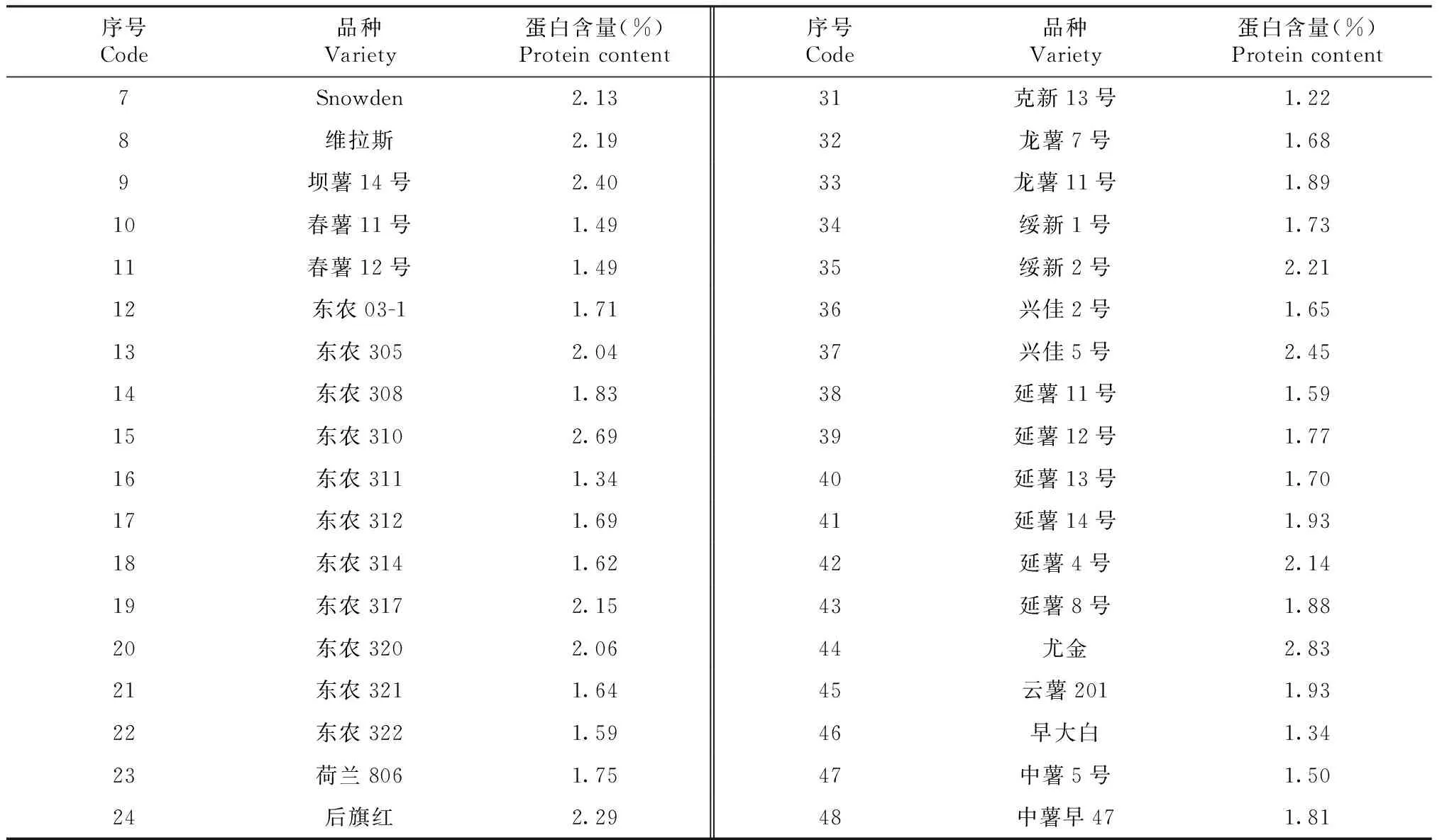
续表1 Continuedtable 1
1.2 试验方法
1.2.1 马铃薯块茎蛋白含量测定 收获后取新鲜马铃薯块茎,切块称取鲜重(A);放入预热至105 ℃的烘箱杀青30 min,80 ℃烘24~72 h至恒重,称取干重(B),干物质含量=B/A×100%;将烘干后的样品粉碎,使用凯氏定氮仪测量全氮含量[14]。
蛋白质含量(%)=全氮值×6.25×干物质含量
(1)
1.2.2 混池构建及DNA提取 F1代分离群体中选取19份极端高蛋白家系(蛋白含量≥2.5%)和24份极端低蛋白家系(蛋白含量≤1.9%)分别构建极端材料池。使用康为世纪的DNA提取试剂盒提取DNA,取新鲜马铃薯叶片加入液氮充分研磨,再加入裂解液充分裂解,旋涡离心。取上清液加入无水乙醇放入吸附柱离心,再加入漂洗液漂洗2次,晾干。用ddH2O溶解DNA,-20 ℃保存。
1.2.3 文库构建测序以及标签的开发和SNP标记检测 亲本池采用全基因组重测序,2个极端混池采用简化测序技术测序,数据由欧易公司、百迈客公司完成评估与过滤。利用五标签串联技术进行测序文库构建,所有样品采用标准型5-NNN-3'接头与酶切标签连接,文库质控合格后在Illumina Hiseq Xten平台进行Paired-end测序。
利用SOAP软件将Raw reads进行过滤,过滤后将Enzyme reads比对到参考序列后进行SNP标记分型。通过相关分析确定与马铃薯蛋白质密切相关的SNP,根据相关阈值确定候选区间,同时,进行基因功能注释和生物途径富集分析。
1.2.4ED值和SNP关联分析 结合F1群体2个极端混池表型及测序后的基因型,利用欧式距离算法,筛选2个混池间存在的显著差异标记,从而评估与蛋白含量相关的区间。BSA项目构建的2个混池间除了目标性状相关位点存在差异,其他位点均趋向于一致,因此非目标位点的ED值应趋向于0。

(2)
式中:Amut为A碱基在突变混池中的频率,Awt为A碱基在野生型混池中的频率;Cmut为C碱基在突变混池中的频率,Cwt为C碱基在野生型混池中的频率;Gmut为G碱基在突变混池中的频率,Gwt为G碱基在野生型混池中的频率;Tmut为T碱基在突变混池中的频率,Twt为T碱基在野生型混池中的频率。ED值越大表明该标记在两混池间的差异越大。
利用2个混池间基因型存在差异的SNP位点,统计各碱基在不同混池中的深度,并计算各位点ED值。为消除背景噪音,对原始ED值进行乘方处理,本项目取原始ED的5次方作为关联值以达到消除背景噪音的功能,然后采用LOESS方法对ED值进行拟合。
1.2.5 标记开发及检测 根据BSA-seq结果,确定控制马铃薯蛋白含量的物理区间。利用亲本全基因组重测序结果,使用Premier Premier 5软件开发目标区间标记。
使用开发的标记在马铃薯F1分离群体及48个四倍体马铃薯品种中进行基因型鉴定,结合各家系及品种蛋白含量,分别分析分离群体及四倍体马铃薯品种基因型与表型的准确度。PCR扩增及产物电泳检测按Yang等[15]的方法进行。
2 结果与分析
2.1 马铃薯亲本及F1分离群体蛋白含量表型分析
母本‘大西洋’块茎的平均蛋白含量为2.32%,具有长势较强、淀粉含量高等特点;父本‘定薯1号’块茎的平均蛋白含量为1.31%,具有花期长、天然结实少等特点。亲本之间平均蛋白含量相差1.77倍(表2)。F1分离群体在各环境中基本符合正态分布(图1)。正态分布检验结果表明,蛋白含量介于1.02%~3.47%,平均变异系数为17.92%。说明,F1分离群体存在广泛的遗传变异,适合进行块茎蛋白含QTL定位。

图1 2019—2020年马铃薯群体的蛋白含量分布Fig.1 Distribution of protein content in potato group from 2019 to 2020

表2 马铃薯群体的蛋白含量Table 2 The protein content in potato group
2.2 测序数据分析和评估
通过测序对马铃薯的2个亲本池、F1群体块茎蛋白含量混池进行序列分析显示,酶切片段长度为300~400 bp。通过伯罗斯-惠勒校准器比对软件Burrow-Wheeler Aligner(BWA)将样本的测序reads与参考基因组进行比对,比对效率正常。从表3可知,父本过滤后的reads数为45 862 067,母本过滤后的reads数为45 544 783,后代极端混池过滤后的reads数分别为45 572 901、45 240 707,平均Q20、Q30分别为97.97%、94.82%,GC含量变异幅度为38.00%~42.00%。表明,测序样本数据量充足,没有明显的A与T、C与G分离现象,测序质量正常,可用于后续的数据分析。

表3 混池测序结果Table 3 Results of mixed pool sequencing
基于比对结果,统计各样本中reads在基因组上各碱基的覆盖深度,并对统计结果进行可视化展示(图2~3)。去除测序深度小于3的标签后,各样品的平均标签数目为129 382,平均测序深度为153.07×,将标签比对至参考基因组,所有样品标签比对率为87.24%~88.34%,建库测序成功,与马铃薯参考基因组比对效率较高,可用于后续变异检测及块茎蛋白含量的基因定位。
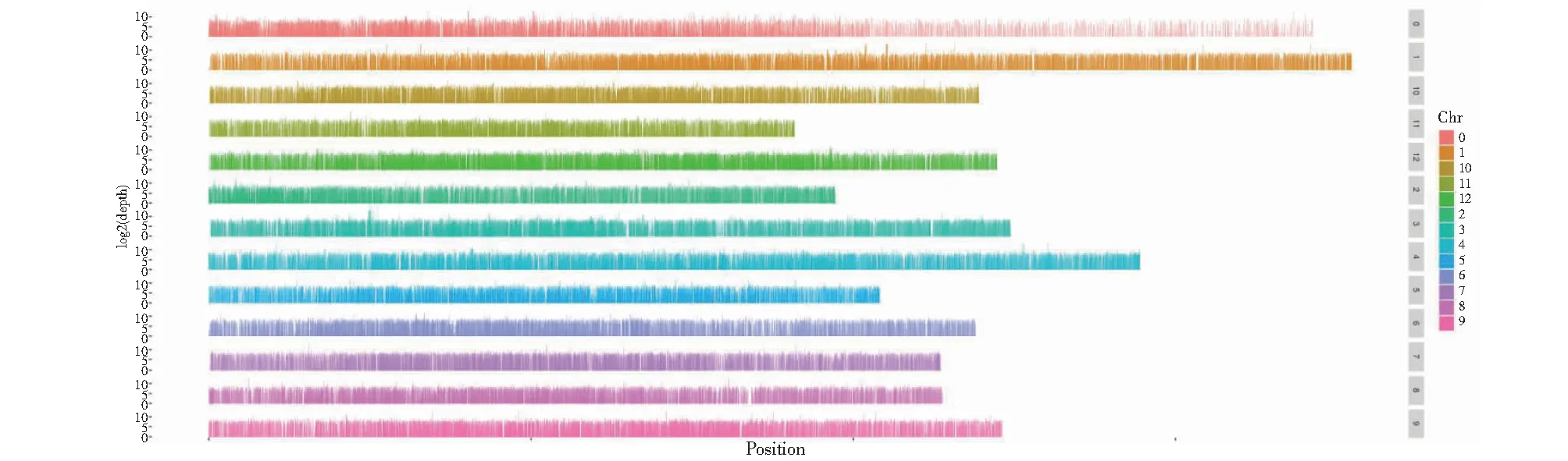
图2 P1样本中reads在基因组上的分布Fig.2 Genomic distribution of reads in P1 samples

图3 P2样本中reads在基因组上的分布Fig.3 Genomic distribution of reads in P2 samples
2.3 马铃薯蛋白质含量关联分析
对SNP、InDel进行过滤,共得到46 103个原始SNP、InDel位点,得到17 306个SNP&InDel位点(图4,表4)。计算各位点ED值并进行乘方,取原始ED值的5次方为关联值(图5),取所有位点拟合值的median+3s为关联阈值,共关联到3个区间。分别为2号染色体18.88~21.59 Mb处,区间大小为2.71 Mb;4号染色体8.30~12.84 Mb处,区间大小为4.54 Mb;4号染色体65.12~66.39 Mb处,区间大小为1.27 Mb(表5)。在这3个区间中存在控制马铃薯块茎蛋白含量的基因。

表4 变异位点染色体分布Table 4 Chromosome distribution of variation site

续表4 Continuedtable 4
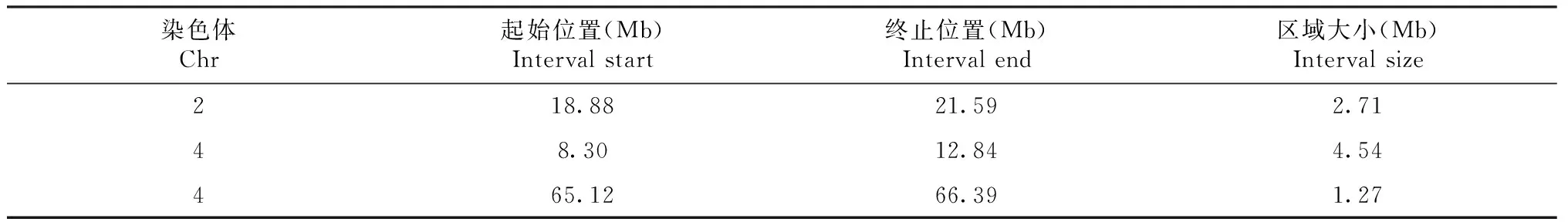
表5 关联区间的位置Table 5 Location of correlation interval

图4 变异位点染色体分布Fig.4 Chromosome distribution of variation sites

横坐标为染色体名称,彩色的点代表每个 SNP 位点的ED值,黑色的线为拟合后的ED值,红色的虚线代表显著性关联阈值,ED值越高,代表该点关联效果越好。取所有位点拟合值的median+3SD作为分析的关联阈值。The abscissa is the chromosome name, the colored point represents the ED value of each SNP site, the black line represents the fitted ED value, and the red dotted line represents the significance association threshold.The higher the ED value, the better the association effect of this point.The median+3SD of all site fit values is taken as the correlation threshold for analysis.图5 ED关联值在染色体上的分布Fig.5 Distribution of ED correlation value on chromosome
2.4 关联区间基因功能注释及马铃薯块茎蛋白含量SSR引物开发
根据定位区域、基因位置及参考基因组信息,共注释到719个候选基因。使用NR、TrEMBL、KEGG、GO、KOG、swissprot、PFAM共7个功能数据库对候选基因进行功能注释。注释基因显著富集在玉米素合成代谢通路,该代谢通路可阻止蛋白质降解。根据双亲重测序信息,使用Primer Premier 5软件分别在3个区间内开发了150对SSR引物(部分SSR引物信息见表6)。
2.5 马铃薯块茎蛋白含量SSR标记检测
以高蛋白的马铃薯品种‘大西洋’和低蛋白的‘定薯1号’基因组DNA为模板,对150对引物的多态性进行PCR筛选。有43对引物能获得相应的目标条带,具有多态性,多态性效率为28.67%(表6)。
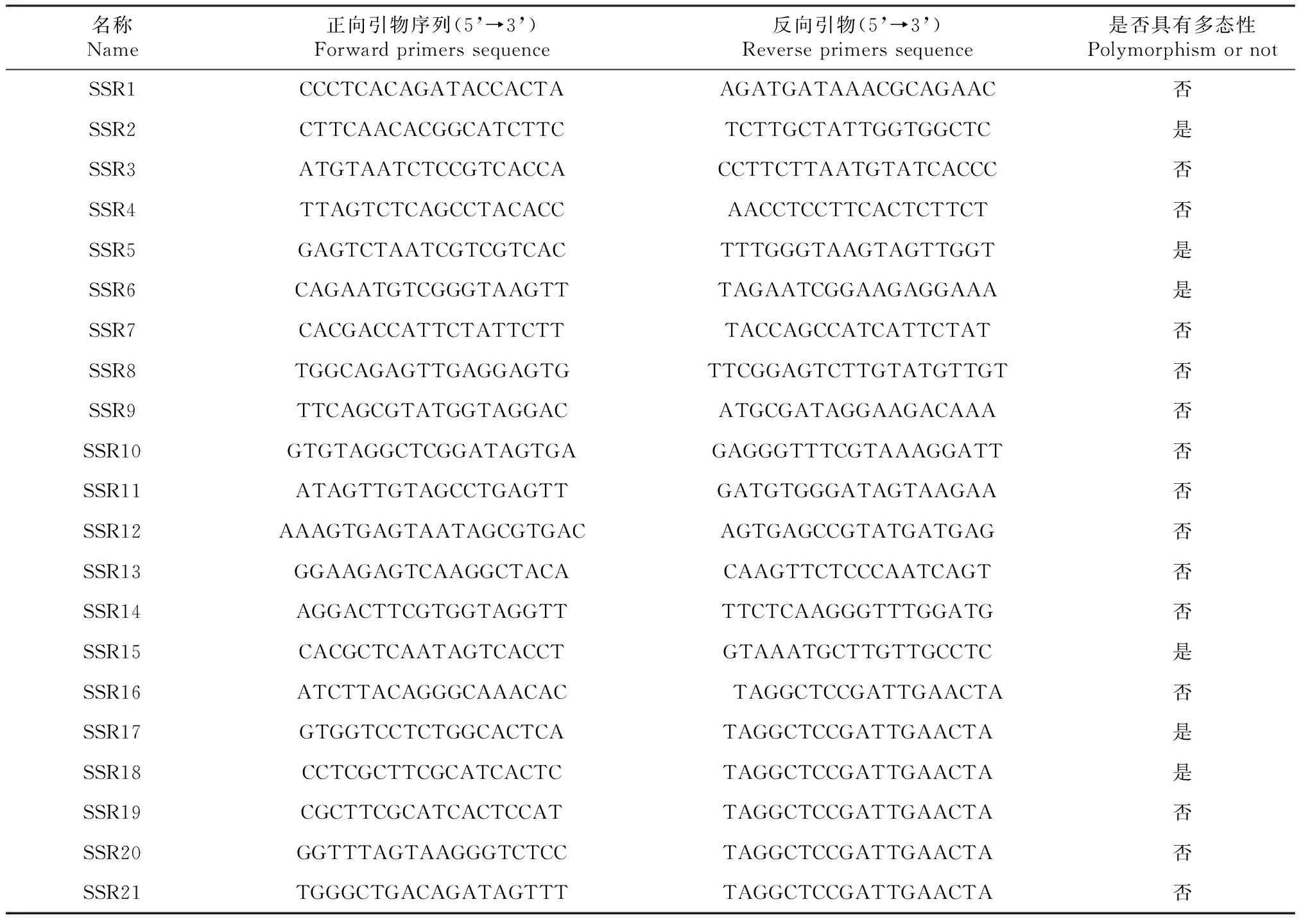
表6 部分SSR引物信息Table 6 Partial SSR primer information
以F1分离群体中的41份高蛋白家系、34份低蛋白家系DNA为模板,进行聚丙烯酰胺电泳,验证引物的多态性。其中,引物SSR15表现出多态性高、条带清晰、重演性好。引物SSR15在41份高蛋白家系中检测出30份高蛋白亲本带型,准确率为73.17%;在34份低蛋白家系中检测出28份低蛋白亲本带型,准确率为82.35%(图6)。

图6 子代马铃薯中的分子标记检测Fig.6 Detection of molecular markers in offspring of potato
在48份四倍体马铃薯品种中,高蛋白品种有14份,占总品种的29.17%;低蛋白品种有8份,占总品种的16.67%。使用引物SSR15对48份马铃薯品种进行检测,在标记为阳性的11个品种中,9个为高蛋白含量,分子标记的检测结果与蛋白含量的对应度达到81.82%;在标记为阴性的11个马铃薯品种中,有6个为低蛋白含量,分子标记的检测结果与蛋白含量的对应度达到54.55%(图7)。引物SSR15能较好地筛选不同马铃薯品种蛋白质含量,可用于马铃薯蛋白含量分子标记辅助育种中,将分子标记SSR15命名为pChr2-4(表7)。

表7 开发的引物序列Table 7 Primer sequence

1.延薯13号,2.延薯8号,3.东农305,4.兴佳2号,5.克新25号,6.东农311,7.兴佳5号,8.龙薯7号,9.东农322,10.东农312,11.延薯12号,12.克新34,13.东农321,14.克新19号,15.延薯11号,16.东农308,17.Desiree,18.东农310,19.克新26号,20.绥新1号,21.克新35,22.克新12号,23.东农317,24.东农314,25.CIP39048,26.中薯早47,27.中薯5号,28.B192,29.Q12,30.东农320,31.Snowden,32.绥新2号,33.B190,34.东农DD412,35.坝薯14号,36.云薯201,37.延薯4号,38.荷兰806,39.尤金,40.早大白,41.春薯11号,42.春薯12号,43.东农03-1,44.维拉斯,45.龙薯11号,46.延薯14号,47.克新13号,48.后旗红。图7 四倍体马铃薯品种中的分子标记检测Fig.7 Detection of molecular markers in tetraploid potato varieties
3 讨 论
马铃薯块茎蛋白是一种优质蛋白,但大多数马铃薯栽培种蛋白含量较低,不适宜优质马铃薯蛋白的开发与利用[16]。因此,挖掘及解析马铃薯块茎发育过程中控制蛋白形成的关键基因,选育高蛋白的马铃薯品种,是培育高蛋白马铃薯品种的关键基础[17]。本研究以‘大西洋’为母本,‘定薯1号’为父本,其中,‘大西洋’种植范围较广,是世界上经典的炸片马铃薯品种之一[18-19],与父本‘定薯1号’相比,具有蛋白含量较高的特点。2个马铃薯亲本杂交构建的F1分离群体中,各家系蛋白含量的变异范围较大,变异系数达到17.92%,是进行马铃薯蛋白相关基因定位的理想群体。
随着测序技术的发展,利用BSA-seq技术进行基因挖掘,已成为快速基因定位的方法之一[20-21]。与传统的分子标记相比,BSA-seq技术具有分辨率高、定位基因速度快、准确度高、稳定性好等特点[22-24]。本研究对马铃薯F1分离群体19个极端高蛋白家系和24个极端低蛋白家系进行了混池测序,共开发129 382个标签,这些标记分布在马铃薯12条染色体上,过滤后得到17 306个具有较高质量的SNP,这些SNP为马铃薯蛋白含量关联分析提供了足够的数据支撑。
与常规育种相比,分子标记辅助育种可加快育种进程,提高育种效率。随着分子标记辅助育种的发展,耐寒[25]、休眠[26]、淀粉含量[27]、植株熟性[28]、抗马铃薯病毒[29]、薯肉颜色[30]、抗晚疫病[31]等很多马铃薯重要性状被标记。本研究利用BSA-seq分别在2号、4号染色体定位了3个与马铃薯蛋白含量相关的区间,区间大小分别为2.71、4.54、1.27 Mb。本研究使用NR、TrEMBL、KEGG、GO、KOG、swissprot、PFAM 7个功能数据库共注释到719个候选基因,发现这些基因显著富集在玉米素的生物合成代谢途径中,该代谢通路可阻止蛋白质降解,可能与提高马铃薯块茎蛋白含量相关。由于注释到的基因较多,无法直接进行候选基因验证,同时,马铃薯物种的特殊性,以回交的方式进行精细定位难度较大,故决定在后续试验中对所有F1家系进行简化基因组测序,并结合转录组、非靶标代谢组学进一步缩小定位区间,挖掘调控马铃薯蛋白含量的候选基因。
本研究根据双亲重测序信息,在3个区间内开发了150对SSR引物,其中43对具有多态性,分子标记pChr2-4特异性最好。使用该引物对F1分离群体75个极端家系及48个四倍体马铃薯品种进行检测,检测结果差异明显、稳定性好,对高蛋白含量马铃薯家系及四倍体马铃薯品种的检测准确率分别为73.17%和81.82%,准确度较高,该标记可用于马铃薯蛋白含量分子标记辅助选择。
4 结 论
本研究用‘大西洋’和‘定薯1号’为亲本构建F1分离群体,选择极端家系进行BSA-seq,将控制蛋白质含量的基因分别定位在2号染色体18.88~21.59 Mb、4号染色体8.3~12.84 Mb及4号染色体65.12~66.39 Mb处。在2号染色体18.88~21.59 Mb处开发了1个与马铃薯蛋白含量紧密连锁的SSR标记pChr2-4。该标记有助于马铃薯块茎蛋白含量分子标记辅助育种。
