抗除草剂棉花GV-2 的分子特征和遗传稳定性分析
2023-04-18陆国清马彩霞孙国清郭惠明程红梅
陆国清 马彩霞 孙国清 郭惠明 程红梅
摘要:分子特征和遗传稳定性是我国转基因植物安全评价流程所需考察的重要数据。利用基因组测序技术对抗除草剂棉花GV-2 的T-DNA 插入位点、拷贝数及侧翼序列进行解析,并采用实时荧光定量PCR(quantitative real time polymerase chain reaction,qRT-PCR)、Southern杂交、胶体金试纸及ELISA(enzyme linkedimmunosorbent assay)方法对连续3代(T3~T5)GV-2转化体中目的基因的表达的稳定性进行验证。结果表明,转化体GV-2中T-DNA以单拷贝形式插入到陆地棉A06染色体2 092 124~2 092 194 bp之间,造成棉花基因组中70 bp DNA的缺失。转化体特异性PCR及Sanger测序结果进一步验证了插入位点的正确性。此外,目的基因及其表达蛋白在不同世代转化体中均可稳定遗传,为转基因棉花GV-2转化体的安全评价提供了有效的支撑。
关键词:转基因棉花;抗除草剂;G10evo;分子特征;遗传稳定性doi:10.13304/j.nykjdb.2021.0581
中图分类号:S562 文献标志码:A 文章编号:10080864(2023)01004208
转基因植物的分子特征主要是指在基因水平、转录水平和翻译水平考察外源插入序列的整合和表达情况[1],这些是我国转基因植物安全评价管理要求中必须提供的信息,对于转基因材料的风险监管至关重要。其中,目的基因在植物基因组中的整合情况是考察的重点。分析外源插入序列整合情况的方法包括转化体特异性聚合酶链式反应(polymerase chain reaction,PCR)、Southern杂交等,其中转化体特异性PCR 的实现要基于PCR的染色体步移技术或高通量的基因组测序技术获得外源序列在植物基因组中的整合位点和侧翼序列。染色体步移是刘耀光院士于1995年发明的一种用于分析T-DNA插入位点的技术,即热不对称交错PCR (thermal asymmetric interlacedPCR,TAIL-PCR),TAIL-PCR是一种用来分离与已知序列邻近的未知DNA 序列的分子生物学技术[2-5]。
基因组测序技术作为近年来解析转基因植物分子特征的新方法,已经在多种植物上得到应用,尤其是粮食作物。Williams-carrier等[6]利用高通量测序技术开发出一套适于玉米转座突变插入位点检测的新方法,该方法为玉米中表型驱动的突变标签鉴定提供了新的工具,同时也适用于其他具有高拷贝转座子的植物。Siddique等[7]利用新一代测序技术鉴定了转基因玉米IE09S034的分子特征。Kovalic 等[8]和Guo等[9]分别利用基因组测序技术分析了转基因大豆(MON17903、MON87704以及GE-J16、ZH10-6)的分子特征。研究表明,利用全基因组测序技术成功鉴定了水稻转化事件的插入位点及侧翼序列[10-12]。Inagaki等[13]利用高通量测序技术成功分析确定了22个拟南芥转化事件中T-DNA的插入位点及侧翼序列。然而,该方法在转基因棉花分子特征检测方面的应用鲜见报道。
遗传稳定性在我国转基因植物安全评价过程中主要是考察目的基因整合的稳定性、表达的稳定性以及目标性状表现的稳定性[1]。Southern杂交由于其结果直观,是检测目的基因整合稳定性的首选方法。实时荧光定量PCR(qRT-PCR)和酶联免疫法(enzyme linked immunosorbent assay,ELISA)在检测目的基因转化体不同世代中转录和翻译水平表达的稳定性方面具有灵敏、高效的优势。对于抗除草剂这一目标性状,我国《转基因生物新品种培育重大专项成果认定指南》中明确指出,转化体至少能够耐受目标除草剂的剂量要达到农业生产上推荐剂量的4倍以上,因而,抗除草剂转化体不同世代的目标性状稳定性也是其是否具有产业化前景的重要前提。
G10evo 基因是已经在玉米和大豆中获得成功应用的抗除草剂基因[1415]。棉花转化体GV-2是通过农杆菌介导的方法将G10evo 基因导入珂字棉312中获得的独立转化事件,具有良好的除草剂抗性[16]。该转化体已于2016年完成了农业转基因植物安全评价的中间实验环节,本研究利用基因组测序技术对GV-2 的T-DNA 插入位点、拷贝数及侧翼序列进行解析,并采用qRT-PCR、Southern 杂交、胶体金试纸及ELISA 方法对连续3代GV-2转化体(T3~T5)中目的基因转录和表達的稳定性进行验证,同时也对不同世代转化体的除草剂抗性开展鉴定,为转基因棉花GV-2转化体安全评价提供有效的支撑。
1 材料与方法
1.1 试验材料
供试植物材料包括源于珂字棉312的转化受体材料R15以及通过农杆菌介导法将G10evo 基因导入R15获得的抗除草剂棉花GV-2,由本实验室创制。转化质粒是以pCambia2301为基础的植物表达质粒,包含2 个分别由木薯叶脉花叶病毒PCsVMV 启动子和花椰菜花叶病毒CaMV35S 启动子驱动的G10evo 基因、1个由CaMV35S启动子驱动的卡那霉素抗性选择标记基因(nptII)[16]。
主要试剂:植物DNA和总RNA提取试剂盒购自天根生化科技(北京)有限公司;反转录试剂盒、荧光定量PCR相关试剂盒购自北京全式金生物技术有限公司;G10-EPSPS胶体金试纸条、ELISA试剂盒购自上海佑隆生物科技有限公司;农达、41%草甘膦异丙胺盐水剂,由美国孟山都公司生产;其余一般化学试剂均购自北京博艾生物技术有限公司。引物由六合华大基因科技有限公司合成。
1.2 试验方法
1.2.1 GV-2的基因组测序及插入位点分析 全基因组测序委托北京利百加生物技术有限公司完成。利用片段化的GV-2 基因组DNA 构建文库,构建好的文库采用PE150 双末端测序法在Illuminia Nova Seq 6000 平台进行测序,测序深度为30× 。用BWA 软件将clean reads 与棉花(NBI_Gh_v1.1.fa)参考基因组比对,根据比对结果确定reads 的insert size 峰值为340 bp,变幅296~383 bp。去除双端都比对上的reads,保留单端比对上的reads以及双端都未比对上的reads,用BWA 软件将保留的reads 与T-DNA 插入序列及棉花基因组比对以获得插入位点及其侧翼序列。
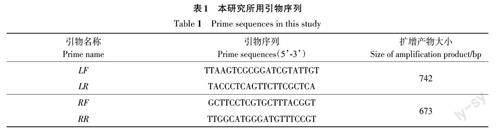
1.2.2 转化体特异PCR 根据基因组测序获得插入位点两端的侧翼序列及T-DNA 的LB 和RB 序列,设计特异的转化体PCR 引物详见表1。PCR程序为:94 ℃ 5 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃30 s,35个循环;72 ℃ 8 min。PCR产物测序后与插入位点侧翼序列进行比对分析。
1.2.3 荧光定量PCR分析 参照试剂盒说明书,分别提取R15及T3~T5代GV-2的根、茎、叶、花组织的总RNA,反转录成cDNA,通过qRT-PCR法分析目的基因在GV-2不同世代植株中转录水平的表达稳定性。荧光定量特异性检测引物及方法参照文献[16]。
1.2.4 Southern杂交分析 参照Clarke[17]的方法,分别选取R15及GV-2 T3~T5代植株的幼嫩叶片组织,采用CTAB 法提取并纯化植物基因组DNA。分别选择Hind Ⅲ和Spe Ⅰ对基因组DNA进行酶切,通过Southern杂交分析目的基因和标记基因在GV-2中的插入表达情况。以G10evo 的353 bp序列片段和nptII 的337 bp序列片段作为探针进行Southern杂交[18]。
1.2.5 目的蛋白的定性与定量分析 于花期分别对R15和转基因GV-2(T3~T5)植株的根、茎、叶、花进行取样,利用G10-EPSPS胶体金快速检测试纸条定性检测目的基因的表达,确定在GV-2不同世代的植株中目的蛋白的稳定性及表达情况;利用ELISA试剂盒对GV-2不同世代植株中目的蛋白进行定量检测,分析不同组织中目的蛋白表达水平。分析方法参照试剂盒说明。
1.2.6 目标性状鉴定 采用盆栽方式,在装有营养土的花盆中种植R15 及T3~T5 代GV-2 棉花种子。待棉花长至2叶1心期,在晴朗的天气,采用手持式喷壶喷施20 mL·L-1农达,使叶面全部喷湿15 d后观察植株生长情况。每个材料处理5盆,每盆不少于5株。
2 结果与分析
2.1 GV-2 的插入位点分析及转化体特异PCR
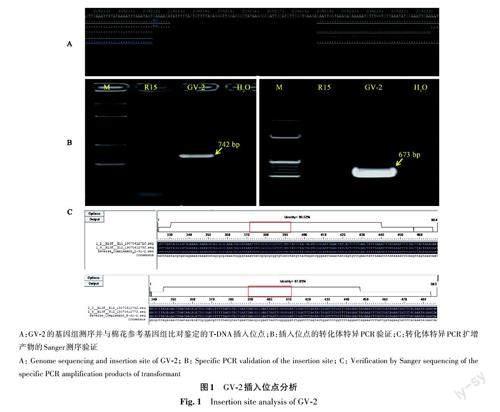
对GV-2进行基因组测序并与棉花参考基因组比对,结果(图1)表明,该转化体仅有1个插入位点,位于陆地棉A06 染色体2 092 124~2 092 194 bp之間,T-DNA的插入删除了原序列中70 bp碱基(图1A)。经检索,插入位点区域无已知编码基因,在陆地棉已发表的转录组中(包括根、茎、叶、花、果实、种子等各种组织器官)均无转录表达痕迹,因此外源基因插入不影响基因组原有功能。利用插入位点侧翼序列及T-DNA左、右边界序列设计的转化体特异引物,能够分别扩增出插入位点左边界742 bp和右边界673 bp的目的片段(图1B),Sanger测序证明,T-DNA左、右边界的侧翼序列与参考基因组的序列一致(图1C),进一步证实了基因组测序获得的GV-2插入位点及侧翼序列的正确性。
2.2 目的基因转录水平的稳定性
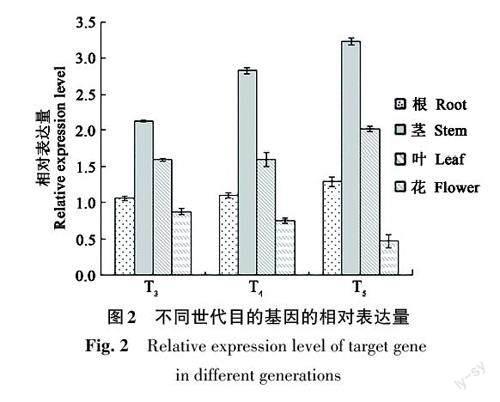
利用qRT-PCR对转基因棉花GV-2不同世代植株的目的基因转录稳定性进行检测,结果(图2)表明,目的基因在不同世代的植株中均可以稳定转录;而在对照R15中未检测到。由此证明,GV-2的草甘膦抗性是由G10evo 基因产生,其转录可以在不同世代间稳定遗传。
2.3 目的基因在不同世代的整合稳定性
分别采用Hind Ⅲ 和Spe Ⅰ 对R15 和GV-2T3~T5 代基因组DNA 进行酶切,利用目的基因(G10evo)及标记基因(nptⅡ)的探针进行杂交,杂交探针在对应基因的相对位置如图3所示。杂交结果(图3B和C)表明,目的基因经2种酶切后分别获得约22和24 kb的条带,标记基因分别获得约3和7 kb的条带。根据插入位点侧翼序列的酶切位点预测Southern杂交时预计产生的条带与实际结果相吻合,表明目的基因与标记基因均以单拷贝的形式整合到棉花基因组中,且能够在GV-2中稳定遗传(T3~T5)。
2.4 目的蛋白翻译水平的稳定性
用胶体金试纸条及ELISA的方法分别定性和定量检测不同世代GV-2中目的蛋白的表达。胶体金试纸条定性检测结果(图4A)显示,目的蛋白在GV-2连续3个世代的不同器官中均有表达,而对照R15中未检测到目的蛋白。根据ELISA试剂盒提供的标准品制备标准曲线,定量检测结果(图4B和C)表明,目的蛋白在茎中的表达量最高,T3~T5代分别为3.30、3.26、3.82 μg·L-1;在叶片中的表达量为2.15、2.13、3.25 μg·L-1;在根中的表达量较低,为0.12~1.12 μg·L-1;在花中表达量最低,T3~T5代分别为0.09、0.08和0.44 μg·L-1。不同世代相同器官中目的蛋白含量差异不显著,进一步表明目的蛋白可以在不同世代及不同器官中稳定表达。
2.5 目标形状的稳定性
对R15和转基因棉花GV-2不同世代植株幼苗按照说明书推荐剂量4倍稀释液喷施,进行草甘膦抗性检测,结果(图5)表明,转基因棉花GV-2不同世代植株生长状态良好,不同世代间差异不显著;对照R15死亡。由此表明,转基因棉花GV-2草甘膦抗性强、遗传稳定性高。
3 讨论
抗除草剂转基因作物的研究和推广在全球农业生产过程中一直处于非常重要的位置。根据国际农业生物技术应用服务组织(InternationalAgricultural Biotechnology Application ServiceOrganization,ISAAA)统计,截至2017 年5 月21日,共有328 个抗除草剂转化事件(这些抗除草剂转化事件均被相关国家授权直接食用、作为添加剂或用于栽培),其中棉花(Gossypiumhirsutum)、大豆(Glycine max)、油菜(Brassicanapus)和玉米(Zea mays)的抗除草剂转化事件300 个,占抗除草剂转化事件总数的91.46%,抗除草剂棉花转化事件39 个。其中,单一抗除草剂性状的转化事件只有5 个,分别是19-51a(杜邦,抗磺酰脲类除草剂)、GHB614(拜耳,抗草甘膦)、LLCotton25(拜耳,抗草铵膦)、MON1445 和MON88913(孟山都,抗草甘膦)[19]。由此可见,转基因抗除草剂棉花转化事件在全球转基因抗除草剂作物中的占比很小,并且基本被跨国公司垄断。截至目前,我国转基因抗除草剂棉花转化事件还没有完成全部安全评价流程。随着全基因组测序技术在水稻、玉米、大豆等作物转基因分子特征检测中的成功应用,将其应用于抗除草剂转基因棉花转化体插入位点及侧翼序列的鉴定也将为加快抗除草剂棉花新品种的培育提供有力的技术支撑。
本实验室培育的抗除草剂棉花GV-2是通过农杆菌介导的方法将外源基因导入陆地棉R15(选育自珂字棉312的易转化品系)中,通过除草剂筛选测试及多代自交获得的纯合且抗性稳定的转化体。研究利用高通量全基因组测序结合生物信息学分析,鉴定出GV-2 中T-DNA 的插入位点及其侧翼序列,结果表明T-DNA以单拷贝的形式插入到陆地棉A06 染色体2 092 124~2 092 194 bp 之间,同时删除了原序列中无功能的70 bp片段。基因组测序数据中部分读序比对到载体骨架上,但数量很少,推测这可能是由于建库过程中质粒污染所致[20]。转化体特异性PCR 及扩增产物的Sanger 测序印证了基因组测序分析结果,Southern杂交再次证明了T-DNA在陆地棉中的单拷贝整合,至此GV-2 的分子特征得到成功解析。
本研究还利用qRT-PCR、Southern 杂交以及ELISA 等技术对目的基因的遗传稳定性进行了分析。在转录水平上,目的基因在各器官的表达量存在一定差异,在茎和叶中的转录水平较高,而在根和花器官中的转录水平较低,分析其原因可能是由于T-DNA 中含有2 个G10evo 基因的表达盒(图3A),其中木薯叶脉花叶病毒启动子的组成表达型是由不同组织特异性结构域引起的,而其中最高启动子表达区域位于维管组织和叶肉细胞中[21]。目的基因在不同世代间的转录没有显著差异,说明其在转录水平的表达非常稳定。此外,GV-2连续3代材料的Southern杂交均一致反映出T-DNA的单拷贝插入形式,说明目的基因在不同世代间整合的稳定性良好。胶体金试纸条检测结果说明目的蛋白在不同世代GV-2中稳定表达,ELISA定量分析不但印证了定性检测结果还发现,目的蛋白在茎和叶中的含量较高,而在花和根中较低,这一结果与基因转录水平的变化趋势相一致,且目的蛋白在不同世代间无显著差异。草甘膦抗性检测反映GV-2棉花目标性状在不同世代间均非常稳定,表明该转化体在生产应用方面具有巨大潜力。總之,本研究为加快抗除草剂棉花的安全评价提供了有力的支撑,为我国抗除草剂棉花新品种突破跨国公司的垄断奠定了基础。
参考文献
[1] 刘培磊,徐琳杰,叶纪明,等.我国农业转基因生物安全管理
现状[J].生物安全学报,2014,23(4):297-300.
LIU P L, XU L J, YE J M, et al .. Administration on the
biosafety of agricultural GMOs in China [J]. J. Biosafety, 2014,
23(4):297-300.
[2] LIU Y G, WHITTIER R F. Thermal asymmetric interlaced
PCR: automatable amplification and sequencing of insert end
fragments from P1 and YAC clones for chromosome walking [J].
Genomics, 1995, 25(3):674-681.
[3] LIU Y G, MITSUKAWA N, OOSUMI T, et al .. Efficient
isolation and mapping of Arabidopsis thaliana T-DNA insert
junctions by thermal asymmetric interlaced PCR [J]. Plant J.,
1995, 8(3):457-463.
[4] LIU Y G, CHEN Y. High-efficiency thermal asymmetric
interlaced PCR for amplification of unknown flanking
sequences [J]. Biotechniques, 2007, 43(5):649-652.
[5] TAN J T, GONG Q Z, YU S C, et al .. A modified highefficiency
thermal asymmetric interlaced PCR method for
amplifying long unknown flanking sequences [J]. Genet.
Genomics, 2019, 46(7):363-366.
[6] WILLIAMS-CARRIER R, STIFFLER N, BELCHER S, et al ..
Use of Illumina sequencing to identify transposon insertions
underlying mutant phenotypes in high-copy mutator lines of
maize [J]. Plant J., 2010, 63(1):167-177.
[7] SIDDIQUE K, WEI J J, LI R, et al .. Identification of T-DNA
insertion site and flanking sequence of a genetically modified
maize event IE09S034 using next-generation sequencing
technology [J]. Mol. Biol., 2019, 61(9):694-702.
[8] KOVALIC D, GARNAAT C, GUO L, et al .. The use of next
generation sequencing and junction sequence analysis
bioinformatics to achieve molecular characterization of crops
improved through modern biotechnology [J]. Plant Genome,
2012, 5(3):149-163.
[9] GUO B F, GUO Y, HONG H L, et al.. Identification of genomic
insertion and flanking sequence of G2-EPSPS and GAT transgenes
in soybean using whole genome sequencing method [J/OL]. Front.
Plant Sci., 2016, 12(7): 1009 [2021-06-15]. https://doi.org/
10.3389/fpls.2016.01009.
[10] WAHLER D, SCHAUSER L, BENDIEK J, et al .. Next-generation
sequencing as a tool for detailed molecular characterisation of
genomic insertions and flanking regions in genetically modified
plants: a pilot study using a rice event unauthorised in the EU [J].
Food Anal. Methods, 2013, 6(6):1718-1727.
[11] PARK D, PARK S H, BAN Y W, et al .. A bioinformatics
approach for identifying transgene insertion sites using whole
genome sequencing data [J]. BMC Biol., 2017, 17(1):67-71.
[12] PARK D, KIM D, JANG G, et al.. Efficiency to discovery
transgenic loci in GM rice using next generation sequencing whole
genome re-sequencing [J]. Genomics Inform., 2015, 13(3):81-85.
[13] INAGAKI S, HENRY I M, LIEBERMAN M C, et al .. Highthroughput
analysis of T-DNA location and structure using
sequence capture [J/OL]. PloS One, 2015, 10(10): e0139672
[2021-06-15]. https//doi.org/10.1371/journal.pone.0139672.
[14] 孫红炜,徐晓辉,李凡,等.转基因玉米双抗12-5-21的抗虫性
及对草甘膦的耐受性 [J].生物安全学报, 2021, 30(1):43-49.
SUN H W, XU X H, LI F, et al .. Insect resistance and
glyphosate tolerance of transgenic maize double-resistance
12-5-21 [J]. J. Biosafety, 2021, 30(1):43-49.
[15] 李娜,曹越平.转基因大豆SHZD32-01对草甘膦的抗性及草
甘膦除草效果研究 [J].大豆科学,2018,37(3):408-414.
LI N, CAO Y P. Study on the resistance of genetically modified
soybean SHZD32-01 to glyphosate and the herbicidal effect of
glyphosate [J]. Soybean Sci., 2018, 37(3):408-414.
[16] 陆国清,王春玲,郝宇琼,等.转G10eve 基因棉花的获得及草
甘膦抗性初探[J].棉花学报, 2018,30(1):21-28.
LU G Q, WANG C L, HAO Y Q, et al .. Obtainment of G10eve
transgenic cotton and preliminary study on glyphosate
resistance [J]. Cotton Sci., 2018, 30(1):21-28.
[17] CLARKE J D. Cetyltrimethyl ammonium bromide (CTAB)
DNA miniprep for plant DNA isolation [J/OL]. Cold Spring
Harbor Protocols., 2009(3):pdb.prot5177 [2021-06-15]. https://
doi.org/10.1101/pdb.prot5177.
[18] GEBBIE L. Genomic southern blot analysis [J]. Mol. Biol.,
2014, 109(9):159-177.
[19] 王園园,王敏,相世刚,等.全球抗除草剂转基因作物转化事
件分析 [J].农业生物技术学报, 2020, 46(11):1703-1710.
WANG Y Y, WANG M, XIANG S G, et al .. Analysis of global
herbicide-resistant transgenic crop transformation events [J]. J.
Agric. Biotechnol., 2020, 46(11):1703-1710.
[20] 马硕,焦悦,杨江涛,等.基因组测序技术解析耐除草剂转基因
水稻G2-7的分子特征[J].作物学报,2020,46(11):1703-1710.
MA S, JIAO Y, YANG J T, et al.. Genome sequencing technology
parsing herbicide resistant transgenic rice molecular characteristics
of the G2-7 [J]. Acta Agron. Sin., 2020, 46(11):1703-1710.
[21] B·贝达格尔, A·德科希科, R·N·比奇,等.木薯叶脉花叶
病毒启动子及其应用: ZL97197430.6. [P]. 19990915.
(责任编辑:张冬玲)
