非洲猪瘟病毒MGF110-5L-6L蛋白诱导宿主细胞翻译阻滞和应激颗粒形成的作用机制
2023-04-10樊帅钟函杨中元何文瑞万博魏战勇韩世充张改平
樊帅,钟函,杨中元,何文瑞,万博,魏战勇,韩世充,张改平
非洲猪瘟病毒MGF110-5L-6L蛋白诱导宿主细胞翻译阻滞和应激颗粒形成的作用机制
樊帅,钟函,杨中元,何文瑞,万博,魏战勇,韩世充,张改平
河南农业大学动物医学院/动物免疫学国家国际联合研究中心,郑州 450046
【】非洲猪瘟(African swine fever,ASF)是由非洲猪瘟病毒(African swine fever virus,ASFV)感染引发的一种猪烈性传染病,是全球公认的养猪业“头号杀手”,至今尚无安全有效的疫苗和药物。病毒作为专性细胞内寄生物,必须通过“劫持”宿主翻译系统为病毒蛋白合成服务。其中翻译起始因子eIF2α作为翻译调控的核心节点,控制细胞应激反应和翻译重编程走向,对病毒毒力、嗜性、致病性及免疫逃逸等具有重要影响,eIF2α磷酸化调控无疑是病毒与宿主细胞竞争翻译资源的重要阵地之一。然而,关于ASFV编码蛋白与eIF2α磷酸化作用关系的认知极度匮乏。【】探究非洲猪瘟病毒MGF110-5L-6L蛋白对宿主细胞翻译阻滞和促进应激颗粒形成的作用机制,为深入揭示非洲猪瘟病毒的致病机制研究提供科学依据。【】在前期利用荧光素酶报告基因载体和绿色荧光报告载体,筛选发现外源表达MGF110-5L-6L极显著上调eIF2α磷酸化水平的基础上。选择猪肺泡巨噬细胞3D4/21和猪肾细胞PK-15作为研究用细胞系,利用质粒转染和特异性化学药物处理等方法,结合免疫印迹和激光共聚焦显微镜等检测技术,验证了MGF110-5L-6L蛋白与eIF2α磷酸化及其下游翻译效应之间的功能关系。随后,通过生物信息学网站预测、亚细胞定位和功能分析等,研究了MGF110-5L-6L蛋白与诱导细胞应激之间的相关性。【】证实外源表达ASFV多基因家族蛋白MGF110-5L-6L能够显著增强细胞内eIF2α磷酸化水平及激活转录因子ATF4表达量,诱导综合应激反应的发生;还可诱导细胞发生内质网应激和未折叠蛋白反应,并通过活化PERK和PKR激酶来诱发eIF2α的磷酸化,进而导致细胞蛋白翻译阻滞和应激颗粒形成。进一步证实,MGF110-5L-6L蛋白含有两个高度保守的半胱氨酸基序,且主要定位于内质网,少量分布于高尔基体、线粒体和溶酶体,还可诱导高尔基体和过氧化物酶体的亚细胞定位及形态出现显著改变,提示其可能通过影响内质网腔中氧化还原稳态、蛋白分泌途径或相关膜性细胞器发生等创造应激条件。【】报道了ASFV早期蛋白MGF110-5L-6L的结构、亚细胞定位及其潜在功能特征,揭示了MGF110-5L-6L蛋白与eIF2α磷酸化和宿主细胞蛋白翻译系统之间的功能关系,为深入认识ASFV的病原生物学与致病机制提供了新的科学参考。
非洲猪瘟病毒;MGF110-5L-6L蛋白;eIF2α磷酸化;翻译阻滞;应激颗粒
0 引言
【研究意义】非洲猪瘟(African swine fever,ASF)是由非洲猪瘟病毒(African swine fever virus,ASFV)引起的一种猪烈性传染病,是全球公认的养猪业“头号杀手”,强毒株引发的超急性和急性感染致死率高达100%[1]。1921年ASF疫情首次报道于肯尼亚,随后疫情传播至撒哈拉以南的非洲地区、欧洲、南美洲、亚洲、大洋洲等多个国家,据世界动物卫生组织最新通报数据显示,截至2022年4月14日,正在暴发ASF疫情的国家/地区有73个(https://www.oie.int/en/ disease/african-swine-fever/),且全球ASF疫情的蔓延和流行态势仍非常严峻。我国作为全球生猪养殖和猪肉消费第一大国,属于ASF疫情重灾区。自2018年8月暴发首例ASF疫情以来,截至目前,全国已累计通报198起疫情,席卷了除台湾以外的所有省份(http://www.moa.gov.cn/gk/yjgl_1/yqfb/),给我国生猪产业构成巨大威胁,且造成了重大的社会、经济以及政治影响。目前,仍无安全有效的ASF疫苗和药物上市,仅依靠严格检疫和扑杀等综合生物安全措施进行防控。世界动物卫生组织将ASF列为法定报告动物疫病,我国也将其列为一类动物疫病。ASFV属于非洲猪瘟相关病毒科(Asfarviridae)非洲猪瘟病毒属(Asfivirus)的唯一成员,属于核质大DNA病毒超家族[2-3]。ASFV病毒粒子结构巨大且复杂,由外向内分别是囊膜、衣壳、内膜、核心壳和基因组[4]。ASFV基因组庞大,为全长约170—194 kb的单分子线性双链DNA,拥有超过150个开放阅读框(open reading frame,ORF),约编码68种结构蛋白和100多种非结构蛋白,参与病毒入侵、基因组复制、DNA修复、转录、翻译调控、病毒组装及免疫逃逸等[2-5]。此外,ASFV基因组两端具有串联重复序列和多基因家族(multigene families, MGFs)构成的可变区,这是导致ASFV基因和表型特征多样性的主要原因[6]。鉴于ASFV复杂的生物学特性,且对其毒力相关基因、复制致病及免疫逃逸/耐受机制等知之甚少,严重制约了安全有效疫苗和药物的研发进度。【前人研究进展】病毒作为一类专性细胞内寄生物,缺乏完整的生命系统,虽携带负责其基因组转录、复制所需的酶,但必须借助细胞的蛋白翻译、修饰加工系统来完成病毒蛋白合成。然而,这一利用过程势必存在高度精准、有效且复杂的“劫持”机制,一方面使得病毒mRNA能够优先享用或独享有限的蛋白翻译资源;另一方面严密调控细胞的一系列应激反应和天然免疫反应,保障病毒能够征服细胞且成功复制[7-9]。大量证据表明,病毒对宿主细胞翻译系统的调控对病毒毒力、细胞嗜性、致病性及免疫逃逸等均有重要影响,且病毒翻译调控策略研究在疫苗种毒改造、抗病毒药物开发和溶瘤制剂创制等方面显示出极大应用价值[8-9]。众所周知,细胞中eIF2α磷酸化水平与综合应激反应(integrated stress response,ISR)、细胞翻译阻滞、应激颗粒(stress granules,SGs)、内质网应激、细胞周期停滞、自噬、凋亡和天然免疫等反应密切相关,参与调控细胞翻译系统的重编程及胞内环境稳态维持[10-13]。因此,eIF2α磷酸化调控可谓是病毒与宿主细胞抢占翻译资源的重要阵地,许多病毒可通过抑制eIF2α发生磷酸化或阶段性利用eIF2α磷酸化创造的应激环境来促进自身翻译复制,这与病毒毒力和嗜性密切相关[9, 14]。ASFV作为“老牌的新兴病毒”,早期研究表明其mRNA与真核细胞大多数mRNAs结构相似,具有5′ 帽子和3′ poly(A)尾结构,利用帽子依赖性翻译起始机制介导病毒蛋白翻译[15]。进一步研究证实,ASFV感染细胞后能够诱导宿主蛋白翻译系统阻滞,抑制宿主细胞的蛋白合成,转而为病毒蛋白翻译服务。其中ASFV对细胞蛋白翻译系统的“劫持”策略包括:增强eIF4F复合体组装及活化;抑制eIF2α发生磷酸化;募集翻译相关因子eIFs、核糖体和线粒体重分布于“病毒工厂”以垄断蛋白翻译资源;调节RNA代谢,包括降解宿主mRNA(D250R脱帽酶)、阻止宿主mRNA出核和限制宿主基因转录等[16-19]。然而,关于ASFV编码蛋白与宿主蛋白翻译系统的调控机制仍存在巨大空白,尤其是与eIF2α磷酸化调控的功能关系研究几乎未见报道。【本研究切入点】近年来,挖掘ASFV毒力/免疫逃逸基因及其作用机制,并据此指导构建新型基因缺失疫苗、亚单位疫苗和活载体疫苗已成为本领域的研究热点及重大需求。聚焦这一科学问题,笔者所在团队以ASFV编码蛋白与eIF2α磷酸化的作用关系为研究切入点。基于激活转录因子4(Activating transcription factor 4,ATF4)的选择性翻译受到eIF2α磷酸化的严格调控这一原理,利用携带ATF4 mRNA翻译调控元件的荧光素酶报告质粒,系统筛选了全套179个ASFV编码蛋白对细胞内eIF2α磷酸化水平的影响。前期结果显示MGF110家族成员上调eIF2α磷酸化水平的功能最为显著。此外,已有研究证实在ASFV感染早期,表达水平最高的病毒基因亦多属于MGF110家族成员,推测其对病毒复制极为重要[20-22]。然而,目前对其功能认知却极度匮乏。【拟解决的关键问题】基于此,本研究靶向挖掘了ASFV MGF110-5L-6L蛋白与eIF2α磷酸化的作用关系,并从病毒蛋白结构、亚细胞定位及潜在功能特征等角度,探讨其与宿主细胞蛋白翻译系统之间的调控关系,为深入认识ASFV的病原生物学特征与致病机制提供新的科学依据。
1 材料与方法
所有试验于2020年10月至2022年5月在河南农业大学动物医学院/动物免疫学国家国际联合研究中心进行。
1.1 细胞及细胞培养
人胚肾细胞(Human embryonic kidney 293T,HEK293T,ATCC#CRL-11268)、猪肾传代细胞(porcine kidney 15,PK-15,ATCC#CCL-33)和猪肺泡巨噬细胞(3D4/21,ATCC#CRL-2843)均来源于美国ATCC细胞库,PK-15细胞和3D4/21细胞由河南农业大学国家动物免疫学国际联合研究中心保存,HEK293T细胞由武汉大学舒红兵院士团队惠赠。细胞均培养于含有10%胎牛血清(FBS)(Invitrogen,美国)、100 U·mL-1青霉素和100 μg·mL-1链霉素的DMEM(Invitrogen)培养基中,并置于5% CO2、37℃条件下培养。
1.2 抗体和化学药物
兔抗FLAG、eIF2α、Phospho-eIF2多克隆抗体购自美国proteintech公司;兔抗ATF4、G3BP1、BIP、PERK、Phospho-PKR、PKR、GCN2多克隆抗体,以及鼠抗FLAG、β-actin单克隆抗体均购自武汉ABclonal公司;鼠抗puromycin单克隆抗体(clone 4G11)购自美国Millipore公司;兔抗Phospho - PERK(Thr980)单克隆抗体购自美国CST公司;兔抗Phospho-GCN2(Thr667)多克隆抗体购自美国Abcam公司;Alexa Fluor 488(AF488)、Alexa Fluor 594(AF594)和HRP标记二抗均购自美国Invitrogen公司。
毒胡萝卜素(Thapsigargin,TG)、PERK抑制剂GSK2606414和ISR抑制剂ISRIB购自美国MedChemexpress(MCE)公司;嘌呤霉素(Puromycin)、亚砷酸钠(Sodium arsenite,Ars)、PKR抑制剂C16和二甲基亚砜(Dimethyl sulfoxide,DMSO)购自美国Sigma - Aldrich公司。
1.3 质粒与质粒构建
根据GenBank公布的ASFV China/2018/AnhuiXCGQ毒株(Accession No. MK128995.1)全基因组序列,由苏州GENEWIZ公司合成ASFV MGF110-5L-6L基因的ORF,并克隆至pCMV-C-Flag骨架载体(Beyotime,中国),获得含有MGF110-5L-6L基因的重组表达质粒;报告基因质粒ATF4-RLuc和ATF4-EGFP交由苏州GENEWIZ公司合成构建,即将包含人源ATF4 mRNA的leader序列及其后面编码区的9个碱基片段(GenBank Accession No. NM_182810.3:1-291)分别克隆到pRL-TK(Promega,美国)和pEGFP-N1(Clontech,美国)骨架载体[23]。
内质网定位质粒pDsRed2-ER、线粒体定位质粒pDsRed2-Mito、高尔基体定位质粒pDsRed-Monomer- Golgi、溶酶体定位质粒pDsRed2-LAMP1和过氧化物酶体定位质粒pDsRed2-Peroxi均购自北京庄盟国际生物基因科技有限公司。
1.4 荧光素酶报告基因检测
待HEK293T细胞单层密度达到70%—80%,利用Lipofectamine 3000(Invitrogen)将ATF4-RLuc报告基因质粒与MGF110-5L-6L表达质粒或FLAG空载体共转染细胞,孵育24 h后,按照说明书操作规程,利用荧光素酶报告基因检测系统(Promega,美国)裂解细胞并检测海肾荧光素酶RLuc的信号强度。
1.5 Western blot分析
煮沸变性的蛋白样品经SDS PAGE电泳后转印至PVDF膜,5% 脱脂乳室温封闭1 h后,4℃孵育一抗(1﹕1 000稀释)过夜,室温孵育HRP标记二抗(1﹕5 000稀释)1 h,加ECL显色液,并置于BioSpectrum 500全自动化学发光成像分析系统(GE Healthcare,美国)观察蛋白条带。
1.6 激光共聚焦(LSCM)检测
1.6.1 亚细胞器定位分析 待PK-15细胞单层密度
达到40%—50%,利用Lipofectamine 3000(Invitrogen)将亚细胞器定位质粒与MGF110-5L-6L表达质粒或FLAG空载体共转染细胞,孵育24 h后,4%多聚甲醛室温固定10 min,0.1% Triton X-100室温透膜处理10 min,5% BSA室温封闭30 min;室温孵育FLAG单抗(1﹕100稀释)1 h,室温避光孵育AF488标记二抗(1﹕1 000稀释)1 h,DAPI(Beyotime)核染5 min,随后利用封片剂(Beyotime)固定于载玻片,并置于Zeiss LSM800激光扫描共聚焦显微镜(Carl Zeiss,美国)下观察。
1.6.2 SGs形成分析 待PK-15细胞单层培养至40%—50%,利用Lipofectamine 3000(Invitrogen)转染MGF110-5L-6L表达质粒或FLAG空载体,孵育24 h后,或利用含有0.5 mmol·L-1亚砷酸钠Ars孵育处理细胞45 min,或不作刺激处理。随后同上执行间接免疫荧光分析,检测用抗体包括鼠抗FLAG(1﹕100稀释)、兔抗G3BP1(1﹕500稀释),以及AF594抗鼠二抗(1﹕500稀释)和AF488抗兔二抗(1﹕500稀释)。
1.7 细胞蛋白翻译效率检测
利用Lipofectamine 3000(Invitrogen)将不同剂量的MGF110-5L-6L表达质粒或FLAG空载体转染PK-15细胞,孵育24 h后,或直接利用5 μg·mL-1嘌呤霉素处理细胞30 min;或利用0.5 mmol·L-1亚砷酸钠Ars预处理空载体转染细胞45 min后再进行嘌呤霉素处理,随后固定或收集细胞,后续如上开展LSCM检测或Western blot分析。
1.8 蛋白分析软件
本文中所用的蛋白质序列分析软件见表1。
1.9 统计分析
使用Image J软件完成荧光细胞计数、荧光共定位分析及蛋白条带灰度值分析,所有统计结果均表示生物统计学意义重复(≥3次)的平均值±标准差。数据统计软件采用IBM SPSS Statistics 22(IBM Corporation,美国),*(<0.05)表示统计学差异显著,**(<0.01)表示统计学差异极显著,n.s.(not significant)表示无显著差异。
2 结果
2.1 外源表达MGF110-5L-6L蛋白诱导eIF2α磷酸化水平显著上调
ATF4作为细胞内胁迫反应响应的“明星级”转录因子,是综合应激反应ISR的关键响应器,因其mRNA 5'-leader中含有两个短抑制上游开放阅读框(uORF)特殊元件(图1-A),使其在elF2α磷酸化后发生选择性地翻译上调,且翻译水平受到eIF2α磷酸化水平的严密调控[10, 23]。基于此,笔者前期利用ATF4-RLuc报告质粒,系统筛选了全套ASFV编码蛋白对eIF2α磷酸化水平的影响(未发表数据)。荧光素酶报告检测结果显示,与DMSO组相比,内质网应激诱导剂TG组的RLuc活性增幅达781%,证实ATF4-RLuc报告质粒的功能有效性;与空载对照组相比,ASFV MGF110-5L-6L表达组的RLuc荧光活性增幅达418%(图1-A)。同时,通过对10个随机视野中ATF4-EGFP荧光报告细胞与所有细胞(DAPI)进行计数和统计分析,发现TG刺激组中荧光细胞比值显著多于DMSO组,约为后者的5.2倍;MGF110-5L-6L表达组的荧光细胞比值约是空载体对照组的2.1倍(图1-B)。利用Western blot进一步证实,在PK-15细胞中过表达MGF110-5L-6L蛋白能够显著上调细胞内eIF2α的磷酸化水平及ATF4的表达量,且呈剂量依赖性(图1-C);随后,选择ASFV野毒和细胞适应毒均可感染的肺泡巨噬细胞3D4/21进行再次验证[32],结果显示与PK-15细胞中的结论一致(图1-D)。以上结果表明,外源表达MGF110-5L-6L蛋白特异性诱导宿主细胞的eIF2α磷酸化水平升高,且具备触发细胞综合应激反应的能力。

表1 蛋白质序列分析软件
2.2 外源表达MGF110-5L-6L蛋白诱导宿主细胞蛋白翻译阻滞
eIF2起始复合体整合了各种应激相关信号来调控宿主细胞翻译系统。在正常情况下,eIF2结合GTP和甲硫酰基-起始转运RNA(Met-tRNAi)形成三元复合体(ternary complex,TC),积极参与43S预起始复合体对AUG的识别、60S亚基招募及翻译起始过程。然而,在应激条件下, eIF2α磷酸化致使eIF2-GDP转换为elF2-GTP过程受阻,限制了TC的可利用性,进而导致翻译起始受阻,细胞整体翻译效率大大降低[10-11](图2-A)。
鉴于MGF110-5L-6L蛋白显著上调eIF2α磷酸化水平,推测其对细胞蛋白翻译效率具有重要影响。基于嘌呤霉素能够掺入新生肽链标记新生蛋白的原理[33],分别利用共聚焦免疫荧光和Western blot试验双重验证MGF110-5L-6L表达对新生蛋白合成效率的影响。激光共聚焦显微镜观察发现,与空载转染组细胞相比,利用eIF2α磷酸化和翻译阻滞的强力诱导剂亚砷酸钠Ars处理细胞,可导致细胞内红色荧光几乎缺失,表明新生蛋白合成量显著减少;同理,MGF110-5L-6L表达细胞中的新生蛋白合成亦显著降低(图2-B)。Western blot结果与免疫荧光结果一致,即与对照组相比,Ars处理组细胞中的新生蛋白条带几乎消失,MGF110-5L-6L表达组细胞中的新生蛋白合成量显著降低,且呈剂量依赖性(图2-C)。为了进一步评估eIF2α磷酸化与MGF110-5L-6L蛋白诱导的细胞翻译阻滞之间的功能关系,利用小分子抑制剂ISRIB进行验证,ISRIB作为eIF2B的靶向激活剂,可特异性恢复eIF2α磷酸化介导的翻译阻滞现象。如图2-D所示,ISRIB处理可显著逆转Ars刺激和MGF110-5L-6L蛋白表达诱导的宿主细胞蛋白翻译阻滞现象。这些结果表明,外源表达MGF110-5L-6L蛋白能够显著抑制宿主细胞蛋白翻译,且该过程依赖eIF2α发生磷酸化。
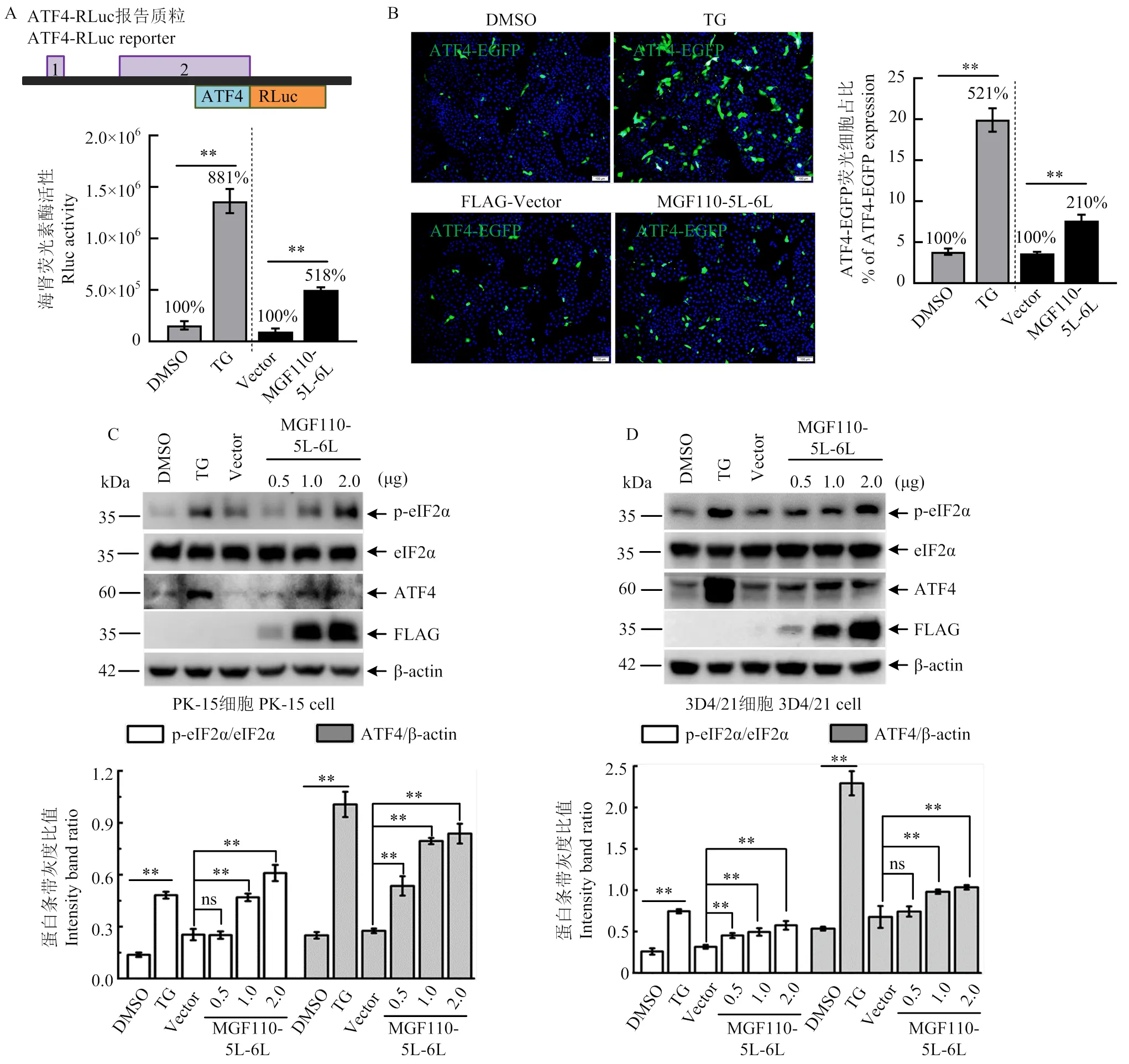
A:ATF4-RLuc报告质粒示意图(上)。将FLAG空载体或MGF110-5L-6L表达质粒与ATF4-RLuc共转染HEK 293T细胞,孵育24 h。在阳性对照组中,细胞经ATF4-RLuc转染18 h后,孵育DMSO或TG (1 μmol·L-1) 6 h,最后进行RLuc活性检测(下);B:将FLAG空载体或MGF110-5L-6L表达质粒与ATF4-EGFP共转染PK-15细胞,孵育24 h;或细胞转染ATF4-EGFP 18 h后,孵育DMSO或TG 6 h,并进行免疫荧光分析;C和D:将不同剂量的MGF110-5L-6L重组质粒或空载体转染PK-15细胞和3D4/21细胞,孵育24 h后,收集细胞裂解物,进行Western blot检测和灰度值分析。图中所示结果源自三次独立重复试验. **:P<0.01,n.s.:差异不显著
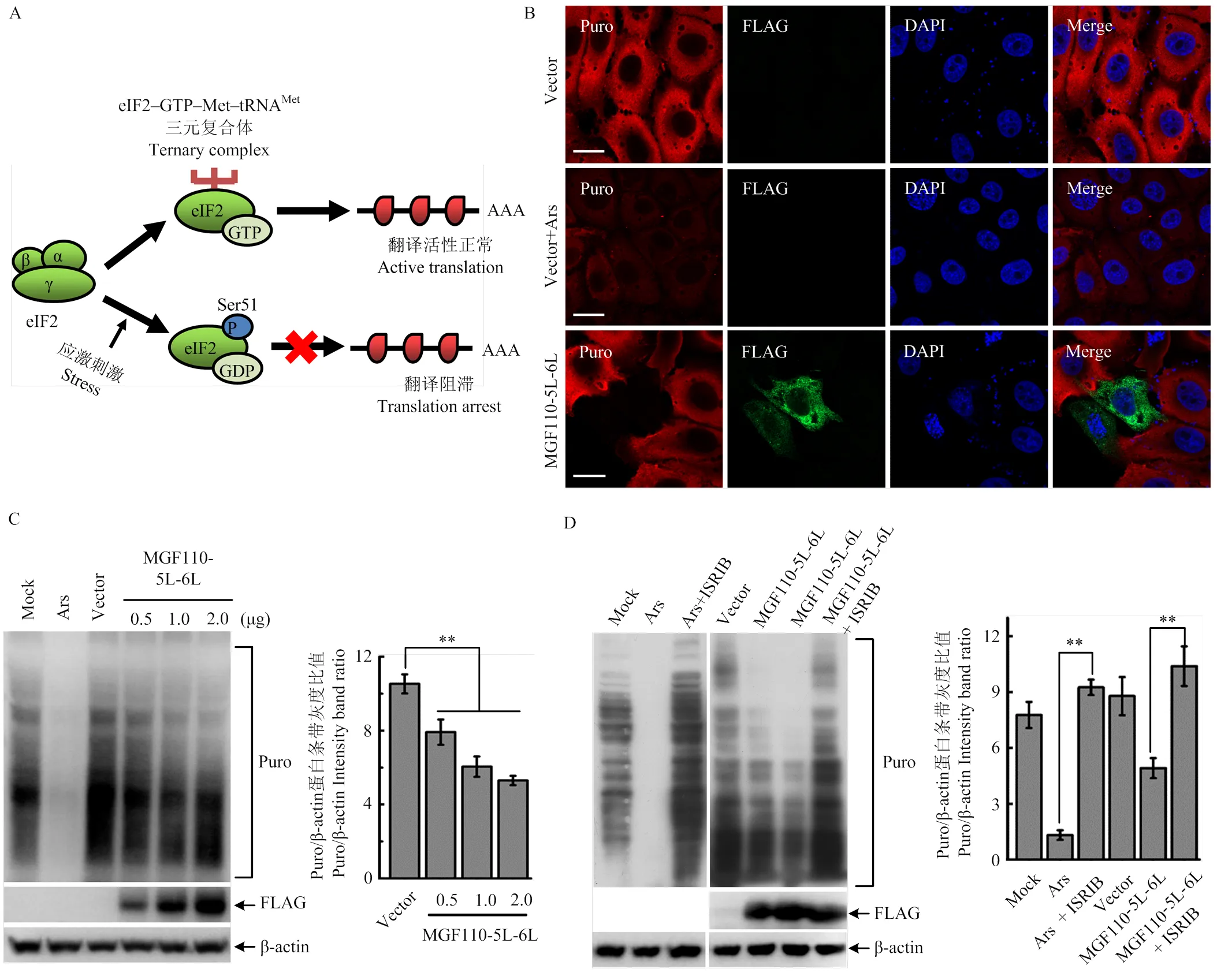
A:eIF2α磷酸化调控宿主细胞翻译活性的模式图;B:将FLAG空载体或MGF110-5L-6L重组质粒转染PK-15细胞,孵育24 h后,利用嘌呤霉素(5 μg·mL-1)处理细胞30 min。随后细胞经固定和透膜处理后,分别孵育嘌呤霉素(红色)和FLAG(绿色)的抗体及相应二抗,细胞核用DAPI染色(蓝色),最后用激光共聚焦显微镜观察。Ars是蛋白翻译阻滞的诱导剂,利用0.5 mmol·L-1Ars预处理空载体转染细胞45 min后进行嘌呤霉素处理;C:将不同剂量的MGF110-5L-6L表达质粒或空载体转染PK-15细胞,孵育24 h后,加入5 μg·mL-1嘌呤霉素处理细胞30 min,随后收集细胞,并进行Western blot检测和灰度值分析。D:在经Ars处理和MGF110-5L-6L表达质粒转染的3D4/21细胞中加入0.5 μmol·L-1的ISRIB,收集细胞样品前,利用嘌呤霉素处理细胞以检测新生蛋白合成量。该结果源自三次独立重复试验. **:P<0.01
2.3 外源表达MGF110-5L-6L蛋白诱导宿主细胞产生应激颗粒(SGs)
宿主细胞暴露于应激情况下,触发eIF2α的磷酸化,导致大多数mRNA翻译受到抑制,而特定的胁迫响应mRNA翻译则增强,这种翻译重编程允许细胞优先将资源用于细胞内稳态恢复。在此期间,翻译起始阻滞以及随后多核糖体的分解导致细胞质内无核糖体结合的mRNA的浓度骤升,催生形成大量的无膜细胞器应激颗粒(SGs)。SGs作为一种高度浓缩的核糖核蛋白颗粒(mRNP),能够拘储与翻译相关的mRNA、eIFs、40S亚基和RNA结合蛋白在内的各种分子,加剧蛋白翻译系统的关闭[10, 34](图3-A)。为探究MGF110-5L-6L蛋白对细胞中SGs形成的影响,笔者选择SG核心网络组分G3BP1作为标记蛋白进行验证。共聚焦免疫荧光检测发现,正常细胞中G3BP1绿色荧光均匀分布于整个细胞质,而MGF110-5L-6L蛋白表达细胞中G3BP1荧光特异地呈现点状聚集,即SGs形成。荧光细胞计数结果显示,与空载对照组相比,MGF110-5L-6L表达组的SGs阳性细胞占比上升至23%;作为阳性对照,Ars处理组的SGs阳性细胞占比高达90%,然而,ASFV DP71L蛋白作为已报道的促eIF2α去磷酸化蛋白[35-36],抑制Ars诱导的SGs形成,DP71L表达组的SGs阳性细胞比例降至56%(图3-B、C)。此外,利用抑制剂ISRIB处理细胞后,发现MGF110-5L-6L蛋白表达细胞中无G3BP1荧光点状聚集,且统计结果显示SGs阳性细胞比例降至3.4%。这些结果表明外源表达MGF110-5L-6L蛋白能够特异地诱导SGs的形成,且该过程依赖eIF2α的磷酸化。
2.4 ASFV MGF110-5L-6L蛋白的序列分析及结构功能域预测
外源表达MGF110-5L-6L蛋白可诱导宿主细胞稳态失调,触发eIF2α发生磷酸化,启动ISR、细胞翻译阻滞和SGs形成等应激反应。为了进一步挖掘MGF110-5L-6L蛋白破坏细胞稳态的机理,我们首先对其氨基酸序列的理化特性、结构和功能域等进行预测分析。众所周知,MGF110基因家族位于病毒基因组的左末端,因存在广泛的密码子重复、丢失或重排事件,使其具有高度的遗传多样性特征[37]。鉴于此,分别选取来自不同年份和不同地区(亚洲、欧洲和非洲)的11株高致病性毒株,并对其MGF110-5L-6L氨基酸序列进行比对分析,发现MGF110-5L-6L蛋白总体上在不同毒株间存在较高的保守性,且均拥有两个保守的半胱氨酸结构域 [C-(X)5-C-(X)2-C-(X)2-C-(X)4-C],且部分羧基端存在内质网滞留信号KDEL基序(图4-A),暗示MGF110- 5L-6L蛋白可能定位于内质网腔,并通过形成二硫键增强腔内的氧化环境。然而,MGF110-5L-6L蛋白在不同毒株中以两种形式存在,其中在亚洲和欧洲为代表的毒株中多以5L-6L融合蛋白形式表达,而在非洲毒株中则以5L和6L两个独立蛋白形式表达,且前者与后者相比,存在3个缺失片段,分别是在5L羧基端缺失23/24个氨基酸,和在6L氨基端分别缺失6个和5个氨基酸(图4-A)。此外,以China/2018/AnhuiXCGQ毒株的MGF110-5L-6L蛋白为例,利用InterProScan和SignalP 5.0预测发现,MGF110-5L-6L蛋白存在潜在的信号肽序列,且位于其氨基端前18个残基(图4-B);利用InterProScan和TMHMM预测发现MGF110-5L-6L蛋白无潜在跨膜区,且ProtScale分析发现其具有极高的亲水性;使用NetNGlyc-1.0预测发现MGF110-5L-6L蛋白在第149位Asn处存在潜在的糖基化位点;使用NetSurfP 2.0和I-TASSER对蛋白二级、三级结构进行预测发现,MGF110-5L-6L蛋白呈复合的α螺旋和β折叠拓扑结构(图4-A、C)。
2.5 ASFV MGF110-5L-6L蛋白的亚细胞定位
MGF110-5L-6L蛋白存在潜在信号肽及内质网滞留信号,可能引导其参与内质网-高尔基体途径,即分泌途径,并被分选至特定的细胞部位发挥功能[38]。众所周知,宿主细胞内膜性细胞器之间的频繁物质交换和功能联系,构成了细胞执行复杂生物学过程的功能网络,其中膜性细胞器包括内质网、高尔基体、线粒体、溶酶体以及过氧化物酶体等。ASFV在感染靶细胞进程中,不可避免地会破坏细胞正常生理状态,引发各种膜性细胞器发生强烈的应激反应[38-40]。为了探究MGF110-5L-6L蛋白在ASFV感染进程中的定位及其潜在功能,分别将MGF110-5L-6L表达质粒与各膜性细胞器定位质粒共转PK-15细胞,以分析其亚细胞定位。通过共聚焦免疫荧光检测及荧光共定位分析发现,MGF110-5L-6L蛋白与内质网定位蛋白的荧光共定位系数为0.92,说明两者几乎完全共定位;与高尔基体定位蛋白、线粒体定位蛋白和溶酶体定位蛋白的荧光共定位系数分别为0.52、0.49和0.55,表明MGF110-5L-6L蛋白与这些蛋白仅部分共定位;而与过氧化物酶体定位蛋白的荧光共定位系数为-0.08,表明其不存在共定位关系。同时,发现MGF110-5L-6L表达诱导细胞内高尔基体和过氧化物酶体的荧光定位及形态发生显著变化。其中,正常细胞中的反式高尔基体网络类似“弯弓”状聚集于细胞核周围,而其在MGF110-5L-6L表达细胞中的分布则显得较为松散且不规则;过氧化物酶体定位蛋白由胞质中规则的点状聚集体形态,转为弥散状分布于整个细胞(图5)。以上结果表明,MGF110-5L-6L蛋白主要定位于内质网,且少量蛋白可能通过分泌途径被转运至高尔基体、线粒体和溶酶体等部位,同时诱导细胞部分膜性细胞器出现形态发生或结构上的变化。
2.6 外源表达MGF110-5L-6L蛋白显著上调eIF2α磷酸化水平的机制
MGF110-5L-6L蛋白是如何打破细胞稳态,细胞又是以何种途径感知应激状态的呢?众所周知,eIF2α磷酸化由四种激酶介导,即PERK(内质网应激)、PKR(dsRNA)、GCN2(氨基酸饥饿)和HRI(血红素不足),4种激酶分别感知不同的应激信号并作出应激性反应(图6-A)。为了探究MGF110-5L-6L蛋白上调eIF2α磷酸化水平的机制,分别在3D4/21和PK细胞中转染MGF110-5L-6L重组质粒,并检测其对不同激酶的蛋白表达量和活化水平的影响。鉴于病毒感染调控HRI活性的报道极为罕见,在此仅检测了PERK、PKR和GCN2等3个激酶的活化情况。Western blot结果显示,与DMSO处理组相比,内质网应激诱导剂TG处理能够诱导BIP蛋白表达量显著升高,且激酶PERK磷酸化水平显著升高,而对激酶PKR和GCN2活化水平的影响不显著。过表达MGF110-5L-6L不仅诱导BIP表达量显著增加,激酶PERK和PKR磷酸化水平亦显著增强,且均呈剂量依赖性,但不影响激酶GCN2的活化水平(图6-B—E)。此外,分别利用PERK抑制剂GSK2606414和PKR抑制剂C16处理细胞,可显著抑制MGF110-5L-6L表达诱导的PERK和PKR活化以及eIF2α磷酸化水平上调(图6-F、G)。综合分析MGF110-5L-6L蛋白的结构、亚细胞定位及功能影响等结果,表明外源表达MGF110-5L-6L蛋白能够通过诱发内质网应激和未折叠蛋白反应,启动PERK和PKR激酶的活化,以触发eIF2α发生磷酸化,进而导致宿主细胞翻译阻滞和SGs的形成。

A:不同ASFV毒株MGF110-5L-6L氨基酸序列比对分析,突出显示了预测的二级结构及保守的半胱氨酸结构域,红色箭头指示为非洲代表毒株MGF110-6L蛋白的首个氨基酸位置;B:信号肽预测;C:蛋白三级结构预测

分别将各亚细胞器定位质粒(内质网pDsRed2-ER,高尔基体pDsRed2-Golgi,线粒体pDsRed2-Mito,溶酶体pDsRed2-LAMP和过氧化物酶体pDsRed2-Peroxi)与FLAG空载体或MGF110-5L-6L表达质粒共转染PK-15细胞,孵育24 h后,细胞经固定和透膜处理,并孵育FLAG(绿色)抗体及相应二抗,细胞核用DAPI染色(蓝色),定位质粒直接表达红色荧光蛋白,最后利用激光共聚焦显微镜观察,并进行荧光共定位分析,包括计算共定位系数R和制作Plot Profile图
3 讨论
3.1 ASFV病毒蛋白翻译策略的研究进展
蛋白翻译是病毒复制过程中最重要且最复杂的过程之一,因自身缺乏翻译系统,病毒必须通过“劫持”细胞的蛋白翻译系统来完成这项工程,该过程不可避免地会干扰细胞的正常生理进程,导致细胞蛋白翻译重编程以及应激反应的发生,以抵抗病毒感染。然而,病毒经过长期进化而来的生存策略,不仅能够保障病毒平稳度过各种应激反应以及抑制天然免疫,还使其能够利用细胞自身的胞内环境稳态维持机制为自身复制创造场地和原始材料[8-9, 41]。
ASFV因基因组庞大、结构复杂,遗传多样性等特征,致使病毒的基因组转录、蛋白翻译和免疫逃逸机制等异常复杂。已有研究表明,ASFV经内吞和巨胞饮途径入侵细胞后,经内体迁移途径和膜融合机制将病毒核心释放至细胞核周围,激活病毒基因表达和DNA复制[42]。ASFV基因表达具有精准的时空特性,感染初期利用病毒自身携带的酶和蛋白因子来完成早期基因的转录,随后借助宿主翻译系统合成病毒早期蛋白,一方面为病毒DNA复制以及中后期基因转录提供所需的酶和转录因子等,另一方面参与调控宿主细胞的应激反应及介导免疫逃逸等。随着ASFV感染进程发展,细胞核周微管组织中心处形成病毒复制的大本营“病毒工厂”,为病毒的蛋白翻译、基因组复制和组装等创造有利条件[3, 16]。然而,目前对ASFV功能蛋白介导上述生存策略的认知极度匮乏,尤其是关于病毒蛋白介导的对宿主细胞翻译系统的调控策略。
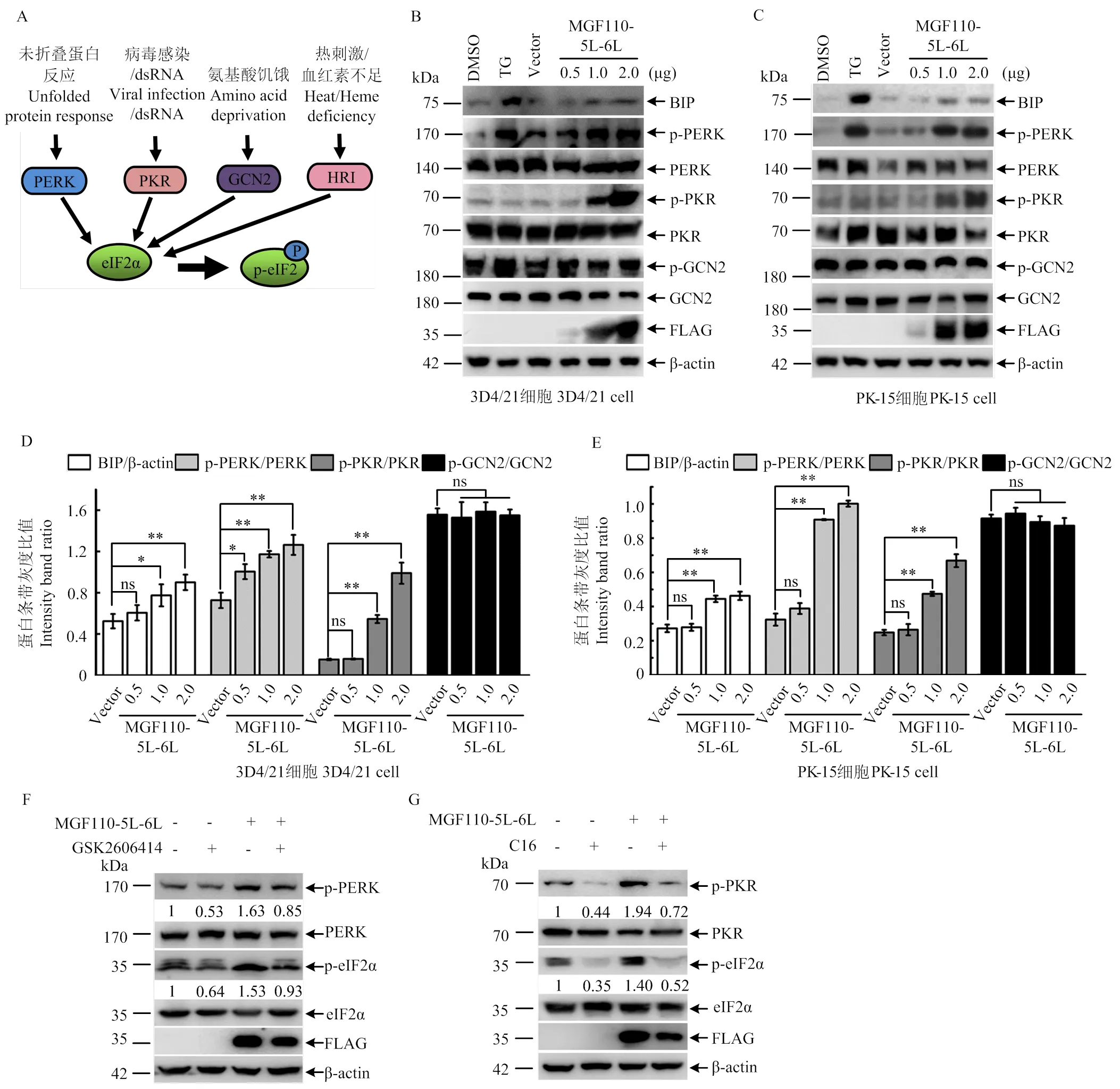
A:PERK,PKR,GCN2,HRI 四种激酶诱导eIF2α磷酸化的模式图;B-E:将不同剂量的MGF110-5L-6L表达质粒或空载体转染3D4/21和PK-15细胞,孵育24 h后,收集细胞裂解物,进行Western blot检测和灰度值分析. 作为阳性对照,利用内质网应激诱导剂TG(1 μmol·L-1)孵育处理细胞6 h。F和G:分别将FLAG空载体或MGF110-5L-6L重组表达质粒转染3D4/21细胞,伴随10 μmol·L-1 GSK2606414或1 μmol·L-1 C16处理细胞,孵育24 h后,收集细胞进行Western blot检测;该结果源自三次独立重复试验. **, P<0.01
3.2 外源表达MGF110-5L-6L蛋白对eIF2α磷酸化及其下游翻译效应调控的影响
eIF2α在翻译起始调控中具有重要功能,且eIF2α磷酸化与翻译阻滞、SG形成、细胞周期停滞、内质网应激等反应密切相关[10-13]。另外,值得关注的是,有报道称ASFV细胞适应性毒株(Ba71V株)在Vero细胞中成功增殖过程中,能够持续阻止eIF2α发生磷酸化[16];而野毒株(ASFV E70株)在感染小鼠单核/巨噬细胞RAW 264.7后能够翻译出病毒早期蛋白,但无法成功增殖,且伴随着eIF2α磷酸化水平的显著升高[19]。基于此,不难推测eIF2α信号通路极有可能是ASFV和宿主细胞决战的重要阵地之一。聚焦这一科学问题,本团队前期以病毒蛋白与eIF2α信号通路之间的作用关系为切入点,系统筛选了全套ASFV编码蛋白对eIF2α磷酸化水平的影响。其中,MGF110-5L-6L作为病毒早期蛋白[43],上调eIF2α磷酸化的水平极为显著。且在ASFV感染整个过程中,MGF110-5L-6L属于表达水平最高的病毒基因之一,推测其在病毒复制过程中具有重要功能。然而,关于其蛋白结构及功能仍未见报道。
在本研究中,我们利用肺泡巨噬细胞3D4/21(ASFV感染细胞系)和猪肾传代细胞PK-15,证实外源表达MGF110-5L-6L蛋白可诱导细胞发生内质网应激且激活未折叠蛋白反应,通过PERK和PKR激酶的活化触发eIF2α的磷酸化,进而诱发ISR、细胞蛋白翻译阻滞和SGs形成等。从理论上来看,应激状态导致细胞翻译重编程和SGs形成对病毒复制是不利的。然而,有研究表明,ASFV早期蛋白pE66L作为一种内质网跨膜蛋白,通过活化PKR/eIF2α通路显著抑制宿主蛋白翻译[44];ASFV晚期蛋白DP71L通过募集磷酸酶PP1诱导eIF2α去磷酸化,解除翻译阻滞以增强病毒蛋白的合成[35-36]。此外,与母本毒株相比,DP71L或pE66L单基因缺失毒株的增殖能力均未受影响,且DP71L基因缺失毒株与母本毒株诱导的eIF2α磷酸化水平无显著差异[35, 44]。进一步结合本团队的筛选结果,发现能够显著上调eIF2α磷酸化水平的多为病毒早期基因,而显著下调eIF2α磷酸化水平的多为病毒晚期基因,推测ASFV对宿主细胞eIF2α磷酸化的调控是全方位且多层次的,病毒可能通过在空间和时间上精准控制不同基因的转录和翻译,进行综合性调控以满足自身不同阶段的增殖需求。其中病毒早期蛋白主要上调eIF2α磷酸化水平,诱发细胞各种应激反应及蛋白翻译系统关闭,抑制抗病毒免疫;随着病毒感染进程的不断发展,中后期蛋白在病毒工厂局部特异地解除eIF2α的磷酸化,以全面促进病毒蛋白的合成。
3.3 外源表达MGF110-5L-6L蛋白与诱发细胞应激反应的相关性分析
最新研究表明,ASFV感染早期可显著诱导未折叠蛋白反应中PERK通路相关基因的表达上调[22]。为了探析早期蛋白MGF110-5L-6L在该过程中的潜在功能,本研究亦对其结构及亚细胞定位进行了剖析。发现MGF110-5L-6L蛋白存在与其他MGF110家族蛋白相似的序列特征[39],即氨基端存在潜在的信号肽,具有高度保守的半胱氨酸基序,且羧基端包含内质网滞留信号序列等。证实MGF110-5L-6L蛋白主要定位于内质网,少量定位于高尔基体、线粒体和溶酶体,且诱导高尔基体和过氧化物酶体定位和形态发生出现显著变化,暗示该蛋白在重塑内质网/高尔基体、内质网-高尔基体膜泡运输、以及过氧化物酶体发生等过程存在独特功能。推测MGF110-5L-6L蛋白可能通过影响内质网腔中氧化还原稳态、蛋白分泌途径或相关膜性细胞器组分来破坏细胞的正常生理功能稳态,进而诱发内质网应激等系列应激反应。然而,ASFV经过长期进化而来的生存策略,可能不仅避免了细胞应激对其复制造成太大影响,反而使其能够利用细胞产生的应激损伤,进而为病毒蛋白的膜向运输、病毒工厂组装和免疫逃逸等过程服务[16-19]。这也与ASFV感染破坏细胞分泌途径中高尔基体反式网络,以及阻挠膜蛋白MHC-I分子呈递至巨噬细胞表面的报道一致[45- 46]。
3.4 MGF110-5L-6L与病毒遗传多样性和表型之间的相关性分析
在不同的高致病性ASFV毒株中,MGF110-5L-6L基因以两种形式表达,分别表达5L-6L融合蛋白或5L、6L两个独立蛋白,且前者较后者缺失多个氨基酸片段;此外,与大多数天然强毒株和母本毒株相比,发现细胞适应性毒株ASFV-P121基因组中的MGF110-5L-6L基因完全缺失,天然致弱毒株OUR T88/3的6L基因完全缺失,且5L基因缺失近43%[47-48],而细胞适应性毒株ASFV-G/VP110和BA71V则保留MGF110-5L-6L基因[49],暗示MGF110-5L-6L属于ASFV的非必需基因,与病毒的遗传多样性和新表型特征发生有关。已有证据表明,同家族成员MGF110-9L与MGF110-5L-6L类似,均在细胞适应传代过程中丢失[47-49],且MGF110-9L与病毒增殖能力与毒力显著相关[50];而MGF110-1L作为所有ASFV毒株的保守基因,单基因缺失后不影响毒株的复制能力与毒力[43]。此外,大量证据表明,同属非必需基因的MGF360和MGF505/530家族成员在决定细胞嗜性,调控病毒毒力、复制、免疫逃逸,以及参与病毒-宿主互作等方面具有重要影响,且被广泛用于基因缺失活疫苗的开发研究[1-3]。
基于本研究证实外源表达MGF110-5L-6L蛋白在调控eIF2α发生磷酸化、细胞翻译重编程和应激反应中的功能特性,结合压力进化过程中MGF110-5L-6L基因表现出的遗传多样性与病毒表型之间的作用关系,推测MGF110-5L-6L在决定病毒宿主细胞范围、复制和毒力方面亦具有巨大潜力。然而,遗憾的是,本单位不具备开展ASFV感染实验的资质。本团队未来将着力与具备活毒操作资质的单位合作,进一步证实MGF110-5L-6L基因与ASFV毒力、嗜性及致病性等方面的功能关系,加深对ASFV病原生物学与致病机制的认识,为疫苗研发提供科学参考。
4 结论
研究发现MGF110-5L-6L蛋白含有两个高度保守的半胱氨酸基序,且主要定位于内质网,少量分布于高尔基体、线粒体和溶酶体。外源表达MGF110-5L-6L蛋白还可诱导高尔基体和过氧化物酶体的亚细胞定位及形态发生显著改变,提示其可能通过影响内质网腔中氧化还原稳态、蛋白分泌途径或相关膜性细胞器发生等,破坏细胞正常生理功能稳态。证实了外源表达MGF110-5L-6L蛋白可诱导宿主细胞发生内质网应激和未折叠蛋白反应,并通过活化PERK和PKR激酶触发eIF2α的磷酸化,进而诱发综合应激反应、导致细胞蛋白翻译阻滞以及应激颗粒形成,为深入探索非洲猪瘟病毒的病原生物学与致病机制提供了新的科学参考。
[1] GALLARDO M C, REOYO A T, FERNÁNDEZ-PINERO J, IGLESIAS I, MUÑOZ M J, ARIAS M L. African swine fever: a global view of the current challenge. Porcine Health Management, 2015, 1: 21. doi:10.1186/s40813-015-0013-y.
[2] ALONSO C, BORCA M, DIXON L, REVILLA Y, RODRIGUEZ F, ESCRIBANO J M, CONSORTIUM I R. ICTV virus taxonomy profile: Asfarviridae. The Journal of General Virology, 2018, 99(5): 613-614. doi:10.1099/jgv.0.001049.
[3] WANG Y, KANG W F, YANG W P, ZHANG J, LI D, ZHENG H X. Structure of African swine fever virus and associated molecular mechanisms underlying infection and immunosuppression: a review. Frontiers in Immunology, 2021, 12: 715582. doi:10.3389/fimmu.2021.715582.
[4] WANG N, ZHAO D M, WANG J L, ZHANG Y L, WANG M, GAO Y, LI F, WANG J F, BU Z G, RAO Z H, WANG X X. Architecture of African swine fever virus and implications for viral assembly. Science, 2019, 366(6465): 640-644. doi:10.1126/science.aaz1439.
[5] ALEJO A, MATAMOROS T, GUERRA M, ANDRÉS G. A proteomic atlas of the African swine fever virus particle. Journal of Virology, 2018, 92(23): e01293-e01218. doi:10.1128/JVI.01293-18.
[6] MALOGOLOVKIN A, KOLBASOV D. Genetic and antigenic diversity of African swine fever virus. Virus Research, 2019, 271: 197673. doi:10.1016/j.virusres.2019.197673.
[7] WALSH D, MOHR I. Viral subversion of the host protein synthesis machinery. Nature Reviews Microbiology, 2011, 9(12): 860-875. doi:10.1038/nrmicro2655.
[8] JAN E, MOHR I, WALSH D. A cap-to-tail guide to mRNA translation strategies in virus-infected cells. Annual Review of Virology, 2016, 3(1): 283-307. doi:10.1146/annurev-virology-100114-055014.
[9] STERN-GINOSSAR N, THOMPSON S R, MATHEWS M B, MOHR I. Translational control in virus-infected cells. Cold Spring Harbor Perspectives in Biology, 2019, 11(3): a033001. doi:10.1101/cshperspect.a033001.
[10] WEK R C. Role of eIF2α kinases in translational control and adaptation to cellular stress. Cold Spring Harbor Perspectives in Biology, 2018, 10(7): a032870. doi:10.1101/cshperspect.a032870.
[11] DONNELLY N, GORMAN A M, GUPTA S, SAMALI A. The eIF2α kinases: Their structures and functions. Cellular and Molecular Life Sciences: CMLS, 2013, 70(19): 3493-3511. doi:10.1007/s00018-012-1252-6.
[12] COSTA-MATTIOLI M, WALTER P. The integrated stress response: from mechanism to disease. Science, 2020, 368(6489): eaat5314. doi:10.1126/science.aat5314.
[13] PAKOS-ZEBRUCKA K, KORYGA I, MNICH K, LJUJIC M, SAMALI A, GORMAN A M. The integrated stress response. EMBO Reports, 2016, 17(10): 1374-1395. doi:10.15252/embr.201642195.
[14] LIU Y, WANG M, CHENG A, YANG Q, WU Y, JIA R, LIU M, ZHU D, CHEN S, ZHANG S. The role of host eIF2α in viral infection. Virology Journal, 2020, 17(1): 112.doi:10.1186/s12985-020-01362-6.
[15] SALAS M L, KUZNAR J, VIÑUELA E. Polyadenylation, methylation, and capping of the RNA synthesizedby African swine fever virus. Virology, 1981, 113(2): 484-491. doi:10.1016/0042-6822(81)90176-8.
[16] CASTELLÓ A, QUINTAS A, SÁNCHEZ E G, SABINA P, NOGAL M, CARRASCO L, REVILLA Y. Regulation of host translational machinery by African swine fever virus. PLoS Pathogens, 2009, 5(8): e1000562. doi:10.1371/journal.ppat.1000562.
[17] QUINTAS A, PÉREZ-NÚÑEZ D, SÁNCHEZ E G, NOGAL M L, HENTZE M W, CASTELLÓ A, REVILLA Y. Characterization of the African swine fever virus decapping enzyme during infection. Journal of Virology, 2017, 91(24): e00990-e00917. doi:10.1128/JVI.00990-17.
[18] BALLESTER M, RODRÍGUEZ-CARIÑO C, PÉREZ M, GALLARDO C, RODRÍGUEZ J M, SALAS M L, RODRIGUEZ F. Disruption of nuclear organization during the initial phase of African swine fever virus infection. Journal of Virology, 2011, 85(16): 8263-8269. doi:10.1128/JVI.00704-11.
[19] SÁNCHEZ E G, QUINTAS A, NOGAL M, CASTELLÓ A, REVILLA Y. African swine fever virus controls the host transcription and cellular machinery of protein synthesis. Virus Research, 2013, 173(1): 58-75. doi:10.1016/j.virusres.2012.10.025.
[20] CACKETT G, MATELSKA D, SÝKORA M, PORTUGAL R, MALECKI M, BÄHLER J, DIXON L, WERNER F. The African swine fever virus transcriptome. Journal of Virology, 2020, 94(9): e00119-e00120. doi:10.1128/JVI.00119-20.
[21] CACKETT G, PORTUGAL R, MATELSKA D, DIXON L, WERNER F. African swine fever virus and host response: transcriptome profiling of the2007/1 strain and porcine macrophages. Journal of Virology, 2022, 96(5): e0193921. doi:10.1128/jvi.01939-21.
[22] ZHENG Y X, LI S, LI S H, YU S X, WANG Q H, ZHANG K H, QU L, SUN Y, BI Y H, TANG F C, QIU H J, GAO G F. Transcriptome profiling in swine macrophages infected with African swine fever virus at single-cell resolution. Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(19): e2201288119. doi:10.1073/pnas.2201288119.
[23] VATTEM K M, WEK R C. Reinitiation involving upstream ORFs regulates ATF4mRNA translation in mammalian cells. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(31): 11269-11274. doi:10.1073/pnas.0400541101.
[24] LARKIN M A, BLACKSHIELDS G, BROWN N P, CHENNA R, MCGETTIGAN P A, MCWILLIAM H, VALENTIN F, WALLACE I M, WILM A, LOPEZ R, THOMPSON J D, GIBSON T J, HIGGINS D G. Clustal W and clustal X version 2.0. Bioinformatics, 2007, 23(21): 2947-2948. doi:10.1093/bioinformatics/btm404.
[25] ROBERT X, GOUET P. Deciphering key features in protein structures with the new ENDscript server. Nucleic Acids Research, 2014, 42(W1): W320-W324. doi:10.1093/nar/gku316.
[26] MITCHELL A L, ATTWOOD T K, BABBITT P C, BLUM M, BORK P, BRIDGE A, BROWN S D, CHANG H Y, EL-GEBALI S, FRASER M I, et al. InterPro in 2019: improving coverage, classification and access to protein sequence annotations. Nucleic Acids Research, 2018, 47(D1): D351-D360. doi:10.1093/nar/gky1100.
[27] ALMAGRO ARMENTEROS J J, TSIRIGOS K D, SØNDERBY C K, PETERSEN T N, WINTHER O, BRUNAK S, VON HEIJNE G, NIELSEN H. SignalP 5.0 improves signal peptide predictions using deep neural networks. Nature Biotechnology, 2019, 37(4): 420-423. doi:10.1038/s41587-019-0036-z.
[28] KROGH A, LARSSON B, VON HEIJNE G, SONNHAMMER E L L. Predicting transmembrane protein topology with a hidden Markov model: Application to complete genomes. Journal of Molecular Biology, 2001, 305(3): 567-580. doi:10.1006/jmbi.2000.4315.
[29] GUPTA R, BRUNAK S. Prediction of glycosylation across the human proteome and the correlation to protein function. Pacific Symposium on Biocomputing, 2002: 310-322.
[30] KLAUSEN M S, JESPERSEN M C, NIELSEN H, JENSEN K K, JURTZ V I, SØNDERBY C K, SOMMER M O A, WINTHER O, NIELSEN M, PETERSEN B, MARCATILI P. NetSurfP-2.0: Improved prediction of protein structural features by integrated deep learning. bioRxiv, 2018, doi:10.1101/311209. doi:10.1101/311209.
[31] ZHENG W, ZHANG C X, LI Y, PEARCE R, BELL E W, ZHANG Y. Folding non-homologous proteins by coupling deep-learning contact maps with I-TASSER assembly simulations. Cell Reports Methods, 2021, 1(3): 100014. doi:10.1016/j.crmeth.2021.100014.
[32] WEINGARTL H M, SABARA M, PASICK J, VAN MOORLEHEM E, BABIUK L. Continuous porcine cell lines developed from alveolar macrophages: Partial characterization and virus susceptibility. Journal of Virological Methods, 2002, 104(2): 203-216. doi:10.1016/S0166-0934(02)00085-X.
[33] SCHMIDT E K, CLAVARINO G, CEPPI M, PIERRE P. SUnSET, a nonradioactive method to monitor protein synthesis. Nature Methods, 2009, 6(4): 275-277. doi:10.1038/nmeth.1314.
[34] IVANOV P, KEDERSHA N, ANDERSON P. Stress granules and processing bodies in translational control. Cold Spring Harbor Perspectives in Biology, 2019, 11(5): a032813. doi:10.1101/cshperspect.a032813.
[35] ZHANG F Q, MOON A, CHILDS K, GOODBOURN S, DIXON L K. The African swine fever virus DP71L protein recruits the protein phosphatase 1 catalytic subunit to dephosphorylate eIF2alpha and inhibits CHOP induction but is dispensable for these activities during virus infection. Journal of Virology, 2010, 84(20): 10681-10689. doi:10.1128/JVI.01027-10.
[36] ROJAS M, VASCONCELOS G, DEVER T E. An eIF2α-binding motif in protein phosphatase 1 subunit GADD34 and its viral orthologs is required to promote dephosphorylation of eIF2α. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(27): E3466-E3475. doi:10.1073/pnas.1501557112.
[37] ZHU Z Z, CHEN H T, LIU L, CAO Y, JIANG T J, ZOU Y Q, PENG Y S. Classification and characterization of multigene family proteins of African swine fever viruses. Briefings in Bioinformatics, 2020, 22(4): bbaa380. doi:10.1093/bib/bbaa380.
[38] GOMEZ-NAVARRO N, MILLER E. Protein sorting at the ER-Golgi interface. The Journal of Cell Biology, 2016, 215(6): 769-778. doi:10.1083/jcb.201610031.
[39] NETHERTON C, ROUILLER I, WILEMAN T. The subcellular distribution of multigene family 110 proteins of African swine fever virus is determined by differences in C-terminal KDEL endoplasmic reticulum retention motifs. Journal of Virology, 2004, 78(7): 3710-3721. doi:10.1128/jvi.78.7.3710-3721.2004.
[40] 丁铲. 病毒感染引起的细胞器应激损伤和选择性自噬. 中国家禽, 2019, 41(10): 1-9. doi:10.16372/j.issn.1004-6364.2019.10.001.
DING C. Virus infection causes cell organelle stress and selective autophagy. China Poultry, 2019, 41(10): 1-9. doi:10.16372/j.issn.1004-6364.2019.10.001. (in Chinese)
[41] MCCORMICK C, KHAPERSKYY D A. Translation inhibition and stress granules in the antiviral immune response. Nature Reviews Immunology, 2017, 17(10): 647-660. doi:10.1038/nri.2017.63.
[42] ANDRÉS G. African swine fever virus gets undressed: New insights on the entry pathway. Journal of Virology, 2017, 91(4): e01906-e01916. doi:10.1128/JVI.01906-16.
[43] RAMIREZ-MEDINA E, VUONO E, PRUITT S, RAI A, SILVA E, ESPINOZA N, ZHU J, VELAZQUEZ-SALINAS L, BORCA M V, GLADUE D P. Development andevaluation of a MGF110-1L deletion mutant in African swine fever strain. Viruses, 2021, 13(2): 286. doi:10.3390/v13020286.
[44] SHEN Z, CHEN C, YANG Y L, XIE Z H, AO Q Y, LV L, ZHANG S F, CHEN H C, HU R L, CHEN H J, PENG G Q. A novel function of African Swine Fever Virus pE66L in inhibition of host translation by the PKR/eIF2α pathway. Journal of Virology, 2020, 95(5): e01872-e01820. doi:10.1128/JVI.01872-20.
[45] MCCROSSAN M, WINDSOR M, PONNAMBALAM S, ARMSTRONG J, WILEMAN T. The trans Golgi network is lost from cells infected with African swine fever virus. Journal of Virology, 2001, 75(23): 11755-11765. doi:10.1128/JVI.75.23.11755-11765.2001.
[46] NETHERTON C L, MCCROSSAN M C, DENYER M, PONNAMBALAM S, ARMSTRONG J, TAKAMATSU H H, WILEMAN T E. African swine fever virus causes microtubule-dependent dispersal of the trans-Golgi network and slows delivery of membrane protein to the plasma membrane. Journal of Virology, 2006, 80(22): 11385-11392. doi:10.1128/JVI.00439-06.
[47] CHAPMAN D A G, TCHEREPANOV V, UPTON C, DIXON L K. Comparison of the genome sequences of non-pathogenic and pathogenic African swine fever virus isolates. The Journal of General Virology, 2008, 89(Pt 2): 397-408. doi:10.1099/vir.0.83343-0.
[48] WANG T, WANG L, HAN Y, PAN L, YANG J, SUN M W, ZHOU P P, SUN Y, BI Y H, QIU H J. Adaptation of African swine fever virus to HEK293T cells. Transboundary and Emerging Diseases, 2021, 68(5): 2853-2866. doi:10.1111/tbed.14242.
[49] KRUG P W, HOLINKA L G, O'DONNELL V, REESE B, SANFORD B, FERNANDEZ-SAINZ I, GLADUE D P, ARZT J, RODRIGUEZ L, RISATTI G R, BORCA M V. The progressive adaptation of a Georgian isolate of African swine fever virus to vero cells leads to a gradual attenuation of virulence in swine corresponding to major modifications of the viral genome. Journal of Virology, 2015, 89(4): 2324-2332. doi:10.1128/JVI.03250-14.
[50] LI D, LIU Y G, QI X L, WEN Y, LI P, MA Z, LIU Y J, ZHENG H X, LIU Z J. African swine fever virus MGF-110-9L-deficient mutant has attenuated virulence in pigs. Virologica Sinica, 2021, 36(2): 187-195. doi:10.1007/s12250-021-00350-6.
African Swine Fever Virus Mgf110-5l-6lInduces Host Cell Translation Arrest and Stress Granule Formation by Activating the Perk/Pkr-eif2α Pathway
FAN Shuai, ZHONG Han, YANG ZhongYuan, HE WenRui, WAN Bo, WEI ZhanYong, HAN ShiChong, ZHANG GaiPing
College of Veterinary Medicine, Henan Agricultural University/International Joint Research Center of National Animal Immunology, Zhengzhou 450046
【】African swine fever (ASF) is an acute, highly contagious, and deadly infectious disease of pigs caused by ASF virus (ASFV), which is currently considered the biggest killer in global swine industry. To date, there is no effective vaccines or antiviral drugs for the prevention or treatment of ASF. As obligate intracellular parasites, the viruses are fully reliant on the host translation machinery to produce the polypeptides that are essential for viral replication. A central mechanism regulating translation initiation involves phosphorylation of the α subunit of eukaryotic initiation factor 2 (eIF2α), which directs host translational control and adaptation to cellular stress. The regulation of eIF2α phosphorylation has been regarded as a critical step for viral infection, with important effects on virulence, tissue tropism, pathogenicity, and immunoevasion. However, the molecular mechanisms by which most of the ASFV-encoded proteins affecting eIF2α phosphorylation have not been well studied. 【】The aim of this study was to explore the mechanism of ASFV MGF110-5L-6L protein on the host cell translation block and promote the formation of stress particles, so as to provide a scientific basis for further revealing the pathogenic mechanism of African swine fever virus.【】 The preliminary screening by luciferase reporter assays identified that ectopic expression of ASFV MGF110-5L-6L, a previously uncharacterized member of the multigene family 110, significantly increased eIF2α phosphorylation levels. To confirm and clarify the potential role of MGF110-5L-6L expression in mediating eIF2α phosphorylation and downstream of translation control, two continuous porcine cell lines, including 3D4/21 (porcine alveolar macrophage) and PK-15 (porcine kidney), were used for the plasmid transfection and/or drug treatment and subjected to immunoblotting or confocal immunofluorescence analysis. To investigate how the ectopic expression of MGF110-5L-6L triggers cellular stress, the structure, subcellular localization and function of the MGF110-5L-6L protein were further characterized by a combination of bioinformatic prediction, immunofluorescence and immunoblotting analysis.【】 Here, it was confirmed that ectopic expression of MGF110-5L-6L remarkably promoted eIF2α phosphorylation and the expression of ATF4, indicating that it functions in the integrated stress response. The subsequent analyses revealed that MGF110-5L-6L expression could trigger the ER stress and activate the unfolded protein response, and the phosphorylation of eIF2α was mediated via PERK and PKR, resulting in the suppression of host translation and stress granule formation. It was further observed that MGF110-5L-6L protein had two highly conserved central cysteine-rich domains and was mainly retained in the endoplasmic reticulum (ER), and also caused a significant reorganization of the subcellular distribution and morphological characteristics of the Golgi and peroxisome, suggesting that it might interfere with ER redox homeostasis, secretory pathway, and other membrane-bound organelles to trigger cellular stress.【】 Together, these results demonstrated a previously uncharacterized role of ASFV MGF110-5L-6L and further defined several molecular interfaces by which ASFV MGF110-5L-6L hijacks the host cell translation, which expanded the view of ASFV in determining the fate of host-pathogen interactions.
African swine fever virus; MGF110-5L-6L protein; eIF2α phosphorylation; translation arrest; stress granules
2022-02-28;
2022-06-30
国家自然科学基金(31941001, 32102655)、河南省重点研发与推广专项(212102110369)
樊帅,Tel:15138698236;E-mail:fshuai9859@163.com。通信作者韩世充,E-mail:hanshichong081@126.com。通信作者张改平,E-mail:zhanggaip@126.com
(责任编辑 林鉴非)
