基于基因组SNP信息分析新疆夏洛莱牛群体的遗传结构和遗传背景初报
2023-03-06蒋烈戈朱婷婷钟发钢蔡国宝
蒋烈戈,彭 健,代 蓉,高 攀,苏 政,刘 鹏,朱婷婷,钟发钢,赵 博,蔡国宝
(1.新疆生产建设兵团第九师农业科学研究所(畜牧科学研究所),新疆 额敏 834601;2.新疆农垦科学院 新疆 石河子 832000;3.新疆生产建设兵团第九师165团农业发展中心,新疆 额敏 834609)
夏洛莱肉牛原产于法国,是世界著名的大型肉牛品种之一。1964年中法两国建交,法国于1965年在北京举办展览会,展出原种夏洛莱肉牛,之后法国总理戴高乐赠送给周总理一批原种夏洛莱肉牛,被调入新疆生产建设兵团第九师165团进行纯繁与杂交利用,以期培育出适应新疆兵团放牧饲养的肉牛新品种。自夏洛莱牛落户新疆生产建设兵团第九师,在近50年的时间里,在塔额盆地已初步形成遗传性能稳定、产肉性能好的新类群。新疆夏洛莱牛新类群个体体型略小于法国原种夏洛莱牛,群体的表型一致性较好,抗逆性强,繁殖性能高,性情温顺,适合放牧饲养和圈养舍饲,能够充分利用塔额垦区的饲料资源[1,2]。目前兵团九师的夏洛莱群体规模近两万头,已经成为九师的特色产业。然而,在夏洛莱牛的杂交改良和选育过程缺乏系统的育种档案资料,存在着群体遗传背景不清、血缘关系不明等问题,影响后续新品种的选育和育种家系的建立,为解决这一难题,同时为适应市场经济发展,提高新疆夏洛莱的生产性能,近年来,新疆生产建设兵团第九师以新疆夏洛莱肉牛选育提高为肉牛产业发展的重点突破方向,开展肉牛种质资源创新利用,通过新疆夏洛莱良种肉牛提纯复壮关键技术研究、九师夏洛莱肉牛遗传结构调查种牛选育等项目,来逐步培育出适应性强且具有较高生产性能的新疆夏洛莱肉牛。
本研究利用基因组高通量检测技术,分析九师夏洛莱杂交新类群核心群中的全基因组水平SNPs变化,结合年龄、性别等信息对新疆夏洛莱群体的遗传多样性进行分析,群体遗传结构、核心群血亲关系,明确系谱信息[3],为群体的血缘关系和遗传背景提供科学的证明,指导组建核心育种群,开展全基因组选育,为新疆夏洛莱肉牛种质资源创新利用打基础。
1 材料与方法
1.1 样品的采集
样品采自新疆生产建设兵团第九师165团5连、8连、金旭养殖合作社等肉牛基地1~9岁生产母牛群体,共采集测定样本为173头新疆夏洛莱成年牛。通过尾根静脉采血,将采集到的血液样本在EDTAK2采血管中保存,同时涂抹血液样品采集卡,样品送至北京康普森生物技术有限公司进行基因组SNP高通量检测,获得上述新疆夏洛莱肉牛群体的全基因组SNP原始检测数据。
1.2 基因芯片检测
检测基因芯片采用NEOGEN公司GGP牛100 K芯片,是一款高密度芯片,大约由100 000个SNPs组成,平均SNP间距29.0 Kb,该芯片主要用于牛遗传评估,全基因组关联研究,定量性状基因座的鉴定和比较遗传学研究。芯片检测试验流程:样本→DNA提取→DNA质检→扩增→片段化→聚沉、晾干→重悬→杂交→洗涤、染色→扫描芯片。
所有样品的gDNA利用琼脂糖凝胶电泳和Nanodrop ND-2000(Themro Scientific)的方法进行定量分析[4],gDNA的浓度调整至50 ng/L。然后,对所有样品进行全基因组扩增,gDNA进行片段化、沉淀和在杂交缓冲液中重悬。将重悬的DNA片段加到芯片上,48℃孵育杂交16~24 h。杂交后,通过清洗去掉非特异性结合的DNA,剩下特异性结合的位点进行单碱基延伸,染色后利用Illumina iScan Reader进行扫描,将iScan系统扫描得到的原始数据导入Illumina官方软件GenomeStudioGenotyping模块中,对原始数据标准化、聚类和基因分型,最后形成数据用于后续分析。
1.3 数据质控
分析之前,首先使用Plink(V1.90)软件对基因型数据进行质控,只保留分型质量最好的位点进行后续分析。根据不同的分析需要,质控标准有所不同,具体分析项目的质控标准见表1。
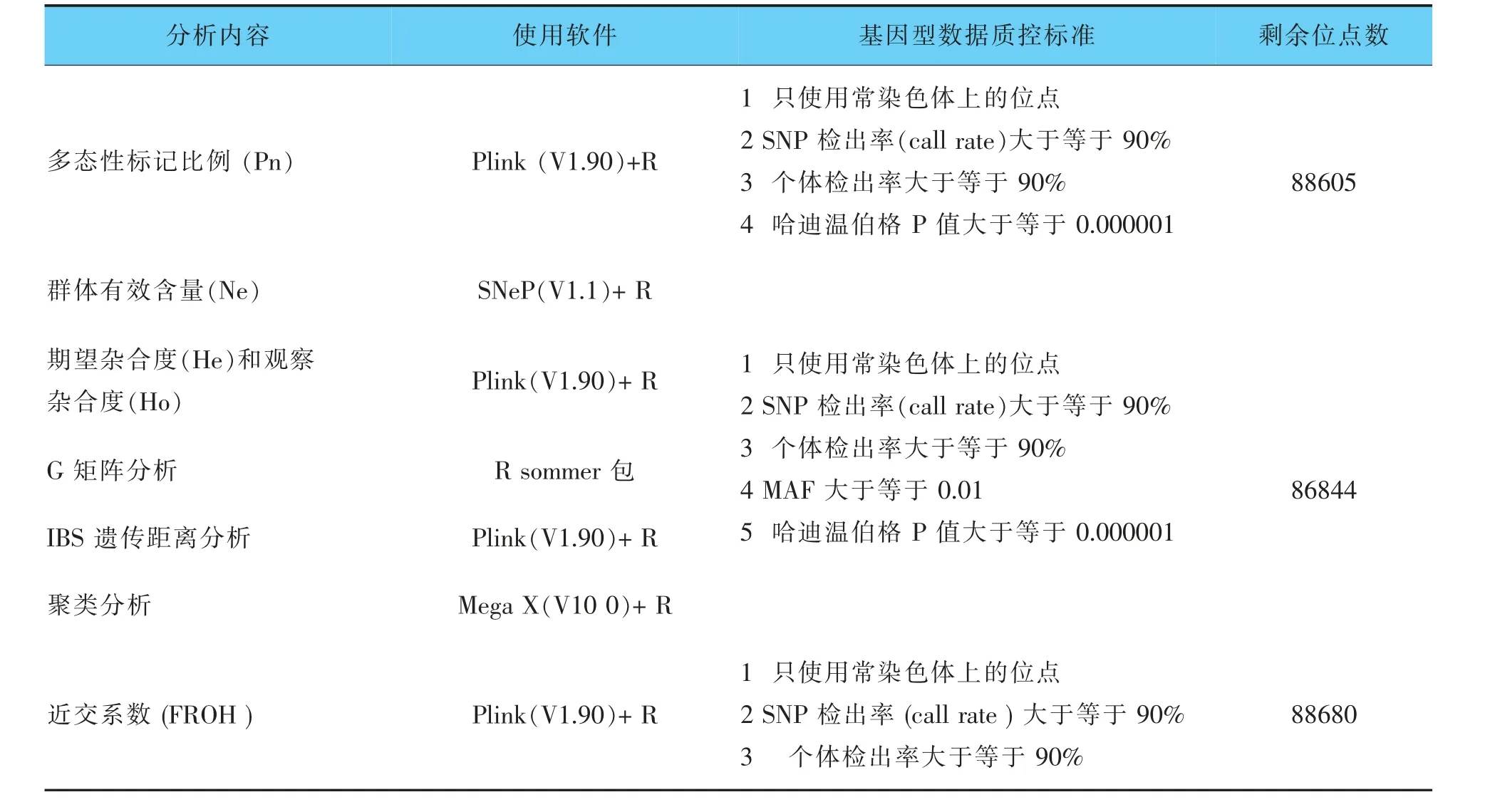
表1 基因型数据质控标准
1.4 数据分析方法
使用Plink(V1.90)和R软件分析多态性标记比例(Pn)、期望杂合度(He)、观察杂合度(Ho)、IBS遗传距离分析和近交系数(FROH),使用SNeP(V1.1)和R软件分析群体有效含量(Ne),使用R sommer包进行G矩阵分析,使用Mega X(V10.0)和R软件进行聚类分析。
2 结果与分析
由于群体有分层,将部分个体当做离群样本进行了剔除,剩余个体数量为173份,剩余样品均为新疆夏洛莱群体,后继分析以新疆夏洛莱群体为主。使用GGP10K芯片对173份牛样本进行了基因分型,173个样本SNP数据平均检出率(Call Rate值)大于90%(99.40%)。
2.1 群体遗传多样性分析
173个新疆夏洛莱群体检测后分析群体中有效群体含量(Ne)、群体的多态性标记比例(Pn),群体的期望杂合度(He)和观察杂合度(Ho)四项数据的分析,具体结果见表2。

表2 群体遗传多样性分析结果
有效群体含量(Ne)决定了群体平均近交系数增量的大小,反映了新疆夏洛莱群体遗传结构中基因的平均纯合速度,是群体遗传学研究的一个重要内容[5,6]。此次检测群体的有效群体含量为28.69,后期可能通过结合不同世代进行分类后分析世代间有效群体含量。群体的观察杂合度(Ho)0.4103低于期望杂合度(He)0.4095,说明现有核心群体的纯度较好。
基于G矩阵的基因组亲缘关系分析能有效解决群体系谱不清或祖代不明对群结构分析的影响,G矩阵分析173头新疆夏洛莱个体间亲缘关系,如图1。

图1 G矩阵分析173牛个体间亲缘关系
在亲缘关系分析结果中,可看出每一个小方格代表两两样本之间的基因组亲缘关系值,方格填充的颜色越接近红色则表示该值越大,即说明两个个体亲缘关系越近;从图1中可以看出,对角线上是每个样本与其本身的亲缘关系值,所以呈现红色;亲缘关系较近的个体会聚类到一起,且他们所在的单元格颜色更接近红色。结果显示大部分个体间的亲缘关系呈中等程度,说明九师165团夏洛莱肉牛有着共同遗传来源,这一群体在科学的人工选育条件下能够保留一定水平的多样性,同时也说明不同的自然环境对夏洛莱群体造成的影响是巨大的。通过亲缘关系理清第九师现有夏洛莱肉牛的资源现状和遗传基础,收集并系统整理它们的家系性能和亲缘关系资料,建立新疆夏洛莱肉牛遗传资料数据库,绘制基础牛群亲缘关系的家系图,可以实现科学引种和合理利用。数据还显示部分个体之间的亲缘关系较近,在以后的选种选配过程中要注意近交风险。
2.2 群体遗传结构分析
为探明检测新疆夏洛莱群体内的亲缘关系,使用邻接法(Neighbor-Joining,NJ),基于IBS遗传距离分析结果对样本进行聚类。聚类分析结果(图2)和亲缘分析结果(图1),将整个群体分为26个家系。新疆夏洛莱群体主成分分析结果显示有18个样本(见图3)和其他所有样本血缘关系较远,被归入“其他”分类中。
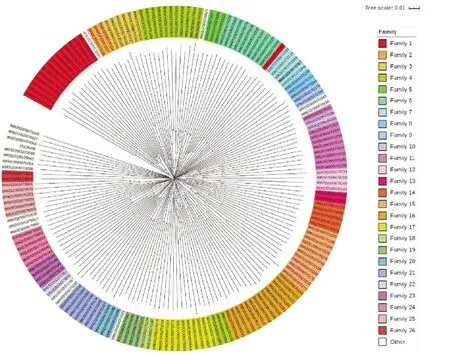
图2 检测样本聚类分析结果
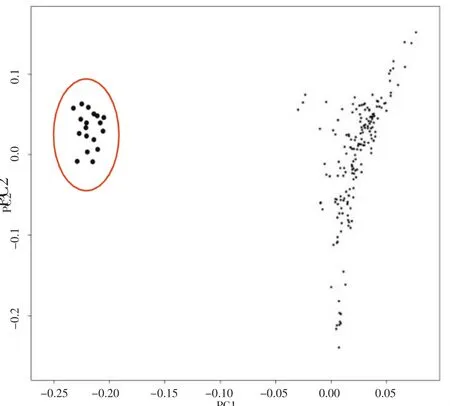
图3 PCA群体分布图
2.3 ROH的近交系数分析
基因组上的长纯合片段(Runs of homozygosity,ROH)在所有群体中广泛存在[7],ROH是个体内纯合基因型的连续片段,它是由于亲代将同源单倍型完整地传递给子代产生的,ROH在基因组中的大小、分布和频率受到自然选择、群体历史和近交水平等诸多因素的影响。随着高通量基因分型技术的使用,基于全基因组水平SNP信息获得ROH分子信息能更准确地估计纯合性,并可以检测过去和最近的近交情况。在173个血缘关系近的样本中,共检测到2 624个ROH片段,范围在0~42个之间,70.5%的个体都含有10个以上ROH片段,个体ROH的平均长度为1 476.17 kb。通过对该群体中每个个体的ROH统计,得到每个个体基于ROH的近交系数值是0.030,见图4。
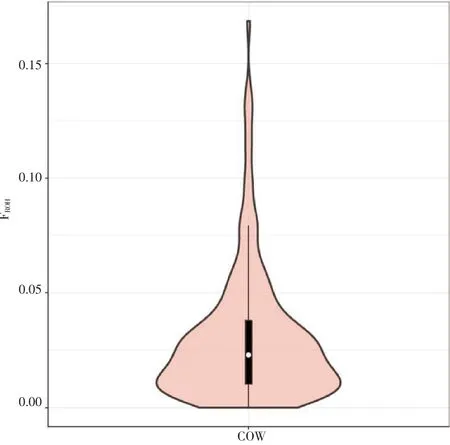
图4 基于ROH的近交系数FROH的分布
2.4 个体亲缘分析
运用GGP100K芯片对173个样本进行了基因分析,通过对分析样本进行个体亲缘关系分析,得到了可能存在亲缘关系的候选个体,部分结果如表3。因为缺少个体的出生日期和性别数据,因此存在两种亲子关系,一种是其他列中的个体是第一列ID1个体的亲本;另一种是其他列中的个体是第一列ID1个体的子代。匹配到2个以上个体的样本,例如表3中999202005070224个体,分别与999202005070222和999202005070248样本存在亲缘关系,既可能是999202005070224的亲本,也可能是999202005070224的子代,后续根据实际情况进行判断。
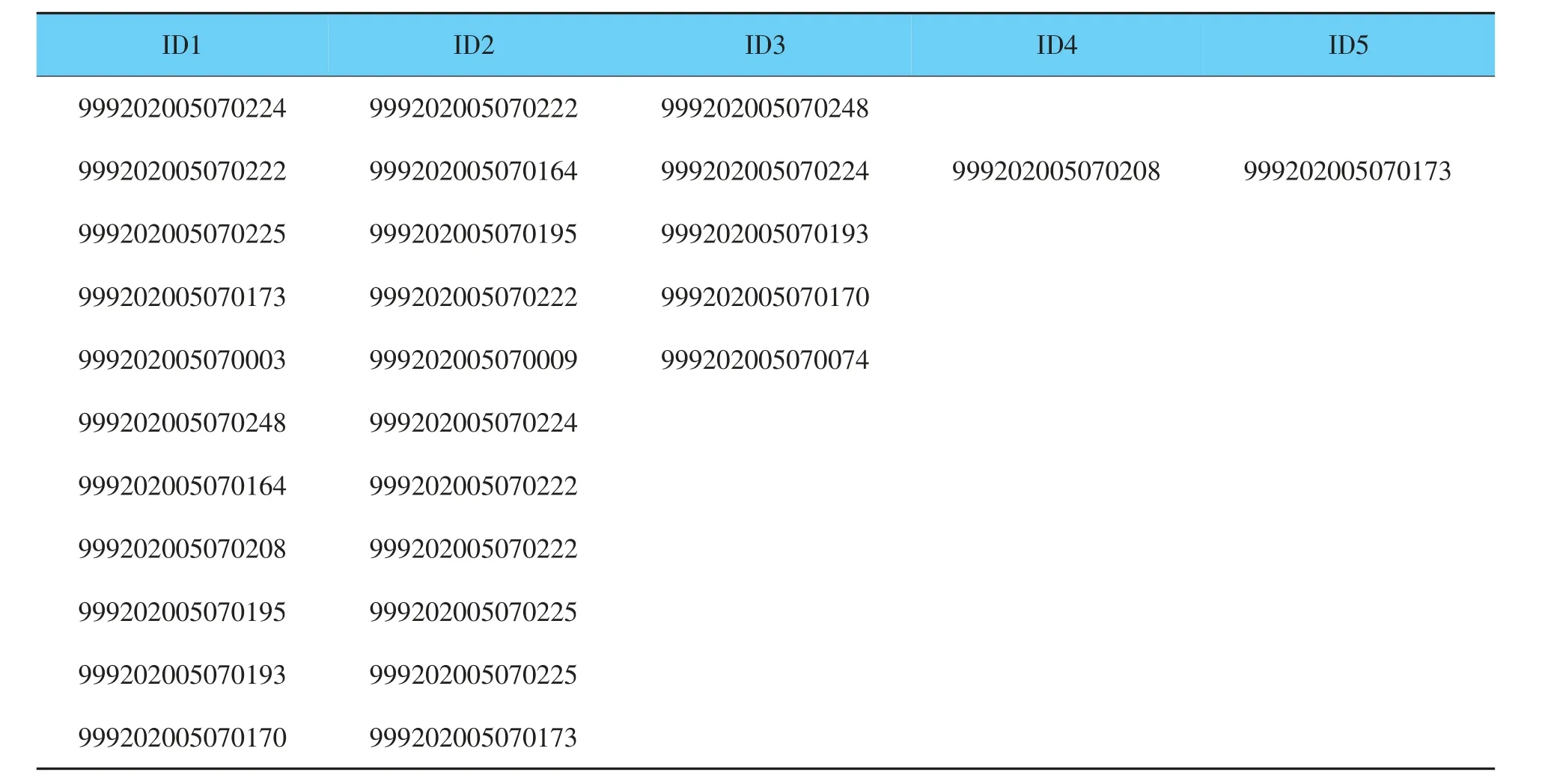
表3 部分样本候选个体亲子关系
3 讨 论
利用基因组高通量检测技术,分析九师夏洛莱杂交新类群核心群中的全基因组水平SNPs变化,新分析新疆夏洛莱肉牛群体内的遗传结构、亲缘关系和育种背景,分析检测结果表明,基因的平均纯合速度适中,如分析该群体世代间有效群体含量,还需要做更深入的研究;173个个体间的亲缘关系呈中等程度,说明九师165团夏洛莱肉牛有着共同遗传来源,可为后期建立新疆夏洛莱肉牛遗传资料数据库丰富基础数据提供指导;该群体近交系数0.030,说明该近交系数过低,这与我们在家系测定时,173头个体达到了26个家系结果一致,说明基因的纯合和稳定结果较差,同时不易暴露有缺点的隐性基因,而对后期的育种造成一定的影响,家系数量过多,也常会出现多峰或偏态现象,不利于优良性状的筛选;在本研究中,测试的样本缺少雄性个体,为明确种公牛与家系的关系,结合生产数据对其种用价值进行科学的评价,可补充种公牛的样本进行核心群全面分析。
4 结 论
本次试验利用全基因组芯片分型技术获得全基因SNP信息进行快速、准确的分析,进而获得实验群体新疆夏洛莱遗传结构、近交系数、个体亲缘关系等数据,实现了在原肉牛群体遗传背景不清、育种资料模糊的情况下对群体结构、亲缘关系和家系组成进行系统科学的分析,也为后续新品种的选种选配提供科学指导,这种方法克服了以前杂交育种时代育种时间较长,分子标记育种时代DNA测序费用高、控制优良性状难度大的缺点,基因组分析运用高通量测序,通过关联分析,对肉牛群体直接进行基因型选择或标记辅助选择,从而提高选种的正确性,缩短世代间隔,同时又不受性别、时间和环境等因素的影响,具有成本低,速度快等优点。
新疆夏洛莱肉牛经过多年的改良和选育,其生长性能、屠宰性能和肉用性能均有不同程度的提高,今后应积极运用现代育种技术,加强对本品种的保护、选育和利用,构建地方特色肉用牛选育体系,不断提高肉牛的生产水平和养殖效益,同时为建立现代新型良种繁育体系奠定坚实基础。
